小黑杨HD-Zip转录因子家族生物信息学及应答盐胁迫分析
2017-11-10张雪梅姚文静姜廷波周博如
张雪梅 姚文静 赵 凯 姜廷波 周博如
(东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040)
小黑杨HD-Zip转录因子家族生物信息学及应答盐胁迫分析
张雪梅 姚文静 赵 凯 姜廷波 周博如*
(东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040)
HD-Zip转录因子基因是植物中特有的一类蛋白家族,在植物生长发育和逆境应答胁迫过程中发挥重要作用。HD-Zip转录因子基因是由高度保守的同源异型结构域(HD)和亮氨酸拉链域(LZ)结构域构成的特殊结构模型。杨树HD-Zip转录因子家族共有63个基因,可被分为HD-ZipⅠ、HD-ZipⅡ、HD-ZipⅢ和HD-ZipⅣ四个亚家族。本文利用RNA-Seq分析了盐胁迫条件下HD-Zip基因家族在小黑杨根、茎、叶等不同组织的基因表达差异,从转录组水平揭示其应答胁迫环境的分子机制,结果表明,盐胁迫下在叶中有25个HD-Zip基因下调表达,21个基因上调表达;茎中有42个基因下调表达,11个基因上调表达;根中有26个基因下调表达,24个基因上调表达。另外,本文根据拟南芥HD-Zip转录因子家族基因的已知功能,预测了杨树HD-Zip转录因子同源基因的功能,并利用生物信息学方法分析了杨树HD-Zip转录因子蛋白序列的保守结构域、氨基酸组成和理化性质等,为进一步研究杨树HD-Zip转录因子基因功能提供参考。
HD-Zip转录因子;非生物胁迫;生长发育;小黑杨;生物信息学
杨树生长速度快、适应性强、分布范围广、材质优良,是重要的绿化、造林和用材树种,主要应用于四旁绿化、农林防护、民用建筑、板料和纸浆制造方面。随着杨树全基因组测序的完成,杨树成为研究植物分子生物学和遗传学上的木本模式植物,在木本植物生长发育和植物抗旱、耐盐、抗低温等非生物胁迫的研究和应用中非常广泛。
转录因子(Transcription factor)也称反式作用因子,是能与真核生物启动子特定DNA序列结合的蛋白质分子,或者是具有已知DNA结合域结构特征的蛋白质分子[1]。转录因子能与基因5′端上游特定序列进行专一性结合,保证目的基因在特定的时间与空间表达。植物中的转录调控能严格控制生物表达过程,从而应答环境胁迫,调节对环境变化的反应。
同源异型域—亮氨酸拉链蛋白(Homeodomain-leucine Zipper,HD-Zip)是植物特有的一类转录因子,属于Homeobox蛋白家族,具有高度保守的HD(Homeodomain,HD)结构域,由61个氨基酸组成[2],可折叠成典型的螺旋—环—螺旋—转角—螺旋的结构,能与DNA特异结合[3],其末端与亮氨酸拉链域(LZ)紧密相连,亮氨酸拉链结构域主要是介导蛋白二聚体的形成[4]。根据氨基酸序列同源性的不同,植物HD-Zip转录因子基因可分为HD-ZipⅠ、HD-ZipⅡ、HD-ZipⅢ和HD-ZipⅣ 4类[5],并且四大亚类均有一定的结构特征和相应的功能特点,HD-ZipⅠ主要包括HD和LZ两个保守结构域,在应答非生物胁迫方面发挥重要作用[6];HD-ZipⅡ亚家族基因除了含有高度保守的HD和LZ序列外,在LZ下游紧挨着LZ结构域还有一个以五个保守氨基酸半胱氨酸(Cys)、脯氨酸(Pro)、丝氨酸(Ser)、半胱氨酸(Cys)和谷氨酸(Glu)命名的N端共有序列,主要负责细胞氧化还原传感[7],该亚家族的基因负责感应光信号并能对植物激素产生应答效应[8];HD-ZipⅢ亚家族成员大多含有START(steroidogenic acute regulatory protein-related lipid transfer)结构域和与START相邻的SAD(START-adjacent domain),除此以外,几乎所有成员在C端均有一个可以结合信号分子来改变HD-ZipⅢ蛋白构象的MEKHLA结构域,该亚家族主要在胚胎形态发生、分生组织调控、侧生器官形成、叶片的极性运输以及生长素的运输等方面发挥重要作用[9~10];HD-ZipⅣ亚家族成员相较于HD-ZipⅢ亚家族缺少了MEKHLA结构域,该亚家族成员主要参与表皮细胞的分化、花青素的积累、根的发育和毛状体的形成[11~12]等。当前已对拟南芥、水稻[13]、苹果、紫花苜蓿等植物的HD-Zip转录因子基因家族有过相关研究,但对杨树的HD-Zip转录因子基因家族介绍较少。
杨树共有63个HD-Zip转录因子基因[14],本文利用转录组测序(RNA-Seq)法比较了杨树在高盐胁迫下分别在根、茎、叶中的相对表达量,从而分析了HD-Zip转录因子基因在不同组织中的差异性表达;并利用生物信息学的方法对杨树的HD-Zip转录因子基因家族成员进行分析,为深度研究杨树转录因子的功能提供依据。
1 材料与方法
1.1 非生物胁迫处理杨树幼苗
从同一株杨树上采集嫩枝,截成15 cm,25℃室温14 h·d-1光照水培两个月,新长出根、茎(顶尖嫩茎处)、叶的杨树苗即为本实验材料。然后分别用水(对照)和0.15 mol·L-1NaCl溶液处理24 h,收集根、茎、叶样品(每个处理有3个生物学重复),液氮速冻、-70℃保存,然后送生物公司提取RNA并利用IlluminaHiSeq 2000基因分析系统进行转录组高通量测序,最后进行RNA测序数据处理[15]。
1.2 杨树HD-Zip转录因子基因的生物信息学分析
从杨树基因组数据库(http://planttfdb.cbi.pku.edu.cn/)中获得了63条转录因子氨基酸序列,从拟南芥基因组数据库(http://www.arabidopsis.org/)中得到48条HD-Zip转录因子氨基酸序列。利用BioEdit对杨树HD-Zip转录因子蛋白序列进行比对分析,利用ClustalX2.1和MEME(Version 4.10.0)对杨树HD-Zip转录因子蛋白序列进行基因保守元件(conserved motifs)的多元比较分析[16]。
将获得的杨树HD-Zip转录因子蛋白氨基酸序列放入ExPaSy在线Protparam软件(http://web/expasy/org/protparam/)进行分析,获得HD-Zip基因的蛋白分子量和等电点等理化属性。用PBIL在线网站(https://npsa-prabi.ibcp.fr/)对63条杨树HD-Zip转录因子蛋白序列的二级结构进行预测分析,并用Swiss-Model程序从四个亚家族中随机抽取3条蛋白序列进行三级结构同源建模。
2 结果与分析
2.1 盐胁迫对杨树HD-Zip转录因子基因表达影响
根据本实验室之前的盐胁迫处理实验,用0.15 mol·L-1盐溶液处理杨树苗24 h后叶片出现干枯现象,故以0.15 mol·L-1NaCl溶液作为胁迫处理杨树的最大浓度。将杨树苗浸泡在水和0.15 mol·L-1NaCl溶液中24 h,分别收集根、茎、叶样品,经转录组测序分析发现,在叶中除了17个基因在盐胁迫条件下没有表达量变化以外,有25个HD-Zip基因下调表达,21个基因上调表达(表1);茎中有10个基因没有表达量变化,42个基因下调表达,11个基因上调表达;根中有13个基因没有表达量变化,26个基因下调表达,24个基因上调表达(图1A),其中,在叶中上调表达2倍以上的有5个,茎中上调表达2倍以上的有5个,最高为5.14倍,根中上调表达2倍以上的有7个,最高为8.25倍。除此以外,Potri.014G103000.1、Potri.001G083700.1和Potri.007G008200.1等5个基因在盐胁迫条件下均在根、茎、叶中显著上调表达,Potri.017G081700.1、Potri.001G372300.1和Potri.008G148200.1等14个基因在根、茎、叶中均显著下调表达(图1B),而在叶中上调4.55倍的Potri.007G029500.1基因在茎和根中表现下调趋势,还有的基因如Potri.009G023600.1、Potri.012G139300.1和Potri.001G229700.1在叶和根中表达量均显著增加,在茎中却呈现下调趋势。结果表明了,杨树HD-Zip转录因子家族各成员在不同的组织中参与植物的抗逆过程,从而实现高盐应答胁迫。
2.2 杨树HD-Zip转录因子家族生物信息学分析
利用BioEdit对63条杨树HD-Zip转录因子蛋白结构域和48条拟南芥HD-Zip转录因子蛋白结构域进行序列比对,根据拟南芥HD-Zip基因家族的分类[17], 将杨树的63个HD-Zip基因同样分成四个亚家族HD-ZipⅠ、HD-ZipⅡ、HD-ZipⅢ和HD-ZipⅣ(表2),从表中可以看出,杨树有22个HD-Zip基因属于HD-ZipⅠ亚家族,17个基因属于HD-ZipⅡ亚家族,8个基因属于HD-ZipⅢ亚家族,16个基因属于HD-ZipⅣ亚家族。
表1HD-Zip家族基因应答盐胁迫的相对表达量
Table1RelativeexpressionofHD-Zipfamilygenesundersaltstress

基因IDGeneID叶中相对表达量Relativeexpressioninleaves茎中相对表达量Relativeexpressioninstems根中相对表达量RelativeexpressioninrootsPotri.006G071600.10.022±0.001-1.383±0.0048.246±0.686Potri.004G020400.10.048±0.007-0.438±0.0170.048±0.003Potri.008G129500.10.108±0.022-0.403±0.0110.912±0.037Potri.018G045100.10.228±0.0010-1.826±0.144Potri.003G052400.10.250±0.0201.045±0.017-0.282±0.025Potri.002G113400.10.336±0.040-1.502±0.069-1.185±0.064Potri.010G112600.10.403±0.0142.912±0.2060.699±0.101Potri.014G045100.10.445±0.016-0.716±0.0321.212±0.026Potri.015G065400.10.477±0.0960.350±0.013-2.969±0.098Potri.007G135100.10.481±0.021-2.785±0.036-1.293±0.132Potri.001G229700.10.606±0.129-0.103±0.0331.791±0.709Potri.007G008200.10.733±0.0480.365±0.0380.357±0.025Potri.012G139300.10.794±0.014-0.355±0.0244.411±0.315Potri.012G070900.11.078±0.0380.845±0.107-1.408±0.521Potri.009G023600.11.420±0.269-0.022±0.0012.389±0.510Potri.002G136400.11.488±0.0330.993±0.0211.488±0.016Potri.012G023700.12.199±0.1892.547±0.0113.805±0.102Potri.002G176300.12.870±0.1052.089±0.195-1.191±0.976Potri.007G029500.14.555±0.138-0.020±0.004-0.144±0.061Potri.001G083700.14.642±0.1313.847±0.5321.209±0.121Potri.014G103000.14.843±0.0595.143±0.3821.116±0.143

图1 盐胁迫条件下杨树HD-Zip家族基因表达 A.HD-Zip家族基因在不同组织中的表达;B.上调表达基因和下调表达基因Fig.1 Expression of HD-Zip family genes in poplar by salt stress A.Expression of HD-Zip family genes in different tissues; B.Up-regulated and down-regulated genes
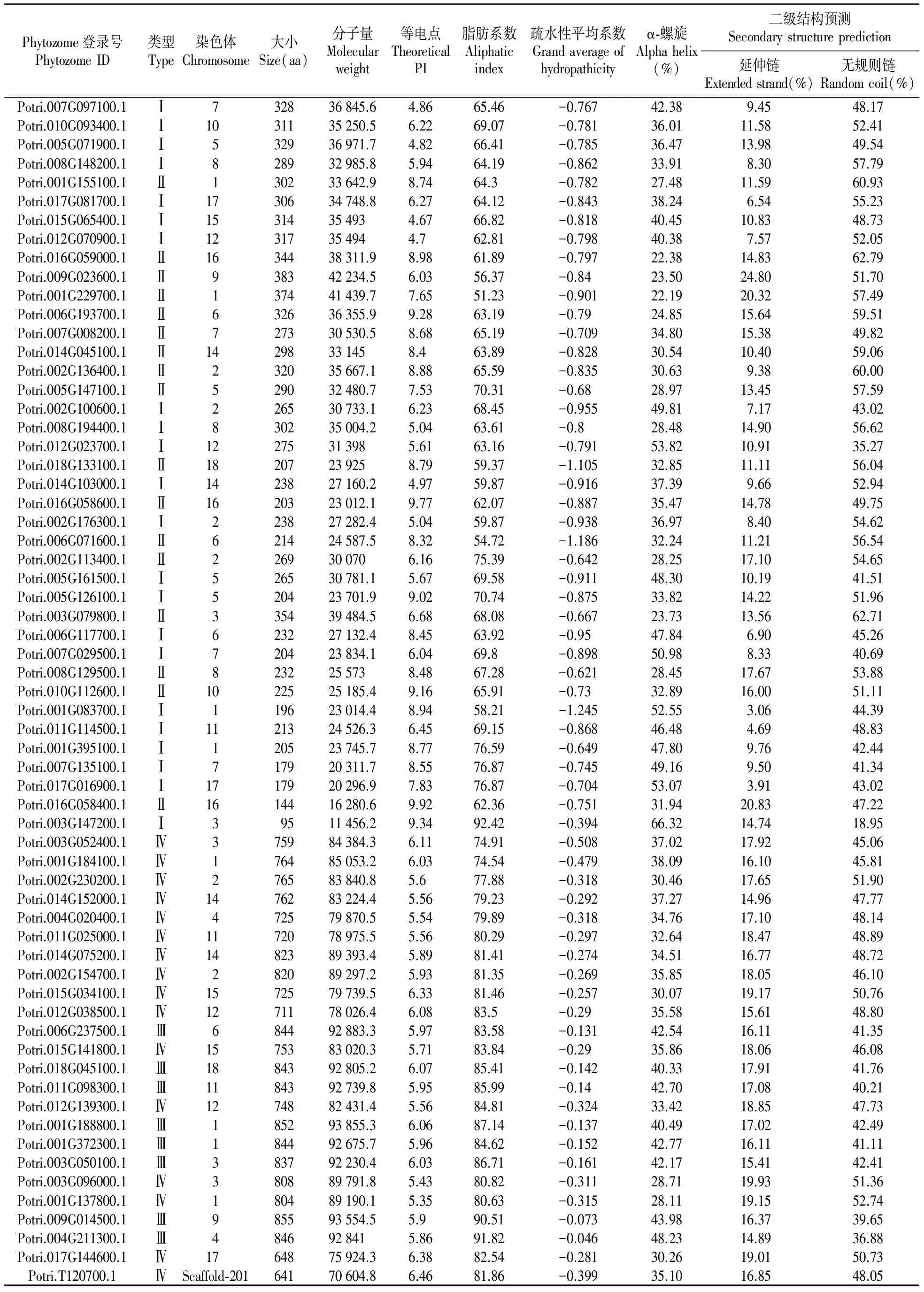
Phytozome登录号PhytozomeID类型Type染色体Chromosome大小Size(aa)分子量Molecularweight等电点TheoreticalPI脂肪系数Aliphaticindex疏水性平均系数Grandaverageofhydropathicityα⁃螺旋Alphahelix(%)二级结构预测Secondarystructureprediction延伸链Extendedstrand(%)无规则链Randomcoil(%)Potri.007G097100.1Ⅰ732836845.64.8665.46-0.76742.389.4548.17Potri.010G093400.1Ⅰ1031135250.56.2269.07-0.78136.0111.5852.41Potri.005G071900.1Ⅰ532936971.74.8266.41-0.78536.4713.9849.54Potri.008G148200.1Ⅰ828932985.85.9464.19-0.86233.918.3057.79Potri.001G155100.1Ⅱ130233642.98.7464.3-0.78227.4811.5960.93Potri.017G081700.1Ⅰ1730634748.86.2764.12-0.84338.246.5455.23Potri.015G065400.1Ⅰ15314354934.6766.82-0.81840.4510.8348.73Potri.012G070900.1Ⅰ12317354944.762.81-0.79840.387.5752.05Potri.016G059000.1Ⅱ1634438311.98.9861.89-0.79722.3814.8362.79Potri.009G023600.1Ⅱ938342234.56.0356.37-0.8423.5024.8051.70Potri.001G229700.1Ⅱ137441439.77.6551.23-0.90122.1920.3257.49Potri.006G193700.1Ⅱ632636355.99.2863.19-0.7924.8515.6459.51Potri.007G008200.1Ⅱ727330530.58.6865.19-0.70934.8015.3849.82Potri.014G045100.1Ⅱ14298331458.463.89-0.82830.5410.4059.06Potri.002G136400.1Ⅱ232035667.18.8865.59-0.83530.639.3860.00Potri.005G147100.1Ⅱ529032480.77.5370.31-0.6828.9713.4557.59Potri.002G100600.1Ⅰ226530733.16.2368.45-0.95549.817.1743.02Potri.008G194400.1Ⅰ830235004.25.0463.61-0.828.4814.9056.62Potri.012G023700.1Ⅰ12275313985.6163.16-0.79153.8210.9135.27Potri.018G133100.1Ⅱ18207239258.7959.37-1.10532.8511.1156.04Potri.014G103000.1Ⅰ1423827160.24.9759.87-0.91637.399.6652.94Potri.016G058600.1Ⅱ1620323012.19.7762.07-0.88735.4714.7849.75Potri.002G176300.1Ⅰ223827282.45.0459.87-0.93836.978.4054.62Potri.006G071600.1Ⅱ621424587.58.3254.72-1.18632.2411.2156.54Potri.002G113400.1Ⅱ2269300706.1675.39-0.64228.2517.1054.65Potri.005G161500.1Ⅰ526530781.15.6769.58-0.91148.3010.1941.51Potri.005G126100.1Ⅰ520423701.99.0270.74-0.87533.8214.2251.96Potri.003G079800.1Ⅱ335439484.56.6868.08-0.66723.7313.5662.71Potri.006G117700.1Ⅰ623227132.48.4563.92-0.9547.846.9045.26Potri.007G029500.1Ⅰ720423834.16.0469.8-0.89850.988.3340.69Potri.008G129500.1Ⅱ8232255738.4867.28-0.62128.4517.6753.88Potri.010G112600.1Ⅱ1022525185.49.1665.91-0.7332.8916.0051.11Potri.001G083700.1Ⅰ119623014.48.9458.21-1.24552.553.0644.39Potri.011G114500.1Ⅰ1121324526.36.4569.15-0.86846.484.6948.83Potri.001G395100.1Ⅰ120523745.78.7776.59-0.64947.809.7642.44Potri.007G135100.1Ⅰ717920311.78.5576.87-0.74549.169.5041.34Potri.017G016900.1Ⅰ1717920296.97.8376.87-0.70453.073.9143.02Potri.016G058400.1Ⅱ1614416280.69.9262.36-0.75131.9420.8347.22Potri.003G147200.1Ⅰ39511456.29.3492.42-0.39466.3214.7418.95Potri.003G052400.1Ⅳ375984384.36.1174.91-0.50837.0217.9245.06Potri.001G184100.1Ⅳ176485053.26.0374.54-0.47938.0916.1045.81Potri.002G230200.1Ⅳ276583840.85.677.88-0.31830.4617.6551.90Potri.014G152000.1Ⅳ1476283224.45.5679.23-0.29237.2714.9647.77Potri.004G020400.1Ⅳ472579870.55.5479.89-0.31834.7617.1048.14Potri.011G025000.1Ⅳ1172078975.55.5680.29-0.29732.6418.4748.89Potri.014G075200.1Ⅳ1482389393.45.8981.41-0.27434.5116.7748.72Potri.002G154700.1Ⅳ282089297.25.9381.35-0.26935.8518.0546.10Potri.015G034100.1Ⅳ1572579739.56.3381.46-0.25730.0719.1750.76Potri.012G038500.1Ⅳ1271178026.46.0883.5-0.2935.5815.6148.80Potri.006G237500.1Ⅲ684492883.35.9783.58-0.13142.5416.1141.35Potri.015G141800.1Ⅳ1575383020.35.7183.84-0.2935.8618.0646.08Potri.018G045100.1Ⅲ1884392805.26.0785.41-0.14240.3317.9141.76Potri.011G098300.1Ⅲ1184392739.85.9585.99-0.1442.7017.0840.21Potri.012G139300.1Ⅳ1274882431.45.5684.81-0.32433.4218.8547.73Potri.001G188800.1Ⅲ185293855.36.0687.14-0.13740.4917.0242.49Potri.001G372300.1Ⅲ184492675.75.9684.62-0.15242.7716.1141.11Potri.003G050100.1Ⅲ383792230.46.0386.71-0.16142.1715.4142.41Potri.003G096000.1Ⅳ380889791.85.4380.82-0.31128.7119.9351.36Potri.001G137800.1Ⅳ180489190.15.3580.63-0.31528.1119.1552.74Potri.009G014500.1Ⅲ985593554.55.990.51-0.07343.9816.3739.65Potri.004G211300.1Ⅲ4846928415.8691.82-0.04648.2314.8936.88Potri.017G144600.1Ⅳ1764875924.36.3882.54-0.28130.2619.0150.73Potri.T120700.1ⅣScaffold⁃20164170604.86.4681.86-0.39935.1016.85 48.05
分析杨树HD-Zip转录因子基因家族的理化性质可知,杨树HD-Zip转录因子基因家族的氨基酸个数在95~855,分布在HD-ZipⅠ和HD-ZipⅡ亚家族的基因氨基酸个数较少,一般在100~400个,而HD-ZipⅠ亚家族中的Potri.003G147200.1仅有95个氨基酸。分布在HD-ZipⅢ和HD-ZipⅣ亚家族中的基因氨基酸个数在641~855,氨基酸个数最多的是HD-ZipⅢ中的Potri.009G014500.1,多达855个。杨树HD-Zip转录因子基因家族蛋白的等电点大多在酸性范围内,脂肪系数均在51.23以上,最高达到92.42,这表明了HD-Zip转录因子家族的蛋白热稳定性较高。其疏水性平均系数均为负数,说明HD-Zip蛋白均表现为亲水性。63个杨树HD-Zip蛋白序列中主要有α-螺旋、延伸链和无规则卷曲结构且三种结构交错排列。利用planttfdb数据库找到63个杨树HD-Zip转录因子氨基酸序列,根据popgenie在线网站上Chromosome Diagram功能,进行染色体定位发现,HD-Zip基因在杨树染色体上呈不均匀分布,在第1、2、3号染色体上分布较多,分别有8、6、5条基因,在其它染色体上分布较少,第4、9、10、16和18号染色体上均只存在2条HD-Zip基因,并且在第13和第19号染色体上不存在HD-Zip基因。HD-Zip基因在染色体上的这种不均衡分布可能跟杨树进化过程中基因的功能特性相关。
利用MEME在线软件确定了20个保守序列(表3),根据20个氨基酸保守序列可将63个杨树HD-Zip基因分成四类,与系统发育树的分类一致。对63条HD-Zip基因分析得到,基序1和基序2编码高度保守的HD结构域,基序17编码亮氨酸拉链结构域(LZ),而基序3、7、8、9、11、12、13、14、16、18可能与HD-ZipⅢ和HD-ZipⅣ中的START结构域的形成有关,基序4、5、6、10、13、15、19、20则可能与仅存在与HD-ZipⅢ中的MEKHLA结构域的编码有关。不同的亚家族包含有不同的保守结构域,这可能与HD-Zip基因蛋白特殊的生物学功能相关。
利用Swiss-Model分别HD-Zip 4个亚家族中的各个成员进行三级结构的同源建模,结果表明,HD-ZipⅠ、HD-ZipⅡ和HD-ZipⅣ亚家族中蛋白的三级结构大体相同,而HD-ZipⅢ亚家族中的蛋白三级结构的构象之间略有不同,这可能与HD-ZipⅢ中基因的特殊保守域MEKHLA相关,从而使HD-ZipⅢ中不同的成员具有不同的功能[18],而且四个亚家族之间的蛋白构型也存在一定的差异。分析其二级结构发现,HD-ZipⅠ亚家族中蛋白的α-螺旋所占比例偏大,HD-ZipⅡ亚家族中蛋白的无规则卷曲结构所占比例偏大,而HD-ZipⅢ和HD-ZipⅣ亚家族中蛋白的延伸链的含量较HD-ZipⅠ和HD-ZipⅡ偏大,因此不同亚家族之间蛋白三级结构的差异可能与蛋白中的α-螺旋、延伸链和无规则卷曲结构的含量有关。
表3HD-Zip基序的长度和保守氨基酸序列
Table3ThelengthandconservedaminoacidsequenceofHD-Zipmotifs

基序Motif长度Length保守氨基酸序列ConservedaminoacidsequenceMotif150LGLRPRQVEVWFQNRRARTKLKQTEVDCEFLKRCCENLTAENRRLQKEVQMotif229RYHRHTARQIQEMEAMFKECPHPDDKQRMMotif329GVAGNYNGALQVMTAEFQVPSPLVPTRENYFVRYCKQMotif450IEPKQIKVWFQNRRCREKQRKEASRLQTVNRKLTAMNKLLMEENDRLQKQMotif541KATGTAVEWVQMPGMKPGPDSIGIVAISHGCTGVAARACGLMotif650PVFTFANQAGLDMLETTLVALQDITLEKIFDDHGRKTLCSEFPQIMQQGFMotif724THQLYRPLISSGMGFGAQRWIATLMotif841TSFWLPVPPKRVFDFLRDENTRNEWDILSNGGVVQEMAHIAMotif941QESCTDSTGSYVIYAPVDISAMNIVLSGGDPDYVALLPSGFMotif1050DVFLLQLCCGMDENAVGTCAELIFAPIDATFADDAPLLPSGFRIIPLDSGMotif1115LPSGCLIQDMPNGYSMotif1229RMVISFCAGVSASTAHTWTTLSGTGADDVMotif1332SLLTVAFQILVNSLPTAKLTVESVETVNNLISMotif1441RSEASRESAVVIMNHVNLVEILMDANQWSTIFCGIVSRAMTMotif1550VLCAKASMLLQNVPPAILLRFLREHRSEWADSGIDAYAAAAVKAGPCSLPMotif1630DKPMIIELAVAAMEELVRMAQMDEPLWMGSMotif1721PATTLTMCPSCERVATTTTSSMotif1841FDEQKLRIENAQLKEELDRVSSIAAKYIGRPISQLPPVQPVMotif1929CLPGGICLSSMGRPVSYERAVAWKVLNEEMotif2029VGLEPTRVAEILKDRPSWFRDCRAVDVLN
3 讨论
HD-Zip转录因子在植物的生长发育过程中发挥重要功能,不同的亚家族转录因子发挥不同的功能,如HD-ZipⅠ蛋白在番茄的生长发育过程中参与果实成熟的调控[19],HD-ZipⅡ亚家族中基因的过表达诱导拟南芥的早花表型[20],HD-ZipⅢ中的成员调控木质部维管束形成[21]、顶端分生组织的发生[22],HD-ZipⅣ亚家族中的基因成员在玉米中过表达后抑制表皮毛的形成[23]等。本文通过转录组测序比较了盐胁迫条件下杨树HD-Zip转录因子基因在根、茎、叶中的差异性表达,找到了盐胁迫下在叶中高表达的21个HD-Zip基因,茎中11个高表达的HD-Zip基因以及根中24个高表达的HD-Zip基因,分析发现Potri.014G103000.1等5个基因经过高盐处理后均显著上调表达,Potri.017G081700.1等14个基因在根、茎、叶中均下调表达,而有的基因则选择性地在不同的组织中高表达,表明了杨树HD-Zip转录因子家族各成员在不同的组织中参与植物的抗逆过程,从而发挥盐胁迫应答作用。
本文利用生物信息学的方法分析了63个杨树HD-Zip转录因子基因的结构及其相关信息,通过与拟南芥HD-Zip转录因子家族中的48个基因进行比较以及对其包含的保守结构域进行分析将其分为四个亚家族,并分析了整个家族中63个基因的理化性质和染色体定位,得到分布在17条染色体上的63个HD-Zip转录因子蛋白大多处于酸性范围内,而等电点值较大的基因蛋白大多含有较多的亮氨酸等碱性氨基酸,有助于形成亮氨酸拉链保守结构域,从而介导蛋白二聚体的形成;通过蛋白二级结构和三级结构的预测分析发现HD-ZipⅠ、HD-ZipⅡ和HD-ZipⅣ亚家族中各成员蛋白结构相似,而HD-ZipⅢ中各成员蛋白结构略有不同,可能与其特殊的结构域有关。统计得到,盐胁迫条件下表现出上调表达趋势的21个HD-Zip转录因子基因除Potri.018G045100.1属于HD-ZipⅢ亚家族外,有3个基因属于HD-ZipⅣ亚家族,而属于HD-ZipⅠ和HD-ZipⅡ亚家族的分别有8个和9个;在根、茎、叶中应答盐胁迫均呈上调表达趋势的基因除Potri.007G008200.1属于HD-ZipⅡ亚家族外,其余都属于HD-ZipⅠ亚家族。这些基因在染色体上呈不均匀分布,其氨基酸个数与其分子量不大,除了Potri.001G083700.1基因的等电点处于碱性范围外,其它基因均处于酸性范围内,其脂肪系数均在58.21以上,蛋白质的热稳定性较高,疏水性平均值在-0.791~-1.245,是亲水性极强的蛋白,这些基因所表现出的较强的胁迫应答能力可能与其特殊的理化性质以及HD-ZipⅠ亚家族中转录因子的HD和LZ结构域有关,而HD-ZipⅢ和HD-ZipⅣ中的START和MEKHLA结构域在胁迫处理环境中可能抑制基因的表达。总之,通过RNA-Seq和生物信息学方法的分析,将进一步为研究杨树HD-Zip转录因子家族蛋白在应答逆境胁迫方面提供有效依据。
1.Yanagisawas.Dof domain proteins:plant-specific transcription factors associated with diverse phenomena unique to plants[J].Plant and Cell Physiology,2004,45(4):386-391.
2.Schena M,Davis R W.HD-Zip proteins:members of anArabidopsishomeodomain protein superfamily[J].Proceedings of the National Academy of Sciences of the United States of America,1992,89(9):3894-3898.
3.Bharathan G,Janssen B J,Kellogg E A,et al.Did homeodomain proteins duplicate before the origin of angiosperms,fungi,andmetazoa?[J].Proceedings of the National Academy of Sciences of the United States of America,1997,94(25):13749-13753.
4.Jain M,Tyagi A K,Khurana J P.Genome-wide identification,classification,evolutionary expansion and expression analyses of homeobox genes in rice[J].The FEBS Journal,2008,275(11):2845-2861.
5.Sessa G,Steindler C,Morelli G,et al.TheArabidopsisAthb-8,-9 and -14 genes are members of a small gene family coding for highly related HD-ZIP proteins[J].Plant Molecular Biology,1998,38(4):609-622.
6.Olsson A S,Engström P,Söderman E.The homeobox genesATHB12 andATHB7 encode potential regulators of growth in response to water deficit inArabidopsis[J].Plant Molecular Biology,2004,55(5):663-677.
7.Tron A E,Bertoncini C W,Chan R L,et al.Redox regulation of plant homeodomain transcription factors[J].Journal of Biological Chemistry,2002,277(38):34800-34807.
8.Sawa S,Ohgishi M,Goda H,et al.TheHAT2 gene,a member of the HD-Zip gene family,isolated as an auxin inducible gene by DNA microarray screening,affects auxin response inArabidopsis[J].The Plant Journal,2002,32(6):1011-1022.
9.Prigge M J,Otsuga D,Alonso J M,et al.ClassⅢ homeodomain-leucine zipper gene family members have overlapping,antagonistic,and distinct roles inArabidopsisdevelopment[J].The Plant Cell,2005,17(1):61-76.
10.Otsuga D,Deguzman B,Prigge M J,et al.REVOLUTAregulates meristem initiation at lateral positions[J].The Plant Journal,2001,25(2):223-236.
11.Nakamura M,Katsumata H,Abe M,et al.Characterization of the class Ⅳ homeodomain-leucine zipper gene family inArabidopsis[J].Plant Physiology,2006,141(4):1363-1375.
12.Luo D L,Oppenheimer D G.Genetic control of trichome branch number inArabidopsis:the roles of theFURCAloci[J].Development,1999,126(24):5547-5557.
13.Agalou A,Purwantomo S,Övernäse,et al.A genome-wide survey of HD-Zip genes in rice and analysis of drought-responsive family members[J].Plant Molecular Biology,2008,66(1-2):87-103.
14.Hu R B,Chi X Y,Chai GH,et al.Genome-wide identification,evolutionary expansion,and expression profile of homeodomain-leucine zipper gene family in poplar(Populustrichocarpa)[J].PLoS One,2012,7(2):e31149.
15.Yao W J,Wang S J,Zhou B R,et al.Transgenic poplar overexpressing the endogenous transcription factor ERF76 gene improves salinity tolerance[J].Tree Physiology,2016,36(7):896-908.
16.Bailey T L,Elkan C.Fitting a mixture model by expectation maximization to discover motifs in biopolymers[J].Proceeding International Conference on Intelligent Systems for Molecular Biology,1994,2:28-36.
17.Aso K,Kato M,Banks JA,et al.Characterization of homeodomain-leucine zipper genes in the fernCeratopterisrichardiiand the evolution of the homeodomain-leucine zipper gene family in vascular plants[J].Molecular Biology and Evolution,1999,16(4):544-552.
19.Lin Z F,Hong Y G,Yin M G,et al.A tomato HD-Zip homeobox protein,LeHB-1,plays an important role in floral organogenesis and ripening[J].The Plant Journal,2008,55(2):301-310.
20.Dezar C A,Giacomelli J I,Manavella P A,et al.HAHB10,a sunflower HD-ZipⅡ transcription factor,participates in the induction of flowering and in the control of phytohormone-mediated responses to biotic stress[J].Journal of Experimental Botany,2011,62(3):1061-1076.
21.Itoh J I,Hibara K I,Sato Y,et al.Developmental role and auxin responsiveness of classⅢ homeodomain leucine zipper gene family members in rice[J].Plant Physiology,2008,147(4):1960-1975.
22.Kim J,Jung J H,Reyes J L,et al.microRNA-directed cleavage ofATHB15mRNA regulates vascular development inArabidopsisinflorescence stems[J].The Plant Journal,2005,42(1):84-94.
23.Vernoud V,Laigle G,Rozier F,et al.The HD-ZIPⅣ transcription factor OCL4 is necessary for trichome patterning and anther development in maize[J].The Plant Journal,2009,59(6):883-894.
National 863 project(2013AA102701)
introduction:ZHANG Xue-Mei(1991—),female,master,mainly engaged in the forestry research of stress resistance.
date:2017-07-14
BioinformaticsandResponsetoSaltStressAnalysisoftheHD-ZipTranscriptionFactorFamilyinPopulussimonii×P.nigra
ZHANG Xue-Mei YAO Wen-Jing ZHAO Kai JIANG Ting-Bo ZHOU Bo-Ru*
(State Key Laboratory of Tree Genetics and Breeding,Northeast Forestry University,Harbin 150040)
The HD-Zip transcription factor family is a kind of special protein in plant, playing crucial roles in plant development processes and abiotic stresses response. The protein family includes a highly conserved homeodomain(HD) and a leucine zipper domain(LZ), forming a special structural model. The HD-Zip transcription factor family, including 63 members, can be classified into 4 subfamilies HD-Zip Ⅰ, HD-Zip Ⅱ, HD-Zip Ⅲ and HD-Zip Ⅳ. RNA-Seq analysis of poplar roots, stems and leaves under salt stress was conducted for the differentially expressed genes in different tissues, and reveal the molecular mechanism of the response to stress environment. Therewere 25 HD-Zip genes down-regulated and 21 HD-Zip genes up-regulated in leaves under salt treatment, 42 genes down-regulated and 11 genes up-regulated in stems, and 26 genes down-regulated and 24 genes up-regulated in roots. According to gene annotations in Arabidopsis thaliana, the functions of HD-Zip transcription factor in poplarwere predicted.The conserved domains, structural characteristics and chemical properties of HD-Zip family members were also studied by bioinformatics, providing reference for gene functions of HD-Zip family in poplar.
HD-Zip transcription factor;abiotic stresses;growth and development;Populussimonii×P.nigra;bioinformatics
国家863课题资助(2013AA102701)
张雪梅(1991—),女,硕士研究生,主要从事林木抗逆机理方面研究。
* 通信作者:E-mail:boruzhou@yahoo.com
2017-07-14
* Corresponding author:E-mail:boruzhou@yahoo.com
S722.3+6
A
10.7525/j.issn.1673-5102.2017.05.011
