日本晴/中嘉早17重组自交系产量性状QTL定位
2017-11-07张应洲罗荣剑圣忠华焦桂爱唐绍清胡培松魏祥进
张应洲,罗荣剑,圣忠华,焦桂爱,唐绍清,胡培松,魏祥进
日本晴/中嘉早17重组自交系产量性状QTL定位
张应洲1, 2,罗荣剑1,圣忠华1,焦桂爱1,唐绍清1,胡培松1,魏祥进1
(1中国水稻研究所/水稻生物学国家重点实验室,杭州 310006;2杭州师范大学生命与环境学院,杭州311121)
【】对产量相关性状进行多年、多环境的QTL分析,寻找能够稳定遗传的产量性状主效QTL,剖析超级早籼稻中嘉早17的高产机理,为选育高产新品种提供有用信息。【】以日本晴×中嘉早17构建的重组自交系群体为研究材料。筛选亲本间多态性SSR标记,对群体各家系进行基因型分析,利用mapmarker/exp 3.0构建分子遗传连锁图谱。群体于2015—2016年,两地三季种植于杭州、海南和杭州,成熟期考察有效穗数、每穗粒数、单株产量、结实率、千粒重、粒长、粒宽和粒厚等产量相关性状。运用Windows QTL Cartographer 2.5检测产量相关性状QTL,运用QTL Network 2.2检测QTL与环境互作效应。【】构建的连锁图谱共包含163对SSR标记,73%的标记父母本基因型比例符合1﹕1理论分离比,23%标记显著偏分离,主要偏向父本中嘉早17,图谱总图距约1 479.4 cM,标记间平均距离约为9.08 cM。3个环境下共检测到46个QTL,分布于除第11染色体外的其他染色体上,贡献率变幅为3.78%—25.45%。共有10个QTL在3个环境下能被重复检测到,分别是控制有效穗数的、、,控制每穗粒数的、,控制结实率的,控制千粒重的,控制粒长的和,控制粒宽的;其中、、、和的增效等位来自亲本日本晴;而、、、和的增效等位来自亲本中嘉早17;除此之外,所检测到的每穗粒数、结实率、粒长和单株产量QTL中大部分增效等位基因均来自中嘉早17。产量性状与环境互作分析显示,控制每穗粒数和、控制结实率的和、控制单株产量的和等6个QTL与环境互作效应显著或极显著。此外,在第1、2、7染色体某区段多个与产量相关的QTL成簇分布。【】以日本晴×中嘉早17构建的重组自交系群体连锁图谱具有丰富的多态性标记,覆盖水稻基因组的93.64%,可较好地满足水稻重要农艺性状QTL定位要求。利用该套群体检测到多个产量相关性状QTL,其中,多数控制每穗粒数、结实率、粒长和单株产量的QTL的增效等位基因均来自中嘉早17。该结果与中嘉早17的每穗粒数、结实率、单株产量、千粒重和粒长等性状显著明显优于日本晴的结果一致,这些产量增效QTL可能是中嘉早17高产、稳产的遗传基础。
水稻;产量性状;QTL;重组自交系;中嘉早17
0 引言
【研究意义】水稻是世界重要粮食作物,世界上1/3以上的人口以水稻为主食,在中国水稻约占粮食播种面积与总产量的27%和35%[1]。作为中国第一大口粮作物,水稻承担了中国约60%人口的口粮。因此,提高水稻产量一直都是水稻育种的重要目标[2]。水稻产量主要由每株穗数、每穗粒数和千粒重等因素构成,这些性状都属于多基因控制的数量性状。因此,发掘控制水稻产量性状的优异资源并对水稻产量性状的遗传机制进行解析,将为高产水稻品种的选育提供重要的指导意义。【前人研究进展】随着分子遗传学的快速发展,水稻产量性状的数量性状位点(QTL)定位与克隆取得了巨大进步。据不完全统计[3-4],目前,单株产量共检测到至少80个QTL,分布于全部12条染色体上,其中第1、7染色体上检测频次较高;有效穗数共检测到207个QTL,分布于全部12条染色体上,其中第1、2、4、7染色体上检测的频次较高;每穗粒数共检测到90多个QTL,分布于除第5染色体外的11条染色体上,其中第2、3、6、7、11染色体上检测的频次较高;结实率共检测到50多个QTL,分布于全部12条染色体上,其中第2、6染色体上检测的频次较高;千粒重共检测到160多个QTL,分布于全部12条染色体上,其中在第1、2、3、5、6、10、11染色体上检测的频次较高。水稻粒型很大程度上直接决定着千粒重,也是研究者们关注的重点。目前,已经定位了100多个粒长QTL、90多个粒宽QTL,这些QTL分布于12条染色体上,其中第1、3、5染色体上检测的频次较高[5-6]。虽然水稻产量性状相关QTL数量众多,但是只有一些主效QTL被克隆。其中,主要包括调控水稻穗粒数的QTL/基因,如[7]、[8]、[9]、[10]、[11]、[12]等;控制水稻粒型或粒重的QTL/基因,如[13]、[14]、[15]、[16]、[17]、[18]、[19]、[20]、[21]和[22]等。【本研究切入点】尽管在水稻产量相关性状分子机理方面已取得长足的进步,然而,由于产量相关性状的复杂性、受多基因调控以及易受环境影响的特点,水稻产量形成的遗传及调控机制仍不清楚,一些精细定位或已克隆的基因在育种实践中难以得到广泛运用。因此,更多的水稻产量相关QTL/基因的定位与克隆将为水稻高产分子育种提供更完备理论基础与基因资源。中嘉早17是2009年通过国家审定的超级早籼稻品种,由于其高产、稳产、适合加工等优点,2010—2017年连续8年被农业部推荐为全国水稻主导品种。目前,年推广面积超过66.67万公顷,已经成为近年来中国南方稻区推广面积最大的水稻品种。但是中嘉早17高产、稳产的遗传机理尚未明确。【拟解决的关键问题】以粳稻品种日本晴和中嘉早17为亲本衍生了一套RIL群体,为RIL群体构建了分子连锁图谱,同时考察RIL群体多年多环境的产量相关性状,检测产量相关性状QTL。以期阐明中嘉早17的高产机理,挖掘水稻新的产量相关QTL/基因,为超高产品种育种提供理论基础和基因资源。
1 材料与方法
1.1 研究材料
以粳稻日本晴为母本,超级稻中嘉早17为父本杂交产生F1,F1自交后得到F2群体,以后各株系均利用单粒传法连续自交直至第10代,最终获得由200个株系组成的重组自交系(RIL)群体。RIL群体2015年7月种植于杭州富阳(2015HZ);2016年1月种植于海南陵水(2016HN);2016年6月种植于杭州富阳(2016HZ)。材料的种植均按亲本和群体每个株系种植64株(8行×8列),株行间距为20 cm×20 cm,区块间距25 cm,单本栽插,田间管理同大田生产。
1.2 连锁图谱的构建
在分蘖盛期采集亲本及RIL群体各家系叶片,按CTAB法提取DNA[23]。针对全基因组选用629对SSR引物(序列来自Gramene网站,由上海英骏生物公司合成)对亲本日本晴和中嘉早17进行多态性筛选。根据多态标记在物理图谱中的位置,挑选出均匀覆盖水稻12条染色体的多态性SSR标记,对RIL群体200个家系进行基因型检测和分析。通过PCR和聚丙烯酰胺凝胶电泳获得SSR标记的基因型分型数据。分别将中嘉早17和日本晴相似的带型标记为“1”和“2”、杂合带和模糊带型分别记为“3”和“0”。应用Mapmaker/EXP3.0对筛选的标记进行连锁分析[24],以LOD值为3.0确定连锁群内标记顺序,采用Kosambi函数将重组值转换成遗传图距离(cM),利用mapdraw 2.1绘制染色体,构建连锁图谱。
1.3 产量相关农艺性状考查
于成熟期,每个家系选取表型基本一致的5个单株(剔除边行),参考Moncada等[25]提出的方法考查8个性状,分别为有效穗数(effective panicles,EP)、每穗粒数(number of grain per panicle,NGPP)、结实率(seed setting rate,SSR)、单株产量(yield per plant,YPP)、千粒重(1 000-grain-weight,TGW)、粒长(grain length,GL)、粒宽(grain width,GW)和粒厚(grain thickness,GT)。以5个单株平均数作为分析的基本数值。
1.4 QTL定位
采用Windows QTL Cartographer 2.5软件[26],以复合区间作图法(CIM),在全基因组范围内检测产量相关性状QTL。以Permutation 1 000次运算确定LOD阈值。当某个位置检测到的LOD值大于LOD阈值时,就认为该位置存在1个QTL。QTL命名法则遵循Blair等[27]的原则。同时,基于MICM法,采用QTLNetwork2.2[28]联合检测3个环境下产量相关性状QTL与环境的互作效应。
2 结果
2.1 日本晴×中嘉早17重组自交系的分子遗传连锁图谱
从629对SSR标记中共检测到246对在亲本间表现多态性,挑选出均匀覆盖水稻12条染色体的163对多态性标记,检测群体家系的基因型,获得163个标记的基因型数据。经卡方检验,73%标记符合1﹕1单基因理论分离比,27%标记显著偏离1﹕1分离比,发生偏分离的标记大部分偏向父本中嘉早17。利用163个SSR标记分析RIL群体基因型,其中有效带型(1,2)占总数的99.25%,杂合带和模糊带型(3,0)仅占0.75%。父本中嘉早17基因型分布频率见图1。在200个株系中,父本基因型频率变幅13%—83%,平均值为56%;在163个标记中,父本基因型频率为37%—85%,平均值为56%。不同个体和标记的基因型频率分布基本符合正态分布,表明该研究群体具有较好的遗传构成。应用Mapmaker/EXP3.0对163个多态性标记间进行连锁分析,构建覆盖12条染色体的连锁图谱(表1)。每条染色体连锁群标记数为9—22个,遗传距离变幅78.0—190.2 cM。遗传连锁图谱总图距为1 479.40 cM,覆盖整个基因组的93.85%,标记间平均图距为9.08 cM,变幅为0.8—26.7 cM,达到QTL作图的要求。
2.2 群体及其亲本产量相关性状表型
对RIL群体及其亲本的8个产量相关农艺性状在3种环境下的表型值统计分析发现,RIL群体各性状表现明显分离,连续分布且有超亲分离现象(表2、图2)。产量相关性状在不同环境下有一定的差异,其中2015HZ和2016HZ的有效穗数平均值少于2016HN;2015HZ和2016HZ的每穗粒数、单株产量和结实率3个性状平均值明显高于2016HN;千粒重、粒长、粒宽、粒厚在3个环境下基本无差异,认为这4个性状受环境的影响较小。RIL群体各性状的峰值和偏值都较小,其中粒长和粒宽3个环境下峰值均>1,每穗粒数和结实率在2015HZ、2016HZ峰值和偏值均>1,而其他性状的峰值介于-0.50—0.55,偏值介于-0.64—0.99,基本符合数量性状遗传的正态分布特点。

表1 遗传图谱信息

图1 父本基因型频率的分布
2.3 产量相关性状之间的相关性
相关性分析表明,RILs群体大部分产量性状之间存在显著的相关性(表3)。部分性状间相关性在3个环境下方向一致。其中,有效穗数与每穗粒数在3个环境中均呈现显著负相关(≤-0.493 );单株产量和每穗粒数及结实率呈显著正相关(≥0.451;≥0.211);千粒重与粒长及粒宽呈显著正相关(≥0.427;≥0.387)。以上结果表明,产量性状是复杂的性状,各性状间关系紧密,相互作用共同影响水稻产量。同时性状间相关系数受环境的影响,不同环境间会出现变化。
2.4 产量相关性状的QTL定位
对3个环境下产量相关性状QTL定位结果见表4和图3。在3个环境下共检测到46个QTL,分布于除第11染色体外的其他染色体不同区间,LOD值介于2.50—15.47,贡献率介于3.78%—25.45%。其中,10个QTL在3个环境下能被重复检测到,分别为、、、、、、、、和。
各产量相关性状QTL具体分布为有效穗数:2015HZ、2016HN和2016HZ 3个环境下共检测到5个QTL,分布于第1、2、4和7染色体,LOD值介于2.79—8.57,贡献率介于3.78%—14.96%。所检测到的QTL中,、、在 3个环境重复检测到,3个环境平均LOD值分别为4.20、5.42和5.19,平均贡献率分别为7.14%、9.20%和8.62%,平均加性效应值分别为-0.70、-0.77和0.84。和增效等位来自日本晴,增效等位基因来自中嘉早17。

表2 RIL群体与亲本间水稻产量性状的表型分析
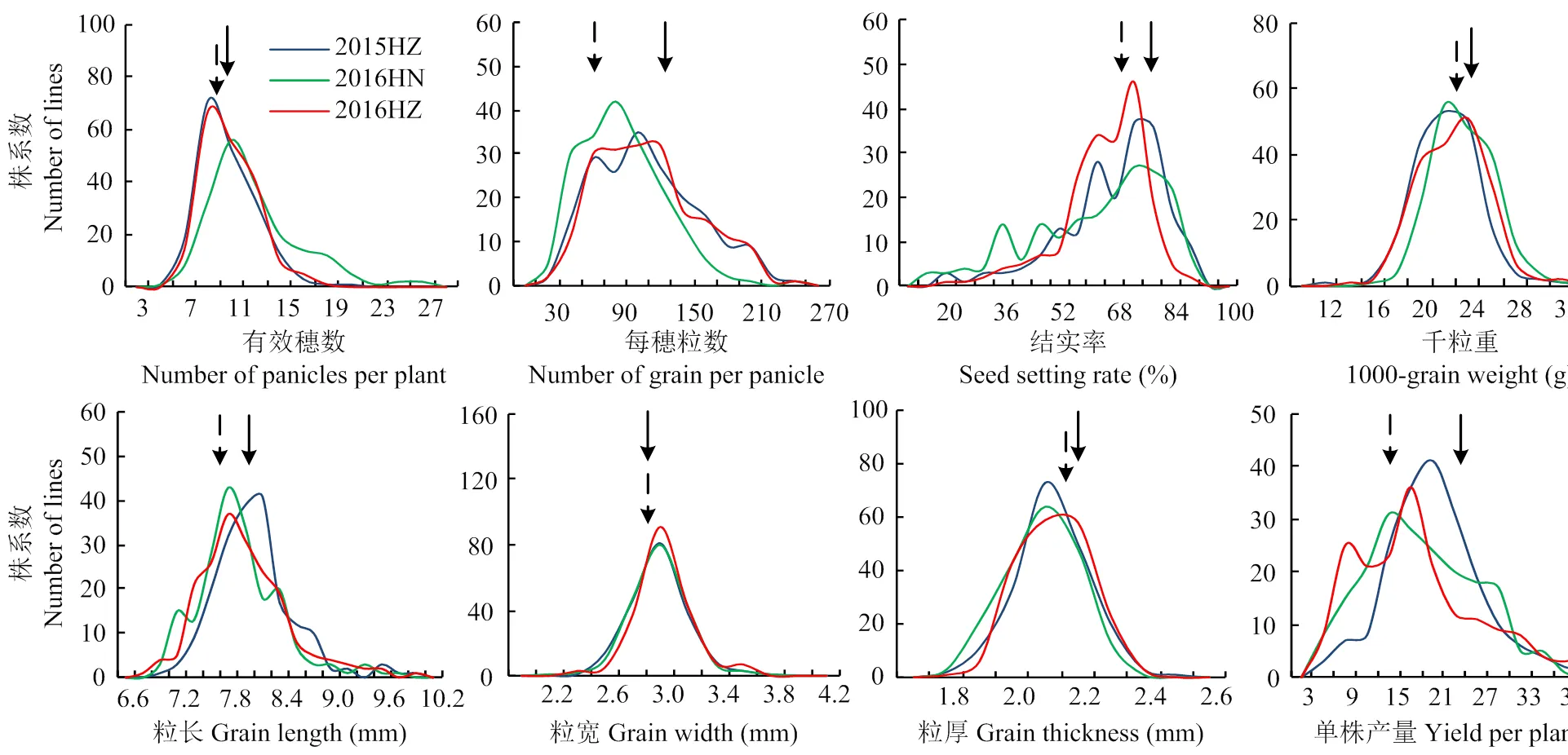
虚线箭头指示日本晴表型平均值;实线箭头指示中嘉早17表型平均值
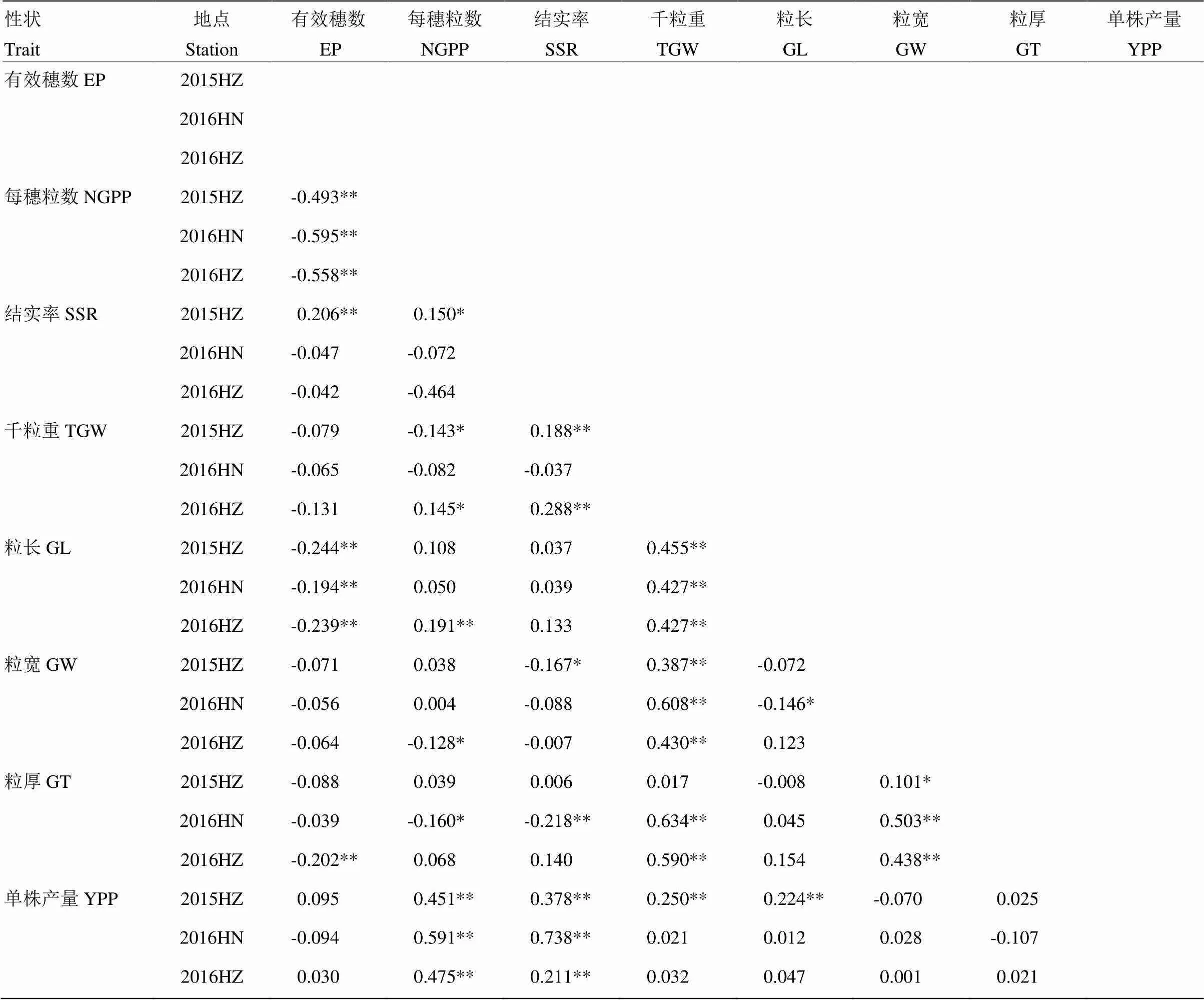
表3 不同产量相关性状间的相关系数
*、**分别表示<0.05、<0.01显著水平。下同
* and** mean significant levels at<0.05 and<0.01, respectively. The same as below
每穗粒数:2015HZ、2016HN和2016HZ 3个环境下共检测到5个每穗粒数QTL,分布于第1、6、7、8和10染色体,LOD值介于2.93—15.47,贡献率介于3.90%—25.45%。其中、在3个环境下重复检测到,平均LOD值分别为5.77和11.83,平均贡献率分别为10.96%和20.09%,平均加性效应值分别为13.62、19.05。增效等位来自中嘉早17,增效等位基因来自日本晴。
结实率:2015HZ、2016HN和2016HZ 3个环境下共检测到7个影响结实率的QTL,分布在第1、5、6、7、8和12染色体上,LOD值介于2.57—7.81,贡献率介于4.84%—14.90%,增效等位基因全部来自中嘉早17。其中在3个环境下重复检测到,平均LOD值为4.71,平均贡献率为9.18%,平均加性效应值为3.01。
千粒重:2015HZ、2016HN和2016HZ 3个环境下共检测到4个千粒重QTL,分别位于第1、2、3和6染色体上,LOD值介于2.50—4.91,贡献率介于6.35%—7.95%。其中只有在3个环境下重复检测到,平均LOD值为4.10,平均贡献率为8.39%,平均加性效应值为-0.80,增效等位基因来自日本晴,而其他千粒重QTL增效等位基因多数来自中嘉早17。
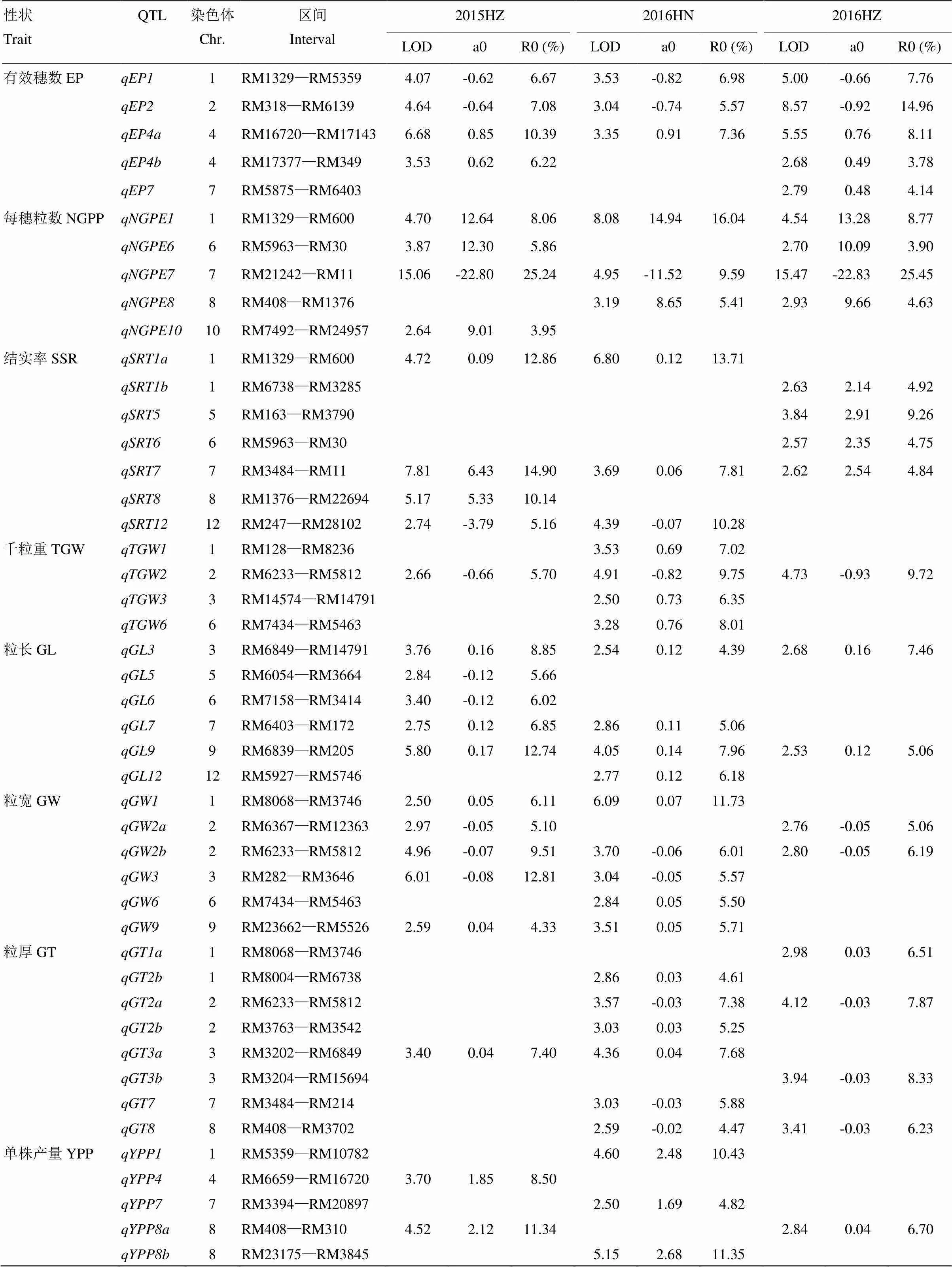
表4 检测到的产量相关性状QTL分布情况

E1:2015杭州;E2:2016海南;E3:2016杭州;“+”和“-”分表代表增效等位基因来自中嘉早17和日本晴
粒长:2015HZ、2016HN和2016HZ 3个环境下共检测到6个粒长QTL,分布在第3、5、6、7、9和12染色体上,LOD值介于2.53—5.80,贡献率介于4.39%—12.74%。其中3个环境下重复检测到和,平均LOD值分别为2.99和4.29,平均贡献率分别为6.90%和8.59%,平均加性效应值分别为0.99和0.14,这两个QTL增效等位基因均来自中嘉早17。
粒宽:2015HZ、2016HN和2016HZ 3个环境下共检测到6个粒宽QTL,分布在第1、2、3、6和9染色体上,LOD值介于2.59—6.09,贡献率介于4.33%—12.81%。其中在3个环境下重复检测到,平均LOD值分别为3.82,平均贡献率分别为7.24%,平均加性效应值分别为-0.06,增效等位基因均来自日本晴。
粒厚:2015HZ、2016HN和2016HZ 3个环境下共检测到8个粒厚QTL,分布于第1、2、3、7和8染色体上,LOD值介于2.59—4.36,贡献率介于4.47%—8.33%。无3个环境下重复检测到的QTL,但是多数QTL增效等位基因来自中嘉早17。
单株产量:2015HZ、2016HN和2016HZ 3个环境下共检测到5个单株产量QTL,分别位于第1、4、7和8染色体上,LOD值介于2.50—5.15,贡献率介于4.80%—11.35%。所检测到的QTL增效等位基因均来自中嘉早17。
2.5 产量性状QTL环境互作
应用QTLNetwork2.2的MCIM分析方法联合检测产量相关性状与环境互作效应(表5),共检测到6个QTL与环境互作效应显著,分别为控制每穗粒数的和;控制结实率的和;控制单株产量的和。与环境互作效应QTL主要集中在第1染色体RM1329—RM600和第7染色体RM214—RM11区间。表明每穗粒数、结实率的和单株产量性状易受环境影响。

表5 产量性状QTL与环境的互作效应
3 讨论
3.1 籼粳交分子连锁图谱
籼稻与粳稻是具有一定生殖隔离的2个主要栽培稻亚种,它们之间在形态性状、农艺性状和生理生化反应等方面存在明显的差异。本研究构建RIL群体选用的亲本为粳稻品种日本晴和籼稻品种中嘉早17。其中中嘉早17连续多年被中国农业部推荐为南方主栽水稻品种,具有高产、稳产等优良特征;而另一亲本日本晴具有最为完整的水稻基因组参考序列。同时中嘉早17的每穗粒数、结实率、单株产量、千粒重和粒长等产量相关显著明显优于日本晴(表2)。另外籼粳亚种间杂交后代基因型具有较大的变幅,因此,利用日本晴和籼稻品种中嘉早17衍生的RIL群体非常适合构建分子连锁遗传图谱开展水稻重要农艺性状QTL定位研究。而本研究结果也显示利用该重组自交系构建的连锁图谱具有丰富的多态性标记(共有163个多态性标记),覆盖水稻基因组的93.64%,可较好地满足产量性状QTL定位需求。
在遗传作图研究中,标记基因型偏分离现象普遍存在。董少玲等[29]以TD70和Kasalath构建的重组自交系,51.8%的标记显著偏分离,偏向父本TD70;沈希宏等[30]用协优9308和协青早B构建的重组自交系48.5%的标记显著偏分离,大部分偏向父本协优9308;徐小飒等[31]以培矮64和93-11构建的重组自交系,50.4%标记显著偏分离,大部分偏向父本培矮64。本研究构建的连锁图谱中有44个标记也出现了显著偏分离,占标记总标记的27.0%,其中大部分偏向父本中嘉早17。目前对偏分离的遗传机制尚不清楚,推测这可能亲本间的亲缘关系、雌雄配子体的选择性、群体构建过程中人工抽样有关。在实际运用中少量的偏分离标记对QTL检测的影响是有限的,会随着QTL间连锁距离的增大而消失[32]。
3.2 QTL定位比较
在不同生态环境下检测农艺性状QTL,在理论上可以减少环境因素的影响,有利于发现在多种环境下稳定表达的主效QTL。本研究在两年三季3个环境下开展了水稻产量相关性状的QTL分析,共检测到了46个QTL。分析发现,位置存在已克隆的控制有效穗数基因[33];而4a在3个环境下均能被检测到,平均贡献率为7.62%,目前尚未发现有关报道。与占小登等[34]发现的有效穗数QTL位于相同的区域。与Chen[35]精细定位的抽穗期和穗粒数QTL在相近的区间。、和区间分别包含已经克隆同时控制抽穗期、株高、穗粒数的多功能基因位点[36]、[11]和[12]。和区间分别包含已经克隆的控制粒长的基因[37][38];和区间分别覆盖已被克隆、报道控制粒宽的功能基因[13]和[16]。上述结果表明,本研究检测到的主效QTL大部分与前人定位或克隆的产量相关基因位置一致,同时本研究也发现了一些新的主效QTL,如控制有效穗数的4a等(图3)。另外,本研究发现的多个产量相关主效QTL其有利于增加产量的等位基因均来自中嘉早17,如控制有效穗数的4a,控制每穗粒数的,控制粒长的和,控制粒长的。单株产量与结实率相关QTL的增效等位基因也大部分来自中嘉早17。该研究结果与中嘉早17的每穗粒数、结实率、单株产量、千粒重和粒长等产量相关明显优于日本晴的结果一致,这些产量增效QTL可能是保证中嘉早17高产稳产的遗传基础。
3.3 产量QTL与环境互作
水稻产量相关性状多属于遗传机制较为复杂的数量性状,其表现型不仅受主效基因和微效基因多基因调控、同时易受环境条件的影响。在不同的群体或者不同环境条件下控制同一性状的QTL表达情况有一定差异,即使同一群体在不同环境下检测到的QTL也不尽一致,因此,环境是影响产量相关性QTL表达的重要因素之一[39]。占小登等[34]在2个环境下共检测到产量相关性状30个,其中10个能在不同环境下重复检测到,5个与环境互作显著;苏相文等[3]在2个环境下共检测到产量相关性状31个,8个能重复检测到,21个对环境敏感;杨占烈等[40]在3个环境条件下检测到26个QTL,4个能重复检测到;本研究利用RIL群体进行产量相关性状QTL定位,3个同环境下检测到46个QTL,其中10个QTL在3个环境下都能被重复检测到。这些能在不同环境中都能检测到的QTL可能与水稻品种的广适性有关。本研究也发现了6个产量性状QTL与环境互作效应显著(表5),这6个QTL主要是调控水稻结实率、每穗粒数及单株产量的QTL,说明这些性状对环境较为敏感。
4 结论
以日本晴×中嘉早17构建的重组自交系群体连锁图谱具有丰富的多态性标记,覆盖水稻基因组的93.64%,可较好地满足水稻重要农艺性状QTL定位要求。利用该套群体检测到多个产量相关性状QTL,其中,多数控制每穗粒数、结实率、粒长和单株产量的QTL的增效等位基因均来自中嘉早17。该结果与中嘉早17的每穗粒数、结实率、单株产量、千粒重和粒长等产量相关显著明显优于日本晴的结果一致,这些产量增效QTL可能是中嘉早17高产、稳产的遗传基础。
[1] 戴农. 水稻生产机械化发展现状、问题与思考. 现代农业装备, 2014(1):16-20.
DAI N. The status, problems and thinking in rice production mechanization., 2014(1):16-20. (in Chinese)
[2] 方福平, 程式华. 论中国水稻生产能力. 中国水稻科学, 2009, 23(6): 539-566.
Fang F P, CHENG S H. Rice production capacity in China., 2009, 23(6): 539-566. (in Chinese)
[3] 苏相文, 高方远, 曹墨菊, 任鄄胜, 陆贤军, 吴贤婷, 刘光春, 任光俊. 利用重组自交系剖析大穗型香稻保持系川香29B产量相关性状的遗传基础. 分子植物育种, 2015, 13(1): 39-50.
Su X W, Gao F Y, Cao M J, REN J S, LU X J, WU X T, LIU G C, REN G J. Genetic basis of the traits related to yield in rice maintainer line Chuan-xiang 29B with large panicle and aroma using recombinant inbred lines., 2015, 13(1): 39-50.
[4] Ikeda M, Kitano H, Matsuoka M... Springer New York, 2013: 227-235.
[5] Huang R, Jiang L, Zheng J, ZHENG J H, WANG T S, WANG H C, HUANG Y, HONG Z L. Genetic bases of rice grain shape: so many genetics, so little known., 2013, 18(4): 218-226.
[6] Zhu Y B, Guo Y C, Liang K J, SUN X L. Progress on the genes controlling grain shape of rice., 2015, 44(1): 1-7.
[7] Ashikari M, Sakakibara H, Lin S, YAMAMOTO T, TAKASHI T, NISHIMURA A, ANGELES E R, QIAN Q, KITANO H, MATSUOKA M. Cytokinin oxidase regulates rice grain production., 2005, 309(5735): 741-745.
[8] Yoshida A, Sasao M, Yasuno N, TAKAGI K, DAIMON Y, CHEN R H, YAMAZAKI R, TOKUNAGA H, KITAGUCHI Y, SATO Y, NAGAMURA Y, USHIJIMA T, KUMAMARU T, LIDA S, MAEKAWA M, KYOZUKA K. TAWAWA1, a regulator of rice inflorescence architecture, functions through the suppression of meristem phase transition., 2013, 110(2): 767.
[9] Huang X Z, Qian Q, Liu Z G, SUN H Y, HE S Y, LUO D, XIA G G, CHU C G, LI J Y, FU X D. Natural variation at the DEP1 locus enhances grain yield in rice., 2009, 41(4): 494.
[10] Jiao Y Q, Wang Y H, XUE D W, WANG J, YAN M X, LIU G F, DONG G J, ZENG D L, LU Z F, ZHU X D, QIAN Q, LI J Y. Regulation of OsSPL14 by OsmiR156 defines ideal plant architecture in rice., 2010, 42(6): 541-544.
[11] Xue W Y, Xing Y Z, Weng X Y, ZHAO Y, TANG W J, WANG L, ZHOU H J, YU S B, XU C G, LI X H, ZHANG Q F. Natural variation in Ghd7 is an important regulator of heading date and yield potential in rice., 2008, 40(6): 761.
[12] Wei X J, Xu J F, Guo H N, JIANG L, CHENG S H, YU C Y, ZHOU Z L, HU P S, ZHAI H Q, WAN J M. DTH8 suppresses flowering in rice, influencing plant height and yield potential stimultaneously., 2010, 153(4): 1747-1758.
[13] Song X J, Huang W, Shi M, ZHU M Z, LIN H X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase., 2007, 39(5): 623.
[14] Shomura A, Izawa T, Ebana K, ENBITANI T, KANEGAE H, KONISHI H, YANO M. Deletion in a gene associated with grain size increased yields during rice domestication., 2008, 40(8): 1023-1028.
[15] Chin J H, Chu S H, Jiang W, CHO Y, BASYIRIN R, DARSHAN S, KOH H. Identification of QTLs for hybrid fertility in inter-subspecific crosses of rice (, L.)., 2011, 33(1): 39-48.
[16] Mao H L, Sun S G, Yao J L, WANG C J, YU S B, XU C G, LI X G, ZHANG Q F. Linking differential domain functions of the GS3 protein to natural variation of grain size in rice., 2010, 107(45): 19579-19584.
[17] Li Y B, Fan C C, Xing Y, XING Y Z, JIANG Y H, LUO L J, SUN L, SHAO D, XU C J, LI C J, XIAO J H, HE Y Q, ZHANG Q F. Natural variation in GS5 plays an important role in regulating grain size and yield in rice., 2011, 43(12): 1266-1269.
[18] Sun L J, Li X J, Fu Y G, ZHU Z F, TAN L B, LIU F X, SUN X Y, SUN X W, XUN C Q. GS6, a member of the GRAS gene family, negatively regulates grain size in rice., 2013, 55(10): 938-949.
[19] Wang S K, Wu K, Yuan Q B, LIU X Y, LIU Z G, LIN X Y, ZENG R Z, ZHU H T, DONG G J, QIAN Q, ZHANG G Q, FU X D. Control of grain size, shape and quality by OsSPL16 in rice., 2012, 44(8): 950.
[20] Ishimaru K, Hirotsu N, Madoka Y, MURAKAMI N, HARE N, ONODERA H, KASHIWAGI T, UJIIE K, SHIMIZU B,ONISHI A, MIYAGAWA H, KATOH E. Loss of function of the IAA-glucose hydrolase gene TGW6 enhances rice grain weight and increases yield., 2013, 45(6): 707.
[21] Qi P, Lin Y S, Song X J, SHEN J B, HUANG W, SHAN J X, ZHU M Z, JIANG M Z, JIANG L W, GAO J P, LIN H X. The novel quantitative trait locus GL3.1 controls rice grain size and yield by regulating Cyclin-T1;3., 2012, 22(12): 1666-1680.
[22] Wang Y X, Xiong G S, Hu J, JIANG L, YU H, XU J, FANG Y X, ZENG L J, XU E, XU J, YE W J, MENG X B, LIU R F, CHEN H Q, JING Y H, JING Y H, WANG Y H, ZHU X D, LI J Y, QIAN Q. Copy number variation at the GL7 locus contributes to grain size diversity in rice., 2015, 47(8): 944-948.
[23] 李金路, 王硕, 于婧, 王玲, 周世良. 一种改良的植物DNA提取方法. 植物学报, 2013, 48(1): 72-78.
Li J L, Wang S, YU J, WANG L, ZHOU S L. A modified method for extracting plant DNA., 2013, 48(1): 72-78. (in Chinese)
[24] Lincoln S, Daly M, Lander E. Constructing genetic maps with MAPMAKER/EXP 3.0. 1992.
[25] Moncada P, Martinez C P, Borrero J, Chatel M, Gauch H, Guimaraes E, Tohme J, McCouch S. Quantitative trait loci for yield and yield components in an×BC2F2population evaluated in an upland environment., 2001, 102(1): 41-52.
[26] Wang S, Basten C J, Zeng Z B. Windows QTL cartographer 2.5 department of statistics. Raleigh, USA: North Carolina State University, 2006.
[27] Blair M W, Panaud O, McCouch S R. Inter-simple sequence repeat (ISSR) amplification for analysis of microsatellite motif frequency and fingerprinting in rice (L.)., 1999, 98(5): 780-792.
[28] Yang J, Hu C C, Ye X Z, YU R D, XIA Z, YE X Z, ZHU J. QTLNetwork-2.1 user manual. Hangzhou: Zhejiang University, 2005.
[29] 董少玲, 张颖慧, 张亚东, 陈涛, 赵庆勇, 朱镇, 周慧丽, 姚姝, 赵凌, 王才琳. 水稻重组自交系分子遗传图谱构建及分蘖角的 QTL检测. 江苏农业学报, 2012, 28(2): 10-16.
Dong S L, Zhang Y L, Zhang Y D, CHEN T, ZHAO Q Y, ZHU Z, ZHOU H L, YAO S, ZHAO L, WANG C L. Construction of molecular genetic linkage map based on a rice RIL populations and detection of QTL for tiller angle., 2012, 28(2): 10-16. (in Chinese)
[30] 沈希宏, 陈深广, 曹立勇, 占小登, 陈代波, 吴明伟, 程式华. 超级杂交稻协优 9308 重组自交系的分子遗传图谱构建. 分子植物育种, 2008, 6(5): 861-866.
Shen X H, Chen S G, Cao L Y, zhan x d, chen d b, wu m w, chenG s h. Construction of genetic linkage map based on RIL population derived from super hybrid rice XY9308., 2008, 6(5): 861-866. (in Chinese)
[31] 徐小飒, 刘喜, 赵志刚, 周裕军, 吴盛阳, 周蓉, 张俊杰, 江玲, 万建民. 培矮64S/93- 11重组自交系分子图谱构建及千粒重QTL检测. 南京农业大学学报, 2011, 34(1): 8-13.
Xu X S, Liu X, Zhao Z G, ZHOU Y J, WU S Y, ZHOU R, ZHANG J J, JIANG L, WAN J M. Construction of genetic linkage map based on a RILs population derived from the hybrid rice Peiai 64S / 93- 11 and detection of QTL for 1000-grain weight., 2011, 34(1): 8-13. (in Chinese)
[32] Zhang L, Wang S, Li H, DENG Q M, ZHENG A P, LI S C, LI P, LI Z L, WANG J K. Effects of missing marker and segregation distortion on QTL mapping in F2 populations., 2010, 121(6): 1071-1082.
[33] Guo S, Xu Y, Liu H, MAO Z, ZHANG C. MA Y, ZHANG q, chong k. The interaction between OsMADS57 and OsTB1 modulates rice tillering via DWARF14., 2013, 4: 1566-1577.
[34] 占小登, 于萍, 林泽川, 陈代波, 沈希宏, 张迎信, 付君林, 程式华, 曹立勇. 利用大粒籼/小粒粳重组自交系定位水稻生育期及产量相关性状 QTL. 中国水稻科学, 2014, 28(6): 570-580.
Zhan X D, Yu P, Lin Z C, CHEN D B, SHEN X H, ZHANG Y X, FU J L, CHENG S H, CAO L Y. QTL mapping of heading date and yield-related traits in rice using recombination inbred lines (RILs) population derived from BG1/XJL., 2014, 28(6): 570-580. (in Chinese)
[35] Chen J H. Fine mapping of qHd1, a minor heading date QTL with pleiotropism for yield traits in rice (L.)., 2014, 127(11): 2515-2524.
[36] Yano M, Harushima Y, Nagamura Y, Kurata N, Minobe Y, Sasaki T. Identification of quantitative trait loci controlling heading date in rice using a high-density linkage map., 1997, 95(7): 1025-1032.
[37] HEANG D, SASSA H. Antagonistic actions of HLH/bHLH proteins are involved in grain length and weight in rice., 2012, 7(2): e31325.
[38] Nakagawa H, Mori M. Short grain1 decreases organ elongation and brassinosteroid response in rice., 2012, 158(3): 1208.
[39] 赵建国, 蒋开锋, 杨莉, 杨乾华, 万先奇, 曹应江, 游书梅, 罗婧, 张涛. 水稻产量相关性状 QTL定位. 中国水稻科学, 2013, 27(4): 344-352.
Zhao J G, Jiang K F, Yang L, Yang Q H, WAN X Q, CAO Y J, YOU S M, LUO Q, ZHANG T. QTL mapping for yield related components in a RIL population of rice., 2013, 27(4): 344-352. (in Chinese)
[40] 杨占烈, 戴高兴, 翟荣荣, 林泽川, 王会民, 曹利用, 程式华. 多环境条件下超级杂交稻协优9308重组自交系群体粒形性状的 QTL分析. 中国水稻科学, 2013, 27(5): 482-490.
Yang Z L, Dai G X, ZHAI R R, LIN Z C, WANG H M, CAI L Y, CHENG S H. QTL analysis of rice grain shape traits by using recombinant inbred lines from super hybrid rice 9308 in multi-environments.2013, 27(5): 482-490. (in Chinese)
(责任编辑 李莉,岳梅)
QTL mapping of yield associated traits of Nipponbare ×Zhongjiazao 17 RIL population
ZHANG Yingzhou1,2, LUO Rongjian1, SHENG Zhonghua1, JIAO Guiai1, TANG Shaoqing1, HU Peisong1, WEI Xiangjin1
(1China National Rice Research Institute/Sate Key Laboratory of Rice Biology, Hangzhou 310006;2College of Life and Environmental Sciences, Hangzhou Normal University, Hangzhou 311121)
【】QTL mapping for yield traits were conducted with phenotype data collected from multi-environments, in order to identify stable QTL for yield traits and analyze the mechanism of high yield of super earlyrice Zhongjiazao 17, which will provide useful information for breeding of new varieties with higher yield and better comprehensive characteristics in rice. 【】A recombinant inbred lines (RILs) population derived from Nipponbare × Zhongjiazao 17 were used as experimental materials. Then a genetic linkage map was constructed by Mapmaker/EXP 3.0 based on the RILs genotypes which analyzed by polymorphism SSR markers. During 2015-2016, the RILs and two parents were grown in the experimental fields in Hangzhou, Hainan and Hangzhou. The agronomic characters including effective panicles, grain number per panicle, seed setting rate, 1 000-grain-weight, grain length, grain width and grain thickness and yield per plant were investigated. QTL mapping for these traits were detected by Windows QTL Cartographer 2.5 and environment interaction effect was detected by QTLNetwork2.2.【】The genetic map was constructed with 163 filtered SSR markers and covered about 1 479.4 cM with an average interval of 9.08 cM. The genotypes of male and female parent of 73.0% makers were segregated as 1﹕1; only 23.0% markers were distorted segregation which was inclined to Zhongjiazao 17.A total of 46 QTLs were mapped on all chromosomes except the 11th with the contribution rate ranged from 3.78% to 25.45%. Ten QTLs, including,,(QTL for effective panicles),,(for grain number per panicle),(for seed setting rate),(for 1 000-grain-weight),,(for grain length),(for grain width) can be detected on all three environments. The increasing effective allele of,,,,and most QTL for grain number per panicle, seed setting rate, grain length and yield per plant come from Zhongjiazao 17. Furthermore, some QTLs about different traits exist as clusters on chromosome 1, 2, 7, respectively. Six yield associated traits QTL were also found can significantly interact with environment. 【】 The linkage map of RIL derived from Nipponbare × Zhongjiazao 17 has abundant polymorphic makers which cover 93.64% of the rice whole genome, so it is very suitable for QTL mapping for important agronomic traits. Many stable QTLs about rice yield traits were detected using this mapping population, and among them, the alleles which can increase yield of many QTL of grain number per panicle, seed setting rate, grain length and yield per plant were from Zhongjiazao 17. The results were consistent with that the most of these yield traits of Zhongjiazao 17 were better than Nipponbare. These increasing effective alleles of yield associated traits QTL may be the genetic basis of stable high yield of Zhongjiazao 17.
rice; yield associated traits; QTL; RILs population; Zhongjiazao 17
10.3864/j.issn.0578-1752.2017.19.002
2017-03-10;接受日期:2017-05-03
国家重点研发计划(2016YFD0101801)
张应洲,E-mail:zhangyingzhoufly@yeah.net。通信作者魏祥进,Tel:0571-63370080;E-mail:weixiangjin@caas.cn
