基于指纹相似度的药物靶点相互作用预测
2017-10-20于亚运刘勇国蒋羽
于亚运 刘勇国 蒋羽


[摘要]药物通过结合人体靶点发挥药效,识别药物靶点相互作用對于药物新功能发现至关重要。该文提出基于分子子结构的靶点指纹特征和基于指纹相似度的药物靶点特征计算方法,构建随机森林分类模型识别和预测药物靶点相互作用,通过酶、离子通道、G蛋白偶联受体和核受体数据集测试并与现有方法对比分析,并将所提模型应用于中药成分靶点相互作用预测,实验结果表明所提方法的有效性。
[关键词]药物靶点相互作用; 分子指纹; 随机森林
Prediction of drugtarget interaction based on fingerprint similarity
YU Yayun1, LIU Yongguo1*, JIANG Yu1, LI Limin2
(1. Knowledge and Data Engineering Laboratory of Chinese Medicine, School of Information and Software Engineering,
University of Electronic Science and Technology of China, Chengdu 610054, China;
2. Sichuan Academy of Chinese Medicine Sciences, Chengdu 610041, China)
[Abstract]Drugs play the pharmacological effects by combining with target proteins. Identification of drugtarget interactions is important for discovering new functions of drugs. In this paper, the target fingerprints based on molecular substructure and the drugtarget similarity based on fingerprints are proposed to a random forestbased classification method, in order to identify the drugtarget interactions. Experiments on enzymes, ion channels, G proteincoupled receptors and nuclear receptors proved the effectiveness of the proposed method. In addition, the proposed method is applied to predict the interactions between ingredients and targets of traditional Chinese medicines.
[Key words]drugtarget interaction; molecular fingerprint; random forest
药物靶点是存在于人体组织细胞内与药物分子相互作用并赋予药物效应的特定蛋白质分子,如受体、酶等[1]。药物通过与靶点相互作用影响靶点的药理作用以达到表型效应[23]。识别药物靶点相互作用对于理解药物的作用机制至关重要,而药物靶点间关系尚未完全明确[4]。目前,通过生物实验手段识别药物靶点相互作用不仅代价高而且耗时长[5]。Mei等认为借助计算机可快速有效预测药物靶点相互作用,提出BLMNII模型从KEGG数据库识别药物D00163,D00506和D05341分别与靶点hsa9971,hsa9970和hsa3174具有相互作用,实现通过计算机技术在有限时间内大规模预测药物靶点相互作用,协助研究人员有效开展生物实验验证[67]。
近年来,研究人员基于计算机技术从不同角度研究药物靶点特征,构建机器学习模型预测药物靶点相互作用[8]。Yamanishi等从KEGG BRITE,BRENDA,SuperTarget和DrugBank数据库搜集药物靶点相互作用数据,根据靶点类型创建酶(enzyme)、离子通道(ion channel)、G蛋白偶联受体(Gproteincoupled receptor)和核受体(nuclear receptor)数据集,基于化学结构和基因序列信息采用统计方法分析药物靶点相互作用,采用ROC曲线下面积(area under curve,AUC)作为评价指标,4个数据集的AUC值分别为0904,0851,0899,0843[9]。van Laarhoven等通过二值矩阵表示药物靶点相互作用,以二值向量表示药物(或靶点)是否与靶点(或药物)具有相互作用,采用高斯函数计算药物(或靶点)间高斯距离矩阵作为高斯相互作用属性(Gaussian interaction profile,GIP)核,提出基于GIP核的正则化最小二乘法分类器预测药物靶点相互作用,算法AUC值分别达到0983,0986,0947,0906[10];Cao等认为药物化学子结构指纹能有效地表示药物,根据药物maccs指纹特征和靶点蛋白的物理化学性质构建药物靶点属性特征,采用支持向量机构建模型预测药物靶点相互作用,算法AUC值分别达到0903 1,0889 1,0846 8,0837 4[5];Hao等采用非线性核融合思想构建正则最小二乘法预测药物靶点相互作用,算法AUC值分别达到0915,0925,0853,0909[11]。上述方法从不同角度构建药物靶点相互作用关系预测模型,对实验数据集获得较高AUC值,表明此类方法能较好预测潜在药物靶点相互作用。endprint
论文采用药物分子子结构特征表示药物指纹特征,根据药物指纹特征和药物靶点相互作用关系设计靶点指纹特征,通过多个相似度计算方法度量药物靶点的指纹相似度,提出基于指纹相似度的随机森林(fingerprint similaritybased random forest,FSRF)模型分析药物靶点相互作用关系。
1数据
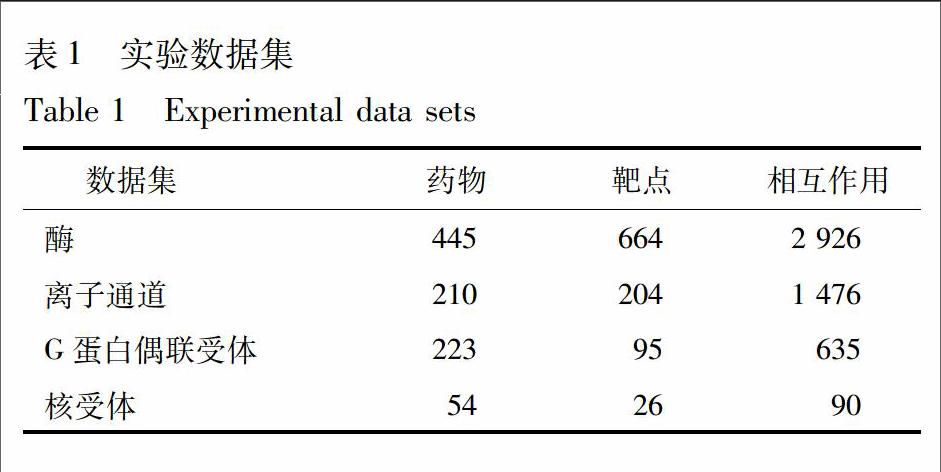
论文采用文献[9]的药物靶点数据集,包含酶、离子通道、G蛋白偶联受体和核受体4个子数据集,数据集的药物数目、靶点数目和药物靶点已知相互作用数目见表1。此外,论文采用DrugBank数据库[12]最新药物靶点相互作用数据验证FSRF模型预测结果的有效性。
3结果
31分类模型评估仿真实验基于Windows 10操作系统,酷睿i3处理器,8GB内存,R软件开发工具。根据药物KEGG标识符提取药物SMILES编码并确定药物指纹特征。为讨论不同药物分子指纹对分类模型的影响,论文采用maccs指纹、pubchem指纹和standard指纹开展药物靶点相互作用分析。将实验数据集中已知相互作用的药物靶点对视为正样本,将未知相互作用的药物靶点对视为负样本,按照正负样本1∶1比例随机无放回选择负样本。以maccs,pubchem和standard分子指纹分别构建分类模型(MFSRF,PFSRF和SFSRF),采用五折交叉验证,通过构建混淆矩阵以计算AUC、准确率(accuracy)、精确度(precision)、召回率(recall)、F度量(Fmeasure)评价指标,分类结果见表2。可见,针对不同数据集论文分类方法AUC值、准确率和F度量均高于095,精确度和召回率接近1,表明基于指纹相似度的随机森林模型能准确识别药物靶点相互作用,其中SFSRF模型分类结果最好。
32分类模型对比分析为与现有方法对比分析,论文选择BLM模型[3]、BGL模型[9]、RLS模型[10]、RLSKF(Max)和RLSKF(Avg)模型[11]、RF(Chem+Bio+net)和RF(Chem+Bio)模型[13]、NetLapRLS模型[14],以AUC指标评价开展对比分析,结果见表3。SFSRF模型对酶、离子通道、G蛋白偶联受体和核受体数据集的AUC值分别达0998,0994,0994,0995,表明该模型具有更高的分类有效性。
33未知药物靶点相互作用预测SFSRF算法的药物靶点相互作用预测结果见表4,其中未知作用数目表示数据集中未知药物靶点相互作用数目,验证数目表示未知相互作用中被DrugBank数据库验证数目,验证比例表示未知相互作用被DrugBank数据库验证比例;预测数目表示SFSRF模型预测存在药物靶点相互作用数目,识别数目表示预测的相互作用中被DrugBank数据库验证的数目,识别比例表示预测的数目被DrugBank数据库验证的比例,识别验证比为识别比例和验证比例的比值,体现SFSRF模型预测未知相互作用的能力。
由表4可见,SFSRF模型的识别验证比达到56~104,表明其能提高未知药物靶点相互作用预测能力。实验数据集共有355 782对未知相互作用,其中666对被DrugBank数据库验证,验证比为0001 87。SFSRF模型预测2 111对,其中60对被DrugBank数据库验证,验证比例为0028 42,识别验证比为152,表明SFSRF模型能显著提升药物靶点相互作用预测水平。此外,分类模型的平均预测率仍较低,分析发现酶数据集中292 554对未知相互作用仅81对被验证,导致未知相互作用预测难度加大;而SFSRF模型预测到1 262对相互作用,其中3对被验证,预测模型能显著缩小验证范围。针对离子通道、G蛋白偶联受体和核受体数据集,SFSRF模型均大幅度压缩预测范围且提升了预测率,对于降低药物研发成本,提高药物靶点预测效率具有重要作用。
34中药成分靶点相互作用预测以杞菊地黄丸为例分析基于SFSRF模型的中药成分靶点相互作用关系。论文通过TCMSP数据库[15]获取杞菊地黄丸的中药成分数据,通過DrugBank数据库获取已批准药物和药物靶点相互作用关系,基于SFSRF模型构建药物靶点关系预测模型,以预测中药成分靶点相互作用关系。为分析杞菊地黄丸对肝肾阴虚证的作用机制,通过筛选DrugBank数据库肾脏疾病治疗靶点形成中药成分靶点的相互作用关系,构建杞菊地黄丸的中药成分靶点网络,见图2,椭圆节点表示中药成分,菱形节点表示靶点。
杞菊地黄丸主要作用于靶点P11274,P02792和P02794,为验证中药成分靶点相互作用关系,从DrugBank数据库获取上述靶点并对靶点功能和已知药物进行分析。结果表明,靶点P11274为肾细胞癌抗原NYREN26,该靶点药物均为抗肿瘤药物。靶点P02792和P02794具有调节发育中肾囊细胞铁摄取功能。靶点P02792的药物为肝脏机能改善剂,
适用于急性肝炎、慢性迁延性肝炎、慢性活动性肝炎,对肝硬化、胆囊炎胆石症亦有效;用于患有慢性肾脏疾病的成人和6岁以上儿童患者接受血液透析和补充促红细胞生成素的治疗。论文21个靶点对应药物中,有13个靶点药物是治疗肝肾相关疾病药物,主要表现为对肾脏机能的改善,对肝炎、肝硬化的治疗,对肾病综合征的治疗,对肝细胞癌、晚期肾细胞癌的治疗等;有3个靶点药物主要用于降低眼内压和治疗视网膜炎。上述结果体现杞菊地黄丸的滋肾养肝明目功效。此外,靶点药物的利尿、补血、消炎镇痛等作用以及对肠胃溃疡、高血压等疾病的治疗与杞菊地黄丸中单味中药的功效存在一致性,如茯苓和泽泻的利水渗湿,山药的补脾养胃、麻醉镇痛,熟地黄的补血滋润等,见表5。
4讨论
论文采用药物指纹特征表示药物属性,根据药物靶点相互作用确定靶点指纹特征,以药物指纹特征与靶点指纹特征相似度为特征,以药物靶点是否具有相互作用作为类属性,基于随机森林算法构建分类模型。仿真实验表明,SFSRF模型能够获得较好分类结果,通过DrugBank数据库对未知作用预测结果验证,表明SFSRF模型具有较好药物靶点未知相互作用预测能力,能够为高效开展生物实验识别药物靶点相互作用提供支持,并将模型有效应用于杞菊地黄丸的中药成分靶点相互作用预测。endprint
[参考文献]
[1]屠鹏飞,曾克武,廖理曦,等 天然活性小分子靶标蛋白识别方法学研究进展[J]. 中国中药杂志,2016,41(1):6.
[2]Tabei Y, Pauwels E, Stoven V, et al. Identification of chemogenomic features from drugtarget interaction networks using interpretable classifiers[J]. Bioinformatics,2012,28(18):i487.
[3]Bleakley K, Yamanishi Y Supervised prediction of drugtarget interactions using bipartite local models[J]. Bioinformatics,2009,25(18):2397.
[4]Takarabe M, Kotera M, Nishimura Y, et al. Drug target prediction using adverse event report systems: a pharmacogenomic approach[J]. Bioinformatics,2012,28(18):i611.
[5]Cao D S, Liu S, Xu Q S, et al. Largescale prediction of drugtarget interactions using protein sequences and drug topological structures[J]. Anal Chim Acta,2012,752:1.
[6]Mei J P, Kwoh C K, Yang P, et al. Drugtarget interaction prediction by learning from local information and neighbors[J]. Bioinformatics,2013,29(2):238.
[7]Ding H, Takigawa I, Mamitsuka H, et al. Similaritybased machine learning methods for predicting drugtarget interactions: a brief review[J]. Brief Bioinform,2014,15(5):734.
[8]刘西,卢朋,左晓晗,等 基于二分图评价模型的网络药物靶标预测改进方法[J]. 中国中药杂志,2012,37(2):125.
[9]Yamanishi Y, Araki M A, Honda W, et al. Prediction of drugtarget interaction networks from the integration of chemical and genomic spaces[J]. Bioinformatics,2008,24(13):i232.
[10]van Laarhoven T, Nabuurs S B, Marchiori E Gaussian interaction profile kernels for predicting drugtarget interaction[J]. Bioinformatics,2011,27(21):3036.
[11]Hao M, Wang Y, Bryant S H Improved prediction of drugtarget interactions using regularized least squares integrating with kernel fusion technique[J]. Anal Chim Acta,2016,909:41.
[12]Wishart D S, Knox C, Guo A C, et al. DrugBank: a knowledgebase for drugs,drug actions and drug targets[J]. Nucleic Acids Res,2008,36:D901.
[13]Cao D S, Zhang L X, Tan G S, et al. Computational prediction of drugtarget interactions using chemical, biological, and network features[J]. Mol Inform,2014,33(10):669.
[14]Xia Z, Wu L Y, Zhou X, et al. Semisupervised drugprotein interaction prediction from heterogeneous biological spaces[J]. BMC Syst Biol,2010,4:S6.
[15]Ru J, Li P, Wang J, et al. TCMSP: a database of systems pharmacology for drug discovery from herbal medicines[J]. J Cheminform, 2014, 6(1):1.
[責任编辑张宁宁]endprint
