反义RNA介导GmFAD2-1B基因沉默增强大豆种子中油酸的高效累积
2017-10-17邢国杰贺红利郭东全杨向东
杨 静 邢国杰 牛 陆 贺红利 杜 茜 郭东全 袁 英 杨向东
吉林省农业科学院农业生物技术研究所 / 吉林省农业生物技术重点实验室, 吉林长春130033
反义RNA介导GmFAD2-1B基因沉默增强大豆种子中油酸的高效累积
杨 静 邢国杰 牛 陆 贺红利 杜 茜 郭东全 袁 英*杨向东*
吉林省农业科学院农业生物技术研究所 / 吉林省农业生物技术重点实验室, 吉林长春130033
油酸含量是评价大豆油食用品质和稳定性的重要指标。本研究采用农杆菌介导转化法, 将反义GmFAD2-1B基因导入栽培大豆品种, 获得油酸含量显著提高的转基因大豆新品系。Southern杂交检测表明, 外源GmFAD2-1B基因片段已导入大豆基因组, 其插入拷贝数为1~5个。qRT-PCR检测表明, 外源GmFAD2-1B主要在大豆种子中表达, 并导致种子中内源GmFAD2-1 mRNA表达水平显著降低, 而根、茎、叶、花组织中内源GmFAD2-1 mRNA表达水平无显著变化。脂肪酸组分分析表明, 12份转基因大豆种子油酸含量为27.38%~80.42%, 其中, L40和L72油酸含量分别为 68.91%~80.42%和 65.98%~80.22%, 较对照品种 Williams 82 (17.8%~22.0%)提高 2.65倍以上, 亚油酸降低至4.84%~14.55%, 饱和脂肪酸降低至 10.34%~11.16%, 但总脂肪和总蛋白含量与对照品种相比没有显著变化。农艺性状分析表明, 转基因大豆在熟期、株高、叶形、花色、结荚高度、百粒重等方面与对照品种也没有显著差异。
大豆; 油酸; GmFAD2-1B; 农杆菌介导转化法
Abstract:The content of oleic acid is usually taken as a crucial indicator of cooking quality and stability for soybean oil. Breeding soybean cultivars with high oleic acid has always been considered as one of the principal aims for soybean improvement. In this study, the anti-sense GmFAD2-1B gene from soybean was transformed into the cultivated soybean by Agrobacterium-mediated method and several soybean transgenic lines with dramatically increased oleic acid content were obtained. Southern blot analysis confirmed integration of the anti-sense GmFAD2-1B fragment into the soybean genome with one to five copies of T-DNA insertions in the selected independent transgenic plants. The results of qRT-PCR showed that the foreign anti-sense GmFAD2-1B was mainly expressed in the transgenic seeds and simultaneously resulted in a significant decrease of the endogenous GmFAD2-1 mRNA, however, there were no significant differences observed for GmFAD2-1 mRNA contents in the root, leaf,stem and flower tissues of the transgenic plants compared to the non-transgenic ones. Analysis of the fatty acid compositions indicated that the content of oleic acid increased to 27.38%–80.42% in twelve transgenic lines. Specifically, in transgenic lines L40 and L72, oleic acid content was raised to 68.91%–80.42% and 65.98%–80.22% respectively, with over 2.65-fold increase compared with the wild type Williams 82 (containing 17.8%–22.0% of oleic oil). Concomitantly, linoleic acid and saturated fatty acid were reduced to 4.84%–14.55% and 10.34%–11.16% respectively in the seeds of transgenic lines L40 and L72. However, there was no significant difference in total oil and protein contents, as well as agronomic traits such as maturity period, plant height, leaf shape, flower color, pod height and weight per 100 seeds between the transgenic lines and non-transformed controls.
Keywords:Soybean; Oleic acid; GmFAD2-1B; Agrobacterium-mediated transformation
大豆(Glycine max L. Merrill)是我国最重要的油料作物和饲料作物之一, 也是人类植物性蛋白和油脂的重要来源。大豆主要加工产品-大豆油约占我国食用植物油总量的 40%左右。大豆油中富含多种人体必需的脂肪酸, 包括硬脂酸(stearic acid, SA,18:0)、棕榈酸(palmitic acid, PA, 16:0)、油酸(oleic acid, OA,18:1Δ9)、亚油酸(linoleic acid, LA, 18:1Δ9,12)和亚麻酸(linolenic acid, ALA, 18:1Δ9,12,15), 其中, 亚油酸约占大豆总脂肪酸的 50%~60%, 亚麻酸约为6%~11%, 油酸含量约为20%~25%[1]。由于亚油酸和亚麻酸不饱和程度较高, 其热稳定性及抗氧化性能较差, 当暴露在空气中或烹调时易发生氧化作用,产生对人体健康有害的氧化产物, 并伴有难闻的气味, 在加工过程中也易产生对人体有害的反式脂肪酸。与之相反, 油酸作为一种单不饱和脂肪酸, 其稳定性高, 富含油酸的食用油可以长时间保存且高温条件下不易氧化变质; 同时, 油酸本身也具有较好的营养和保健功能, 在营养学界常被称为安全脂肪酸, 其含量是评价大豆油食用品质和稳定性的重要指标。高油酸也一直是我国大豆育种的重要目标之一。
在种子发育过程中, 质体中的脂肪酸合成酶复合物催化乙酰CoA合成脂肪酸, 并进入细胞质中合成三酯酰甘油[2]。作为LA合成的前体, OA是多不饱和脂肪酸合成途径中最关键的步骤。Δ12-脂肪酸脱饱和酶(delta-twelve fatty acid desaturase 2 enzyme,FAD2, EC 1.3.1.35)是催化这一生化过程的关键酶,决定种子中多不饱和脂肪酸的含量[3]。Okuley等[3]于1994年首次从拟南芥中克隆了FAD2基因, 之后国内外陆续从大豆[4]、油菜[5]、向日葵[6]、花生[7]、棉花[8]中克隆了多个 FAD2基因。研究表明, 植物FAD2基因家族各成员的功能有所不同[4,5]。目前已从大豆中分离了 3个 FAD2基因, 即 FAD2-1A、FAD2-1B和 FAD2-2。研究表明, FAD2-1A和FAD2-1B主要在大豆胚胎发育过程中表达, 而FAD2-2则为组成型表达[9]。大豆 FAD2-1A和FAD2-1B序列相似程度极高, 二者编码蛋白仅有24个氨基酸存在差异[9]。Tang等[10]研究表明, FAD2-1A和FAD2-1B对环境温度的敏感程度不同, FAD2-1A对温度具有较强的敏感性。进一步研究表明,FAD2-1蛋白185位丝氨酸(serine-185)磷酸化状态可能在调控FAD2-1稳定性方面发挥重要的作用[10]。
抑制或过表达 FAD2-1基因, 可以显著改变油酸/亚油酸比例[11-12]。Pham等[11]研究发现, FAD2-1A和 FAD2-1B双突变体大豆的油酸含量极显著提高(80%)。Li等[12]对大豆 FAD2-1B基因研究发现, 在正常的野生型酵母细胞中不存在亚油酸, 但在过表达FAD2-1B基因的酵母细胞中能够积累一定量的亚油酸。Zhang等[13]通过反义RNA介导转录后基因沉默抑制大豆内源 FAD2基因的表达, 获得了油酸含量高达51.71%的转基因大豆。Wang等[14]利用RNAi技术抑制大豆 FAD2基因, 获得了油酸含量为71.5%~81.9%的大豆品系。Haun等[15]利用人工核酶技术(Transcription activator-like effector nucleases,TALEN)对大豆内源FAD2-1A和FAD2-1B进行定点突变, 获得了油酸含量达 80%的转基因大豆。本研究采用农杆菌介导法, 拟将大豆β’-伴球蛋白基因启动子BCSP 驱动的反义GmFAD2-1B基因导入栽培大豆品种, 筛选高油酸转基因大豆新材料, 为下一步高油酸转基因大豆新品种培育及产业化奠定基础。
1 材料与方法
1.1 试验材料
栽培大豆品种Williams 82由吉林省农业科学院大豆研究所提供; 双元表达载体 pCAMBIA3300和根癌农杆菌(Agrobacterium tumefaciens)菌株EHA105由本实验室保存; 大肠杆菌DH5α购自北京TIANGEN公司; KOD FX高保真酶和ExTaq MasterMix聚合酶分别购自日本TOYOBO和北京康为试剂公司; 限制性内切酶Hind III、Sac I、Xba I购自美国Thermo Fisher Scientific公司; Hybood TM-N+尼龙膜和杂交检测试剂盒(DIG High Prime DNA Labeling and Detection Starter Kit II)分别购自美国Amersham和Roche公司; RNA提取试剂盒EasyPure PlantRNA Kit, 反转录试剂盒 TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix,qRT-PCR检测试剂盒 TransStart Top Green qPCR SuperMix均购自北京全式金公司; 除草剂 Basta购自北京鼎国生物公司, 草胺膦购自美国Sigma公司;QuickStix PAT/BAR转基因检测试纸购自美国EnviroLogix公司; 遗传转化试验中所用的培养基基础成分、植物激素、抗生素等购自美国 PhytoTech和 Sigma公司; 基因及引物委托南京金斯瑞生物有限公司合成。
1.2 植物表达载体构建
根据大豆基因组数据库(http://soybase.org/sbt/)序列信息, 利用 KOD FX高保真酶从栽培大豆Williams 82基因组中分离 β’-伴球蛋白基因启动子BCSP。扩增引物为 CongF1/CongR1 (5′-GCAAGCTT TGCTTGGATTTGGACCAGACTTG-3′, 5′-GCTCTA GATTAAATTATTTATTAAGGTATACTAGGATATTG-3′), 扩增片段长度为875 bp。采用Hind III/Xba I双酶切, BCSP构建到质粒 pCAMBIA3300中, 获得pCAMBIA 3300-BCSP。根据大豆GmFAD2-1B基因序列(GenBank登录号为XM_003555831), 委托南京金斯瑞生物有限公司合成GmFAD2-1B基因5′-UTR及部分编码序列, 合成序列长度为801 bp。采用Xba I/Sac I双酶切, 将GmFAD2-1B基因片段反向构建到pCAMBIA3300-BCSP质粒中, 并链接到BCSP启动子(该启动子可以驱动外源基因在种子中的高效特异性表达)下游, 构建植物表达载体pCAMBIA3300-BCSP-GmFAD2-1B (图1-A)。植物筛选标记为草胺膦抗性基因 BAR。采用冻融法, 将载体质粒导入农杆菌菌株EHA105以备转化。
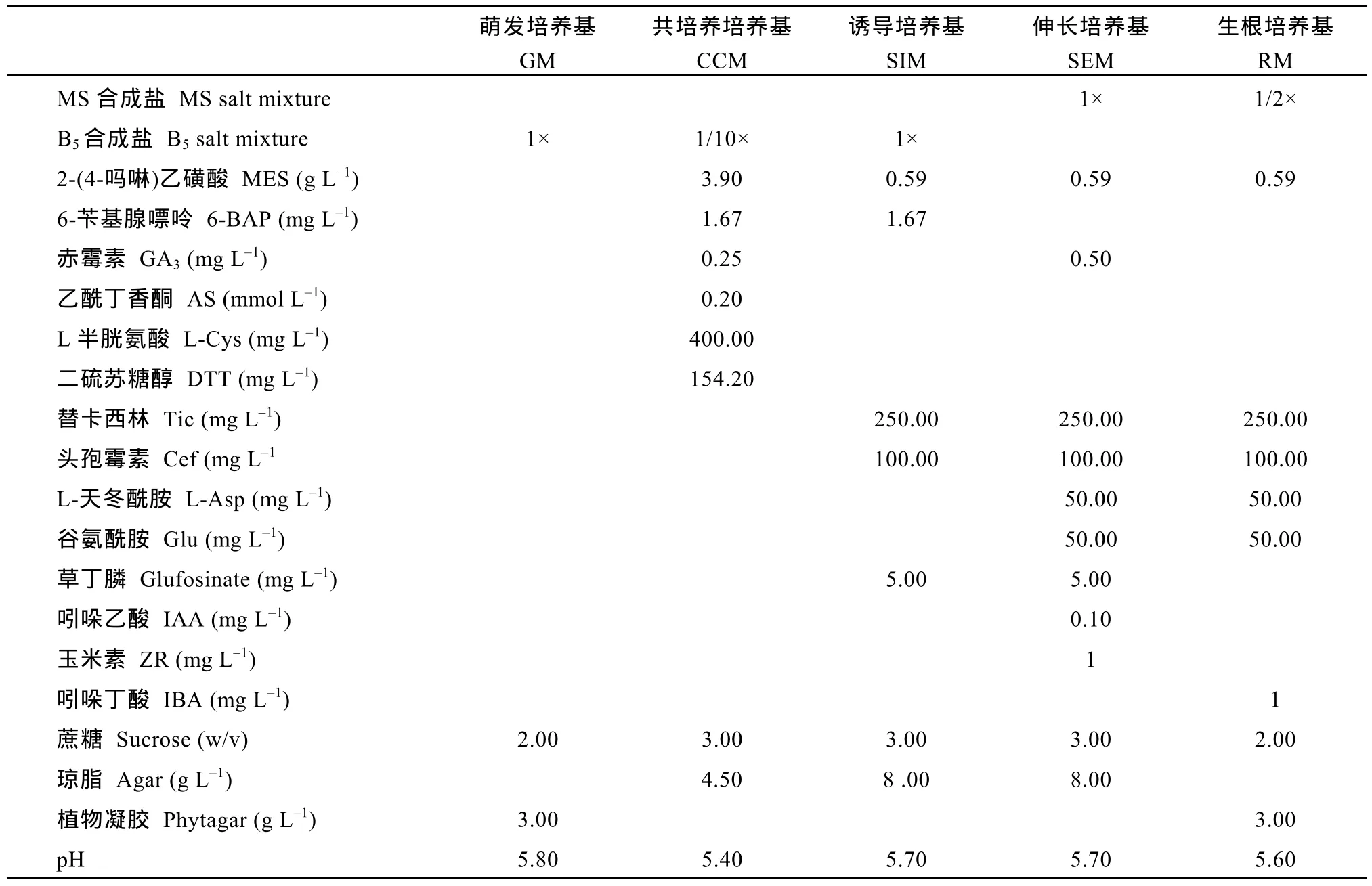
表1 大豆遗传转化中使用的培养基及成分Table 1 Mediums and compositions in soybean transformation
1.3 农杆菌介导大豆遗传转化
采用农杆菌介导法进行大豆遗传转化[16], 转化受体品种为Williams 82, 试验中用到的培养基见表1。将在萌发培养基GM中培养24 h后的大豆种子沿种脐部位剖开, 并在子叶节位置轻微划伤, 然后置农杆菌菌液中侵染30 min。将侵染后的外植体转移至共培养培养基CCM中, 于23℃条件下暗培养4 d。将外植体近轴面朝上转移至诱导培养基 SIM中,25℃, 16 h/8 h光暗条件下培养2周左右。然后将外植体取出, 切除多余的下胚轴部分, 转移至诱导培养基SIM中继续培养2~4周。将外植体上产生的丛生芽转移至芽伸长培养基SEM中培养, 培养条件为25℃, 16 h/8 h光暗周期。待抗性芽长至3~5 cm时将其切下, 并用1 mg L–1IBA浸泡30 s, 然后转至生根培养基RM中继续培养。待抗性芽长出健壮的根时,移栽至温室中生长结实(图1-B)。
1.4 PCR检测和除草剂喷施鉴定
参照产品说明书对移栽至温室中的T0代转化再生植株进行PAT/BAR试纸检测。采用DNA快速提取法提取植株叶片DNA[17], 根据表达载体中上游启动子 BCSP和反义 GmFAD2-1B序列设计引物Fad2F1/ Fad2R1 (5′-GGAAAGCAACCATATCAGCA TATCAC-3′, 5′-GTTCTCCAAGGTTGCATTCTTACTGG-3′); 内参基因 Gmactin检测引物为 Act-F1/Act-R1 (5′-TTGACTGAGCGTGGTTATTCC-3′, 5′-G ATCTTCATGCTGCTGGGTG-3′), 预期扩增大小分别为752 bp和403 bp。2对引物PCR扩增条件均为95℃ 5 min; 94℃ 30 s, 58℃ 30 s, 72℃ 1 min, 共30个循环; 72℃延伸10 min。扩增产物经1.0%琼脂糖凝胶电泳检测。对 T1~T4代转基因株系采用喷施除草剂 Basta (有效成分为草胺膦)和PCR检测进行筛选。待大豆幼苗长出第1组三出复叶时, 喷施500 mg L–1Basta。7~14 d 后拔除对除草剂 Basta敏感的植株; PCR检测引物及检测方法同上。获得的纯合转基因株系用于后续研究。
1.5 Southern杂交及qRT-PCR分析
选取T2代PCR阳性大豆植株进行Southern杂交检测。采用高盐CTAB法提取转基因大豆和对照品种叶片总DNA[18]。利用Hind III分别酶切基因组总DNA (约50 μg)。采用高盐转移法, 将酶切片段转移至 Hybood TM-N+尼龙膜上。探针扩增引物为BarF1/BarR1 (5′-GCACCATCGTCAACCACTACATC GAG-3′, 5′-TGAAGTCCAGCTGCCAGAAACCCAC-3′)。利用DIG随机引物标记试剂盒标记探针, 探针制备及杂交程序根据Roche公司的DIG High Prime DNA Labeling and Detection Starter Kit II试剂盒说明书进行。杂交温度为 42℃, 于 2×SSC (含 0.1%SDS), 37℃条件下洗膜 2次, 每次 5 min; 0.5×SSC(含0.1% SDS), 66℃条件下洗膜2次, 每次15 min。然后利用BCIP/NBT底物化学显色。
利用EasyPure Plant RNA Kit提取T3代转基因大豆根、茎、叶、花、种子样品RNA。利用TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix反转录试剂盒合成 cDNA第一链。采用TransStart Top Green qPCR SuperMix试剂盒进行qRT-PCR检测, 大豆内源 GmFAD2-1B检测引物为qFad2F1/qFad2R1 (5′-ATGCCTTGCCTCACTATGAT TC-3′, 5′-GCACTCTCTTGCTTCTCTCCA-3′), 内参基因Gmactin检测引物为qAct-F1/qAct-R1 (5′-ATCT TGACTGAGCGTGGTTATTCC-3′, 5′-GCTGGTCCT GGCTGTCTCC-3′)。反应程序为 94℃ 30 s; 94℃ 5 s,60℃ 30 s, 72℃ 10 s, 共40个循环。根据内参基因表达量, 计算目标基因相对表达量(2–ΔΔCT), 每个样品重复3次。
1.6 品质分析及农艺性状调查
委托吉林省农业科学院农业质量标准与检测技术研究所检测转基因大豆品质。采用气相色谱法检测脂肪酸组分, 采用国家标准 GB/T6432-1994和 GB/T 5512-2008检测总脂肪(%)及总蛋白含量(%)。农艺性状调查内容包括生育期、叶形、花色、株高、种皮色、单株分枝数、单株节数、结荚高度、百粒重和种脐色。
2 结果与分析
2.1 转基因大豆的获得及遗传稳定性检测
根据大豆GmFAD2-1B基因序列选取801 bp反义序列(含 5′-UTR及部分编码序列), 该序列与同源基因GmFAD2-1A相似度为99.8%。上游启动子为大豆种子特异型β’-伴球蛋白基因启动子BCSP[19]。采用农杆菌介导的大豆子叶节转化法, 将构建的基因载体导入大豆栽培品种Williams 82中。共转化外植体1600多块, 经过分化、筛选和再生, 获得耐5 mg L–1草铵膦再生植株 74株。PAT/BAR试纸(图 1-C)和PCR检测结果表明, 其中49株能扩增出目的条带,扩增条带大小为 752 bp, 与预期大小一致, 而对照非转基因植株则没有相应的条带, 表明外源基因已导入受体大豆品种, PCR阳性转化率为3.04%。转基因植株移栽至温室后能够正常开花结实, 且在表型上与对照非转基因植株无明显差异。对 T1~T4代转基因大豆喷施除草剂结果表明, 在500 mg L–1Basta处理条件下, 非转基因大豆很快出现叶片黄化或枯死现象, 而转基因植株则表现正常(图 1-D)。连续 4代PCR检测及除草剂喷施结果表明, 外源基因在转基因大豆不同世代中能够稳定遗传。
2.2 外源基因整合及表达分析
对T2代转基因大豆植株进行Southern杂交检测结果表明, 转基因大豆均可以检测到杂交信号, 且杂交片段均大于3.8 kb (表达载体中Hind III位点至左边界序列), 而对照非转基因大豆则没有杂交条带出现, 表明外源基因片段已整合到大豆基因组中(图2)。在检测的转基因植株中, 外源T-DNA插入拷贝数为1~5个, 其中1~2个拷贝的比例为66.7%。采用qRT-PCR技术对T3代转基因植株外源基因表达情况进一步分析表明, 转基因大豆L40和L72根、茎、叶、花中内源Gmfad2-1基因表达水平与对照品种相比没有显著变化, 而在种子中的表达水平分别降低了3.91倍和2.50倍(图3), 表明外源反义Gmfad2-1B基因主要在大豆种子中表达, 并导致内源 Gmfad2-1基因表达水平的显著下降, 也表明大豆β’-伴球蛋白基因启动子 BCSP可以驱动外源基因在大豆种子中的特异性表达。
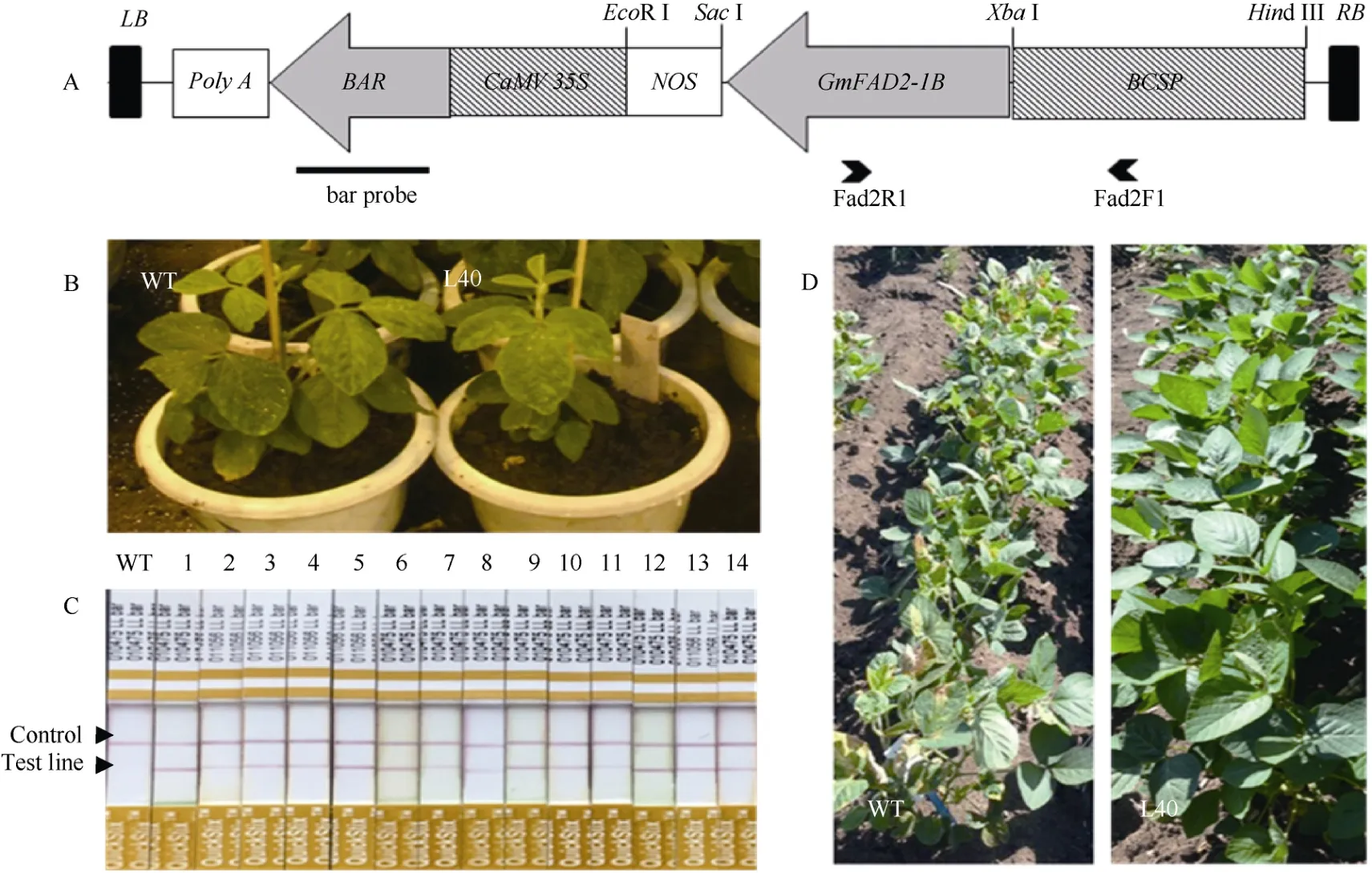
图1 植物表达载体示意图及转基因大豆的获得Fig. 1 Schematic representation of the construct and generation for the transgenic soybean

图2 转基因大豆Southern杂交检测Fig. 2 Southern blot analysis of the transgenic plants
2.3 转基因大豆品质分析及农艺性状调查
采用气相色谱法对T3代转基因大豆脂肪酸组成分析表明, 在检测的13份转基因大豆中, 12份油酸含量 27.38%~80.42% (表 2), 极显著高于对照品种Williams 82 (22.00%), 其中油酸含量较高的转基因大豆材料为 L40和 L72, 分别为 80.40%和 80.22%,较对照品种提高 2.65倍, 表明反义 RNA介导大豆GmFAD2-1B基因沉默显著增强了油酸在大豆种子中的高效累积。对T4代转基因大豆进一步分析表明,6份转基因大豆油酸含量为 59.63%~68.91% (表 3),其中L40和L72油酸含量分别为68.91%和65.98%,尽管其含量较T3代有所降低, 但仍极显著高于对照品种(17.81%), 提高 2.70~2.87倍, 表明高油酸性状在转基因大豆不同世代间能够稳定遗传。不同世代间油酸含量的差异可能与不同年份间的环境温度变化有关[1,10]。结合不同世代检测结果, L40和L72油酸含量分别为 68.91%~80.42%和 65.98%~80.22%,较对照品种Williams 82提高2.65倍以上, 亚油酸降低至 4.84%~14.55%, 饱和脂肪酸降低至 10.34%~11.16%, 其脂肪酸组成接近橄榄油。

图3 转基因大豆GmFAD2-1基因qRT-PCR表达分析Fig. 3 Expression analysis of the GmFAD2-1 in the transgenic soybean by qRT-PCR
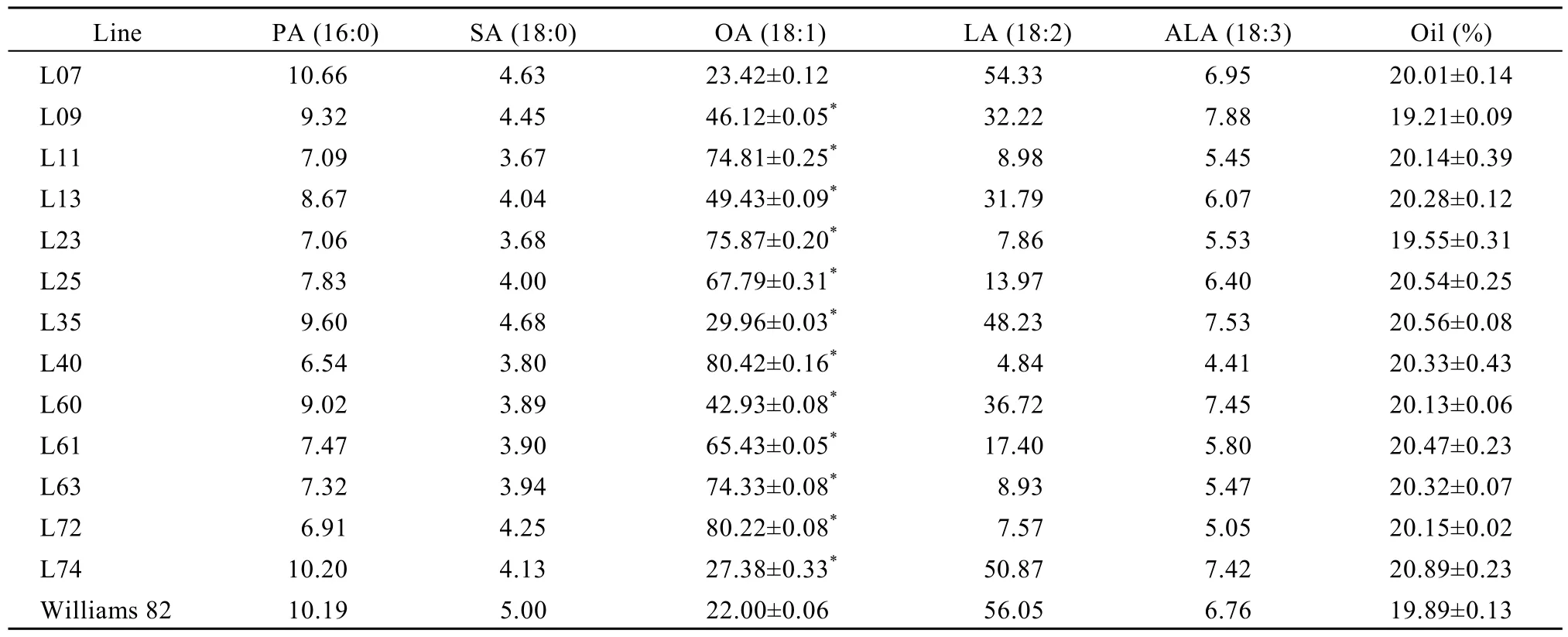
表2 T3代转基因大豆脂肪酸组分Table 2 Fatty acid compositions in T3transgenic soybean seeds
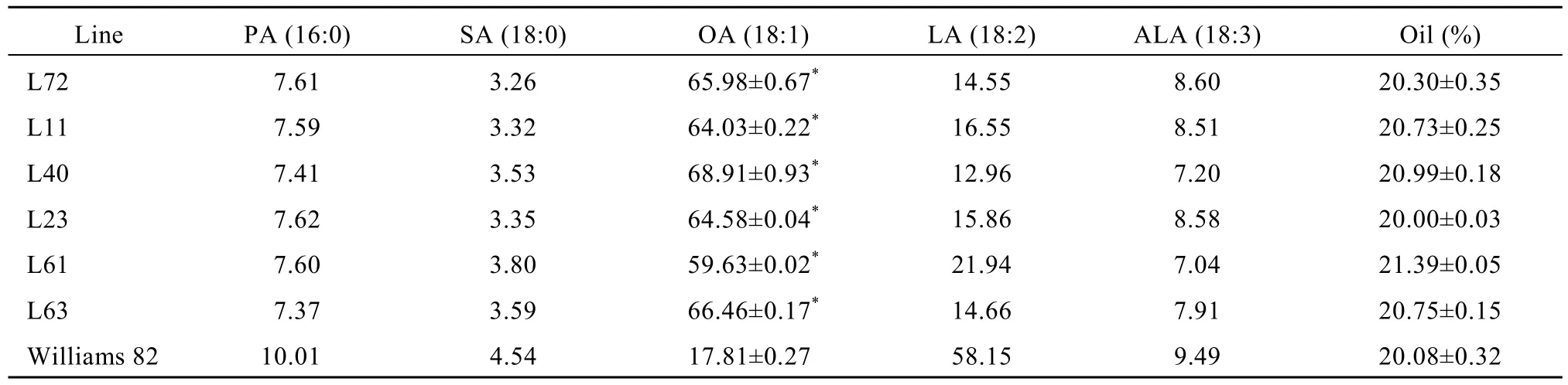
表3 T4代转基因大豆脂肪酸组分Table 3 Fatty acid compositions in T4transgenic soybean seeds
对T4代转基因大豆蛋白和脂肪含量检测结果表明, 转基因大豆L40和L72其蛋白和脂肪含量分别为 38.30%~38.85%和 20.30%~20.99%, 与对照品种Williams 82无显著差异(表4), 表明反义RNA介导大豆 GmFAD2-1B基因沉默没有显著影响转基因大豆种子中总蛋白和总脂肪的积累。另外, 对 T4代转基因大豆田间表型性状调查结果表明, L40和L72转基因大豆株系在叶形、花色、种皮色、种脐色、株高、节数、结荚高度、生育期等方面与对照品种Williams 82没有明显差异(表 4), 表明外源反义GmFAD2-1B基因的表达对转基因大豆的表型性状没有非特异影响。
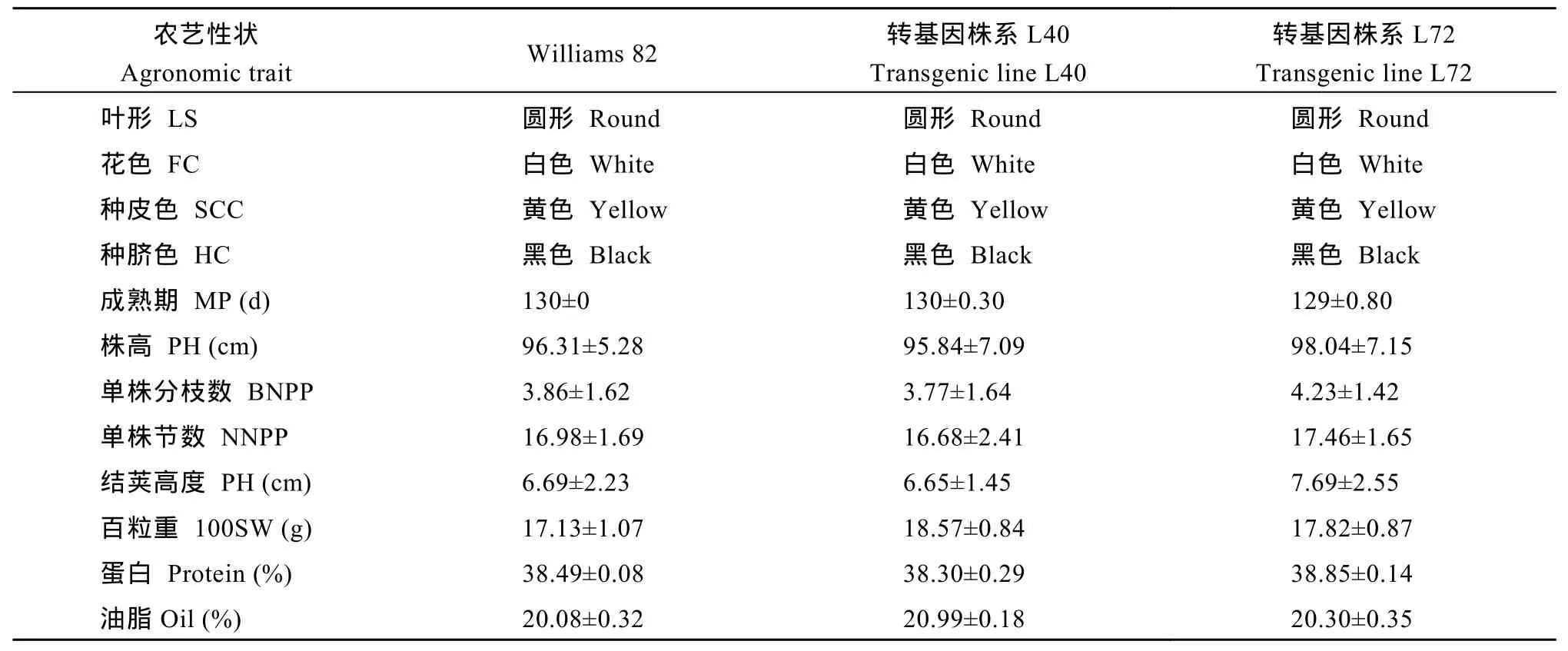
表4 转基因大豆田间表型性状Table 4 Agronomic traits of the transgenic lines in field conditions
3 讨论
长期以来, 高油酸育种一直是大豆品质性状改良的重要内容之一。尽管目前已筛选出多个油酸含量(30%~70%)较高的大豆种质资源[20], 但由于高油酸与产量性状之间的紧密连锁[21], 利用常规育种选育的高油酸大豆品种尚未在生产中广泛应用。转基因技术为高油酸转基因大豆新品种选育提供了强有力的技术手段。已有的研究表明, 通过基因沉默及基因编辑技术抑制大豆种子中多不饱和脂肪酸合成途径中的关键酶基因 FAD2的表达, 可以有效抑制多不饱和脂肪酸的合成, 促进种子中 OA 的累积[13-15]。本研究采用反义RNA介导大豆GmFAD2-1B基因沉默, 获得了油酸含量达 68.91%~80.42%和 65.98%~80.22%的转基因大豆, 其脂肪酸组成接近橄榄油,表明利用反义RNA技术介导GmFAD2-1B基因沉默可以显著增强大豆种子中油酸的高效累积。本研究获得的高油酸转基因大豆在技术指标上接近于美国杜邦公司研发的高油酸大豆品种(Plenish, 油酸含量>75%), 具有重要的育种应用价值。
大豆种子中 FAD2-1A和FAD2-1B具有相似的功能, 只有同时抑制 2个同源基因的表达, 才能显著提高 OA的含量水平[11]。本研究选用的反义GmFAD2-1B与 GmFAD2-1A相似度为 99.8%,qRT-PCR检测表明, 转基因大豆种子中内源GmFAD2-1表达水平较对照品种显著降低(3.91倍和2.50倍), 表明外源反义FAD2-1B基因显著抑制了大豆内源FAD2-1A和FAD2-1B基因的表达, 从而促进了种子中油酸的高水平积累。
本研究发现, 不同世代转基因大豆油酸含量存在明显差异(表2和表3), 其原因可能与不同年份间环境温度差异有关。已有的研究表明, 大豆发育过程中, 环境温度对种子中油酸的积累具有重要的影响。在一定范围内, 温度越高, 越易促进油酸的积累[1]。另一方面, 尽管转基因大豆油酸含量在不同年份间存在差异, 但从变化趋势上来说, 转基因大豆油酸含量增加幅度与对照品种相比变化并不大(2.65~2.87倍), 表明高油酸性状在转基因大豆不同世代间能够稳定遗传。另外, 本研究获得的转基因大豆除脂肪酸组成发生变化外, 其总蛋白、总油脂含量及其他农艺性状如熟期、株高、叶形、花色、结荚高度、百粒重等方面与对照品种也没有显著差异, 表明外源反义FAD2-1B基因的整合与表达并没有对转基因大豆产生非特异性的影响。
4 结论
采用农杆菌介导法, 获得了油酸含量显著提高的转基因大豆新品系。外源GmFAD2-1B基因片段已导入大豆基因组中, 并在种子中特异表达。12份转基因大豆种子油酸含量为 27.38%~80.42%, 极显著高于对照品种Williams 82, 其中L40和L72油酸含量分别为 68.91%~80.42%和 65.98%~80.22%, 较对照品种提高 2.65倍以上, 亚油酸降低至 4.84%~14.55%, 饱和脂肪酸降低至 10.34%~11.16%, 但总脂肪和总蛋白含量没有显著变化。另外, 转基因大豆在农艺性状方面与对照品种没有显著差异。
[1]Clemente T E, Cahoon E. Soybean oil: genetic approaches for modification of functionality and total content. Plant Physiol,2009, 151: 1030–1040
[2]Sari A R, Thomas G, Christoph B, John B O. Contrapuntal networks of gene expression during Arabidopsis seed filling. Plant Cell, 2002, 14: 1191–1206
[3]Okuley J, Lightner J, Feldmann K, Yadav N, Lark E, Browse J.Arabidopsis FAD2 gene encodes the enzyme that is essential for polyunsaturated lipid synthesis. Plant Cell, 1994, 6: 147–158
[4]Li L Y, Wang X L, Gai J Y, Yu D. Molecular cloning and characterization of a novel microsomal oleate desaturase gene from soybean. Plant Physiol, 2007, 64: 516–526
[5]Lee K R, Sohn S I, Jung J H, Kim S H, Roh K H, Kim J B, Suh M C, Kim H U. Functional analysis and tissue-differential expression of four FAD2 genes in amphidiploid Brassica napus derived from Brassica rapa and Brassica oleracea. Gene, 2013,531: 253–262
[6]Hongtrakul V, Slabaugh M, Knapp S J. A seed specific Δ12oleate desaturase is duplicated, rearranged, and weakly expressed in high oleic acid sunflower lines. Crop Sci, 1998, 38: 1245–1249
[7]López Y, Nadaf H L, Smith O D, Fritz A K. Isolation and characterization of the Δ12-fatty acid desaturase in peanut (Arachis hypogaea L.)and search for polymorphisms for the high oleate trait in Spanish market-type lines. Theor Appl Genet, 2000, 101: 1131–1138
[8]Zhang D, Irma L P, Stacy J P, Mongkol N, Purnima N, Sylvia W W, Robert M P, Kent D C. Identification and expression of a new delta-12 fatty acid desaturase (FAD2-4) gene in upland cotton and its functional expression in yeast and Arabidopsis thaliana plants. Plant Physiol Biochem, 2009, 47: 462–471
[9]Heppard E P, Kinney A J, Stecca K L, Miao G H. Developmental and growth temperature regulation of two different microsomalω-6saturase genes in soybeans. Plant Physiol, 1996, 110: 311–319
[10]Tang G Q, Novitzky W P, Griffin H C, Huber S C, Dewey R E.Oleate desaturase enzymes of soybean: evidence of regulation through differential stability and phosphorylation. Plant J, 2005,44: 433–446
[11]Pham A T, Lee J D, Shannon J G, Bilyeu K D. Mutant alleles of FAD2-1A and FAD2-1B combine to produce soybeans with the high oleic acid seed oil trait. BMC Plant Biol, 2010, 10: 195
[12]Li L Y, Wang X L, Gai J Y, Yu D Y. Isolation and characterization of a seed-specific isoform of microsomal omega-6 fatty acid desaturase gene (FAD2-1B) from soybean. DNA Sequence, 2008.19: 28–36
[13]Zhang L, Yang X D, Zhang Y Y, Yang J, Qi G X, Guo D Q,Xing G J, Yao Y, Xu W J, Li H Y, Li Q Y, Dong Y S. Changes in oleic acid content of transgenic soybeans by antisense RNA mediated posttranscriptional gene silencing. Int J Genom, 2014,921–950
[14]Wang G L, Xu Y N. Hypocotyl-based Agrobacterium-mediated transformation of soybean (Glycine max) and application for RNA interference. Plant Cell Rep, 2008, 27: 1177–1184
[15]Haun W, Coffman A, Clasen B M, Demorest Z L, Lowy A, Ray E, Retterath A, Stoddard T, Juillerat A, Cedrone F, Mathis L,Voytas D F, Zhang F. Improved soybean oil quality by targeted mutagenesis of the fatty acid desaturase 2 gene family. Plant Biotechnol J, 2014, 12: 934–940
[16]杨向东, 牛陆, 张伟, 杨静, 杜茜, 邢国杰, 郭东全, 李启云,董英山. RNAi介导SMV-P3基因沉默增强大豆对花叶病毒病的抗性. 作物学报, 2016, 42: 1647–1655 Yang X D, Niu L, Zhang W, Yang J, Du Q, Xing G J, Guo D Q,Li Q Y, Dong Y S. RNAi-mediated SMV-P3 silencing increases soybean resistance to soybean mosaic virus. Acta Agron Sin,2016, 42: 1647–1655 (in Chinese with English abstract)
[17]Edwards K, Johnstone C, Thompson C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis. Nucl Acids Res, 1991, 19: 1349
[18]Tel-Zur N, Abbo S, Myslabodski D, Mizrahi Y. Modified CTAB procedure for DNA isolation from epiphytic cacti of the genera Hylocereus and Selenicereus (Cactaceae). Plant Mol Biol Rep,1999, 17: 249–254
[19]Yoshino M, Nagamatsu A, Tsutsumi K, Kanazawa A. The regulatory function of the upstream sequence of the beta-conglycinin alpha subunit gene in seed-specific transcription is associated with the presence of the RY sequence. Genes Genet Syst, 2006,81: 135–141
[20]Alt J L, Fehr W R, Welke G A, Shannon J G. Transgressive segregation for oleate content in three soybean populations. Crop Sci,2005, 45: 2005–2007
[21]Scherder C W, Fehr W R. Agronomic and seed characteristics of soybean lines with increased oleate content. Crop Sci, 2008, 48:1755–1758
Antisense RNA-mediated GmFAD2-1B Gene Silencing Enhances Accumulation of Oleic Acid in Transgenic Soybean Seeds
YANG Jing,*XING Guo-Jie, NIU Lu, HE Hong-Li, DU Qian, GUO Dong-Quan, YUAN Ying*, and YANG Xiang-Dong
Agro-Biotechnology Institute, Jilin Academy of Agricultural Sciences / Jilin Provincial Key Laboratory of Agricultural Biotechnology, Changchun 130033, China
10.3724/SP.J.1006.2017.01588
本研究由国家转基因生物新品种培育科技重大专项(2016ZX08004-003)和国家自然科学基金项目(31671764)资助。
This study was supported by the National Major Project for Developing New GM Crops (2016ZX08004-003) and the National Natural Science foundation of China (31671764).
*通讯作者(Corresponding authors): 杨向东, E-mail: xdyang020918@126.com, Tel: +86-431-87063044; 袁英, E-mail: yuanying2003@sohu.com
联系方式: E-mail: jinggyang@126.com, Tel: +86-431-87063244
): 2017-03-20; Accepted(接受日期): 2017-07-19; Published online(网络出版日期): 2017-08-01.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20170801.1840.014.html
