草莓胶孢炭疽菌CFEM候选效应子的生物信息学鉴定及其侵染过程中的转录分析
2017-10-09张丽勍邹小花何成勇高清华
张丽勍, 段 可, 邹小花, 何成勇, 高清华
(上海市农业科学院林木果树研究所, 上海 201403)
草莓胶孢炭疽菌CFEM候选效应子的生物信息学鉴定及其侵染过程中的转录分析
张丽勍, 段 可, 邹小花, 何成勇, 高清华*
(上海市农业科学院林木果树研究所, 上海 201403)
病原真菌通常分泌效应子到寄主组织中调控寄主的生理过程,从而有利于其侵染。CFEM(common in several fungal extracellular membrane)蛋白是真菌所独有的,且与致病性密切相关。本研究利用Pfam数据库对草莓胶孢炭疽菌全基因组进行搜索,鉴定获得22个CFEM蛋白。对CFEM蛋白的信号肽、跨膜结构域和亚细胞定位进行分析,结果表明仅有8个CFEM蛋白为分泌蛋白。对CFEM分泌蛋白在不同侵染阶段的转录情况进行转录组学及RT-PCR分析,结果显示8个CFEM蛋白在侵染后不同时期均有表达。其中,1个CFEM分泌蛋白于附着胞形成期特异表达,2个于活体寄生阶段特异表达,2个于死体寄生阶段特异表达。综合上述分析结果,预测这8个分泌蛋白可能为草莓胶孢炭疽菌的效应子。本研究为深入解析植物病原真菌CFEM效应子提供了理论依据。
胶孢炭疽菌; 效应子; CFEM蛋白
由炭疽菌属Colletotrichum真菌引起的草莓炭疽病是草莓上的一种重要病害。草莓炭疽病危害严重年份草莓死苗率达80%,草莓减产达50%以上[1]。胶孢炭疽菌Colletotrichumgloeosporioides是草莓炭疽病的主要病原之一,为半活体营养型真菌。
在植物病原真菌与寄主植物共进化的过程中,病原真菌的致病性与寄主的抗性之间一直发生着相互作用[2]。病原真菌通过分泌一系列的效应子来调控寄主植物的抗性[3]。效应子是一类分泌蛋白,必须被分泌并转运到寄主细胞的特定部位才能发挥其功能。真菌的效应子具有以下特点: 序列特异性高,不同物种间序列相似性小;氨基酸序列短,一般不超过300 个氨基酸残基;含有信号肽,同时不含跨膜区; 富含半胱氨酸[4]。效应子依据其功能不同可以分为两类,一类效应子通过抑制植物的抗性来促进病原真菌的侵染(称为效应子诱发的感病性);另一类效应子则能够间接或直接地被寄主植物识别,导致效应子触发的免疫。2006年,一篇关于卵菌(oomycete)全基因组的文献中提出了一个概念,即真菌类生物或真菌本身可能有成百上千种效应子。自此开始,大量的真菌全基因组数据被发掘,各种不同的生物信息学手段被用于精确的分析和定义效应子[5]。
2013年,胶孢炭疽菌的全基因组测序工作完成。Gan等[6]对胶孢炭疽菌基因组的分泌蛋白进行了生物信息学分析,预测其含有2 042个分泌蛋白,其中755个为少于300个氨基酸的小分泌蛋白(small secretory proteins,SSP)。汪倩等[7]利用生物信息学手段发现胶孢炭疽菌基因组中存在稻瘟菌效应子BASP2的同源蛋白CgBASP2。对该蛋白编码基因进行功能研究发现,与野生型相比CgBASP2基因缺失突变株不能对寄主产生致病性,说明其在致病过程中发挥着重要的作用。何芬[8]对利用生物信息学筛选到的胶孢炭疽菌LysMs (Lysin Motifs)型候选效应子CgLysM,进行了亚细胞定位,发现CgLysM在分生孢子萌发过程中有向芽管顶端(或附着胞)富集的倾向。这些报道表明,高通量测序和生物信息学手段结合功能学试验能够有效预测并鉴定出真菌效应子。随着高通量测序技术的革新、生物信息学的快速发展以及胶孢炭疽菌全基因组信息的公布,胶孢炭疽菌-草莓的互作研究工作将被极大地推动。
CFEM(common in several fungal extracellular membrane)是真菌独有的一种蛋白结构域,含有8个半胱氨酸[9]。CFEM蛋白结构域最初是在稻瘟病菌Magnaporthegrisea中发现的,含有该结构域的ACI1蛋白在稻瘟病菌致病过程中起着至关重要的作用。此外,在白假丝酵母Candidaalbicans中发现的含CFEM结构域的CSA1和稻瘟病菌中的Pth11蛋白均与致病性相关[10]。目前在胶孢炭疽菌中尚未见CFEM效应子的相关报道。本研究利用NCBI网站(http:∥www.ncbi.nlm.nih.gov/)公布的胶孢炭疽菌基因组数据和注释信息,从全基因组水平上鉴定胶孢炭疽菌中含有CFEM蛋白结构域的成员,采用生物信息学手段进行分析,并结合转录组学数据及RT-PCR分析结果,预测胶孢炭疽菌CFEM的候选效应子,为进一步研究CFEM效应子的功能奠定基础。
1 材料与方法
1.1 材料
胶孢炭疽菌C.gloeosporioides的全基因组蛋白质数据来自NCBI网站。试验所用草莓为栽培草莓品种‘久香’,取自上海市农业科学院林木果树研究所草莓种质资源圃。
1.2 方法
1.2.1 CFEM 蛋白的生物信息学分析
利用蛋白质家族数据库Pfam (http:∥pfam.xfam.org/family/)对胶孢炭疽菌基因组编码的CFEM蛋白进行分析,E-value 设为0.001。
通过SignalP 4.1(http:∥www.cbs.dtu.dk/services/SignalP/)预测各CFEM蛋白的N端信号肽;通过TMHMM Server v. 2.0(http:∥www.cbs.dtu.dk/services/TMHMM/)预测各CFEM蛋白的跨膜结构域;通过TargetP 1.1 Server(http:∥www.cbs.dtu.dk/services/TargetP/)预测各CFEM蛋白的亚细胞定位。
1.2.2 系统发育树构建
通过ClustalX 软件对炭疽属真菌蛋白CFEM氨基酸序列进行多重序列比对,利用MEGA 6.0中的邻近法构建系统发育树,bootstrap值设置为1 000。
1.2.3 CFEM蛋白在胶孢炭疽菌侵染草莓叶片不同阶段中的转录组学分析
取生长健壮、长势一致的草莓植株于人工气候培养箱培养。试验设置处理组和对照组,每组5株草莓植株,设3次重复。处理组采用孢子浓度为1×106个/mL的孢子悬浮液喷雾法接种草莓叶片,对照组采用纯培养胶孢炭疽菌。将对照组及接种后三个阶段:附着胞形成阶段(inplantaappressoria)、活体营养阶段(biotrophic phase)及死体营养阶段(necrotrophic phase)的叶片分别取样[9],提取RNA。RNA提取方法参见OMEGA Plant RNA Kit试剂盒使用说明。提取的RNA样本经琼脂糖凝胶电泳及 Thermo Scientific NanoDrop 2000分光光度计检测。检测合格的 RNA 样品送上海瀚宇生物技术有限公司进行转录组测序。
1.2.4 基因编码CFEM蛋白的转录分析
对这三个阶段受侵染的草莓叶片和阴性对照(接种等量无菌水的草莓叶片)分别提取RNA,反转录为cDNA。反转录参见PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒使用说明。扩增引物由生工生物工程(上海)股份有限公司合成。RT-PCR方法扩增得到侵染时期三个不同阶段的编码CFEM蛋白基因。CFEM蛋白基因克隆片段均构建至pMD18-T(购自宝生物工程有限公司)并送生工生物工程(上海)股份有限公司测序,测序结果与CFEM蛋白基因序列完全一致。
2 结果与分析
2.1 草莓胶孢炭疽菌CFEM 蛋白的鉴定
Pfam数据库是一个包含了大量多重序列校准,以及“隐藏式马尔科夫模型”的巨大集合,涵盖了许多蛋白质家族。草莓胶孢炭疽菌基因组基因共编码15 381个蛋白,利用蛋白质家族数据库Pfam (http:∥pfam.xfam.org/)对胶孢炭疽菌基因组编码的CFEM蛋白进行分析,共获得22个CFEM蛋白(表1)。
表1草莓胶孢炭疽菌CFEM蛋白
Table1TheinformationofCFEMproteinsinColletotrichumgloeosporioidesonstrawberry
2.2 草莓胶孢炭疽菌CFEM 蛋白信号肽分析
效应子由真菌分泌到胞外后,由信号肽引导其进行跨膜转运。因此,本文首先对胶孢炭疽菌中所有的CFEM 蛋白信号肽预测,初步确定草莓胶孢炭疽菌CFEM分泌蛋白的组成。由SignalP 4.1 Server在线软件对CFEM蛋白进行信号肽预测分析的结果显示,22个CFEM蛋白中有21个CFEM蛋白N端氨基酸序列具有信号肽,其信号肽序列为N 端16~25个氨基酸(表2)。一些内在膜蛋白、膜结合蛋白和各种胞内的细胞器蛋白同样含有信号肽,但不是效应子。因此,需进一步对CFEM蛋白进行亚细胞定位分析,以排除上述蛋白。
表2草莓胶孢炭疽菌各CFEM蛋白信号肽分析1)
Table2BioinformaticanalysisofsignalpeptidesofCFEMproteinsinColletotrichumgloeosporioides

蛋白序列号ProteinID信号肽Signalpeptide信号肽切割位点Cleavagesite蛋白序列号ProteinID信号肽Signalpeptide信号肽切割位点CleavagesiteCGGC5_2461.1 +21/22CGGC5_1173.1-CGGC5_13949.1+19/20CGGC5_10073.1+20/21CGGC5_1956.1 +17/18CGGC5_9409.1+24/25CGGC5_13478.1+18/19CGGC5_5966.1+22/23CGGC5_13200.1+16/17CGGC5_5833.1+16/17CGGC5_11628.1+19/20CGGC5_621.1 +26/27CGGC5_1525.1 +19/20CGGC5_7948.1+20/21CGGC5_11293.1+22/23CGGC5_908.1 +19/20CGGC5_11153.1+20/21CGGC5_9429.1+21/22CGGC5_11062.1+16/17CGGC5_1574.1+19/20CGGC5_10469.1+20/21CGGC5_15026.1+21/22
1) “+” 表示该蛋白具有信号肽;“-”表示该蛋白不具有信号肽。 “+” indicates the protein with signal peptide sequences; “-” indicates the protein without signal peptide sequences.
2.3 草莓胶孢炭疽菌CFEM蛋白亚细胞定位分析
本文利用TargetP软件对21个具有信号肽的CFEM蛋白进行亚细胞定位分析。分析结果表明,21个CFEM蛋白通过分泌途径分泌至胞外的得分值≥0.677。其中,除CGGC5_11628.1、CGGC5_11293.1和CGGC5_15026.1定位可信度略低(RC=3)外,其余18个CFEM蛋白的定位预测可信度皆较高(RC≤2)。因此,21个具有信号肽的CFEM均为分泌蛋白,可通过草莓胶孢炭疽菌分泌途径分泌至胞外(表3)。
表314个具信号肽的CFEM蛋白的亚细胞定位分析1)
Table3SubcellularlocalizationpredictionofCFEMproteinsinColletotrichumgloeosporioides

蛋白序列号ProteinID细胞器定位得分值SubcellularlocalizationpredictionvaluemTPSPOtherLocRC蛋白序列号ProteinID细胞器定位得分值SubcellularlocalizationpredictionvaluemTPSPOtherLocRCCGGC5_2461.10.0510.9670.026S1CGGC5_10073.10.0450.9170.057S1CGGC5_13949.10.0950.9180.027S1CGGC5_9409.10.0780.9200.022S1CGGC5_1956.10.0230.9650.080S1CGGC5_5966.10.0330.9560.029S1CGGC5_13478.10.0210.9470.130S1CGGC5_5833.10.0650.8830.087S2CGGC5_13200.10.0250.9520.070S1CGGC5_621.10.0600.9640.014S1CGGC5_11628.10.2540.8400.017S3CGGC5_7948.10.1120.8950.062S2CGGC5_1525.10.0220.9890.017S1CGGC5_908.10.0400.9650.022S1CGGC5_11293.10.1550.6770.119S3CGGC5_9429.10.0710.9390.021S1CGGC5_11153.10.0330.8670.095S2CGGC5_1574.10.0160.9700.058S1CGGC5_11062.10.0480.8470.155S2CGGC5_15026.10.2440.8050.011S3CGGC5_10469.10.0320.8460.091S2
1) mTP:线粒体定位;S:分泌途径;SP:该蛋白含有信号肽,可通过分泌途径分泌至胞外,为分泌蛋白;Other:其他定位;Loc:预测定位结果。RC:可信度等级从1~5,估算亚细胞定位预测最大值和次值之间的差距(diff)。1:diff≥0.800;2:0.800>diff≥0.600;3:0.600>diff≥0.400;4:0.400>diff≥0.200;5:diff<0.200。RC值越低,定位越准确,RC=1定位预测最可靠。 mTP: Mitochondrion localization; S: Secretion pathway; SP: Signal peptide; Other: Any other localization; Loc: Prediction of localization.RC: Reliability class, from 1 to 5, where 1 indicates the strongest prediction.RCis a measure of the size of the difference (diff) between the highest (winning) and the second highest output scores. There are 5 reliability classes, defined as follows: 1:diff≥0.800; 2: 0.800>diff≥0.600; 3: 0.600>diff≥0.400; 4: 0.400>diff≥0.200; 5:diff<0.200. Thus, the lower the value ofRC, the safer the prediction.
2.4 草莓胶孢炭疽菌CFEM分泌蛋白跨膜结构域分析
由于分泌蛋白和跨膜蛋白均含有疏水区域,因此很难将两者区分开来。因此,为了避免跨膜蛋白和分泌蛋白相混淆,选用TMHMM软件对21个含信号肽的CFEM蛋白进行跨膜结构域的预测,剔除跨膜结构域>1的蛋白。结果表明21个含信号肽的CFEM蛋白中有13个CEFM蛋白均含有跨膜结构域,说明其为跨膜蛋白。而其余8个CEFM蛋白不含有跨膜结构域,说明它们属于CFEM分泌蛋白(表4)。
表4草莓胶孢炭疽菌CFEM蛋白跨膜结构域分析
Table4BioinformaticanalysisoftransmembraneofCFEMproteinsinColletotrichumgloeosporioides

蛋白序列号ProteinID跨膜结构域数目NumberofpredictedTMHs跨膜结构域位置PositionoftransmembranedomainTM1TM2TM3TM4TM5TM6TM7TM8CGGC5_2461.16113~132152~174187~209219~241262~284299~321CGGC5_13949.187~29 104~126139~161181~203215~237265~287300~322342~364CGGC5_1956.10CGGC5_13478.10CGGC5_13200.10CGGC5_11628.17103~120135~157177~199209~231252~274294~316329~351CGGC5_1525.17100~122135~157179~201214~236260~282295~317337~359CGGC5_11293.10CGGC5_11153.10CGGC5_11062.10CGGC5_10469.10CGGC5_10073.12101~123187~206
续表4Table4(Continued)

蛋白序列号ProteinID跨膜结构域数目NumberofpredictedTMHs跨膜结构域位置PositionoftransmembranedomainTM1TM2TM3TM4TM5TM6TM7TM8CGGC5_9409.187~24110~128141~163183~205218~240265~287300~322342~364CGGC5_5966.175~22102~119139~156171~193219~241256~278290~312CGGC5_5833.10CGGC5_621.16149~171191~213233~255270~292305~327342~364CGGC5_7948.17108~130143~162186~208220~242266~288301~323343~365CGGC5_908.182~2195~117130~152172~194214~236256~275288~310325~347CGGC5_9429.1599~118131~153173~195208~230290~308CGGC5_1574.1793~115128~147172~194207~229253~275288~306321~343CGGC5_15026.174~2134~5379~101114~136159~181193~215235~257
2.5 炭疽菌属CFEM蛋白系统进化分析
MEGA建树结果表明,尽管以8个CFEM为靶标蛋白绘制的进化树总体来说有较高的相似性,但各自之间仍存在差异。进化树大体上分为2个大分支,其中分支一由C.simmondsii、C.fioriniae、C.nymphaeae、C.salicis和本研究中的C.gloeosporioides组成,分支二由C.tofieldiae、C.incanum、C.higginsianum、C.sublineola和C.graminicola组成。有趣的是,在分支一中,C.simmondsii、C.fioriniae、C.nymphaeae和C.salicis同源性较高,被划分到一个小分支;而本研究中的C.gloeosporioides则与上述4种炭疽菌同源性相对较低,被独立分到另一小分支。
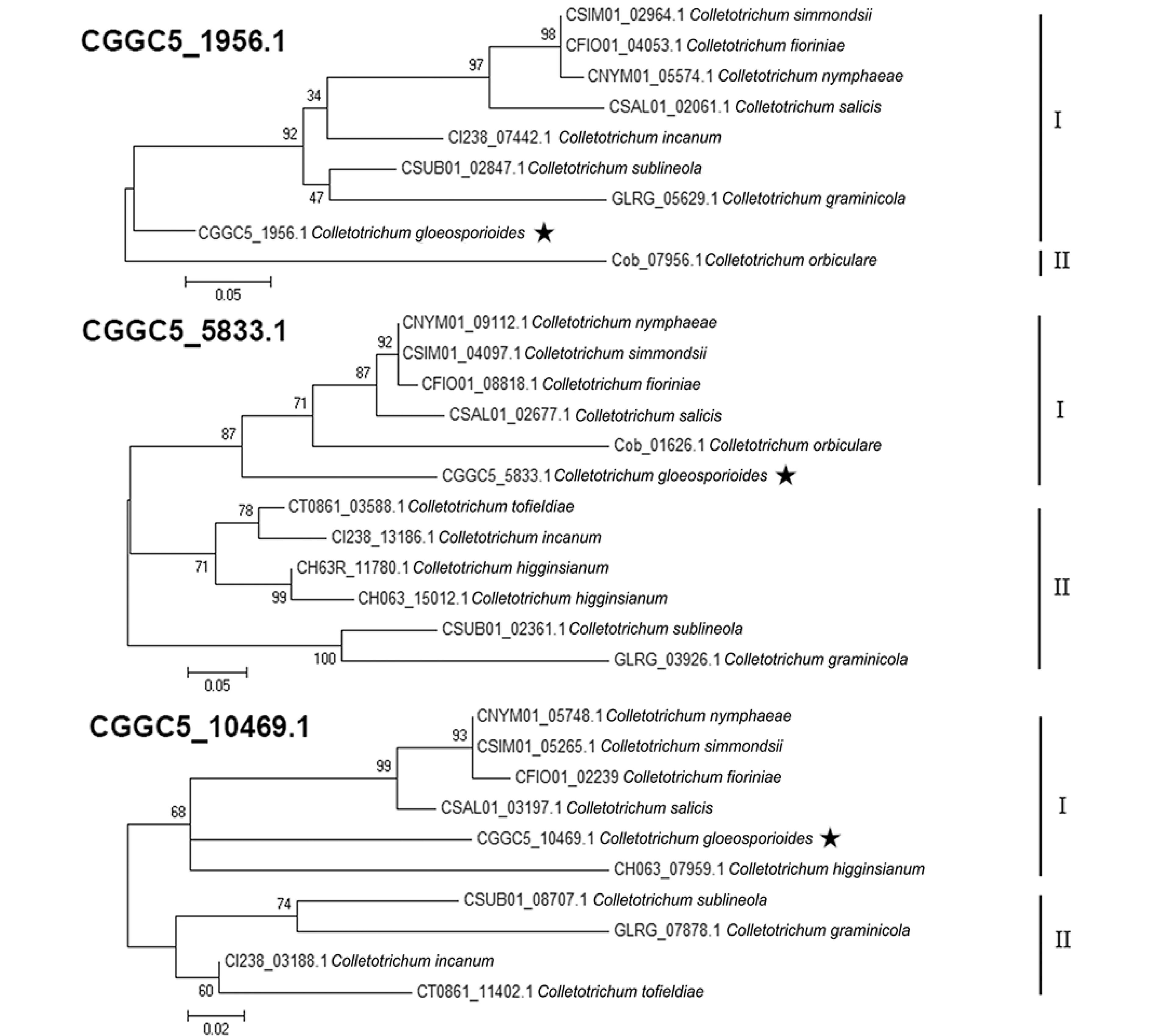
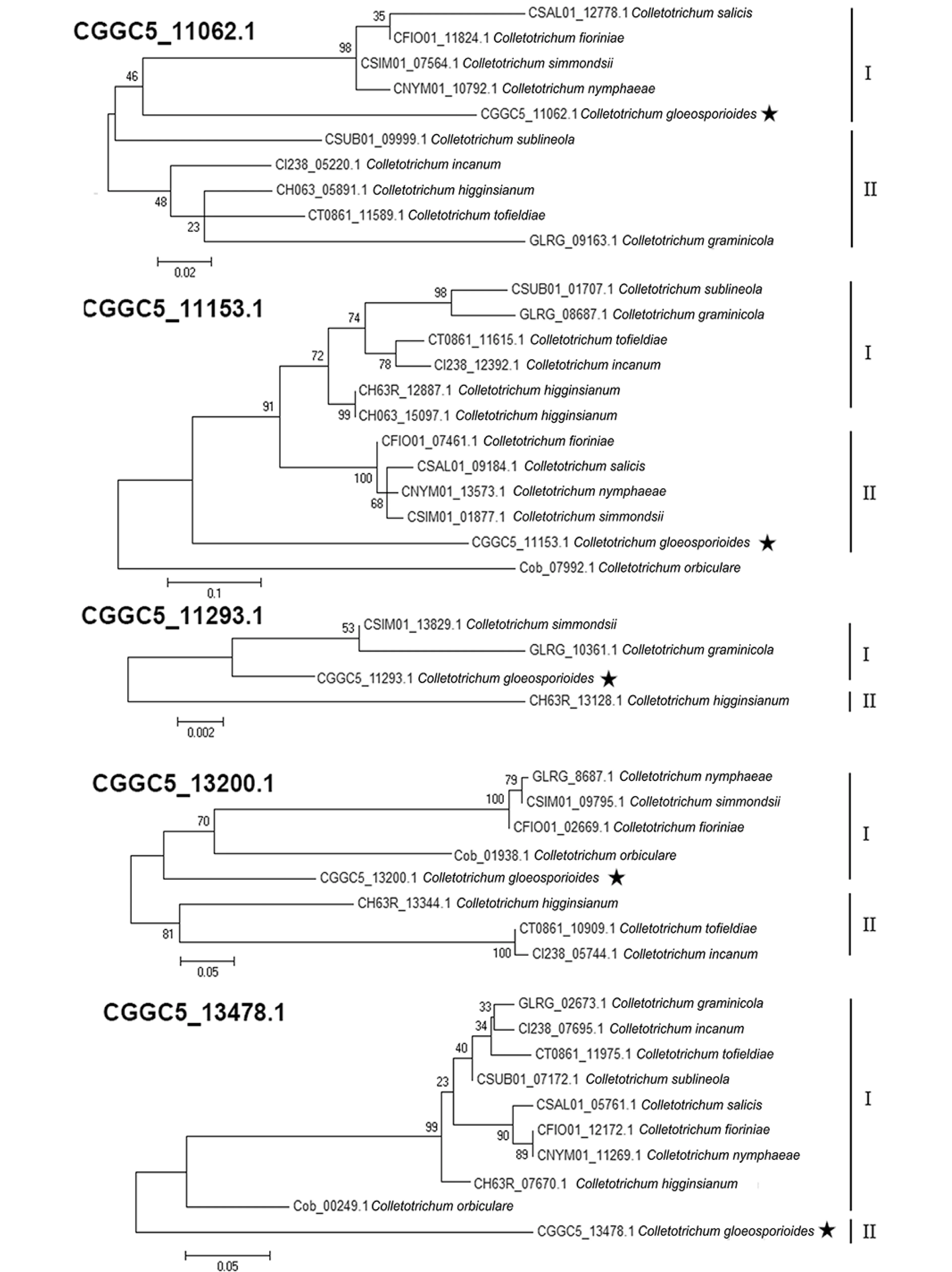
图1 CFEM蛋白系统发育树Fig.1 The phylogenetic tree of CFEM proteins
2.6 CFEM基因在胶孢炭疽菌侵染草莓叶片不同阶段中的转录组学分析
效应子由病原真菌在侵染过程中泌出,因此,在侵染阶段表达的分泌蛋白更有可能与病菌的致病性密切相关,为此,本研究以纯培养和在植物组织内侵染的3个不同阶段的胶孢炭疽菌为研究对象,利用转录组学技术进行分析,比较8个CFEM分泌蛋白编码基因在不同环境下的差异表达。图2为转录组测序各样本总RNA 的提取质量检测:28S rRNA和18S rRNA条带较清晰,满足测序需要。转录组测序结果表明,8个CFEM蛋白编码基因中,CGGC5_13478.1和CGGC5_11153.1在侵染后3个阶段均上调表达,其中CGGC5_13478.1在侵染后3个时间点上调均大于80倍。CGGC5_13200.1在附着胞形成阶段表达显著上调,在活体营养阶段和死体营养阶段表达量则有一定程度下调。相较之下,CGGC5_11293.1和CGGC5_10469.1在活体和死体营养阶段均有表达。在活体营养阶段特异性上调表达的基因有CGGC5_11602.1和CGGC5_5833.1,其中CGGC5_5833.1上调倍数达90倍。而仅有一个CFEM蛋白编码基因,CGGC5_1956.1,在死体营养阶段上调表达,与对照组相比上调约34倍(图3)。这些结果说明,尽管8个CFEM蛋白编码基因在胶孢炭疽菌侵染均有上调表达,但表达的时间点和程度有较大差异,说明这些CFEM分泌蛋白在胶孢炭疽菌侵染过程中发挥不同的作用。
图2 转录组测序RNA样本琼脂糖凝胶检测Fig.2 Agarose gel electrophoresis of total RNA
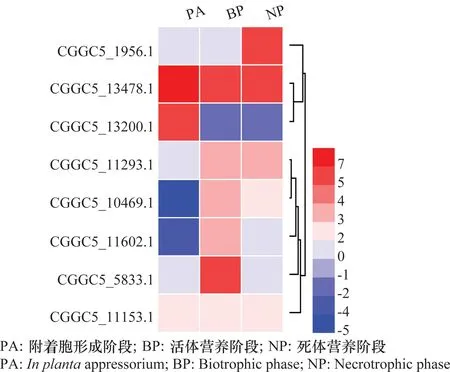
图3 CFEM分泌蛋白编码基因差异表达热图Fig.3 Heat map showing expression levels of CFEM protein-coding genes
2.7 草莓胶孢炭疽菌CFEM分泌蛋白的RT-PCR分析
采用RT-PCR方法对8个CFEM分泌蛋白在胶孢炭疽菌在不同侵染阶段的表达情况进行进一步验证,结果显示,8个CFEM分泌蛋白在侵染的3个不同阶段均有表达(表5)。其中,CGGC5_13200.1在附着胞形成阶段特异表达;CGGC5_11602.1和CGGC5_5833.1在活体营养阶段特异表达,在此阶段,胶孢炭疽菌在寄主体内形成初级菌丝;CGGC5_1956.1在死体营养阶段特异表达,此阶段,次级侵染菌丝形成并诱导寄主细胞死亡。CGGC5_13478.1和CGGC5_11153.1在3个阶段均有表达。CGGC5_11293.1和CGGC5_10469.1在活体和死体营养阶段均有表达,与转录组测序结果一致。因此,8个CFEM分泌蛋白皆可在胶孢炭疽菌侵染阶段表达,预测其均为胶孢炭疽菌CFEM效应子。
表5CFEM分泌蛋白的转录分析1)
Table5TranscriptionalanalysisofColletotrichumgloeosporioidessecretoryproteins

蛋白序列号ProteinID3个侵染阶段Allthreephases附着胞形成阶段Inplantaappressoriumphase活体营养阶段Biotrophicphase死体营养阶段Necrotrophicphase阴性对照NegativecontrolCGGC5_1956.1+CGGC5_13478.1+CGGC5_13200.1+CGGC5_11293.1++CGGC5_11153.1+CGGC5_11062.1+CGGC5_10469.1++CGGC5_5833.1+
1) “+” 表示CFEM候选效应子在该阶段表达。 “+” indicates that this CFEM candidate effector is expressed in this phase.
3 讨论
目前有关半活体营养型真菌如何应对寄主植物的免疫反应及其如何调控活的寄主植物细胞的机制尚不明确[13]。然而近年来的研究发现,与植物和动物致病细菌一样,植物病原真菌也能够产生和分泌效应子。这些效应子能够与寄主植物互作且在病原真菌致病的过程中起着至关重要的作用[14-15]。
自从在粗球孢子菌Coccidioidesimmitis中首次发现CFEM蛋白以来,一些新的CFEM蛋白也从不同的真菌中鉴定出来。这些含有CFEM蛋白的真菌分别为稻瘟病菌Magnaporthegrisea、白假丝酵母C.albicans、近平滑假丝酵母C.parapsilosis、烟曲霉Aspergillusfumigatus和尖孢镰刀菌Fusariumoxysporum[9,17-19]。对CFEM蛋白的功能研究发现,这类蛋白在真菌的某些生理过程中起作用,可能与致病性相关。Ling等[20]将尖孢镰刀菌F.oxysporum中12个不同的专化型进行了分析,发现每个专化型平均含有16个CFEM蛋白,其中某些CFEM蛋白含有DR基序。包含DR基序的CFEM蛋白(CFEM_DR蛋白)在尖孢镰刀菌菌株间非常保守,相比其他真菌,CFEM_DR蛋白的数量在尖孢镰刀菌中更多。对CMEF_DR蛋白的表达分析发现,编码CMEF_DR蛋白基因在孢子和菌丝阶段表达量明显高于其他阶段,且在接种寄主3 d后,表达量显著升高。由此说明CMEF_DR蛋白在定殖和侵染寄主过程中发挥着重要的作用。井忠英等[9]对禾谷炭疽菌C.graminicola中的CFEM蛋白进行了生物信息及转录分析。结果表明,禾谷炭疽菌含有32个CFEM蛋白,且其中22个为分泌蛋白。这22个分泌蛋白在病菌侵染时期的3个阶段均有表达。预测这22个CFEM分泌蛋白为禾谷炭疽菌致病相关的效应子。
高通量测序技术的快速发展及全基因组测序真菌数量的大幅增加,为在病原真菌中鉴定效应子提供了有力的保障。有关草莓胶孢炭疽菌效应子的报道十分有限[21],其尚无CFEM效应子的相关研究。由于缺乏一致的序列特征或结构折叠,真菌的效应子预测是一个难题。
本文利用Pfam数据库,对胶孢炭疽菌的基因组进行分析,获得22个含有CFEM结构域的蛋白。效应子通常经内质网或高尔基体分泌,由此需要疏水的N末端信号肽[22]。迄今为止,经研究证实的真菌效应子均含有该信号肽[23]。本文采用SignalP、TargetP及TMHMM软件对22个CFEM蛋白进行了分析,综合结果分析预测,8个CFEM蛋白为胶孢炭疽菌的分泌蛋白。对其利用MEGA进行系统发育树的构建,结果表明,胶孢炭疽菌CFEM蛋白不属于分支二,在分支一中又单独成一支,与已报道的禾谷炭疽菌中的CFEM分泌蛋白亲缘关系相对较远。说明与其他炭疽菌相比,胶孢炭疽菌CFEM蛋白有其自身的特点。由于效应子与病原真菌侵染相关,因此,本文采用转录组测序和RT-PCR技术对8个CFEM分泌蛋白在3个不同侵染时期的转录表达进行了研究,结果表明,8个CFEM分泌蛋白均在胶孢炭疽菌的侵染阶段表达,但表达的阶段和程度有较大差异。其中CGGC5_13478.1在侵染后3个时间点上调均大于80倍,说明其可能在胶孢炭疽菌整个侵染过程中都发挥重要作用。除此之外,CGGC5_5833.1在活体营养阶段上调表达高达90倍,说明其可能在活体营养阶段发挥重要作用。除此之外,其他CFEM基因在不同时间点均有上调表达。综合以上结果预测,8个CFEM分泌蛋白有可能是胶孢炭疽菌的效应子。本实验室将在后续试验中,对这些推测加以求证,明确CFEM蛋白效应子的功能,为抗病育种奠定坚实基础。
[1] Liu Xie, Zhang Jingze, Wan Yao, et al. Identification ofColletotrichumspp. isolated from strawberry in Zhejiang Province and Shanghai City, China [J]. Journal of Zhejiang University-Science B(Biomedicine & Biotechnology), 2010, 11(1):61-70.
[2] Hadwiger L A, George N. Agrios, Plant Pathology [M]. 5th ed. Amsterdam: Elsevier Academic Press, 2005.
[3] Stergiopoulos I, Wit P J G M D. Fungal effector proteins [J]. Annual Review of Phytopathology, 2009, 47(1):233-263.
[4] Jones J D G, Dangl J L. The plant immune system [J]. Nature, 2006, 444(7117):323-329.
[5] Kamoun S. A catalogue of the effector secretome of plant pathogenic oomycetes [J]. Annual Review of Phytopathology, 2006, 44(1):41-60.
[6] Gan P, Ikeda K, Irieda H, et al. Comparative genomic and transcriptomic analyses reveal the hemibiotrophic stage shift ofColletotrichum, fungi [J]. New Phytologist, 2013, 197(4):1236-1249.
[7] 汪倩, 何朝族, 罗红丽. 橡胶树胶孢炭疽病菌CgBASP2基因敲除突变体构建及其致病力分析[J]. 热带生物学报, 2015, 6(1): 41-46.
[8] 何芬. 橡胶树炭疽病菌效应蛋白基因CgLysM的RNAi突变体构建及其蛋白的亚细胞定位[D]. 海口:海南大学, 2014.
[9] Kulkarni R D, Kelkar H S, Dean R A. An eight-cysteine-containing CFEM domain unique to a group of fungal membrane proteins[J]. Trends in Biochemical Sciences, 2003, 28(3):118-121.
[10] Zhang Zhenna, Wu Qinyi, Zhang Guizhi, et al. Systematic analyses reveal uniqueness and origin of the CFEM domain in fungi [J]. Scientific Reports, 2015, 5:13032.
[11] 井忠英, 王国梁, 刘文德. 禾谷炭疽菌CFEM效应子的生物信息学鉴定与转录分析[J]. 植物保护, 2016, 42(1): 81-86.
[12] Mendgen K, Hahn M. Plant infection and the establishment of fungal biotrophy[J]. Trends in Plant Science, 2002, 7(8): 352-356.
[13] Horbach R, Navarro-Quesada A R, Knogge W, et al. When and how to kill a plant cell: Infection strategies of plant pathogenic fungi [J]. Journal of Plant Physiology, 2011, 168(1): 51-62.
[14] Koeck M, Hardham A R, Dodds P N. The role of effectors of biotrophic and hemibiotrophic fungi in infection [J]. Cellular Microbiology, 2011, 13(12): 1849-1857.
[15] Dodds P N, Rafiqi M, Gan P H P, et al. Effectors of biotrophic fungi and oomycetes: pathogenicity factors and triggers of host resistance [J]. New Phytologist, 2009, 183(4): 993-1000.
[16] Kamoun S. The secretome of plant-associated fungi and oomycetes [M]∥ Deising H B.Plant relationships. Berlin: Springer, 2009:173-180.
[17] Vleeshouwers V G, Oliver R P. Effectors as tools in disease resistance breeding against biotrophic, hemibiotrophic, and necrotrophic plant pathogens [J]. Molecular Plant-Microbe Interactions: MPMI, 2014, 27(3): 196-206.
[18] Weissman Z, Kornitzer D. A family of Candida cell surface haem-binding proteins involved in haemin and haemoglobin-iron utilization[J]. Molecular Microbiology, 2004, 53(4): 1209-1220.
[19] Vaknin Y, Shadkchan Y, Levdansky E, et al. The threeAspergillusfumigatus, CFEM-domain GPI-anchored proteins (CfmA-C) affect cell-wall stability but do not play a role in fungal virulence [J]. Fungal Genetics & Biology, 2014, 63(1): 55-64.
[20] Ling Jian, Zeng Feng, Cao Yuexia, et al. Identification of a class of CFEM proteins containing a new conserved motif inFusariumoxysporum[J]. Physiological & Molecular Plant Pathology, 2015, 89: 41-48.
[21] Stephenson S A, Hatfield J, Rusu A G, et al. CgDN3: An essential pathogenicity gene ofColletotrichumgloeosporioidesnecessary to avert a hypersensitive-like response in the hostStylosanthesguianensis[J]. Molecular Plant-Microbe Interactions, 2000, 13(9):929-941.
[22] Heijne G V. The signal peptide [J]. Journal of Membrane Biology, 1990, 115(3): 195-201.
[23] Presti L L, Lanver D, Schweizer G, et al. Fungal effectors and plant susceptibility[J]. Annual Review of Plant Biology, 2015, 66: 513-545.
(责任编辑: 田 喆)
BioinformaticidentificationandtranscriptionalanalysisofColletotrichumgloeosporioidescandidateCFEMeffectorproteins
Zhang Liqing, Duan Ke, Zou Xiaohua, He Chengyong, Gao Qinghua
(ResearchInstituteofForestryandFruitTrees,ShanghaiAcademyofAgriculturalSciences,Shanghai201403,China)
Plant pathogens secrete effector proteins into host tissues to regulate host physiological processes and promote its infection. Common in several fungal extracellular membrane (CFEM) proteins is unique to fungi and plays an important role in pathogenesis. In our study, we obtained 22 CFEM proteins ofColletotrichumgloeosporioidesby searching Pfam database. Then, we conducted a bioinformatics analysis on signal peptide, transmembrane domain and protein subcellular localization of these 22 CFEM proteins. The results showed that only 8 of them were secreted proteins. The transcriptome and RT-PCR analysis of these 8 CFEM proteins at different stages of infection were performed. Among them, one protein was expressed ininplantaappressorium stage, two were expressed in biotrophic stage and one was expressed in necrotrophic stage. Collectively, the 8 CFEM proteins were predicted to be the candidate effectors ofC.gloeosporioides. Our findings provide a theoretical basis for in-depth analysis for the function of CFEM effectors in plant pathogenic fungi.
Colletotrichumgloeosporioides; effector; CFEM protein
S 436.639
: ADOI: 10.3969/j.issn.0529-1542.2017.05.006
2016-10-08
: 2017-01-05
国家自然科学基金青年基金(31501592);上海市科委自然科学基金项目(14ZR1436800);上海市科委现代农业领域重点科技项目(16391901400);上海市科委基础研究重点项目(14JC1405400);上海市瓜果产业技术体系(沪农科产字(2017)第1号)
* 通信作者 E-mail: qhgao20338@sina.com
