基于深度卷积网络和结合策略的乳腺组织病理图像细胞核异型性自动评分
2017-09-08周超徐军*罗波
周 超 徐 军* 罗 波
1(南京信息工程大学江苏省大数据分析技术重点实验室,南京 210044)2(华中科技大学附属武汉市中心医院病理科,武汉 430014)
基于深度卷积网络和结合策略的乳腺组织病理图像细胞核异型性自动评分
周 超1徐 军1*罗 波2
1(南京信息工程大学江苏省大数据分析技术重点实验室,南京 210044)2(华中科技大学附属武汉市中心医院病理科,武汉 430014)
细胞核异型性是评估乳腺癌恶性程度的一个重要指标,主要体现在细胞核的形状、大小变化、纹理和质密度不均化。提出基于深度学习和结合策略模型的乳腺组织细胞核异型性自动评分模型。该模型使用3个卷积神经网络,分别处理每个病例的3种不同分辨率下的组织病理图像,每个网络结合滑动窗口和绝对多数投票法,评估每个病例同一种分辨率下的图像的分值,得到3种分辨率下的评分结果。使用相对多数投票法,综合评估每个病例的最终细胞核异型性评分结果。为评估模型对细胞核异型性评分的有效性,利用训练好的模型对124个病例的测试图像进行自动评分,并把其评分结果与病理医生的评分结果作比较,进行性能评估。该模型的评分正确率得分为67分,其结果在现有的细胞核异型性评分模型中准确率排名第二。此外,该模型的计算效率也很高,平均在每张×10、×20、×40分辨率下图像的计算时间分别约为1.2、5.5、30 s。研究表明,该细胞核异型性评分模型不仅具有较高的准确性,而且计算效率高,因此具备潜在的临床应用能力。
细胞核异型性;深度卷积网络;绝对多数投票法;相对多数投票法
引言
美国癌症协会(ACS)2016年发布的数据显示,乳腺癌、肺癌和结直肠癌依次居全球女性恶性肿瘤发病率前三位,占所有新发病例的一半,乳腺癌独占29%[1]。目前,我国乳腺癌发病率位居女性恶性肿瘤之首,但相比肺癌、胃癌和肝癌,其预后效果最好[2]。乳腺癌早诊断、早治疗,其5年无病生存率可达90%以上。组织病理学分析作为乳腺癌诊断的“金标准”,可评估乳腺癌的恶性程度,从而指导临床医生制订治疗方案[3]。
苏木素- 伊红染色(H&E)是临床组织病理分析中应用最为广泛的一种染色方式,H&E图像可用于组织学分级,评估乳腺癌的分化程度,进而评估其恶性程度[4]。病理医生通常是通过研究和统计视角下细胞的形态和分布,进行人工评分。由于组织病理图像具有高度复杂性,因此人工分析具有较大的难度。文献[5]中的研究表明,由于人工分析方法具有很强的主观性,不同的病理医生在相同的客观条件下在人工评分上具有较大的不一致性。人工分析除了容易受主观和环境因素的影响之外,其过程也是十分耗时费力的,人力代价很高,而计算机辅助诊断技术可以弥补人工分析的缺陷。图1(a)~(c)分别为乳腺组织病理图像中低、中、高3种等级的细胞异型性组织病理图像。这三幅图像内容无序,目标众多,背景复杂,因此对于图像处理算法来说具有较大的难度,如果运用传统的计算机分析算法很难自动地区分3种类型的图像。
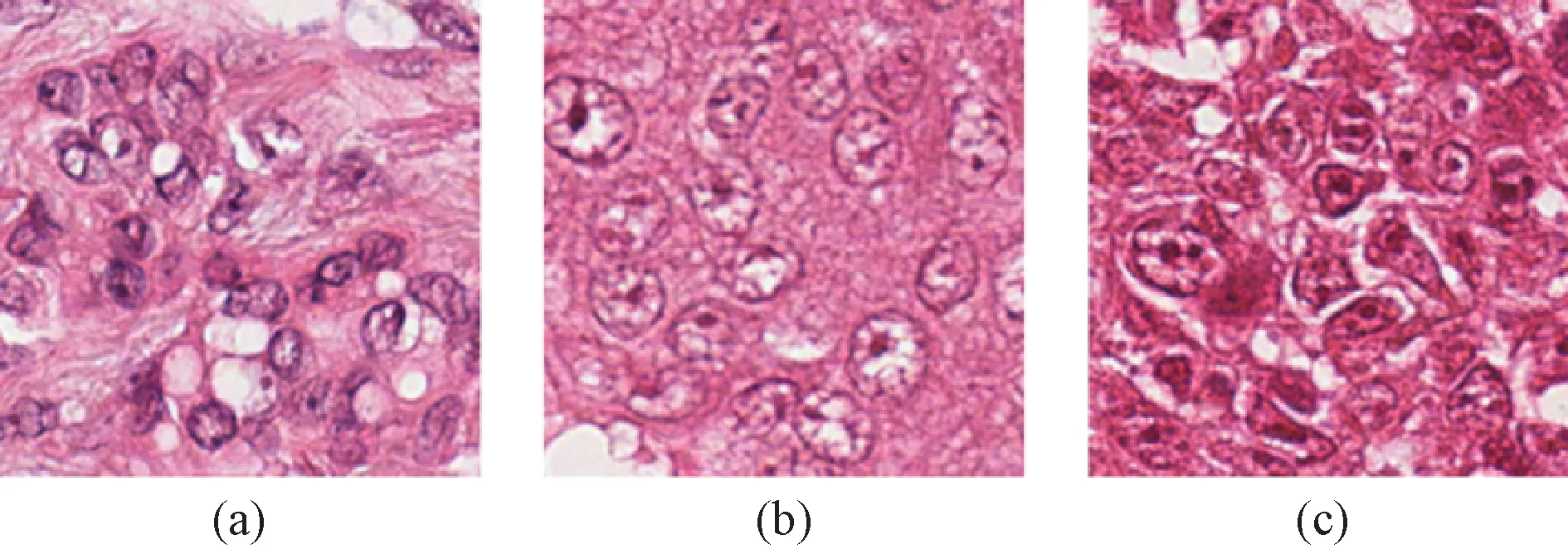
图1 具有不同细胞核异型性等级的乳腺组织病理图像。(a)~(c) 细胞核异型性等级为1~3的组织病理图像块Fig.1 Pathological nuclear atypia images of different scores. (a)~ (c) Nuclear atypia score values 1~ 3
根据国际卫生组织发布的诊治指南,乳腺癌恶性程度的诊断采用最广泛的是诺丁汉分级系统(NGS)[6]。这套系统主要是依据乳腺组织病理图像的3个指标:乳腺导管(腺管)的形成程度、有丝分裂数、细胞核异型性。腺管的形成程度是评估肿瘤形成腺管结构的百分比,所占比例越大,乳腺癌等级越低;有丝分裂相记数评估在显微镜400倍放大倍数下核分裂像计数,数量越多,乳腺癌等级越高;细胞核异型性评估肿瘤细胞核与正常乳腺上皮细胞核的差异性,差异越大,乳腺癌等级越高。每一项指标恶性程度由低到高的分值范围是1~3 分,因此每个患者3个指标综合起来的分值范围是3~9。 患者的评分分值越接近1,表明癌症的恶性程度越低,因此患者的治疗和预后的效果越好;相反,恶性程度越高,治疗和预后的效果也越差。因此,精确地确定NGS系统的3个指标分值,在临床中至关重要。近些年来,随着组织切片数字扫描技术的快速发展[7],以及计算机处理高分辨率组织病理图像的能力的增强[8],计算机辅助分析组织病理片切片领域的研究非常活跃。针对乳腺癌诺丁汉分级系统3个指标的计算机辅助自动评分,2012和2014年的国际模式识别大会(International Conference on Pattern Recognition,ICPR)[9],以及2013 年的国际医学图像处理与计算机辅助干预(the International Conference on Medical Image Computing and Computer Assisted Intervention,MICCAI)国际大会[10]发布了针对第二个指标(有丝分裂次数)的竞赛[11]。在MICCAI 2013 的竞赛中,基于深度卷积网络的方法[12]获得了竞赛的第一名。2014年,ICPR大会继续发布了以组织病理图像自动分类为基础的细胞核异型性(诺丁汉分级系统的第三个指标)自动评分的竞赛,内容为乳腺组织病理图像中细胞核异型性的定量评估。细胞核异型性作为乳腺癌分级的三大指标之一,一直就受到研究人员的高度重视。文献[13]详细地描述了细胞核异型性的评定标准。细胞核异型性的评分标准为:
1)1分。如图1(a)所示,细胞核较小,与正常细胞核相比,在尺寸上有略微增大,有正常的轮廓,细胞核染色质分布均匀。
2)2分。如图1(b)所示,与正常细胞核相比尺寸要大,可见泡状核,核仁清晰,大小及形态较一致。
3)3分。如图1(c) 所示,细胞核多形性程度增加,细胞核明显增大,核仁清楚,且可见奇异细胞核。
近年来,由于计算机视觉和机器学习等领域研究的快速发展,推动了组织病理图像的分析领域研究的发展。许多高性能的计算机视觉算法被应用于处理病理图像,并取得了不错的效果。在文献[14]中,Adnan等使用了区域协方差描述子来实现对细胞核异型性的图像评分。近几年,深度学习[15]在图像的自动分类和识别方面取得了巨大成功[16],因此受到了学术界和工业界的高度关注。深度学习以数据驱动为基础,而且是端对端的学习模式,因此在图像处理和分析方面获得极大的成功。由于组织病理图像具有高度的复杂性,因此与传统的方法相比,基于深度学习的分析方法能够体现出明显的优势,能够使高效地分析数字病理组织图像向前迈进一大步[17- 18]。文献[17]采用了深度学习的方法,自动、快速、准确地检测组织病理图像中的细胞核。结果表明,与传统的细胞核检测方法相比,深度学习方法具有较高的鲁棒性和准确性。
本研究的主要目标是:构建一个基于深度卷积网络和结合策略的模型,以实现组织病理图像中细胞核异型性的自动评分。为了实现这一目标,使用深度卷积网络结合滑动窗口的方法,高效精确地分析病理图像,同时毫不遗漏地扫描和评估尺寸较大的组织病理切片。运用绝对多数投票法,评估每个分辨率下病理切片的分值,最后运用相对多数投票法,综合不同分辨率下的分值,对每个病例做最后的投票,以决定评分的分值。
接下来的内容如下:详细介绍本研究提出的乳腺组织病理图像细胞核异型性自动评分模型,分析该评分算法的实验设计及实验结果,最后对该项工作进行讨论和总结。
1 方法
1.1 乳腺癌细胞核异型性评分模型
本研究提出的乳腺癌细胞核异型性评分模型的整体框架和流程如图2所示,主要的思路是:针对每个病例同一个视角、3种不同分辨率(10、20、40倍)下的组织病理图像,分别输入3个深度卷积网络进行训练,从而得到3个在不同分辨率下的分类模型。然后,利用已经训练好的深度卷积网络,结合滑动窗口的方法,分别处理每个分辨率下的乳腺病理切片原图,得到一张图像很多子集的得分。采用绝对多数投票法,处理同一分辨率下所有子集的得分,得出这个分辨率下图像的得分;使用相对多数投票法,对每个病例3个分辨率下绝对多数投票法的结果进行投票,得到每个病例的最终得分[19]。 该算法具体流程如图2所示。

图2 基于深度卷积网络和结合策略的乳腺组织病理图像细胞核异型性评分模型流程Fig.2 The flowchart of nuclear atypia grading based on deep convolutional networks and combination strategy on breast histopathology
1.1.1 深度卷积网络
深度卷积网络作为最初的人工神经网络模型前馈神经网络的一种,在近些年得到了长足的发展,特别是在图像处理和语音识别方面。作为可训练的多层网络结构,卷积阶段包括卷积层、非线性变换层、下采样层(即池化层)[20]。
卷积层通过卷积核来提取图像的特征,如图3中特征提取虚线框所示,这是基于局部感受野的概念。每个卷积核提取输入特征图上所有位置的特定特征,实现同一个输入特征图上的权值共享。为了提取输入特征图上不同的特征,就使用不同的卷积核进行卷积操作。非线性变换层将卷积层提取的特征进行非线性映射。传统的卷积神经网络采用的是饱和非线性函数来进行非线性映射,包括sigmoid、tanh 或者softsign。 最近提出的卷积神经网络中,经常使用非饱和非线性函数ReLU。在模型进行反向传播、梯度下降的时候,ReLU要比传统的饱和非线性函数的收敛速度更快,这样训练网络时具有很高的效率。
池化层是对每个特征图进行独立的操作,目前使用较多的是采用平均池化或者最大池化两种操作。平均池化是在选定领域窗口内计算像素的均值,而最大池化是计算选定区域窗口的最大值,一般窗口平移的步长为1。特征图通过池化操作之后,特征图的分辨率降低,却保留了有效的特征,同时池化也达到了降维的目的。特征提取完成之后,将最后一层的输出特征图后连接全连接层和分类器,如图3 中预测分类虚线框所示。
深度卷积网络使用原始的图像像素直接作为网络的输入,与传统的图像识别算法不同,避免了复杂的人工特征的提取[21];权值共享可以减少权值的数量,从而降低网络的计算复杂度;局部感受野的使用,使得整个网络所观测到的特征具有平移和旋转不变性。网络的输出直接对应于类别,这样的端对端的网络模型极大提高了识别精度。
1.1.2 滑动窗口
为了将模型运用到细胞核异型性分类当中,并且考虑到组织切片中组织结构的复杂性、多样性、遍布性和无规律性,本研究采用滑动窗口的方法[17]遍历整张图像。利用所选的滑动框,从切片图像的左上方开始,从左往右、从上到下依次滑动,每滑动一次都对窗口内的图像块进行判断,判断每一个窗口所属的等级(即评分)。在本实验中,使用的窗口大小为256×256,滑动的步长为窗口大小的1/5。在滑动窗口扫过图像边缘时会出现边界效应,即角点处的像素出现真空状态,在本研究中使用镜像填塞(即像素围绕图像边界)进行镜像反射,解决了边界效应问题。
1.1.3 投票法
在本研究中,当使用滑动窗口的方法来处理每个病例的单个分辨率图像时,每一个分辨率下的图像会得到很多子图像块的预测结果,通过投票法,结合这些子图像块的结果,用来预测这个分辨率下整张图像的结果。在这个阶段,选择使用绝对多数投票法[22],完成单个分辨率下的整张图片评分的任务。
绝对多数投票法的表决策略为
(1)
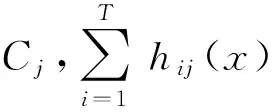
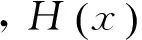
在对单个分辨率下图像的评分完成之后,结合3个分辨率下的评分来确定图像最终的结果。本研究采用相对多数投票法的结合策略来做最终决策。相对多数投票法[23]即为预测得票最多的标记,若同时有多个标记获得最高票,则从中随机选取一个。
相对多数投票法的表决策略为
(2)
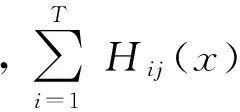
1.2 实验设计
1.2.1 实验数据
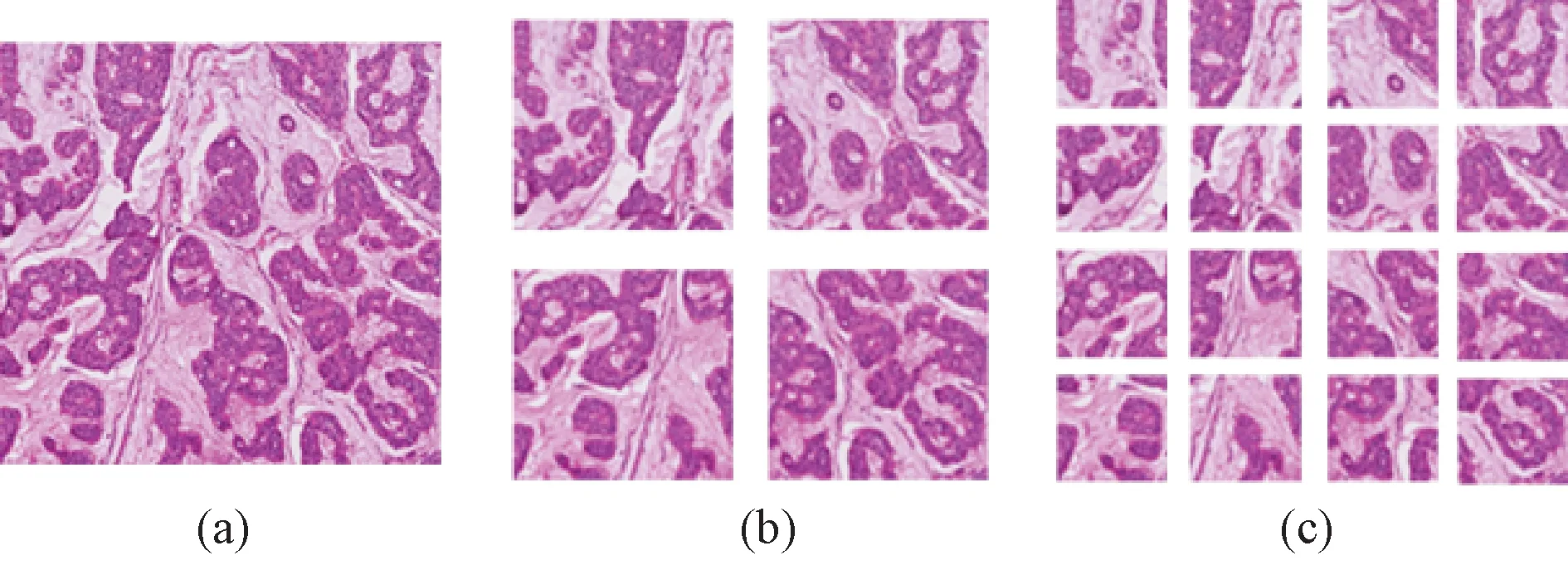
图4 同一个视角下组织病理图像的3种分辨率下的图像样例。(a)×10分辨率下的图像,将(a)分割4×4图像块得到(b),(b)中的每个小块的尺寸和(a)的尺寸是一样的。同样,(c) 中的每块图像块是由(b)中每个图像块的1/4,尺寸和(a)的尺寸是一样的Fig. 4 Pathological images of three resolutions under the same view. (a) At ×10 magnification; (b) At ×20 magnification; (c)At ×40 magnification. Each ×10 frame is subdivided into four frames at ×20 magnification. Each ×20 frame is also subdivided into four frames at ×40 magnification. Each piece of (b) and (c) has the same size of (a).
使用的数据是由2014年国际模式识别会议公布的竞赛提供,是由组织者从法国巴黎的萨伯特慈善医院病理科挑选出的乳腺病理组织图像,并且这些数据都是由2~3名经验丰富的病理医生共同标注。数据中每个病例的图像都包含3种分辨率下的图像,分辨率分别是×10、×20和×40,每个病例的3个分辨率的图片大小为769像素×688像素、1 539像素×1 376像素和3 078像素×2 752像素(见图4)。 在实验过程中,随机分别从3种不同分辨率下训练集中截取256×256的图像块,构建实验所用的包含3种不同分辨率的数据集。在训练集中,分辨率为×10、×20、×40的数据集分别约有2、4、8万张。除此之外,在训练神经网络的时候,对现有数据进行了数据扩容的操作,主要包括对每张图像的旋转和镜像操作。除了训练图片,竞赛还提供124个病例的测试图片,每个病例包括3张不同分辨率的乳腺病例组织图像,用来检验自动评分模型的泛化能力。需要指出的是:该数据集只提供了训练集的评分分值,并没有提供测试集的评分分值。为了测试算法的性能,竞赛组织者要求参与者把对每张测试集的评分分值发给组织者,由组织者提供算法的自动评分准确率。
1.2.2 实验环境
实验使用的硬件设备如下:处理器,Intel(R) Core(TM) i7- 3770 CPU @ 3.40 GHz;内存(RAM),16.0 GB;独立显卡,GeForce GTX Titan X;系统类型,64 位window 7操作系统,Ubuntu 14.04;开发工具,Matlab R2014a,Caffe 框架[24]。
1.2.3 实验设置
所有训练病例的3个不同分辨率对应3个数据集,数十万个的图片,直接送入Caffe的框架中,训练3个对应于不同分辨率的深度卷积网络。在训练过程中,为了提高训练的效率和准确度,实验深度卷积网络的训练是在Alexnet[16]网络参数上微调进行的,见表1。Alexnet 网络模型是2012年Imagenet 图像分类大赛获得第一名的参赛模型,也是深度学习在图像处理方面的完美展示。该模型是由数百万张的图像上训练出来的,具有很强的鲁棒性。众所周知,深度卷积网络是以数据驱动为基础的方法,数据集的大小直接影响模型的能力,这也是选择这个模型的重要原因。
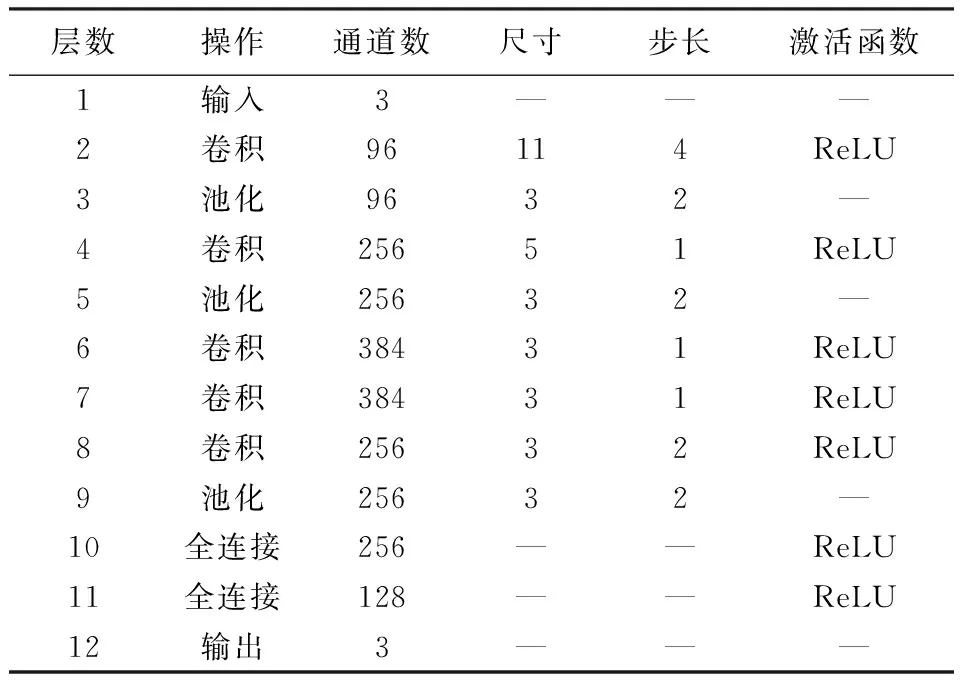
表1 所使用的深度卷积网络结构及其参数设置Tab.1 The structure details and parameters of deep convolutional networks in experiment
乳腺病理组织图像采取滑动窗口的方法,输入到相对应的深度卷积网络中,以判断每个窗口的分数。根据病理医生的给病理组织切片评分的经验,使用绝对多数投票法来推测输入图片的异型性分数。对于每个病例的3个不同分辨率下图像的3个预测结果,使用相对多数投票法来决策该病例的最终预测结果。
1.3 实验评估方法
在实验测试阶段,使用上述方法对测试图片进行细胞核异型性评分,得到每张测试图片的评分。由于2014 年ICPR 竞赛未公布测试图片的专家标记(即病理医生的评分),为了检验本模型的分级能力,笔者将每个病例图像的自动评分分值发给了竞赛的组织者Ludovic Roux,由他来评价本方法自动评分的准确性。Ludovic Roux 评估的标准采取了计分制,记不同模型预测输入组织病理图像得分为p,病理医生给这张图片给分为g,这张图片的评估得分为S,评估标准如下:
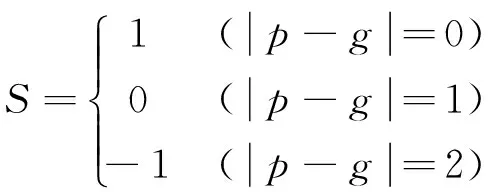
(3)
最后,计算所有测试病例的评估分数总和,得到此模型的预测测试数据的总分。
2 结果
利用所提出的方法,对2014年国际模式识别会议竞赛提供的124病例的测试图片进行细胞核异型性评分。图5是由Ludovic Roux提供的本方法和迄今为止评分准确率排名前几位的算法评估分值(points)的对比结果。结果表明,所提出方法的评分准确率目前排名第二,领先于除了华威大学的其他算法。由图中的结果可以看出,本方法的评估结果是67分,明显地优于其他大部分的算法,这也证实本方法在细胞核异型性评分上的有效性。

图5 乳腺组织细胞核异型性评分结果Fig.5 Comparison result diagram of nuclear atypia grading
为了验证结合多个分辨率图像信息对模型的影响,本方法除了与其他算法作对比,还对不同分辨率下的结果进行对比。图6中×10、×20、×40分别对应于各自分辨率下的得分结果,即各自分辨率下绝对多数投票的评分结果,而本方法是结合上述3个分辨率下的结果、再进行相对多数投票的评分结果。如图6所示,所提出的利用投票法对多个分辨率下的评分结果进行综合评分的方法,在实际测试过程中增强了算法对细胞核异型性评分的能力;同时也说明,结合不同分辨率下的信息,在一定程度上可以提高模型对问题的分析能力。
在实验过程中,本方法在计算机时间上具有一定的优势,在给每一张分辨率分别为×10、×20、×40的图像自动评分时,计算时间分别约为1.2、5.5、30 s。这说明,所采用的方法不仅评分准确率高,而且计算机速度快,因此具备了在实际临床应用中对组织病理图像自动评分的可能性。
3 讨论
从图5中可以看出,本研究的方法在乳腺组织细胞核异型性测试中的表现要比除了华威大学的其他方法有明显优势。这些方法都遵循着一个流程:首先对感兴趣区域(细胞核区域)进行分割,然后对分割出的感兴趣区域进行特征提取,再利用这些特征训练分类器来实现最终的等级评分。如陕西师范大学的方法[25],利用局部阈值和形态学方法分割细胞核,进而对细胞核提取了包括尺寸、梯度和纹理在内的上百维特征来训练细胞核异型性评分的分类器。同样,华威大学的方法[14]也是遵循这个流程,不同的是华威大学的方法对同一幅图像提取了两次特征,第一次是基于图像整体提取像素级的特征,第二次是基于感兴趣区域提取像素级的特征。而本研究的方法并未使用传统的人工特征,采用与评分结果相结合的自学习特征,即将特征提取阶段和分类器训练阶段合二为一,由分类器的表现来决定特征提取器从图像中提取什么样的特征。一个好的特征描述有利于寻找不同类型图像的区分度和同种类型图像的相似度,从而使分类器的精度得以提高。深度学习的出现正好提供了一个特征学习和图像分类相结合的可行性方案,这样的结合使得学习的特征极大地提高了分类器的能力,也使得计算机辅助的乳腺癌细胞核异型性病理图像分级完全有可能帮助医生制订治疗方案。有了好的特征提取器之后,感兴趣区域的选择同样会影响着细胞异型性的分析。在细胞核异型性程度较高的图像中,细胞核并没有完整的细胞边界而且经常出现扭曲重叠的问题,这都会影响评分的准确率。本研究提出的用滑动窗口从图像中提取图像小块特征的方法可以克服这些问题,并且可以提取到细胞核周边信息的特征,这有利于区分细胞核异型性的等级。
除此之外,在乳腺组织细胞核异型性的分析问题中,结合多分辨率下的信息是非常重要的,这符合病理医生观察病理切片的流程,同时可以有效地利用数据信息。本方法采用结合策略,处理3个不同分辨率下的细胞核异型性评分结果,以确定最终结果。从图6中前3个横条可以看出,×20分辨率下的评分准确率最高,而本方法综合3个分辨率下的评分结果要优于这个分辨率下的结果,这说明低分辨率下的整体信息和高分辨率下的细节信息对细胞核异型性的评分都有着深远影响,无论丢弃了哪种信息都会导致最终的评分结果准确率下降,所以本方法结合多个分辨率下的信息,可以有效地提高细胞核异型性评分的准确率。综上所述,本研究采用深度卷积网络和传统联合决策相结合的方法,充分利用实验数据来驱动深度算法解决细胞核异型性评分问题。传统的图像处理算法很难有效地分析高度复杂的病理组织图像,而深度学习方法的出现和快速发展给研究人员提供了新的研究方向和实验工具,这对病理图像的研究乃至整个病理学的发展都具有至关重要的影响。
4 结论
本研究提出了一种对乳腺组织病理图像细胞核异型性进行评分的深度模型。该模型综合了深度卷积网络和两种结合策略,其中深度卷积网络分别处理每个病例的多分辨率的病理组织图像,采用结合策略处理深度卷积网络的预测结果,本研究使用了两种结合策略,使用绝对多数投票法,得到每个病例每个分辨率下的细胞核异型性的得分,再使用相对多数投票法得到每个病例的最终得分。通过实验数据的测试,模型能够对乳腺细胞核异型性组织图像很好地分级。由于使用了卷积神经网络处理病理组织图像,提高了特征对图像的表达能力,加上端到端的分类器训练方式,再结合多分辨率下的信息,使得本方法具有优良的细胞核异型性评分结果。
所提出的方法在细胞核异型性评分上取得了一定的成果,对于以后的工作还有许多实践经验,可为同类研究提供参考。在算法上,越有效的特征所具有解决问题的能力就会越强,本研究是结合多分辨率下的结果,而未来的研究内容是结合多分辨率下的图像特征,利用这种融合后特征,对细胞核异型性进行评分。
(致谢:该实验的评估工作是由2014年国际模式识别会议竞赛的组织者Ludovic Roux协助实现的。)
[1] Jemal A, Siegel R, Xu Jun, et al. Cancer statistics [J]. Ca Cancer Journal for Clinicians, 2012, 62(1):10- 29.
[2] Chen Wanqing, Zheng Rongshou, Baade PD, et al. Cancer statistics in China [J].Ca Cancer Journal for Clinicians, 2016, 66(2):115- 132.
[3] 龚磊, 徐军, 王冠皓,等. 基于多特征描述的乳腺癌肿瘤病理自动分级[J]. 计算机应用, 2015, 35(12):3570- 3575.
[4] 陈佳梅, 屈爱平, 王林伟,等. 计算机图像分析挖掘乳腺癌病理预后新指标[J]. 生物物理学报, 2014, 30(7):517- 530.
[5] Robbins P, Pinder S, Klerk ND, et al. Histological grading of breast carcinomas: A study of inter observer agreement [J]. Human Pathology, 1995, 26(8):873- 879.
[6] Basavanhally AN, Ganesan S, Agner S, et al. Computerized image- based detection and grading of lymphocytic infiltration in HER2+ breast cancer histopathology [J]. IEEE Transactions on Biomedical Engineering, 2010, 57(3):642- 653.
[7] May M. A better lens on disease [J]. Scientific American, 2010, 302(5):74- 77.
[8] Bourzac K. Software: The computer will see you now [J]. Nature, 2013, 502(7473):92- 94.
[9] Roux L, Racoceanu D, Loménie N, et al. Mitosis detection in breast cancer histological images An ICPR 2012 contest [J]. Journal of Pathology Informatics, 2013, 4(1):4- 8.
[10] Cruz- Roa A, Basavanhally A. Mitosis detection in breast cancer pathology images by combining handcrafted and convolutional neural network features [J]. Journal of Medical Imaging, 2014, 1(3):1- 8.
[11] Veta M, Diest PJV, Willems SM, et al. Assessment of algorithms for mitosis detection in breast cancer histopathology images [J]. Medical Image Analysis, 2015, 20(1):237- 248.
[13] Irshad H. Automated mitosis detection in histopathology using morphological and multi- channel statistics features [J]. Journal of Pathology Informatics, 2013, 4:1- 10.
[14] Khan AM, Sirinukunwattana K, Rajpoot N. A global covariance descriptor for nuclear atypia scoring in breast histopathology images [J]. Surface Science, 2015, 19(5):1637.
[15] Hinton GE, Salakhutdinov RR. Reducing the dimensionality of data with neural networks [J]. Science, 2006, 313(5786):504- 507.
[16] Krizhevsky A, Sutskever I, Hinton GE. Imagenet classification with deep convolutional neural networks [J]. Advances in Neural Information Processing Systems, 2012, 25(2):2012- 2022.
[17] Xu Jun, Xiang Lei, Liu Qingshan, et al. Stacked sparse autoencoder (SSAE) for nuclei detection on breast cancer histopathology images [J]. IEEE Transactions on Medical Imaging, 2015, 35(1):119- 130.
[18] Xu Jun, Luo Xiaofei, Wang Guanhao, et al. A deep convolutional neural network for segmenting and classifying epithelial and stromal regions in histopathological images [J]. Neurocomputing, 2016, 191:214- 223.
[19] Basavanhally A, Ganesan S, Feldman M, et al. Multi- field- of- view framework for distinguishing tumor grade in ER+ breast cancer from entire histopathology slides [J]. IEEE Transactions on Biomedical Engineering, 2013, 60(8):2089 - 2099.
[20] Koushik, Jayanth. Understanding convolutional neural networks [J].Eprint Arxiv ,25 May,2016 [Epub ahead of print].
[21] Zeiler MD, Fergus R. Visualizing and understanding convolutional networks [M]// Fleet,Computer Vision- ECCV 2014. Cham: Springer International Publishing, 2014:818- 833.
[22] Alpaydin E. Combining pattern classifiers: methods and algorithms[J]. IEEE Transactions on Neural Networks, 2007, 18(3):963- 964.
[23] 周志华. 机器学习[M]. 北京:清华大学出版社, 2015:171- 196.
[24] Jia Yangqing, Shelhamer E, Donahue J, et al. Caffe: convolutional architecture for fast feature embedding [J]. Eprint Arxiv, 2014:675- 678.
[25] Chen Lu. Automated image analysis of nuclear atypia in high‐power field histopathological image [J]. Journal of Microscopy, 2015, 258(3): 233- 240.
A Deep Convolutional Networks and Combination Strategy for Automated Nuclear Atypia Grading on Breast Histopathology
Zhou Chao1Xu Jun1*Luo Bo2
1(School of Information and Control, Nanjing University of Information and Technology, Nanjing 210044, China)2(Department of Histopathology, The Central Hospital of Wuhan, Affiliated Hospital of TongJi Medical College Huazhong University of Science and Technology, Wuhan 430014, China)
Nuclear atypia is one of important factors in Nottingham Grading System (NGS) for evaluating the aggressiveness of breast cancer. The nuclear atypia is mainly manifested in change of the nuclear shape, size, texture and uneven density. However, histologic image has complicated nature that makes the automated nuclei atypia grading a pretty difficult task. In the paper we integrated deep convolutional neural networks and combination strategy for automated nuclei atypia grading. Firstly, the histologic patches with three different resolutions were cropped into same size for training three convolutional neural networks models, respectively. During the testing, a sliding window technique was employed to choose image patches and feed to the trained DCNN. Then the majority voting was used to evaluate the grade of the image under each resolution. Finally, plurality voting was employed to evaluate the score based on three different resolutions. The proposed model got 67 points in the test set, ranking the 2nd comparing with all of current methods with good performance. Moreover, the proposed approach was computationally efficient. The average computational time on each images with the resolution of ×10, ×20, ×40 were 1.2, 5.5, and 30 seconds, respectively, indicating that the proposed approach can be applied in clinical routine procedure for automated grading of nuclei atypia on histologic images.
nuclei atypia; convolutional neural networks; majority voting, plurality voting
10.3969/j.issn.0258- 8021. 2017. 03.003
2016-05-06, 录用日期:2016-05-27
国家自然科学基金(61273259);江苏省“六大人才高峰”高层次人才项目(2013-XXRJ-019);江苏省自然科学基金(BK20141482)
R318
A
0258- 8021(2017) 03- 0276- 08
*通信作者(Corresponding author),E- mail: xujung@gmail.com
