翘嘴鲌生长激素(GH)基因与侧翼区的克隆及分析
2017-09-03刘士力贾永义蒋文枰迟美丽赵金良顾志敏傅建军
刘士力,贾永义,蒋文枰,迟美丽,程 顺,赵金良,顾志敏,*,傅建军
(1.浙江省淡水水产研究所 农业部淡水渔业健康养殖重点实验室/浙江省淡水水产遗传育种重点实验室,浙江 湖州 313001; 2.上海海洋大学 水产与生命学院,上海 201306; 3.中国水产科学研究院 淡水渔业研究中心,农业部淡水渔业与种质资源利用重点实验室,江苏 无锡 214081)
翘嘴鲌生长激素(GH)基因与侧翼区的克隆及分析
刘士力1,2,贾永义1,蒋文枰1,迟美丽1,程 顺1,赵金良2,顾志敏1,*,傅建军3
(1.浙江省淡水水产研究所 农业部淡水渔业健康养殖重点实验室/浙江省淡水水产遗传育种重点实验室,浙江 湖州 313001; 2.上海海洋大学 水产与生命学院,上海 201306; 3.中国水产科学研究院 淡水渔业研究中心,农业部淡水渔业与种质资源利用重点实验室,江苏 无锡 214081)
采用保守引物进行PCR成功扩增了编码翘嘴鲌生长激素的基因,该基因全长5 966 bp,其中转录单元长1 648 bp,由5个外显子和4个内含子组成。5′端和3′端侧翼序列长度分别为2 282 bp和2 036 bp,分别包含 (AAT)8和(TTC)5T(TAA)8的微卫星序列。上游区域包含TATA框,还有一些重要的转录因子结合位点,如Pit-1、Pit-1a、CREB、AP1、GR、HNF-3、HNF-3B等转录因子。翘嘴鲌的5个外显子长度分别为64、140、117、162 和255 bp。推测的阅读框为603 bp,编码由22个氨基酸的信号肽和178个氨基酸成熟肽组成的多肽。在这个多肽中发现了5个保守的半胱氨酸残基(Cys71、Cys135、 Cys173、Cys190和Cys198)和2个可能的N-糖基化位点(145th和197th)。翘嘴鲌GH氨基酸序列与团头鲂完全相同,与草鱼只有一个氨基酸残基的差异,构建的鱼类进化树符合基本分类地位。翘嘴鲌生长激素基因4个内含子长度分别为229、103、565 和103 bp。相对外显子来说,种间内含子变异较大,其中第三内含子变异最大。该结果为进一步研究翘嘴鲌GH基因的表达、功能及其转录调控特征奠定了分子基础。
翘嘴鲌;生长激素基因;克隆;序列分析
生长激素(growth hormone,GH)是由动物垂体前叶分泌的一种蛋白类激素,是影响动物生长性状的主效基因[1],不仅具有提高饲料转化率[2],促进肌肉中的蛋白质合成[3],加速鱼类骨骼纵向生长[4]的重要作用,同时也参与了精子的发生和卵母细胞的成熟[5]。作为生产性能的候选基因,已经有不少学者在猪、牛、鸡等物种上做过研究,并发现了一些与重要经济性状相关的多态位点[6-8]。研究表明,GH基因编码氨基酸序列高度保守,适合鱼类高层分支进化的研究[9-10]。在GH基因启动子区域也存在cAMP应答元件(cAMP response element, CRE)和垂体特异性转录因子1 (pituitary-specific transcription factor-1, Pit-1)的结合位点。CRE含有回文序列TGACGTCA,或者TGACG模体[11-13],可以与活化后的CRE结合蛋白(CRE-binding protein, CREB)结合[14]。CREB主要对cAMP等信号发生应答反应,通过自身磷酸化实现其调节转录功能。对于虹鳟(Oncorhynchusmykiss)[15]启动子的研究表明,其含有TGACG模体。草鱼(Ctenopharyngodonidella)[16]中含有类似CRE的结构(TGACC),与虹鳟有1个碱基的差异,在莫桑比克罗非鱼(Oreochromismossambicus)[17]中还没有明确的CRE,但它们均由cAMP调节。这表明不同鱼类GH转录调控机制是有差别的,具有较高的可变性。
Pit-1是垂体中一种重要的转录调控因子,对于垂体GH、PRL 和TSH 的合成具有重要的调节作用。对于虹鳟[15]、金头鲷(Sparusaurata)[18]、草鱼[16]和莫桑比克罗非鱼[19]GH启动子中Pit-1结合位点的研究表明其在脊椎动物中是保守的,其可能在生长激素转录中具有关键作用。莫桑比克罗非鱼中cAMP对于GH表达的调节是Pit-1依赖的,当离GH基因最近的Pit-1结合位点碱基发生突变时失去作用[17]。微卫星和小卫星存在于数种鱼GH基因的启动子或内含子中,包括尖吻鲈(Latescalcarifer)[20]、牙鲆(Paralichthysolivaceus)[21]和金头鲷(Sparusaurata)[22]等。正是这些序列重复数目和长度的不同导致GH基因出现了复杂的多态性。当串联重复序列位于基因的调控区域时,他们可以直接影响其表达,从而影响引起这些基因数量性状的变化[23]。此外,二核苷酸重复序列可以形成另外的DNA结构,如Z-DNA[24]被证明是与多个基因的转录活性有关。
翘嘴鲌(CulteralburnusBasilewsky)隶属于鲤科(Cyprinida)、鲌亚科(Culterinae)、鲌属(Culter),是鲌亚科中体型最大的一种鱼类,其肉白而细嫩,味美而不腥,具有重要的经济价值。翘嘴鲌又称翘嘴红鲌、白条鱼、大白鱼等,广泛分布于中国各大水系[25]。此外,翘嘴鲌是以活鱼为主食的凶猛肉食性鱼类,对于维持淡水水域生态系统的稳定具有重要作用,具有良好的生态价值。近年来,翘嘴鲌养殖规模的不断扩大,浙江省养殖面积已达到2 000 hm2以上。开展翘嘴鲌的育种具有重要的经济意义,采用分子标记技术为辅助手段可以提高育种效率。已有一些翘嘴鲌微卫星引物已被开发[26-27],但对于明确的重要功能基因中的遗传标记的研究较少。本研究采用T-A克隆了翘嘴鲌GH基因全序列,通过对其核苷酸序列的比较分析,以期为翘嘴鲌人工选育奠定技术基础,同时为鲌亚科鱼类起源及进化机制研究提供理论依据。
1 材料与方法
1.1 材料
试验用翘嘴鲌采自浙江省淡水水产研究所综合实验基地,剪取少量尾鳍,用无水乙醇于-20 ℃保存备用。
主要试剂:PCR 反应试剂和pMD18-T 载体购自宝日医生物技术(北京)有限公司;胶回收试剂盒、大肠埃希菌 (Escherichiacoli) DH5α、氨苄和异丙基硫代半乳糖苷购自天根生化科技(北京)有限公司;用于DNA提取的试剂购自生工生物工程(上海)股份有限公司。
1. 2 方法
1.2.1 DNA 提取
采用苯酚-氯仿法提取样本DNA。用1%琼脂糖凝胶检测DNA完整性,提取DNA原液于-20 ℃保存备用。
1.2.2 引物设计
利用本实验室获得翘嘴鲌转录组中GH的mRNA序列在GenBank中进行Blast比对,结果表明与团头鲂(AF463498)和草鱼(X60419)的相似度较高,根据其保守区域设计7对特异性引物(表1)。引物由生工生物工程(上海)股份有限公司合成。
1.2.3 PCR 反应体系
PCR反应体系为25 μL: 10×Buffer (含Mg2+) 2.5 μL,dNTPs (各2.5 mmol·L-1) 2.0 μL,模板DNA (50 ng·μL-1) 1.0 μL,上游、下游引物( 10 μmol ·L-1) 各0.5 μL,Taq聚合酶(5 U·μL-1) 0.2 μL,灭菌超纯水补足体系。
PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸3 min,共32个循环;72 ℃延伸10 min;4 ℃保存。
1.2.4 克隆及测序
PCR 产物送生工生物工程(上海)股份有限公司测序,如序列中包含微卫星和Poly结构导致测序不完整,克隆后再进行测序。
1.2.5 序列分析
利用软件ContigExpress将所获得的DNA片段和已知的mRNA序列拼接在一起;利用在线软件Alibaba 2.1分析启动子区域顺式调控元件。GH基因的mRNA及编码的氨基酸序列按照刘士力等[28]的方法进行分析。内含子的相似度通过GenBank的Blast功能计算。微卫星和小卫星的查找分别通过SSRhunter 1.3和在线软件Repfind进行。
表1 翘嘴鲌GH基因克隆PCR引物序列
Table 1 Primers used forGHgene cloning ofCulteralburnus

引物Primer序列(5'﹣3')Sequence(5'﹣3')产物长度Length/bp用途UsageP1F:CGGTAGACAGTCATCAGAATR:CCACAACCATCCAATCAATT6641-604P2F:GCTTCCATTCCGATTGTAACR:GTCGCACGGGTATATTTCTA139710-1406P3F:AATGAAGTCCTTAGCAATGCR:ATCACATCCATACCTCTAGC11461214-2359P4F:CTTAGTGCCAACAACATCATR:GCTCTTCTGTGTTTCATCTT12192107-3325P5F:GTTATCAAGGAGGACAACCTR:ATACAGCAGACACATTGGAT15883203-4790P6F:CTCCCATATCTAAACCCTACTTR:GCTGAATACACGACTCCTAA12914423-5713P7F:ACATCCTTCCAGAATCCTTCR:CAACCTACGCTACCATCTAA8685160-5966
2 结果与分析
2.1GH基因与5′侧翼区的克隆、测序及鉴定
经PCR扩增产物的电泳条带清晰,无杂带,片段长度在800~2 000 bp,与对应的目的序列十分接近,据此可初步确定获得正确的目的片段。测序拼接获得全序列5 966 bp。其碱基组成为:A+T 占47. 26% ,C+G 占52. 74%。
通过与GenBank中团头鲂(Megalobramaamblycephala)(登录号:AF463498)和草鱼GH基因的CDS序列(登录号:AY157496)比对分析,相似度在90%以上,由此可以认为所获得的序列为翘嘴鲌GH基因序列。将该序列提交GenBank 数据库,获得登录号KX925976。
分析表明,该基因DNA序列中转录单元长1 648 bp,无微卫星和小卫星序列;5′端和3′端侧翼序列长度分别为2 282 bp和2 036 bp,分别包含 (AAT)8和(TTC)5T(TAA)8的微卫星序列。翘嘴鲌GH基因转录单元包含4个内含子、5个外显子。预测的内含子均以GT开始,以AG 结束,符合真核生物外显子与内含子之间的剪接规律。其中4个内含子大小分别为229、103、565和103 bp,5个外显子长度分别为64、140、117、162和255 bp;mRNA序列全长为738 bp,5′-非翻译区(5′ UTR)为54 bp,3′-非翻译区(3′ UTR)为51 bp,开放阅读框区(ORF)为633 bp,编码由210个氨基酸残基组成的蛋白质多肽。
2.2GH启动子的分析
获得的GH启动子(图1),转录起始位点A在翻译起始密码子ATG上游54 bp。以转录起始位点A为+1位,-28~-23为TATA-box,此外还有2个TATA-box位于-147~-142和-310~-305。但在TATA盒附近没有发现典型的CAAT盒。翘嘴鲌GH基因5端序列与团头鲂、草鱼的相似度分别为94%和92%。Sun等[16]对于草鱼生长激素基因启动子的研究表明,其基本活性是由启动子中-986至-742的序列维持,而黄体生成素受体的激活响应序列位于-616至-572这一区域。通过将这一序列进行比对发现,其核苷酸序列完全相同。
AliBaba 2.1分析GH启动子序列发现,GH启动子含有多个转录调控位点,包括2个Pit-1、4个Pit-1a、2个CREB、5个AP1、8个GR、4个HNF-3、2个HNF-3B、1个SP1和1个MEF-2的结合位点。
2.3GH基因外显子及其编码氨基酸序列的结构比较
将鲤科7种鱼类和鮰科斑点叉尾鮰(Ictaluruspunctatus)的GH基因比较发现,露斯塔野鲮(Labeorohita)和斑点叉尾鮰由于第四外显子长度和表2中所列的鱼类不同,导致了编码氨基酸长度的差异。表2中所列其他鲤科鱼类编码区序列长度均在603 bp左右,编码210个氨基酸。
通过预测,翘嘴鲌GH蛋白质相对分子质量为23.61 ku,理论等电点(isoelectric point,pI)为5.96,分子式C1038H1683N287O317S11。其中,亮氨酸(Lue)含量最高为10.7%。带负电荷氨基酸残基(Asp+Glu) 66个,带正电荷氨基酸残基(Arg+Lys) 57个。脂肪族氨基酸指数为88.45。经过蛋白质序列分析,翘嘴鲌GH基因氨基酸预测无无跨膜结构。翘嘴鲌生长激素成熟肽序列中包含由22个氨基酸组成的信号肽。在Asn155和Asn207位置上存在2个糖基化位点(天冬酰胺-天冬氨酸-丝氨酸和天冬酰胺-半胱氨酸-苏氨酸)。
通过NCBI上的BLAST蛋白质相似性分析,翘嘴鲌GH基因cDNA 序列与团头鲂编码的氨基酸序列完全相同,而与草鱼编码的氨基酸序列只有一个氨基酸残基差异,同源性分别为100%和99.5%。鲤科鱼类GH氨基酸序列高度同源;翘嘴鲌和其他鲤科鱼类一样,均具有5个的半胱氨酸残基(Cys71、Cys145、Cys183、Cys200、Cys208),其中的4个半胱氨酸残基非常保守,参与了二硫键(Cys71和Cys183,Cys200和Cys208)。它们对GH的正常折叠、维持空间结构以及发挥生理功能有重要的作用。
2.4 翘嘴鲌GH氨基酸序列和其他物种的比较分析
利用MEGA5. 0 等软件,对本研究获得的翘嘴鲌GH 氨基酸序列及GenBank数据库中获得的团头鲂、草鱼等15种鱼类GH氨基酸序列,构建NJ系统进化树(图3)。在NJ系统进化树中,翘嘴鲌等鲤科鱼类聚集在一起,然后与鮰科的斑点叉尾鮰聚成一支,露斯塔野鲮是鲤科中与翘嘴鲌关系最远的鱼类。除鳗鲡 (Anguillajaponica)单独为一支外,其他用作参考的鱼类聚成一支。该基因的系统进化关系与传统的物种进化地位基本一致。

推测的转录因子结合位点用单下划线和阴影表示;转录起始位点用粗体加方框表示;翻译起始密码子ATG用粗体加双下划线表示Putative transcription factor binding sites are single-underlined and shaded; the transcription initiation site is in bold and boxed; the translation start codon ATG is in bold and double-underlined图1 翘嘴鲌GH基因5′侧翼区序列及部分潜在转录因子结合位点预测Fig.1 Sequence of the 5′-flanking region of the Culter alburnus GH gene and partial prediction of transcription factor binding sites
2.5GH基因内含子的分析
相对于外显子,GH基因内含子存在相对较大的差异。虽然鲷科斑点叉尾鮰也包含4个内含子,5个外显子。但其4个内含子均与翘嘴鲌没有显著的相似性,而且其第二和第四内含子长度与翘嘴鲌相差悬殊。在目前已公布的序列中,团头鲂与翘嘴鲌最为相似,第一、二内含子的相似度分别为88%和95%;第三、四内含子完全一致。第一内含子中主要差异在于存在2个缺失,长度分别为15 bp和9 bp。团头鲂、草鱼、鲢(Hypophthalmichthysmolitrix)这三种鱼和翘嘴鲌的内含子长度较为接近,这三种鱼的4个内含子与翘嘴鲌的平均相似度分别为88%、87%、86%和92%。露斯塔野鲮,是鲤科中与翘嘴鲌差异较大的鱼类,其第三内含子的长度是翘嘴鲌的2.8倍左右,但Blast覆盖度仅为12%。此外,在鲤科的这几种鱼类的内含子中没有发现微卫星和小卫星。
表2 翘嘴鲌与其他鱼类GH基因外显子和内含子大小的对比
Table 2 Size comparison of the exon and intron ofGHgene amongCulteralburnusand other fish species
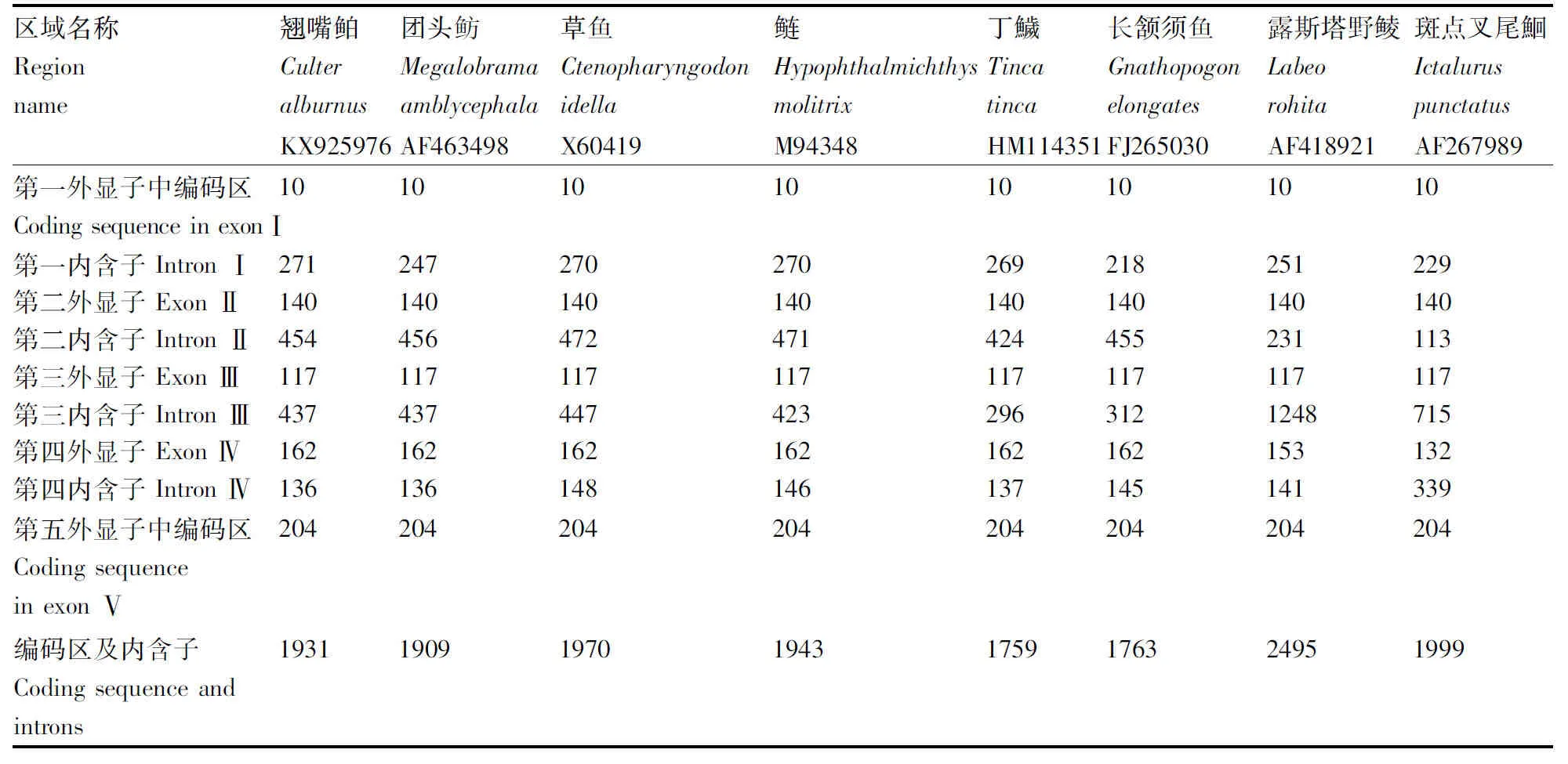
区域名称Regionname翘嘴鲌Culteralburnus团头鲂Megalobramaamblycephala草鱼Ctenopharyng-odonidella鲢Hypophthalmich-thysmolitrix丁鱥Tincatinca长颔须鱼Gnathopogonelongates露斯塔野鲮Labeorohita斑点叉尾鮰IctaluruspunctatusKX925976AF463498X60419M94348HM114351FJ265030AF418921AF267989第一外显子中编码区CodingsequenceinexonⅠ1010101010101010第一内含子IntronⅠ271247270270269218251229第二外显子ExonⅡ140140140140140140140140第二内含子IntronⅡ454456472471424455231113第三外显子ExonⅢ117117117117117117117117第三内含子IntronⅢ4374374474232963121248715第四外显子ExonⅣ162162162162162162153132第四内含子IntronⅣ136136148146137145141339第五外显子中编码区CodingsequenceinexonⅤ204204204204204204204204编码区及内含子Codingsequenceandintrons19311909197019431759176324951999

“*”表示完全保守的氨基酸位点;“:”表示不完全保守位点;“.”表示半保守位点;下划线表示糖基化位点;半胱氨酸用黑框表示.Fully conserved residues are marked with an asterisk; incompletely conservative and semiconservative positions are marked with colons and periods respectively; N-glycosylation sites are underlined; Cys residues are boxed图2 翘嘴鲌GH与其他鱼类GH氨基酸序列比较Fig.2 Alignment of the amino acid sequences of C. alburnus GH and GHs from other fish
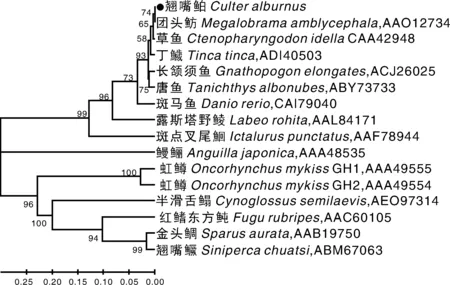
图中枝上的数据代表置信度,黑圆点是本研究得到的翘嘴鲌的GH氨基酸序列Numbers on nodes indicate bootstrap values. The amino acid sequence of C. alburnus GH is marked with a black dot图3 基于GH基因氨基酸序列构建的翘嘴鲌及其他鱼类系统发育树Fig.3 Phylogenetic tree of C. alburnus and other fish based on GH amino acid sequences
3 讨论
生长激素基因是物种进化中相对保守的基因,目前对鱼类GH基因序列研究发现,一部分鱼类由5个外显子和4个内含子组成[29-31],一部分鱼类包含6个外显子和5个内含子[22,32-33],还有少部分鱼类由4个外显子和3个内含子组成[34-35]。现有的GH基因数据表明,鲤科中除了鳙鱼(Aristichthysnobilis)和唐鱼(Tanichthysalbonubes)外,其余鱼类均属于第一种。鳙鱼和唐鱼中第一外显子编码区长150 bp,对应其他鲤科鱼类第一外显子编码区和第二外显子的长度之和。在对包含翘嘴鲌在内的由5个外显子和4个内含子组成的7 种鲤科鱼类的GH基因序列结构比较发现,外显子长度基本相同,内含子大小存在一定程度的差异,露斯塔野鲮是鲤科中与翘嘴鲌差异较大的鱼类,其第三内含子的长度是翘嘴鲌的2.8倍左右。鲤科这7种鱼类中除了亲缘关系较远的露斯塔野鲮[36]第四外显子略有差异外,其余外显子的长度完全一致。对于8 科15种鱼类GH基因编码区序列进行对比分析,鲤科鱼类的GH基因编码区序列同源性在89%~100%,与其他不同科的鱼类序列同源性在65%~82%,这表明同一科鱼类之间,生长激素基因编码区序列具有相对较高的同源性,不同科鱼类的生长激素基因序列的同源性明显下降。
GH基因的多态性主要集中在内含子中,如Almuly等[37]发现金头鲷GH基因非编码区内存在小卫星和微卫星[18]的重复序列,对于内含子1中小卫星saGHFIM的研究表明具有长内含子的片段会抑制GH基因表达的活性。鲁双庆等[32]在鳜属(Siniperca)三种鱼GH第二内含子中的相同位置均发现了“AG”微卫星序列。翘嘴鲌GH基因5′端序列也比较保守,与团头鲂和草鱼的相似度分别为94%和92%。还有两个关键区域序列与草鱼完全一致[16]。虽然在翘嘴鲌内含子中未发现微卫星和小卫星。但在5端启动子中发现了(AAT)8微卫星序列。在同属鲤科的团头鲂中也发现了(AAT)11微卫星序列。在鲷科中也发现了类似的现象,金头鲷[18]中与第二个Pit-1的结合位点相邻处存在(CA)n微卫星序列,而且等位位点高达11个,极有可能会调节启动子的活性,影响GH基因的表达从而与生长性状关联。在同属鲷科的真鲷[38](Pagrusmajor)类似位置也发现了(CA)n微卫星序列,但在对一个野生群体进行检测时未发现多态性。
翘嘴鲌与各物种在GH基因编码区核苷酸序列上的差异表明,尽管翘嘴鲌与其他鱼类在GH基因核苷酸水平上具有一定的保守性,但由于不同物种间起源、进化过程中的变化,导致它们在DNA 水平上产生差异,这为从分子水平上研究翘嘴鲌与其他鱼类物种间的起源及进化关系等提供了理论基础。虽然唐鱼只有4个外显子,但在GH氨基酸序列构建的NJ 系统进化树中,其仍然与鲤科鱼类聚集在一起。除了用于系统进化分析外,完善的翘嘴鲌GH基因序列为启动子和内含子功能的进一步研究打下了基础。
[1] MØLLER N, COPELAND K C, NAIR K S. Growth hormone effects on protein metabolism [J].EndocrinologyandMetabolismClinicsofNorthAmerica, 2007, 36(1): 89-100.
[2] CAVARI B, FUNKENSTEIN B, CHEN T T, et al. Effect of growth hormone on the growth rate of the gilthead seabream (Sparusaurata) and the use of different constructs for production of transgenic fish [J].Aquaculture, 1993, 111(1/4): 189-197.
[3] SAKAMOTO T, SHEPHERD B S, MADSEN S S, et al. Osmoregulatory actions of growth hormone and prolactin in an advanced teleost [J].GeneralandComparativeEndocrinology, 1997, 106(1): 95-101.
[4] FORSYTH I A, WALLIS M. Growth hormone and prolactin-molecular and functional evolution [J].JournalofMammaryGlandBiologyandNeoplasia, 2002, 7(3): 291-312.
[5] GAC F L E, BLAISE O, FOSTIER A, et al. Growth hormone (GH) and reproduction: a review [J].FishPhysiologyandBiochemistry, 1993, 11(1): 219-232.
[6] CHUNG E R, LEE C S. A previously unreported DraI polymorphism within the regulatory region of the bovine growth hormone gene and its association with growth traits in Korean Hanwoo cattle [J].AnimalGenetics, 2004, 35(2): 152-158.
[7] 王文君, 陈克飞, 任军, 等. 中外不同猪品种生长激素基因遗传多态性检测[J]. 农业生物技术学报, 2003, 11(1): 103-104. WANG W J, CHEN K F, REN J, et al. Detection of the Polymorphisms of growth hormone gene in Chinese and European pig breeds [J].JournalofAgriculturalBiotechnology, 2003, 11(1): 103-104. (in Chinese with English abstract)
[8] 聂庆华, 张细权, 杨关福, 等. 鸡生长激素基因的 PCR-RFLPs 分析 [J].农业生物技术学报, 2001, 9(3): 282-285. NIE Q H, ZHANG X Q, YANG G F, et al. PCR-RFLPs analysis of chicken growth hormone gene [J].JournalofAgriculturalBiotechnology, 2001, 9(3): 282-285. (in Chinese with English abstract)
[9] BERNARDI G, D’ONOFRIO G, CACCIO S, et al. Molecular phylogeny of bony fishes, based on the amino acid sequence of the growth hormone [J].JournalofMolecularEvolution, 1993, 37(6): 644-649.
[10] RUBIN D A, DORES R M. Obtaining a more resolute teleost growth hormone phylogeny by the introduction of gaps in sequence alignment [J].MolecularPhylogeneticsandEvolution, 1995, 4(2): 129-138.
[11] MONTMINY M R, GONZALEZ G A, YAMAMOTO K K. Regulation of cAMP-inducible genes by CREB [J].TrendsinNeurosciences, 1990, 13(5): 184-188.
[12] NICHOLS M, WEIH F, SCHMID W, et al. Phosphorylation of CREB affects its binding to high and low affinity sites: implications for cAMP induced gene transcription [J].TheEMBOJournal, 1992, 11(9): 3337-3346.
[13] MEYER T E, HABENER J F. Cyclic adenosine 3′, 5′-monophosphate response element binding protein (CREB) and related transcription-activating deoxyribonucleic acid-binding proteins [J].EndocrineReviews, 1993, 14(3): 269-290.
[14] MONTMINY M R, GONZALEZ G A, YAMAMOTO K K. Characteristics of the cAMP response unit [J].Metabolism, 1990, 39(9):6-12.
[15] ARGENTON F, BERNARDINI S, PUTTINI S, et al. A TGACG motif mediates growth-hormone-factor-1/pituitary-transcriptional-activator-1-dependent cAMP regulation of the rainbow trout growth-hormone promoter [J].EuropeanJournalofBiochemistry, 1996, 238(3): 591-598.
[16] SUN C, HE M, KO W K W, et al. Mechanisms for luteinizing hormone induction of growth hormone gene transcription in fish model: Crosstalk of the cAMP/PKA pathway with MAPK-and PI3K-dependent cascades [J].MolecularandCellularEndocrinology, 2014, 382(2): 835-850.
[17] SEKKALI B, BELAYEW A, BORTOLUSSI M, et al. Pit-1 mediates cell-specific and cAMP-induced transcription of the tilapia GH gene [J].MolecularandCellularEndocrinology, 1999, 152(1/2): 111-123.
[18] ALMULY R, POLEG-FANIN Y, GORSHKOV S, et al. Characterization of the 5′ flanking region of the growth hormone gene of the marine teleost, gilthead sea breamSparusaurata: analysis of a polymorphic microsatellite in the proximal promoter [J].FisheriesScience, 2005, 71(3): 479-490.
[19] SEKKALI B, BRIM H, MULLER M, et al. Structure and functional analysis of a tilapia (Oreochromismossambicus) growth hormone gene: activation and repression by pituitary transcription factor Pit-1 [J].DNAandCellBiology, 1999, 18(6): 489-502.
[20] YOWE D L, EPPING R J. A minisatellite polymorphism in intron III of the barramundi (Latescalcarifer) growth hormone gene [J].Genome, 1996, 39(5): 934-940.
[21] KANG J H, LEE S J, PARK S R, et al. DNA polymorphism in the growth hormone gene and its association with weight in olive flounderParalichtysolivaceus[J].FisheriesScience, 2002, 68(3): 494-498.
[22] ALMULY R, CAVARI B, FERSTMAN H, et al. Genomic structure and sequence of the gilthead seabream (Sparusaurata) growth hormone-encoding gene: identification of minisatellite polymorphism in intron I [J].Genome, 2000, 43(5): 836-845.
[23] KASHI Y, KING D, SOLLER M. Simple sequence repeats as a source of quantitative genetic variation [J].TrendsinGenetics, 1997, 13(2): 74-78.
[24] WÖLFL S, MARTINEZ C, RICH A, et al. Transcription of the human corticotropin-releasing hormone gene in NPLC cells is correlated with Z-DNA formation [J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 1996, 93(8): 3664-3668.
[25] 乐佩琦, 罗云林. 鲌亚科鱼类系统发育初探(鲤形目: 鲤科) [J].水生生物学报, 1996, 20(2): 182-185. YUE P Q, LUO Y L. Preliminary studies on phylogeny of subfamily Cultrinae (Cypriniformes: Cyprinidae) [J].ActaHydrobiologicaSinica, 1996, 20(2): 182-185. (in Chinese with English abstract)
[26] LIU S L, GU Z M, JIA Y Y, et al. Isolation and characterization of 32 microsatellite loci for topmouth culter (CulteralburnusBasilewsky) [J].GeneticsandMolecularResearch, 2014, 13(3): 7480-7483.
[27] LIU S L, GU Z M, ZHAO J L, et al. Characterization of microsatellites and repetitive flanking sequences (ReFS) from the topmouth culter (CulteralburnusBasilewsky) [J].BiochemicalSystematicsandEcology, 2015, 63(5): 127-135.
[28] 刘士力, 张爱菊, 练青平, 等. 泥鳅和大鳞副泥鳅细胞色素P450c17-Ⅰ(CYP17-Ⅰ)基因的克隆及组织表达分析[J]. 浙江农业学报, 2016,28(11): 1853-1861. LIU S L, ZHANG A J, LIAN Q P, et al. Cloning and tissue expression analysis of the cytochromeP450c17-Ⅰ(CYP17-Ⅰ) gene from mud loach (Misgurnusanguillicaudatus) and large-scale loach (Paramisgurnusdabryanus) [J].ActaAgriculturaeZhejiangensis, 2016,28(11): 1853-1861. (in Chinese with English abstract)
[29] HO W K K, WONG M W, CHAN A P Y. Cloning and sequencing of the grass carp (Ctenopharyngodonidellus) growth hormone gene [J].BiochimicaetBiophysicaActa, 1991, 1090(2): 245-248.
[30] HONG Y, SCHARTL M. Sequence of the growth hormone (GH) gene from the silver carp (Hypophthalmichthysmolitrix) and evolution of GH genes in vertebrates [J].BiochimicaetBiophysicaActa, 1993, 1174(3): 285-288.
[31] TANG Y, LIN C M, CHEN T T, et al. Structure of the channel catfish (Ictaluruspunctatus) growth hormone gene and its evolutionary implications [J].MolecularMarineBiologyandBiotechnology, 1993, 2(4): 198-206.
[32] 鲁双庆, 刘峰, 刘臻, 等.三种鳜鱼(Siniperca)生长激素基因的克隆及序列比较[J]. 海洋与湖沼, 2008, 39(4): 354-361. LU S Q, LIU F, LIU Z, et al. Comparison in cloning and sequence of growth hormone gene in three species of genusSiniperca[J].OceanologiaetLimnologiaSinica, 2008, 39(4): 354-361. (in Chinese with English abstract)
[33] MA Q, LIU S F, ZHUANG Z M, et al. Genomic structure, polymorphism and expression analysis of the growth hormone (GH) gene in female and male half-smooth tongue sole (Cynoglossussemilaevis)[J].Gene, 2012, 493(1): 92-104.
[34] 郁二蒙, 叶星, 白俊杰, 等. 唐鱼生长激素基因的克隆及序列分析 [J]. 安徽农业科学, 2008, 36(23): 9914-9917. YU E M, YE X, BAI J J, et al. Cloning and sequence analysis of growth hormone gene fromTanichthysalbonubes[J].JournalofAnhuiAgriculturalSciences, 2008, 36(23): 9914-9917. (in Chinese with English abstract)
[35] 贡成良, 曹广力, 薛仁宇, 等. 编码鳗鲡生长激素基因的序列与结构 [J]. 水产学报, 2002, 26(4): 295-300. GONG C L, CAO G L, XUE R Y, et al. Sequence and structure of encoding eel growth hormone gene [J].JournalofFisheriesofChina, 2002, 26(4):295-300. (in Chinese with English abstract)
[36] RAJESH R, MAJUMDAR K C. A comparative account of the structure of the growth hormone encoding gene and genetic interrelationship in six species of the genusLabeo[J].FishPhysiologyandBiochemistry, 2007, 33 (4): 311-333.
[37] ALMULY R, SKOPAL T, FUNKENSTEIN B. Regulatory regions in the promoter and first intron ofSparusauratagrowth hormone gene: Repression of gene activity by a polymorphic minisatellite [J].ComparativeBiochemistryandPhysiology-PartD:GenomicsandProteomics, 2008, 3(1): 43-50.
[38] SAWAYAMA E, TAKAGI M. Isolation and characterization of tandem repeat sequences in the growth hormone gene of the red seabream,Pagrusmajor(Temminck & Schlegel, 1843) [J].JournalofAppliedIchthyology, 2015, 31(4): 762-766.
(责任编辑 张 韵)
Cloning and sequence analysis of growth hormone gene and its flanking region in topmouth culter (CulteralburnusBasilewsky)
LIU Shili1,2, JIA Yongyi1, JIANG Wenping1, CHI Meili1, CHENG Shun1, ZHAO Jinliang2, GU Zhimin1,*, FU Jianjun3
(1.AgricultureMinistryKeyLaboratoryofHealthyFreshwaterAquaculture/KeyLaboratoryofFreshwaterAquaticAnimalGeneticandBreedingofZhejiangProvince,ZhejiangInstituteofFreshwaterFisheries,Huzhou313001,China; 2.CollegeofFisheriesandLifeScience,ShanghaiOceanUniversity,Shanghai201306,China; 3.KeyLaboratoryofFreshwaterFisheriesandGermplasmResourcesUtilization,MinistryofAgriculture,FreshwaterFisheriesResearchCenter,ChineseAcademyofFisherySciences,Wuxi214081,China)
Using conserved primers and PCR, a gene encoding topmouth culter (CulteralburnusBasilewsky) growth hormone (CaGH) was amplified and sequenced. The gene spans 5 966 bp, including 2 282 bp of 5′- and 2 036 bp of 3′-flanking sequences and a 1.7-kb transcription unit comprised of five exons and five introns. (AAT)8and (TTC)5T(TAA)8microsatellite sequences were found in the 5′- and 3′-flanking regions, respectively. The upstream region contains TATA boxes, and binding sites of important transcription factors such as Pit-1, Pit-1a, CREB, AP1, GR, HNF-3, and HNF-3B. The five exons inCulteralburnuswere 64 bp, 140 bp, 117 bp, 162 bp and 255 bp in length, respectively. The complete coding sequence was 603 bp and encodes a protein with a 22 amino acid signal peptide and a 178 amino acid mature peptide. Five conserved Cys residues (Cys71, Cys135, Cys173, Cys190, and Cys198) and two possible sites of N-glycosylation (residues 145 and 197) were detected in the GH polypeptide. The amino acid sequence of GH inCulteralburnuswas identical to that inMegalobramaamblycephala, and only one amino acid residue differs fromCtenpharyngodonidellus. The phylogenetic relationships among the GH amino acid sequences in fish were in accord with traditional classification. The lengths of the four introns inCulteralburnusGHgene were 229 bp, 103 bp, 565 bp and 103 bp, respectively. The variation of the introns among species was greater than that of the exons, and the variation of the third intron is the highest. These results provided the molecular basis for study of function and transcriptional regulation of theGHgene inC.alburnus, as well as the temporal expression in different developmental stages and at various nutritional levels.
Culteralburnus; growth hormone gene; clone; sequence analysis
10.3969/j.issn.1004-1524.2017.08.08
2017-03-17
浙江省农业新品种选育重大科技专项(2012C12907-7, 2016C02055-1)
刘士力(1985—),男,湖北洪湖人,博士研究生,主要从事水生动物遗传育种研究。E-mail: liushili1212@126.com
*通信作者,顾志敏,E-mail: guzhimin2006@163.com
S96;Q349+.55
A
1004-1524(2017)08-1281-09
刘士力,贾永义,蒋文枰,等. 翘嘴鲌生长激素(GH)基因与侧翼区的克隆及分析[J].浙江农业学报,2017,29(8): 1281-1289.
