分子标记在我国番茄抗病育种的应用研究进展
2017-09-03陈远松朱晓伟龚翔宇梅至诚
陈远松,朱晓伟,龚翔宇,梅至诚,祝 彪
(浙江农林大学 农业与食品科学学院,生物种业研究中心,浙江 临安 311300)
分子标记在我国番茄抗病育种的应用研究进展
陈远松,朱晓伟,龚翔宇,梅至诚,祝 彪*
(浙江农林大学 农业与食品科学学院,生物种业研究中心,浙江 临安 311300)
抗病育种是当前番茄育种的一个重要方向,分子标记技术已成为番茄抗病育种中的重要方法之一。分子标记技术很大程度上克服了表型选择的局限性,能极大地缩短育种周期,已广泛应用于番茄的辅助育种。文章基于献计量分析的方法,综述了分子标记技术在中国番茄抗病育种应用上的研究现状,介绍了常用几种分子标记的特点,概述了番茄主要病害抗病分子标记使用现状及进展,并对存在的问题进行了讨论和分析,指出今后的发展方向。
番茄病害;抗病育种;分子标记
番茄品种丰富、种植广泛、营养价值高,富含具有抗癌效果的番茄红素等抗氧化物质[1],但其病害种类繁多、危害严重。目前,生产上对番茄病害的防治主要依赖于喷施化学药剂,这不但会带来食品安全问题,而且增加成本,污染环境。此外,随着病菌和害虫对药物的抗性逐渐增强,杀菌剂、杀虫剂的功效也大大减弱。因此,在番茄育种中培育优良的抗病品种显得尤为重要。
此前,绝大多数番茄品种是通过表型筛选和传统育种得到的。随着分子标记和分子标记辅助选择技术的出现,番茄遗传育种研究已进入一个新的时代。Rick等[2]最早在番茄育种领域应用 DNA 分子标记技术进行初探,并获得很大的突破。目前,分子标记技术已广泛用于遗传图谱的构建以及番茄许多重要农艺性状的基因和QTL的鉴定,包括抗病虫、抗非生物胁迫,花和果实发育相关特性等[3-6]。该技术还被用于几个重要经济性状的辅助育种,尤其是抗病性上[5,7]。对于番茄的许多简单的抗病性状,分子标记辅助育种不但快于传统育种,而且更便宜、更有效。分子标记技术可以从DNA水平上准确、快速地鉴定抗病位点,显著提高育种效率[3]。目前,番茄中超过35种病原体的抗性基因已被识别和定位[8-9]。
本文采用文献计量分析的方法,从文献时间分布、文献来源地区分布、文献来源机构、各类分子标记使用次数分析和各种番茄病害文献数量分析等几个方面对国内番茄抗病分子标记的应用现状进行研究分析,以期为番茄的抗病育种提供参考。
1 数据来源与研究方法
1.1 文献获取
期刊和学位论文获取:以《中国知识资源总库(CNKI)》为数据源。采用高级检索类型,以 “主题”为检索项,以“番茄”并含“标记”为检索词,采用“精确”匹配为检索式,文献检索时间范围为2000年1月1日至2015年12月31日,检索库范围为期刊、特色期刊和学位论文。
专利文献获取:以《国家知识产权局互联网检索数据库》为数据源,利用Soopat专利搜索平台,在中国专利栏输入“番茄标记”,检索范围设为“发明、实用新型、发明授权”。
1.2 统计分析方法
首先建立Excel数据库,然后根据分析需求,分别建立不同的工作表和不同的字段,再利用数据库的强大支撑功能,运用文献计量法进行研究。同时,还运用了内容分析法和比较分析法,见表1。
2 结果与分析
2.1 分子标记在我国番茄抗病育种中的运用现状
根据上述方法检索得到期刊文献360篇、学位论文445篇、专利153篇,其中226篇涉及番茄性状相关分子标记。上述文献中,抗病相关的占71%;抗逆性的占7%;果实品质、产量的占11%;品种、纯度鉴定的占7%;雄性不育、单性结实的占4%(图1)。显然,分子标记在抗病性状的研究上应用最多。排除了与分子标记无关和分子标记在其他方面应用等文献以及数据不全的期刊和学位论文,排除了不在2000—2015年的专利、与分子标记无关和分子标记在其他方面应用等文献以及数据不全的专利,最终得到番茄抗病分子标记相关期刊文献85篇,学位论文46篇,专利29篇,总计160篇。
2.2 文献的年份分布
由图2可知,2000—2015年文献的发表数量总体呈上升趋势,这表明科研工作者对分子标记在番茄抗病育种中的研究热度增加且不断深入。期刊和专利发表量基本呈现稳定上涨的趋势,而学位论文在不同年份间波动较大。从总体数量上来看,期刊最多,学位论文第二,专利最少。
表1 研究方法汇总
Table 1 Study methods

研究方法Studymethods研究目的Studyaims文献计量法Bibliometricanalysis文献年份分布Yearlydistributionofliterature分析国内番茄抗病分子标记研究热度变化Analysisontheyearlychange文献地域分布Regionaldistributionofliterature分析比较不同地区研究状况Analysisonthesituationsofdifferentregions文献来源机构分析Researchinstitutedistributionofliterature分析比较不同机构的研究状况Analysisonthesituationsofdifferentresearchinsti-tutes内容分析法Contentanalysis分析文献的研究内容Contentanalysis比较分析法Comparativeanalysis不同类别文献量的比较分析Comparativeanalysis
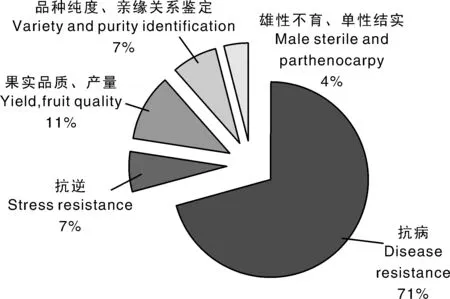
图1 涉及番茄性状相关分子标记文献分布Fig.1 The distribution of molecular markers used in tomato breeding
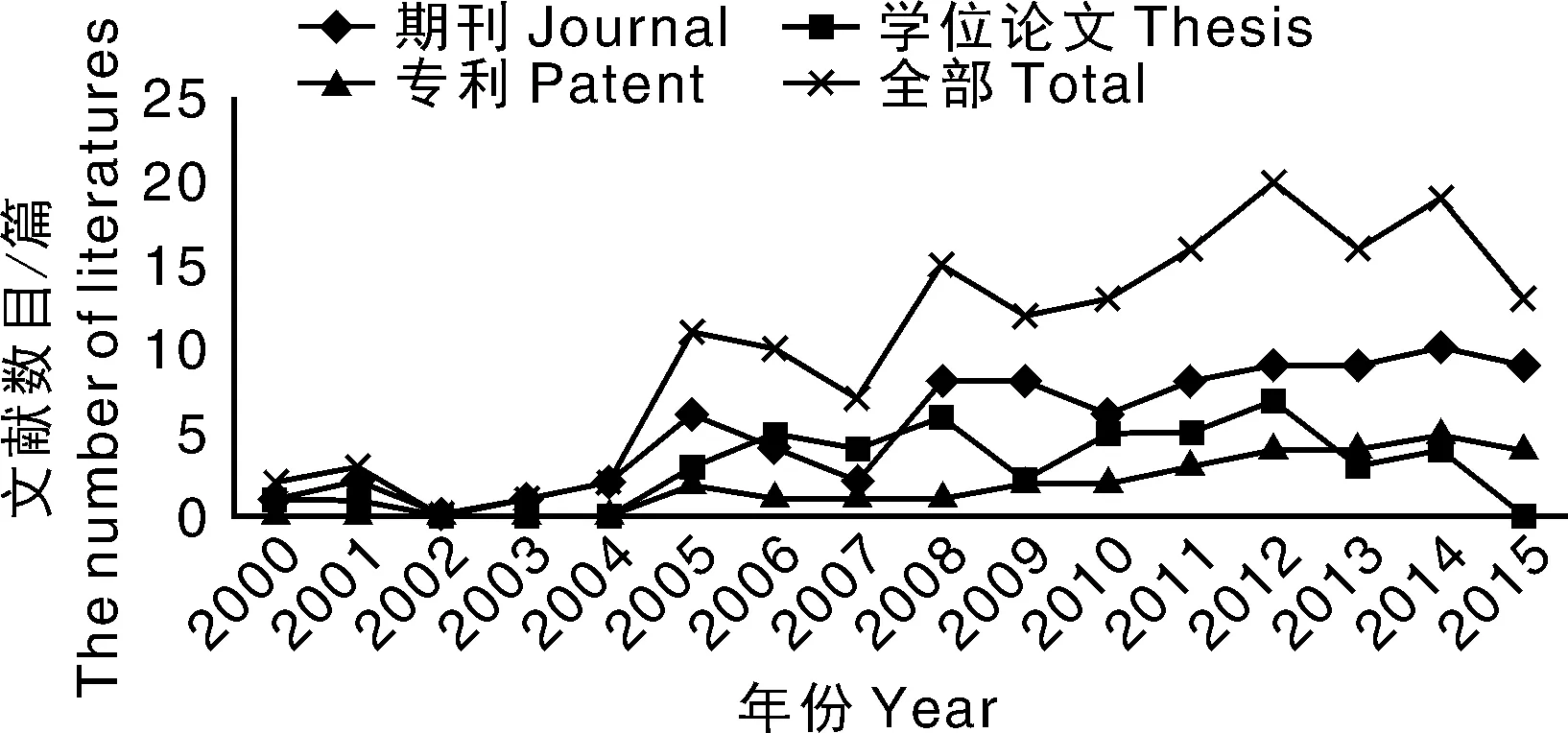
图2 2000—2015年国内番茄抗病分子标记相关文献数量折线图Fig.2 The literature number of molecular markers related with tomato disease resistance during 2000 to 2015
2.3 文献来源分析
2.3.1 文献数目地域分析
文献的地域分布整体上表现为东部多于西部,北方多于南方,主要集中于东北农业大学,以中国农业科学院为代表的北京周边科研机构与大学,浙江、江苏地区的大学与农业科学院。其中东北农业大学十分热衷于番茄抗病分子标记的研究与运用,131篇学位论文和期刊论文中有35篇来自东北农业大学。
2.3.2 几种主要病害研究的地域分布
根据图3可以发现,番茄抗病分子标记研究与番茄产地以及主要病害发生的地理分布密切相关,如北方地区对番茄花叶病毒病(ToMV)和根结线虫病(root knot nematodes)的研究较多,而南方地区对此研究较少;对番茄花叶病毒病的研究以中国农业科学院为主,但其对番茄黄化曲叶病毒病(TYLCV)的研究较少;对青枯病(bacterial wilt)分子标记相关研究较少,主要集中在浙江大学和浙江省农业科学院等南方地区科研机构;东北农业大学对番茄主要病害相关分子标记都有所研究。
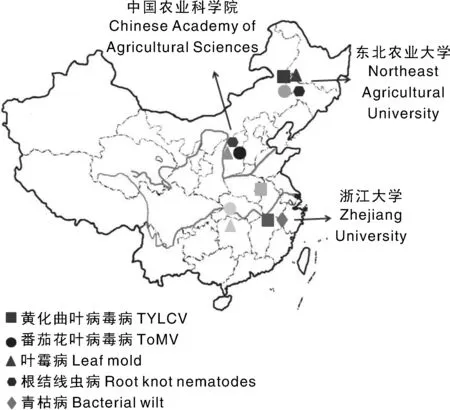
图3 几种主要病害相关文献来源的地域分布Fig.3 The regional distribution of the study of molecular markers used in the tomato disease resistance breeding
2.4 番茄抗病育种中应用的主要分子标记分析
从图4可以看出,特定序列扩增(sequence characterized amplified regions,SCAR)标记的使用次数最多。扩增片段长度多态性(amplified fragment length polymorphism,AFLP)标记、简单重复序列标记(simple sequence repeat,SSR)、酶切扩增多态性序列标记(cleaved amplified polymorphism sequences,CAPS)使用次数也较多,仅次于SCAR标记。进一步研究表明,番茄不同病虫害所使用的分子标记不同。
图5表明,番茄花叶病毒病主要使用SCAR标记,很少使用其他标记;青枯病主要使用AFLP标记;根结线虫病主要使用SCAR标记和CAPS标记;叶霉病(leaf mold)很少使用SCAR标记;SSR标记的病害比例与AFLP标记很相似,实际上SSR标记和AFLP标记经常联合使用。
从表2可以看出,分子标记在番茄抗病育种中的使用情况与他们自身的特点密切相关。限制性片段长度多态性(restriction fragment length polymorphism,RFLP)技术最主要的缺点是由于它的产生是由碱基突变致使限制性酶切位点的获得或失去,故多态位点少,信息量小。随机扩增多态性DNA(random amplified polymorphism DNA,RAPD)技术受影响的因素多,实验重复性和稳定性差。CAPS分子标记技术依赖于已发现的突变酶切点。AFLP技术需要使用同位素检测。SSR标记必须依赖测序设计引物。单核苷酸多态性(single nucleotide polymorphism,SNP)标记建立在大量测序或基因芯片的基础上。相较其他标记,SCAR标记具有多态性高、稳定性和重复性好、使用简单、费用较低等明显优势,所以它在番茄抗病研究上的使用最多。然而实际研究中可能影响分子标记使用的因素有很多,如分子标记开发难度、已开发的标记数目、标记的使用效果和标记的使用成本等都会影响标记使用。
2.5 番茄主要病害的分子标记使用情况分析
由图6得知,分子标记在黄化曲叶病毒病上的运用远多于其他病害。叶霉病和根结线虫病也是运用分子标记较多的病害[10-13]。黄化曲叶病毒病使用分子标记最多的原因可能与其病害危害严重、防治难度大,且传统育种进展缓慢、困难大,黄化曲叶病毒病抗性是一种质量性状、使用分子标记育种操作难度低且抗病效果明显有关[3]。目前,已经定位到的黄化曲叶病毒病抗病基因有Ty-1、Ty-2、Ty-3、Ty-4和Ty-5,开发的标记种类和数目较多且经过验证[14-19]。
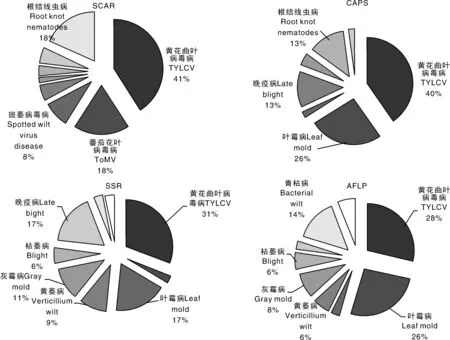
图5 常用几种标记相关病害占比Fig.5 The proportion of different kinds of molecular markers used in tomato disease resistance breeding
表2 几种主要分子标记特点
Table 2 The characteristics of different kinds of molecular markers
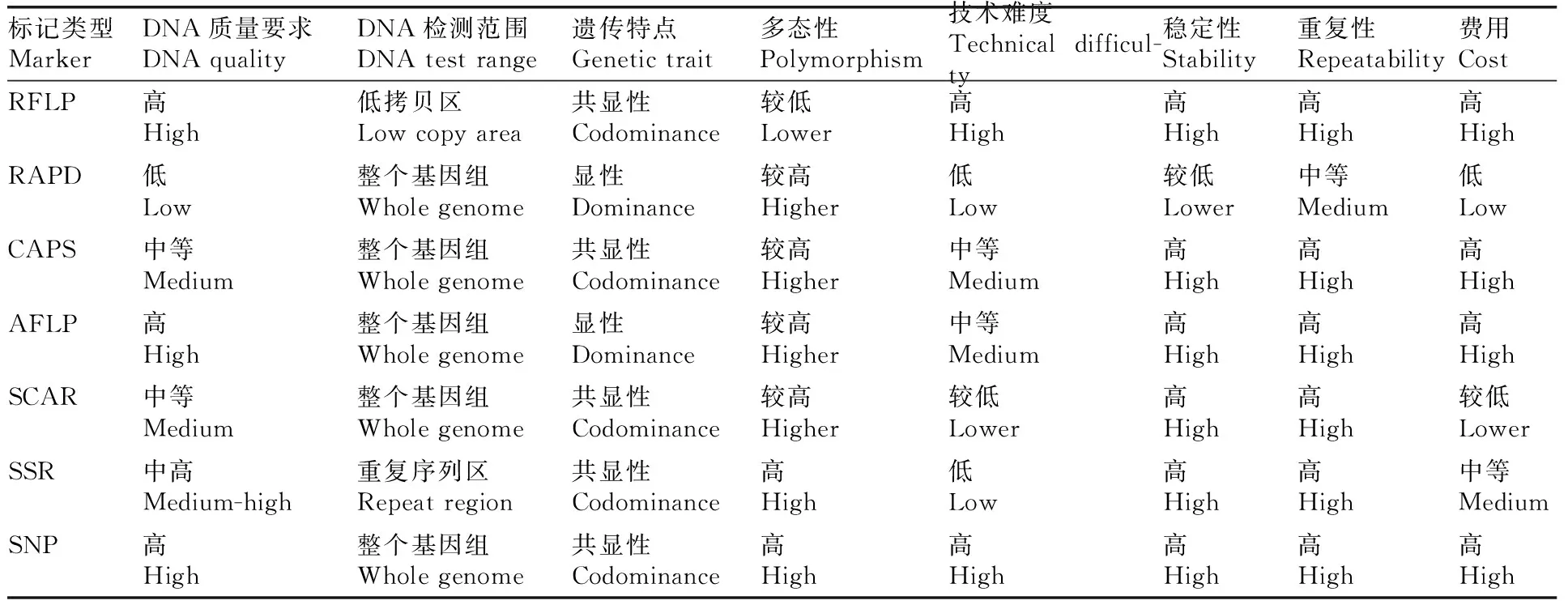
标记类型MarkerDNA质量要求DNAqualityDNA检测范围DNAtestrange遗传特点Genetictrait多态性Polymorphism技术难度Technicaldifficul-ty稳定性Stability重复性Repeatability费用CostRFLP高High低拷贝区Lowcopyarea共显性Codominance较低Lower高High高High高High高HighRAPD低Low整个基因组Wholegenome显性Dominance较高Higher低Low较低Lower中等Medium低LowCAPS中等Medium整个基因组Wholegenome共显性Codominance较高Higher中等Medium高High高High高HighAFLP高High整个基因组Wholegenome显性Dominance较高Higher中等Medium高High高High高HighSCAR中等Medium整个基因组Wholegenome共显性Codominance较高Higher较低Lower高High高High较低LowerSSR中高Medium-high重复序列区Repeatregion共显性Codominance高High低Low高High高High中等MediumSNP高High整个基因组Wholegenome共显性Codominance高High高High高High高High高High
由图7可知,不同病害使用的标记具有较大差异,黄化曲叶病毒病使用最多的4种标记也是总体使用最多的分子标记,使用量最大的SCAR标记在叶霉病上却使用较少,根结线虫病很少使用AFLP和SSR标记。
3 讨论
分子标记技术已成为番茄育种中重要的工具之一。本文通过文献计量分析的方法对我国番茄抗病分子标记的研究进展进行了分析,结果表明,分子标记技术在番茄抗病育种上的应用正在快速增加[20-21]。国内番茄抗病分子标记的研究具有明显的地域性特点,这与番茄栽培产地实际和国内主要农业研究机构的地域分布基本吻合。各类型的分子标记都在广泛使用,其中SCAR标记的使用量最大。不同病害使用分子标记情况不同,黄化曲叶病毒病是当前比较热门的研究对象。从已发表文献看,目前国内对于番茄抗病分子标记的研究主要集中在科研机构,国内育种公司在这方面的研究还不足,与国外的大公司仍有不小的差距。即使是国内在番茄抗病分子标记研究方面基础较好的单位,目前的主要工作还在于对现有标记的研究和应用上,很少有国内自主开发的新标记。随着分子标记技术在番茄育种上的应用越来越多,自主开发新标记和育种公司参与分子标记的应用研究将会是我国未来对番茄抗病分子标记研究和应用的主要方向。

图6 番茄主要病害使用分子标记技术的情况Fig.6 The utilization of molecular markers used in major disease in tomato breeding
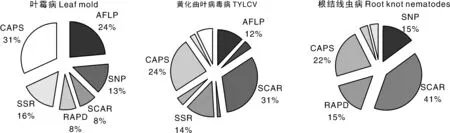
图7 使用分子标记技术最多的三种主要病害分析Fig.7 The analysis of the usage of molecular markers in three major diseases in tomato
[1] GIOVANNUCCI E. Tomatoes, tomato-based products, lycopene, and cancer: Review of the epidemiologic literature[J].TheJournaloftheNationalCancerInstitute, 1999, 91(4): 317-331.
[2] RICK C M, ZOBEL R W, FOBES J F. Four peroxidase loci in red-fruited tomato species: genetics and geographic distribution[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 1974, 71(3): 835-839.
[3] AGRAMA H A, SCOTT J W. Quantitative trait loci for tomato yellow leaf curl virus and tomato mottle virus resistance in tomato[J].JournaloftheAmericanSocietyforHorticulturalScience, 2006, 131(2): 267-272.
[4] 董淑芳,王孝宣,高建昌,等. 番茄粉红色果实相关基因的dCAPS和InDel标记开发与应用[J].中国蔬菜,2016(1):24-29. DONG S F, WANG X X, GAO J C, et al. Development and application of dCAPS and InDel markers in pink tomato fruit related genes[J].ChinaVegetables, 2016(1):24-29. (in Chinese with English abstract)
[5] KIM H J, JUNG J, LEE J M, et al. Development of functional molecular marker system to differentiate multiple disease resistances in tomatoes[C]//Plant & Animal Genome ⅩⅩⅢ, San Diego, CA,2015.
[6] PALLAVI N, SHASHANK S S, LAJJA V, et al. Molecular screening of tomato (SolanumlycopersicumL.) genotypes for resistance alleles against important biotic stresses[J].JournalofAppliedandNaturalScience, 2016, 8 (3): 1654-1658.
[7] YANG W, FRANCIS D M. Genetics and breeding for resistance to bacterial diseases in tomato[M]// RAZDAN M K, MATTOO A K. Genetic improvement of solanaceous crops. Vol 2. Enfield: Science Publishers, 2006: 379-419.
[8] FOOLAD M R, PANTHEE D R. Marker-assisted selection in tomato breeding[J].CriticalReviewsinPlantSciences, 2012, 31(2): 93-123.
[9] JEMIN L, CHANGSIK O, INHWA Y. Molecular markers for selecting diverse disease resistances in tomato breeding programs [J].PlantBreeding&Biotechnology, 2015,3(4):308-322.
[10] 王欣莹, 宋静,张冬冬.我国番茄灰霉病文献计量分析[J].湖北农业科学, 2011, 50(11):2369-2370. WANG X Y,SONG J,ZHANG D D. Bibliometric analysis of literatures onBotrytiscinerea[J].HubeiAgriculturalSciences, 2011, 50(11):2369-2370. (in Chinese with English abstract)
[11] WANG A X, MENG F J, XU X Y, et al. Development of molecular markers linked toCladosporiumfulvumresistant gene Cf-6 in tomato by RAPD and SSR methods[J].HortScience, 2007, 42(1): 11-15.
[12] YAGHOOBI J, YATES J L, WILLIAMSON V M. Fine mapping of the nematode resistance gene Mi-3 inSolanumperuvianumand construction of aS.lycopersicumDNA contig spanning the locus[J].MolecularGeneticsandGenomics, 2005, 274(1): 60-69.
[13] KIM B, HWANG I S, LEE H J, et al. Combination of newly developed SNP and InDel markers for genotyping the Cf-9, locus conferring disease resistance to leaf mold disease in the tomato[J].MolecularBreeding, 2017, 37(5):59.
[14] JI Y, SCHUSTER D, SCOTT J. Ty-3, a begomovirus resistance locus near the tomato yellow leaf curl virus resistance locus Ty-1 on chromosome 6 of tomato[J].MolecularBreeding, 2007, 20(3): 271-284.
[15] JI Y, SCOTT J W, SCHUSTER D J. Molecular mapping of Ty-4, a new tomato yellow leaf curl virus resistance locus on chromosome 3 of tomato[J].JournaloftheAmericanSocietyforHorticulturalScience, 2009,134(2): 281-288.
[16] JI Y F, SCOTT J W, SCHUSTER D J. Toward fine mapping of the tomato yellow leaf curl virus resistance gene Ty-2 on chromosome 11 of tomato[J].HortScience, 2009, 44(3): 614-618.
[17] MENDA N, STRICHLER S R, MUELLER L A. Advances in tomato research in the post-genome era[J].PlantBiotechnology, 2013, 30(3): 243-256.
[18] CHEN H M, LIN C Y, YOSHIDA M, et al. Multiplex PCR for detection of tomato yellow leaf curl disease and root-knot nematode resistance genes in tomato (SolanumlycopersicumL.)[J].InternationalJournalofPlantBreeding&Genetics, 2015, 9(2):44-56.
[19] JUNG J, KIM H J, LEE J M, et al. Gene-based molecular marker system for multiple disease resistances in tomato against Tomato yellow leaf curl virus, late blight, and verticillium wilt[J].Euphytica, 2015, 205(2):599-613.
[20] 陈宝玲,甘桂云,王先裕,等. 126份番茄材料的抗性基因分子标记检测[J]. 中国蔬菜,2016(6):34-41. CHEN B L, GAN G Y, WANG X Y, et al. Molecular marker detection of resistant genes from 126 tomato varieties[J].ChinaVegetables, 2016(6):34-41.
[21] HANSON P, LU S F, WANG J F, et al. Conventional and molecular marker-assisted selection and pyramiding of genes for multiple disease resistance in tomato[J].ScientiaHorticulturae, 2016, 201:346-354.
(责任编辑 张 韵)
Research advance of molecular marker-assisted selection in tomato disease resistance breeding in China
CHEN Yuansong,ZHU Xiaowei,GONG Xiangyu,MEI Zhicheng,ZHU Biao*
(ResearchCenterofBio-BreedingIndustry,SchoolofAgricultureandFoodScience,ZhejiangA&FUniversity,Lin’an311300,China)
Disease resistance breeding is an important direction of tomato breeding. Molecular marker technology largely overcomes the limitations of phenotypic selection, greatly shortens the breeding cycle, and it has been widely used in tomato breeding. This paper reviewed the history and recent advances of molecular marker technology used in tomato disease resistance breeding in China. The characteristics of the main molecular markers and the main diseases in tomato disease resistance breeding were analyzed.
tomato disease; disease resistance breeding; molecular marker
10.3969/j.issn.1004-1524.2017.08.25
2017-02-23
浙江省大学生科技创新活动计划暨新苗人才计划项目(2016R412006);浙江省教育厅科研项目(Y201432633);浙江省“三农六方”科技协作计划项目(SN2014-2);浙江农林大学生物种业研究中心项目(2013SWZY1-5)
陈远松(1992—),男,贵州遵义人,主要研究方向为蔬菜栽培学。E-mail: 1291929934@qq.com
*通信作者,祝彪,E-mail: billzhu@zafu.edu.cn
S436
A
1004-1524(2017)08-1415-06
陈远松,朱晓伟,龚翔宇,等. 分子标记在我国番茄抗病育种的应用研究进展[J].浙江农业学报,2017,29(8): 1415-1420.
