卵巢癌诊断新基因HELQ的生物信息学分析
2017-08-28李亚萍邹余粮
李亚萍,邹余粮,张 琳,黄 新,秦 勇
(1.西安市中心医院妇产科;陕西 西安 710003;2.西安交通大学第一附属医院妇产科,陕西 西安 710061;3.西安市中心医院神经内科;陕西 西安 710003;4.西安市中心医院普通外科;陕西 西安 710003)
卵巢癌诊断新基因HELQ的生物信息学分析
李亚萍1,邹余粮2,张 琳3,黄 新4,秦 勇4
(1.西安市中心医院妇产科;陕西 西安 710003;2.西安交通大学第一附属医院妇产科,陕西 西安 710061;3.西安市中心医院神经内科;陕西 西安 710003;4.西安市中心医院普通外科;陕西 西安 710003)
目的 本研究拟采用生物信息学工具分析卵巢癌易感基因HELQ所编码蛋白的结构及结构与功能的关系,并初步探讨其对肿瘤发生的影响。方法 采用ExPASy服务器中的工具分析HELQ蛋白的理化特征;分别采用PSORTⅡ和TMHMM预测HELQ蛋白的亚细胞定位和跨膜拓扑结构;采用SignalP 4.0预测HELQ的信号肽;采用ScanProsite和SMART分析HELQ的功能结构域以及结构域分区,采用PSIPRED和I-TASSER预测HELQ蛋白的二级和三级结构以及配体结合位点;最后采用STRING 10.0预测HELQ与其他蛋白质的交互作用。结果 HELQ主要定位在细胞核(47.8%)和细胞质(39.1%)中,有4个保守结构域DEXDc、HELICc、HHH_5和PRK02362,并包含解旋酶ATP结合区和解旋酶C端结合区两个功能结构域;成功建立了HELQ的二级和三级结构模型,其中二级结构中α螺旋、无规则卷曲及折叠结构比例分别为54.0%、31.0%和6.0%。三维配体结构分析结果可见,HELQ包含一个酶活性中心位点His341,催化Ile333、Lys335、Tyr337、Gln340、Pro360、Thr361、Ser362、Gly363、Gly364、Lys365、Thr366、Leu367、Glu464和Ala711等结合位点与配体ATP结合。交联蛋白质分析发现HELQ可能与RAD51、POLA1、PLON、FANCD2、DHX8等蛋白存在交互作用。结论 HELQ具有ATP依赖性解旋酶活性,参与DNA损伤的修复过程,可能是其低表达时引起卵巢癌等恶性肿瘤发生的机制。
HELQ;肿瘤;结构;功能;生物信息学。
卵巢癌是女性常见癌症的第二大杀手,早期难以诊断,且预后效果较差,5年生存率约37%[1]。因此,对卵巢癌发生的相关基因的研究,能够更好地为早期诊断和靶向治疗提供理论依据。HELQ基因是2013年在小鼠体内发现的抑制卵巢癌的新基因,该基因缺失时会增加小鼠患卵巢癌的风险。研究发现,小鼠体内HELQ基因的缺失,不仅影响小鼠的生殖,同时增加小鼠发生恶性肿瘤的风险,HELQ基因的拷贝数减少一个,致使卵巢癌发生的风险提高2倍[2]。已有研究表明,HELQ基因可以修复细胞增殖时DNA复制过程中发生的DNA损伤,如果该基因发生缺陷或缺失,DNA错误复制的可能性会增加,从而提高恶性肿瘤发生的几率[3]。因而作者推测,如果HELQ基因在人类和小鼠中的作用一致,未来或可通过筛查HELQ基因来诊断和治疗女性卵巢癌。但纵观国内外研究,关于该基因的研究尚不够成熟,该基因表达蛋白的结构及功能也尚未明确,因此,本研究利用生物信息学工具分析和预测HELQ蛋白的结构特征及其构效关系,并探讨该基因增加卵巢癌发病风险的分子机制,以期为卵巢癌的诊断和治疗开发新的途径。
1 材料与方法
1.1 基因序列
NCBI数据库中获取HELQ基因的核苷酸序列,其Genbank序号为AF436845.1,Gene ID:113510,定位于染色体4q21上。HELQ基因又称HEL308,全长3 591bp,ORF finder分析HELQ基因序列包含17个开放阅读框,其中ORF3编码基因的全长,共1 101个氨基酸。
1.2 生物信息学分析
利用ExPASy服务器[4]中的ProtParam、Compute pI/Mw和ProtScale软件预测HELQ蛋白的理化性质:氨基酸组成、分子质量、等电点、疏水性/亲水性、不稳定系数及脂肪系数等;采用PSORTⅡ(http://www.genscript.com/psort.html)预测HELQ蛋白的亚细胞定位;TMHMM软件分析HELQ的跨膜拓扑结构;SignalP 4.0 Server预测HELQ的信号肽[5];利用Scan Prosite分析HELQ蛋白的功能结构域[6];使用NCBI保守结构域数据库分析蛋白序列的保守结构域[7];利用PSIPRED序列分析分析HELQ蛋白的二级结构[8],利用蛋白质结构建模工具I-TASSER预测HELQ的三维结构模型,并分析其配体结合位点及其配体[9-11];使用STRING 10.0分析HELQ蛋白质与其他蛋白质交联的相互作用[12]。
2结果
2.1 HELQ的理化性质分析
Prot Param计算HELQ蛋白的分子量约为124 175.3Da,理论等电点为6.12,氨基酸组成分布见表1,其中酸性氨基酸多于碱性氨基酸,且亮氨酸(Leu)比例最高(12.4%);不稳定指数为45.55,故推测HELQ应为不稳定的酸性蛋白。ProtScale软件对HELQ进行疏水性分析结果如图1所示,并计算出其疏水性指数平均值为-0.317,脂肪族指数为92.34,说明其为亲脂性蛋白。

图1 氨基酸疏水性直观图(以0为界限,正值表示疏水性,负值表示亲水性)
Fig. 1 Pictorial diagram of hydrophobicity of amino acids (Bounded to 0, hydrophobicity was represented by positive number, and hydrophilicity was represented by negative value)
表1 氨基酸组成分布
Table 1 Distribution of amino acids
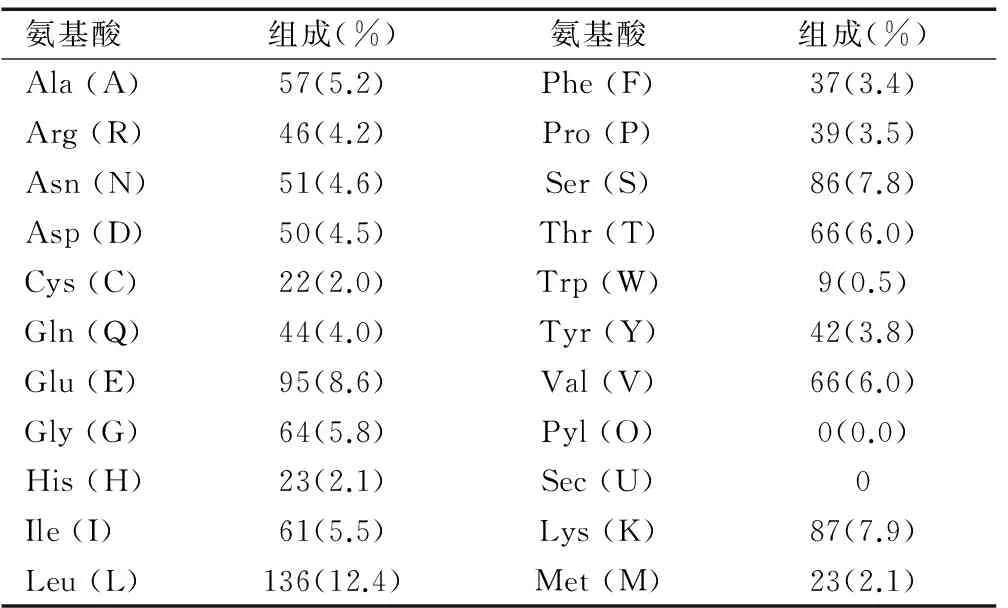
氨基酸组成(%)氨基酸组成(%)Ala(A)57(5.2)Phe(F)37(3.4)Arg(R)46(4.2)Pro(P)39(3.5)Asn(N)51(4.6)Ser(S)86(7.8)Asp(D)50(4.5)Thr(T)66(6.0)Cys(C)22(2.0)Trp(W)9(0.5)Gln(Q)44(4.0)Tyr(Y)42(3.8)Glu(E)95(8.6)Val(V)66(6.0)Gly(G)64(5.8)Pyl(O)0(0.0)His(H)23(2.1)Sec(U)0Ile(I)61(5.5)Lys(K)87(7.9)Leu(L)136(12.4)Met(M)23(2.1)
2.2 亚细胞定位及结构域分析
PSORTⅡ软件对HELQ蛋白的亚细胞定位分析表明:HELQ最大可能分布在细胞核(47.8%)和细胞质(39.1%)中,另外也可能出现在线粒体及囊泡分泌系统(高尔基体、内质网等结构)中(见表2),因而-NN给出的预测结论为HELQ应定位于细胞核中。TMHMM和SignalP4.0的预测结果发现,HELQ蛋白没有跨膜结构域及信号肽,说明该蛋白可能不是跨膜蛋白及内分泌蛋白,而可能是代谢调控蛋白或转录因子。
采用ScanProsite软件预测HELQ蛋白的功能结构域(图2A),预测结果显示HELQ包含两个功能结构域,分别是Helicase_ATP-Bind_1(解旋酶ATP结合区A)和Helicase_Cter(解旋酶C端结合区),其功能分别是结合和水解ATP。通过查找NCBI 保守结构域数据库,发现HELQ序列有4个保守结构域(图2B),分别是DEAD(解旋酶家族成员之一)、HELICc(解旋酶C端超级结构域)、HHH-5(复合螺旋结构域)和PRK02362(ski2_like结构域),其中DEAD参与各种RNA代谢,包括核转录、前信使RNA剪接、核糖体合成、核质运输、翻译、RNA衰变及细胞器基因的表达。
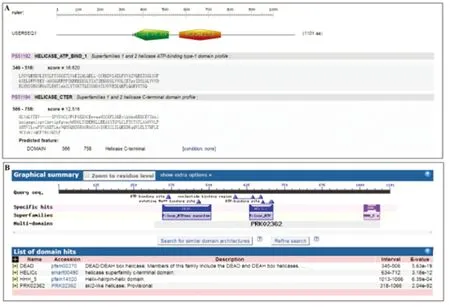
图2 结构域的预测(A:ScanProsite预测HELQ蛋白的功能结构域,B:NCBI保守结构域数据库中预测HELQ的保守结构域)
表2 HELQ蛋白的亚细胞定位预测结果

Table 2 Predicted results of subcellular localization of HELQ protein
2.3 HELQ蛋白结构分析
利用PSIPRED预测HELQ蛋白的二级结构(见图3),结果显示HELQ蛋白结构主要由α-螺旋(54.0%)和无规则卷曲(31.0%)、β-折叠(6.0%)组成。采用I-TASSER对HELQ蛋白的三维结构进行建模结果如图4所示。同时利用3DLigandsite建立HELQ蛋白三维配体结合模型,发现其可能结合的配体为ATP,结合位点为Ile333、Lys335、Tyr337、Gln340、360Pro、361Thr 、Ser362、Gly363、Gly364、Lys365、Thr366、Leu367、464Glu、711Ala,且主要分布在Helicase_ATP-Bind_1功能结构域;另外,结合位点中心区域的His341为HELQ蛋白的酶活性中心位点(图5),因此,推测HELQ的活性依赖于与ATP结合从而提供的能量。

图3 二级结构预测
Fig. 3 Prediction of secondary structure
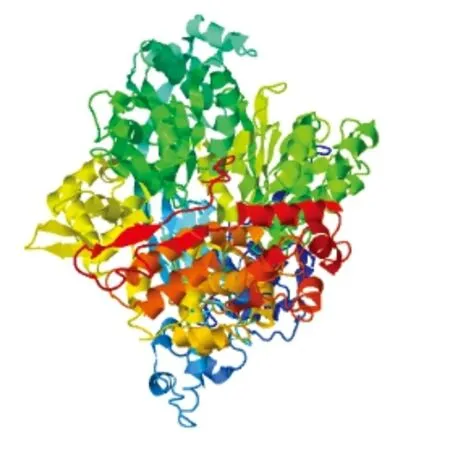
图4 HELQ三维空间结构
Fig. 4 Three-dimensional structure of HELQ protein
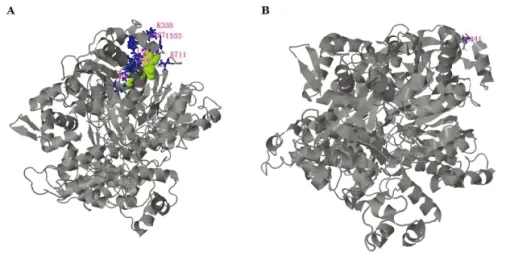
图5 三维空间配体结合位点及活性中心预测模型(A:HELQ配体结合位点模型,B:HELQ活性中心预测模型)
Fig. 5 Prediction models of ligand binding site and active center in three-dimensional space (A: model of ligand binding sites of HELQ, B: prediction models of active center of HELQ)
2.4 HELQ-蛋白交联网络
利用STRING 10.0交互式数据库进行蛋白质交联分析,发现与HELQ相互作用的蛋白质主要有RAD51、POLA1、PLON、FANCD2、DHX8 等(见图6 )。其中RAD51具有DNA依赖的ATP酶活性,参与同源重组和双链断裂时的DNA损伤修复的激活;PLON是一种DNA聚合酶;FANCD2参与DNA双链断裂的修复过程,维持染色体的稳定。而HELQ与这些蛋白存在交互作用,因此推测HELQ的功能作用与这些蛋白相似,也与DNA复制有关,即可能参与修复细胞增殖时DNA复制过程中所发生的的DNA损伤。
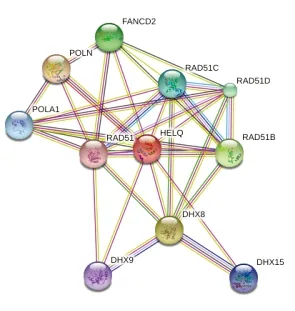
图6 HELQ蛋白的交联作用
Fig. 6 Cross-linked action HELQ protein
3讨论
3.1 HELQ蛋白结构域及其空间结构的分析
DNA修复是细胞对DNA损伤后的一种反应,可使DNA结构恢复、重新执行原来的功能。若DNA的损伤未被完全消除,且细胞能够耐受这种损伤继续生存,那么该DNA损伤有很大几率会在适合的条件下显示出来,如导致细胞癌变。因此,DNA损伤修复与肿瘤的发生密切相关。DNA解旋酶可以修复核苷酸的错配,双链断裂及同源重组交叉反应等DNA损伤,对于维持机体基因及染色体的稳定至关重要。不同的解旋酶如RuvAB、RecBCD、RecQ1等,修复不同的DNA损伤[13-14]。HELQ也是DNA解旋酶之一,编码解旋酶蛋白HEL308。Moldovan等[15]研究发现,人的HEL308缺失可以引起DNA复制过程受阻,且其他研究也进一步证实HEL308是一种依赖ATP的酶,具有3′-5′的DNA解旋酶活性,参与细胞的增殖过程,是一种癌症相关基因[16]。
本研究在此基础上对HELQ的结构及其功能进行了分析预测,结果表明:HELQ包含Helicase_ATP_Bind 1和 Helicase_Cter两个中心位置,功能分别是结合和水解ATP,行使解旋酶的活性;NCBI 保守结构域数据库也发现HELQ包含与DNA双螺旋结合的HHH-5(复合螺旋结构域)、解旋酶家族特征结构域DEAD以及解旋酶C端超级结构域HELICc,同样证实了HELQ具有解旋酶功能。HELQ蛋白三级结构及三维配体结合模型构建结果表明,其结合的配体为ATP, 且结合位点及酶活性中心位点均位于Helicase_ATP-Bind_1功能结构域中,进一步证实了HELQ的解旋酶功能,且其酶活性依赖于ATP供能,而后参与DNA的损伤修复。
3.2 HELQ-蛋白交联分析
蛋白质交联网络分析发现与HELQ相互交联的蛋白均具有修复DNA损伤的功能,其中RAD51协同同系产物(如RAD51B、RAD51D)在DNA损伤的末端形成核蛋白丝,催化断裂的DNA双链与同源DNA姐妹链进行链间重组交换,从而维持基因的稳定性[17]。RAD51是DNA 同源重组修复过程中的关键酶,与BRCA1、BRCA2相互协作,干扰DNA损伤修复,引起基因组的不稳定性,诱导乳腺癌等多种肿瘤的发生[18-19]。PLON是由POLH基因编码的DNA聚合酶ν,修复紫外辐射导致的DNA嘧啶二聚体,使DNA的复制正常进行[16]。FANCD2在应对DNA损伤时第561位的赖氨酸单泛素化,在核中特定位点与FA通路下游相关修复蛋白共定位,修复损伤DNA。FANCD2基因是FA通路的关键基因,若缺失则导致DNA损伤修复功能障碍,引发癌症[20-21]。HDX8是一种依赖ATP的RNA解旋酶。因此推测HELQ 也具有解旋酶的功能。
3.3 HELQ与卵巢癌发生的相关性
已有研究表明人类HELQ基因在卵巢、精巢、心脏及骨骼肌均表达,且HELQ与生殖细胞的发育密切相关。HELQ基因发生突变后,可以引起男性出现少精、唯支持细胞综合症、女性卵巢功能不全而不孕等[22-23]。越来越多的研究集中于对HELQ功能与DNA复制相关性的探索,Ward等[24]研究发现HELQ联合RAD51修复减数分裂时DNA双链的断裂;Luebben等[25]研究发现哺乳动物的HELQ具有维持基因稳定的功能,但HELQ与肿瘤发生发展的关系需进一步确证。Liu等[26]研究发现HELQ可以激活与DNA损伤相关的CHK1-RAD51,抑制骨肉瘤的生长;敲除HELQ基因导致骨肉瘤的增殖和迁移能力增强,但其机制尚不明确。本研究通过生物信息学分析发现HELQ具有依赖ATP的DNA解旋酶活性,参与DNA的损伤修复,因此推测人类HELQ的缺失,可能引起DNA损伤修复功能障碍,因此导致恶性肿瘤尤其是生殖系统肿瘤如卵巢癌的发生。
本研究对HELQ蛋白的结构及功能进行了分析,初步推测其可能参与肿瘤发生发展过程中的DNA损伤修复过程,从而影响恶性肿瘤细胞的增殖以及肿瘤治疗过程中的敏感性。因此推测该基因可能是卵巢癌等肿瘤的易感基因之一,并对其发生机制甚至治疗均起着关键作用,可以作为进一步研究卵巢癌等恶性肿瘤的实验对象之一。
[1]Wagener J, Seybold U.MRSA-hygiene management, diagnostics and treatment[J].Dtsch Med Wochenschr, 2014,139(13):643-651.
[2]Adelman C A, Lolo R L, Birkbak N J,etal. HELQ promotes RAD51 paralogue-dependent repair to avert germ cell loss and tumorigenesis[J]. Nature, 2013, 502(7471): 381-384.
[3]Dolgova E V, Alyamkina E A, Efremov Y R,etal. Identification of cancer stem cells and a strategy for their elimination[J]. Cancer Biol Ther, 2014, 15(10): 1378-1394.
[4]Artimo P, Jonnalagedda M, Arnold K,etal. ExPASy: SIB bioinformatics resource portal[J].Nucleic Acids Res, 2012,40(W1):W597-W603.
[5]Petersen T N, Brunak S, von Heijne G,etal. SignalP 4.0: discriminating signal peptides from transmembrane regions[J]. Nat Methods, 2011, 8(10): 785-786.
[6]Sigrist C J, de Castro E, Cerutti L,etal. New and continuing developments at PROSITE[J].Nucleic Acids Res, 2012, 41(D1): D334-D347.
[7]Marchler-Bauer A, Bo Y, Han L,etal. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures[J]. Nucleic Acids Res, 2017,45(D1):D200-D203.
[8]Buchan D W, Minneci F, Nugent T C,etal. Scalable web services for the PSIPRED Protein Analysis Workbench[J]. Nucleic Acids Res, 2013, 41(W1): W349-W357.
[9]Yang J, Yan R, Roy A,etal. The I-TASSER Suite: protein structure and function prediction[J]. Nat methods, 2015, 12(1): 7-8.
[10]Yang J, Zhang Y. I-TASSER server: new development for protein structure and function predictions[J]. Nucleic Acids Res, 2015, 43(W1): W174-W181.
[11]Roy A, Kucukural A, Zhang Y. I-TASSER: a unified platform for automated protein structure and function prediction[J]. Nat protoc, 2010, 5(4): 725-738.
[12]Szklarczyk D, Franceschini A, Wyder S,etal. STRING v10: protein-protein interaction networks, integrated over the tree of life[J]. Nucleic Acids Res, 2014,43(D1):D447-D452.
[13]Spies M.DNA helicases and DNA motor proteins[M].Advances in experimental medicine and biology:v.767.New York:Springer, 2013:973:V-VII.
[15]Moldovan G L, Madhavan M V, Mirchandani K D,etal. DNA polymerase POLN participates in cross-link repair and homologous recombination[J]. Mol Cell Biol, 2010, 30(4): 1088-1096.
[16]Tafel A A, Wu L, McHugh P J. Human HEL308 localizes to damaged replication forks and unwinds lagging strand structures[J]. J Biol Chem, 2011, 286(18): 15832-15840.
[17]Wang A T, Kim T, Wagner J E,etal. A dominant mutation in human RAD51 reveals its function in DNA interstrand crosslink repair independent of homologous recombination[J]. Mol Cell, 2015, 59(3): 478-490.
[18]彭亚婷, 梅金红. Rad51 与肿瘤关系的研究进展[J]. 临床与实验病理学杂志, 2011, 27(1): 86-89.
[19]高丽丽, 朱长乐. 乳腺癌中 Chk2, p53 和 Rad51 的表达及意义[J]. 临床与实验病理学杂志, 2014, 30(3): 308-311.
[20]刘超, 高军丽, 乐贻军, 等. FANCD2 在结直肠癌中的表达及其与预后的关系[J]. 胃肠病学, 2014, 19(7): 404-407.
[21]Lambert M W, Sridharan D, Zhang P. FANCF, a Fanconi anemia core complex protein involved in monoubiquitination of FANCD2, also has a role with nuclear alpha spectrin in DNA interstrand crosslink repair[J]. Cancer Res,2016;76(14 Suppl):Abstract nr 2758.
[22]Wang W, Zhao S, Zhuang L,etal. The screening of HELQ gene in Chinese patients with premature ovarian failure[J]. Reprod Biomed Online, 2015, 31(4): 573-576.
[23]Pelttari L M, Kinnunen L, Kiiski J I,etal. Screening of HELQ in breast and ovarian cancer families[J].Fam Cancer, 2016,15(1):19-23.
[24]Ward J D, Muzzini D M, Petalcorin M I,etal. Overlapping mechanisms promote postsynaptic RAD-51 filament disassembly during meiotic double-strand break repair[J]. Mol Cell, 2010, 37(2): 259-272.
[25]Luebben S W, Kawabata T, Akre M K,etal. Helq acts in parallel to Fancc to suppress replication-associated genome instability[J]. Nucleic Acids Res, 2013, 41(22): 10283-10297.
[26]Liu D N, Zhou Y F, Peng A F,etal. HELQ reverses the malignant phenotype of osteosarcoma cells via CHK1-RAD51 signaling pathway[J]. Oncol Rep, 2017,37(2):1107-1113.
[专业责任编辑:吕淑兰]
Bioinformatics analysis of a new diagnostic marker of ovarian cancer HELQ
LI Ya-ping1, ZOU Yu-liang2, ZHANG Lin3, HUANG Xin4, QIN Yong4
(1.Department of Gynecology and Obstetrics, Xi’an Central Hospital, Shaanxi Xi’an 710003, China; 2.Department of Obstetrics and Gynecology, First Affiliated Hospital of Xi'an Jiaotong University, Shaanxi Xi’an 710061, China;3.Department of Neurology, Xi’an Central Hospital, Shaanxi Xi’an 710003, China;4.Department of General Surgery, Xi’an Central Hospital, Shaanxi Xi’an 710003, China)
Objective To analyze the structure of protein encoded by ovarian cancer susceptibility gene HELQ and relationship between the structure and function by using bioinformatics tools and to discuss its effect on occurrence of tumors. Methods Tools of ExPASy Server were employed to analyze the physicochemical properties of HELQ protein. Its subcellular localization and transmembrane topology were predicted by PSORTⅡand TMHMM, respectively. Signal peptide of HELQ was predicted by SignalP 4.0. ScanProsite and SMART were used to analyze function domain and domain partition of HELQ. PSIPRED and I-TASSER were employed to predict secondary structure and three-dimensional model and find ligandbinding sites of HELQ protein. And STRING 10.0 was used to predict the interaction of HELQ with other proteins. Results HELQ protein was mainly located in nucleus (47.8%) and cytoplasm (39.1%). HELQ had 4 conserved domains, consisting of DEXDc, HELICc, HHH_5 and PRK02362, and contained 2 function domains, helicase_ATP binding and helicase_C terminal binding. Secondary structure and three-dimensional model were constructed. In secondary structure, proportion of α helix, random coils and β-structure was 54.0%, 31.0% and 6.0%, respectively. Results of three-dimensional model analysis found that HELQ had an enzyme activity site His341, catalyzing binding of binding sites appeared in Ile333, Lys335, Tyr337, Gln340, Pro360, Thr361, Ser362, Gly363, Gly364, Lys365, Thr366, Leu367, Glu464 and Ala711 and ligand ATP. Result of cross-linked protein analysis discovered that HELQ might have interaction with RAD51, POLA1, PLON, FANCD2 and DHX8. Conclusion HELQ possesses ATP dependent helicase activity and participate in DNA repair process. Its low expression is possible pathogenic mechanism of ovarian cancer or other malignant tumor.
HELQ; tumor; structure; function; bioinformatics
2017-05-11
李亚萍(1974-),女,副主任医师,在读博士,主要从事妇科肿瘤研究。
邹余粮,主任医师。
10.3969/j.issn.1673-5293.2017.07.010
R711.7
A
1673-5293(2017)07-0781-05
