青海BYDV-GAV青稞分离物基因组克隆及结构特征分析
2017-08-16李廷芳吴淑华季英华赵文浩周益军郭青云
李廷芳,吴淑华,季英华,赵文浩,周益军,郭青云
(1.青海大学农牧学院,青海西宁 810016; 2.江苏省农业科学院植物保护研究所/江苏省食品质量安全重点实验室-省部共建国家重点实验室培养基地,江苏南京 210014; 3.青海省农林科学院/农业部西宁作物有害生物科学观测实验站/青海省农业有害生物综合治理重点实验室,青海西宁 810016)
青海BYDV-GAV青稞分离物基因组克隆及结构特征分析
李廷芳1,2,3,吴淑华2,季英华2,赵文浩2,周益军2,郭青云1,3
(1.青海大学农牧学院,青海西宁 810016; 2.江苏省农业科学院植物保护研究所/江苏省食品质量安全重点实验室-省部共建国家重点实验室培养基地,江苏南京 210014; 3.青海省农林科学院/农业部西宁作物有害生物科学观测实验站/青海省农业有害生物综合治理重点实验室,青海西宁 810016)
大麦黄矮病毒(Barley yellow dwarf viruses,BYDVs)病是一种在世界范围内发生危害的禾谷类作物病毒病害。2014年笔者在对青海重要作物病毒病调查时发现田间青稞作物上有疑似BYDVs危害的症状,利用BYDVs引物对田间采集的9份典型症状病样进行了RT-PCR检测,结果均扩增到目的条带(约300 bp),随机抽选样品进行序列测定,BLAST分析结果显示其与BYDV-GAV同源性最为接近(99.2%)。为进一步明确该分离物的分类地位及基因组结构特征,根据已测定的BYDV-GAV序列设计全长扩增引物,利用分段法克隆了其全基因组序列,发现其全长为5 683 bp,具有典型的黄症病毒属的特征,编码6个ORF,有4个非编码区(UTR)。基因组序列聚类进化分析结果显示,其与BYDV-GAV聚类于同一分支,与中国陕西渭南小麦分离物亲缘关系较近。以上结果说明,该分离物是BYDV-GAV的一个分离物,这是青海BYDV-GAV青稞分离物全基因组的首次报道。
大麦黄矮病毒;青稞;BYDV-GAV;青海
大麦黄矮病毒(Barley yellow dwarf viruses,BYDVs)是一种在世界范围内发生危害的植物病毒,主要由蚜虫以持久非增殖方式传播,可侵染小麦、大麦、燕麦、玉米等150多种禾本科植物,伴随叶片黄化和植株矮缩等发病症状,影响作物的产量和品质[1-3]。由BYDVs危害麦类作物引起黄矮病早在1951年于美国加州被报道过,而在我国于20世纪60年代在陕西、甘肃小麦上才开始出现该病危害,后陆续扩散蔓延,目前已成为北方麦类作物上的流行性病害,受害小麦常减产40%左右[4-5]。根据病毒血清学、基因组结构及传播介体的专化性等特征,BYDVs分为BYDV-PAV、-MAV、-PAS、-GAV、-RPV、-GPV、-RMV、-SGV等种[6-7],分类上它们都属于黄症病毒科的成员,其中BYDV-PAV、-MAV和-PAS属于黄症病毒属(Luteovirus)的确定种,BYDV-GAV属黄症病毒属暂定种,BYDV-RPV(新名称Cereal yellow dwarf virus-RPV,CYDV-RPV)属于马铃薯卷叶病毒属(Polerovirus)的确定种,BYDV-GPV、-RMV和-SGV属黄症病毒科成员,暂时未列入任何属[8]。目前,我国报道的有4种,分别为BYDV-GAV、-GPV、-PAV和-RMV[9-11]。其中,BYDV-GPV在血清学上与其他病毒不相关[11-12],是我国特有种[9, 13-14],主要由禾谷缢管蚜(Rhopalosiphumpadi)和麦二叉蚜(Schizaphisgraminum)以持久非增殖方式传播[15]。BYDV-GAV主要由麦二叉蚜(S.graminum)和麦长管蚜(Sitobionavenae)传播[16],与BYDV-MAV血清学相关[10]。
青稞是禾本科大麦属的一种裸大麦(HordeumvulgareL.var.nudumHook-.F.),在青海、甘肃、四川、西藏、云南等高原地区有广泛种植,青海种植面积已达到39.3×103hm2,是当地重要粮食作物之一[17-19]。近年来,黄矮病在青稞上的危害在各种植区屡有报道,造成青稞产量减少,局部地区甚至有绝收的报道[18]。但我国针对青稞黄矮病的研究还相对较少,目前尚未有针对该类病毒的全基因组序列相关报道。我国BYDVs的种类较多,开展此类工作有利于明确病毒种类、指导生产防控。笔者对2014年在青海地区青稞作物上采集的9份典型病样利用BYDVs引物进行了RT-PCR检测,经 BLAST分析结果显示其与BYDV-GAV同源性最为接近(99.2%)。为进一步明确该分离物的基因组结构特征及分类地位,利用分段法克隆其全基因组序列后对序列结构特征及系统进化关系进行了分析。
1 材料与方法
1.1 材 料
1.1.1 样 品
2014年6月采集青海省海东市田间表现为叶片黄化、植株轻微矮缩的发病青稞叶片,于-70 ℃保存备用。
1.1.2 化学试剂
Trizol Reagent购自北京康为世纪生物技术有限公司;反转录试剂盒PrimerScriptTMRT Master Mix和DNA LATaq酶购自大连宝生物公司;其他常用化学试剂均为国产分析纯。
1.2 方法
1.2.1 样品总RNA的提取
取典型症状样品0.1 g,采用Trizol法提取总RNA。将RNA溶于35 μL 0.1%DEPC水中,检测RNA的含量和质量后,于-20 ℃保存备用。
1.2.2 BYDVs的RT-PCR检测
样品RNA反转录采用PrimeScriptTMRT Master Mix反转录试剂盒(TakaRa)进行,反应体系:5×PrimeScriptTMRT Master Mix buffer 2 μL,500 ng样品RNA,最终DEPC水定容10 μL。反应条件:37 ℃反应15 min,85 ℃ 5 s终止反应。以合成的cDNA为模板,进行PCR检测,引物(PAVL1/PAVR1)及反应体系参照Deb等[20]。扩增结束后,取5 μL PCR产物,采用1.0%琼脂糖凝胶电泳进行检测,培清JS-680D全自动凝胶成像系统记录结果,编号为14Qh1-1和14Qh8-1的阳性样品送金斯瑞生物技术有限公司进行序列测定。
1.2.3 BYDVs青海分离物基因组的克隆
根据1.2.2中BYDVs检测结果及相应的序列分析结果设计BYDVs青海分离物的全基因组扩增引物,利用分段克隆法扩增其全基因组,其中BYD-1sbF(5′-GGCCTGCAGGTAGTGAAG ATTGACCATCTCACAAAAG-3′)与BYD-1R(5′-GCGGTTATTTCTACGGCCTACT-3′)组合(退火温度54 ℃)扩增第一个片段14Qh8-1F(2 888 bp),BYD-2bsF(5′-GGTGTACATTAG CTCTCTCCTACTTYAT-3′)与BYD-2xmR(5′-CCCCCGGGGGGTTGCCGAACTGCTCTTT-3′)组合(退火温度55 ℃)扩增第二个片段14Qh8-1R(2 892 bp)。PCR反应体系:PrimeSTAR Max 12.5 μL,cDNA 1 μL,上下游引物(10 μmol·L-1)各1.5 μL,最终DEPC水定容至25 μL。反应程序:95 ℃预变性5 min;95 ℃变性45 s,54/55 ℃退火45 s,72 ℃ 延伸3.5 min,35个循环;72 ℃延伸10 min。扩增结束后,PCR产物经1.0%琼脂糖凝胶电泳,由Axygen DNA凝胶回收试剂盒分离纯化,回收产物连接PMD18-T载体,热击法转化大肠杆菌Trans10,挑选阳性克隆,送金斯瑞生物技术有限公司进行序列测定。
1.2.4 序列分析
测序结果BLAST分析由NCBI网站上的BLAST程序(http://www.ncbi.nlm.nih.gov/BLAST/Blast.cgi)完成,序列拼接及同源性分析分别由DNASTAR软件的Seqman、MegAlign完成,序列多重比对、聚类分析及进化树构建采用MEGA 6.0软件的临近法(Neighbor-Joining)完成,进化树的可信度使用1 000次自导复制验证[21]。
2 结果与分析
2.1 田间病株症状
田间发病青稞植株叶尖多表现明显的黄化,有些甚至出现红化(图1),早期侵染的植株会伴有矮化症状,影响青稞抽穗,对产量影响较大。在调查过程中还发现,虽然田块间发病程度存在差异,但各调查田块中均有植株发病,矮化症状也零星见到,暗示该病害在当地青稞上已呈常发态势。

图1 青海省海东市受BYDVs侵染的青稞病株症状Fig.1 Symptom of the hulless barley infected by BYDVs in Haidong, Qinghai province
2.2 病样的RT-PCR检测结果
参照Deb等[20]的方法对田间采集的9份典型症状样品中的BYDVs进行了RT-PCR分子检测,结果(图2)显示,BYDV-PAV引物扩增到了约300 bp的目的片段,与预期目的条带大小符合。为了进一步明确检测到的BYDVs种类,随机抽选样品(14Qh1-1和14Qh8-1)进行序列测定,序列BLAST分析结果显示,它们与BYDV-GAV(EU402390)的同源性最高,相似性达99.2%。
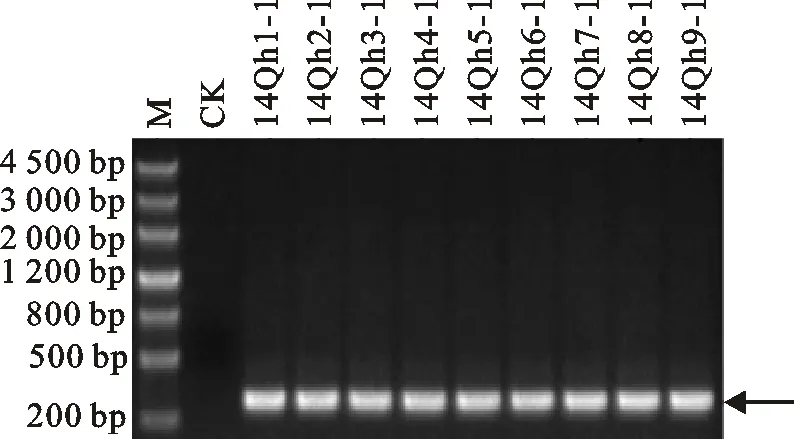
M:核酸分子量标准 DL4500;14Qh1-1至14Qh1-9为9份感病青稞样品; CK为健康青稞样品;箭头所指为目的片段。下同。
M: DNA marker DL4500; 14Qh1-1-14Qh1-9 indicated nine samples of infected hulless barley; CK indicated samples of healthy hulless barley;The target fragments is pointed with an arrow;The same in Fig.3.
图2 引物PAVL1/PAVR1的PCR扩增结果
Fig.2 Results of PCR amplified with primers PAVL1/PAVR1
2.3 全基因组序列克隆结果
以典型样品14Qh8-1总RNA为模板,利用分段的两对引物BYD-1sbF/BYD-1R和BYD-2bsF/BYD-2xmR进行RT-PCR,分别扩增到两条大小约2.8 kb的特异性目的片段(图3)。片段回收纯化后连接pMD18-T载体,PCR法筛选阳性克隆,送测序公司测定序列。
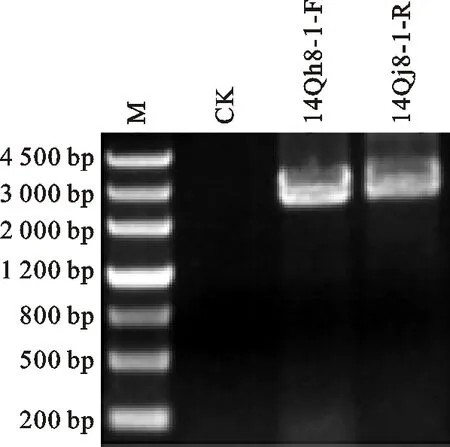
14Qh8-1-F和14Qh8-1-R: 用引物BYD-1sbF/BYD-1R和BYD-2bsF/BYD-2xmR扩增出的片段。
14Qh8-1-F and 14Qh8-1-R indicated the target fragments amplified with BYD-1sbF/BYD-1R and BYD-2bsF/BYD-2xmR, respectively.
图3 引物BYD-1sbF/BYD-1R和BYD-2bsF/BYD-2xmR的PCR扩增结果
Fig.3 Results of PCR amplified with primers BYD-1sbF/BYD-1R and BYD-2bsF/BYD-2xmR
2.4 基因组结构分析
利用Seqman软件将2.3测定的序列进行拼接,获得14Qh8-1的全基因组序列。序列分析结果显示,14Qh8-1的基因组全长为5 683 bp,编码6个ORF,ORF1(140~1 159 bp)编码一个含339个氨基酸、分子量约39 kDa的蛋白P1;ORF2与ORF1会通读(140~2 742 bp),编码一个含867个氨基酸、分子量约99 kDa的融合蛋白P1-P2,是病毒复制酶(RNA-dependent RNA polymerase,RdRp);ORF3(2 856~3 455 bp)编码一个含199个氨基酸、分子量约22 kDa的蛋白P3,是病毒的外壳蛋白(Coat protein,CP);ORF3内部含有一个较小的阅读框ORF4(2 893~3 357 bp),编码一个含154个氨基酸、分子量约17 kDa的蛋白P4,是病毒的运动蛋白(Movement protein,MP);ORF3、ORF4、ORF5(2 856~4 832 bp)会以通读的方式编码一个含659个氨基酸、分子量约72 kDa的蛋白P3-P5,是病毒的蚜传相关蛋白(Aphid transmission protein);ORF6(1 939~5 070 bp)编码一个含43个氨基酸、分子量约4.3 kDa的蛋白P6。除6个ORF外,14Qh8-1还含有4个非编码区(Untranslated regions),其中3′和5′末端各一个,一个位于RdRp与CP之间,另一个位于蚜传蛋白与P6之间。
2.5 同源性及聚类进化分析
对14Qh8-1进行BLAST分析可知,其与BYDV-GAV的同源性最高(98.9%)。将14Qh8-1与已报道的黄症病毒属相关病毒及其分离物(结合黄症病毒属分类标准[8])及BYDVs在我国的发生种类进行同源性分析,结果表明,14Qh8-1与中国报道的BYDV-GAV同源性最高(97.0%~98.9%)。将14Qh8-1与黄症病毒属的成员聚类进行系统进化分析,结果也显示,14Qh8-1聚类到BYDV-GAV分支中(图4)。说明14Qh8-1是BYDV-GAV的一个分离物。
在系统进化关系上,BYDV-GAV分离物聚类到3个簇(图4):陕西渭南分离物(05WN2、05WN3、05WN5)、陕西咸阳杨凌分离物(05YL6)、陕西宝鸡扶风分离物(FF1)及38W、30W等分离物聚类于一个簇(Cluster I);陕西咸阳杨凌分离物(YL4)单独形成第二个簇(Cluster II);陕西宝鸡凤翔分离物(04FX1)、陕西渭南分离物(05WN7、WN5)、陕西安康分离物(AK2)、甘肃天水分离物(TS1)聚类于第三个簇(Cluster III)。其中,14Qh8-1聚类于第一个簇(Cluster I),与陕西渭南分离物(05WN5、05WN2、05WN3)分离物的同源性较近,与其他分离物的同源性相对较远。
3 讨 论
大麦黄矮病毒是危害麦类作物的一类重要病毒病,目前我国报道的有四种[9],其中,BYDV-GAV是根据Rochow[22]系统命名的一个中国分离物,分类上属于黄症病毒属的暂定种。近年来的调查结果显示,该病毒在我国很多地方上升为当地大麦黄矮病毒的优势种群[11]。本文对青海分离的一个青稞分离物14Qh8-1全基因组序列进行了克隆测序,发现其为BYDV-GAV的一个分离物。参照Deb等[20]的方法对样品进行分子检测时,采用的引物包括PAVL1/PAVR1(检测BYDV-PAV)、MAVL1/MAVR1(检测BYDV-MAV)、SGVL2/SGVR2(检测BYDV-SGV)、RMVL1/RMVR2(检测BYDV-RMV)和RPVL/RPVR(检测CYDV-RPV),结果发现,PAVL1/PAVR1和MAVL1/MAVR1都有检出目的条带(部分结果未呈现),而测序结果显示序列既不是BYDV-PAV,也不是BYDV-MAV,而都与BYDV-GAV同源性最高,说明这两对引物在BYDV-GAV上也有匹配位点,而针对14Qh8-1序列的分析结果也印证了这一结论(图5)。除Deb等[20]的方法外,笔者也采用马铃薯卷叶病毒属[23]及黄症病毒属[24]的通用引物进行了检测,结果发现,黄症病毒属的通用引物也可以检测到目的条带,测序结果也显示其与BYDV-GAV(EU402389)的同源性最高(99.8%)。
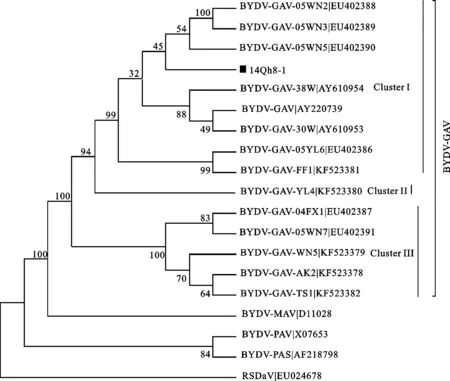
■:本试验测定的序列;各枝上的数字是1 000次Bootstrap自导复制的置信度。
■: The sequence studied in this research;Bootstrap confidence values of each branch are indicated above the branch.
图4 根据病毒核苷酸序列一致性构建的系统进化树
Fig.4 Phylogenetic tree based on nucleotide sequence identities ofLuteovirus

图5 14Qh8-1引物结合位点的分析Fig.5 Analysis of the possible binding sites of primers on 14Qh8-1
目前在我国发生的四种BYDVs中,已有多个全基因组序列得到了克隆[10-11, 25-26],这些工作不仅为我国BYDVs的分类提供了基础,也为针对病毒培育抗性品种及提出有针对性的防控策略奠定了基础,但这些研究大多集中在小麦等作物上,青稞上的研究就相对较少,虽然也有报道显示大麦黄矮病毒也可以侵染青稞[18-19],但尚未见到相关基因组报道。14Qh8-1是第一个报道的BYDV-GAV青稞分离物基因组。在对其与国内BYDV-GAV分离物聚类分析时发现,它们共同聚类于一个大的分枝,该分枝虽然可以分为三个簇,但各分离物在进化关系上既没有表现出地理相关性,也没有表现出寄主相关性。分析可能的原因有两个:第一,BYDV-GAV目前仅在我国有报道,其基因组序列相对保守(同源性超过97.0%),可能未出现地理隔离或者寄主专化型;第二,目前有报道基因组的BYDV-GAV分离物数量有限,主要集中在陕西、甘肃等西北地区,而寄主目前主要集中在小麦上,样本量小可能是导致规律不明显的原因。在BYDV-GAV第一个簇中,14Qh8-1与陕西渭南分离物(05WN5、05WN2、05WN3)相对近缘,暗示了陕西渭南可能是该病毒的来源。
大麦黄矮病毒自1960年在陕西、甘肃的小麦上发现以来,给小麦的生产造成了严重的损失,成为制约小麦产业发展的重要限制因素之一,因此,在生产上,BYDVs在小麦上的危害一直备受关注。但是在一些其他寄主作物上,则关注相对比较少。有研究结果显示,BYDVs能侵染150多种禾本科植物,这些寄主植物一方面是BYDVs的潜在危害对象,另一方面又是BYDVs的中间寄主,在BYDVs周年循环中是重要的病毒侵染来源。本研究明确了侵染青海青稞的病毒为BYDV-GAV,翟 浩等[27]在2010年对青海麦类作物上BYDVs调查时也发现BYDV-GAV是当地优势的大麦黄矮病毒种群,而青稞在当地是重要的农作物,因此,生产上应当密切关注该病毒在青稞等作物上的危害,加大监控力度,尽早开展青稞抗BYDV-GAV种质资源筛选,防止BYDVs在青稞等作物上造成更大的危害。
[1]GRAY S,GILDOW F E.Luteovirus-aphid interactions [J].AnnualReviewofPhytopathology,2003,41:539.
[2]MILLER W A,RASOCHOVA L.Barley yellow dwarf viruses [J].AnnualReviewofPhytopathology,1997,35:167.
[3]ROCHOW W F.Barley yellow dwarf virus: phenotypic mixing and vector specificity [J].Science,1970,167(3919):875.
[4]OSWALD J W,HOUSTON B R.A new virus disease of cereals,transmitted by aphid [J].PlantDiseaseReporter,1951,35:471.
[5]周广和.世界大麦黄矮病研究进展[J].世界农业,1989(7):36.
ZHOU G H.The study of barley yellow dwarf virus research in the world [J].WorldAgriculture,1989(7):36.
[6]ROCHOW W F.Comparative diagnosis of BYDV by serological and aphid transmission tests [J].PlantDiseaseReporter,1979(63):426.
[7]ROCHOW W F.Biological properties of four isolates of barley yellow dwarf virus [J].Phytopathology,1969,59(11):1580.
[8]KING A M Q,ADAMS M J,CARSTENS E B,etal.Virus taxonomy-9th report of the international committee on taxonomy of viruses [R].Pittsburgh:Academic Press,2012:1338.
[9]周广和,张淑香,钱幼亭.小麦黄矮病毒4种株系鉴定与应用[J].中国农业科学,1987,20(4):7.
ZHOU G H,ZHANG S X,QIAN Y T.Identification and application of four strains of wheat yellow dwarf virus [J].ScientiaAgriculturaSinica,1987,20(4):7.
[10]晋治波,王锡锋,常胜军,等.大麦黄矮病毒GAV基因组全序列测定及其结构分析[J].中国科学(C辑:生命科学),2003,33(6):505.
JIN Z P,WANG X F,CHANG S J,etal.The complete nucleotide sequence and its organization of the genome of barley yellow dwarf virus-GAV[J].ScienceinChina(SeriesC:LifeScience),2003,33(6):505.
[11]LIU F,WANG X,LIU Y,etal.A Chinese isolate of barley yellow dwarf virus-PAV represents a third distinct species within the PAV serotype [J].ArchivesofVirology,2007,152(7):1365.
[12]贾 力,吴茂森,张文蔚,等.大麦黄矮病毒PAV中国分离物外壳蛋白基因的克隆及同源性分析[J].植物病理学报,2001(3):219.
JIA L,WU M S,ZHANG W W,etal.Cloning and sequencing of the coat protein gene for a BYDV-PAV Chinese isolate [J].ActaPhytopathologicaSinica,2001(3):219.
[13]ZHOU G H,CHENG Z M,QIAN Y T,etal.Serological identification ofLuteovirusesof small grains [J].PlantDisease,1984,68:710.
[14]周广和,张淑香,ROCHOW W F.一种由麦二叉蚜和麦长管蚜传播的小麦黄矮病毒株系的鉴定[J].植物病理学报,1986(1):19.
ZHOU G H,ZHANG S X,ROCHOW W F.One wheat yellow dwarf virus strain,transmissible by aphid vectors ofSchizaphisgraminumandSitobionavena[J].ActaPhytopathologicaSinica,1986(1):19.
[15]WANG H,WU K,LIU Y,etal.Integrative proteomics to understand the transmission mechanism of barley yellow dwarf virus-GPV by its insect vectorRhopalosiphumpadi[J].ScientificReports,2015,5:10971.
[16]LIU Y,SUN B,WANG X,etal.Three digoxigenin-labeled cDNA probes for specific detection of the natural population of barley yellow dwarf viruses in China by dot-blot hybridization [J].JournalofVirologicalMethods,2007,145(1):22.
[17]梁珠英.试论藏区青稞产业发展现状及对策措施[J].青海农林科技,2014(3):41.
LIANG Z Y.Present situation and countermeasures on Tibetan barley industry development [J].ScienceandTechnologyofQinghaiAgricultureandForestry,2014(3):41.
[18]刘仁建.青稞黄矮病发生规律及防治方法[J].西藏农业科技,2015,37(2):34.
LIU J R.The outbreak regularity and control methods of barley yellow dwarf [J].TibetAgriculturalTechnology,2015,37(2):34.
[19]梅 红,木德伟,李学毅,等.云南青稞种质抗大麦黄矮病抗性鉴定研究[J].云南大学学报,2008,30(S1):25.
MEI H,MU D W,LI X Y,etal.The resistence identification study of Yunnan barley germplasm against barley yellow dwarf [J].JournalofYunnanUniversity,2008,30(S1):25.
[20]DEB M,ANDERSON J M.Development of a multiplexed PCR detection method for barley and cereal yellow dwarf viruses,wheat spindle streak virus,wheat streak mosaic virus and soil-borne wheat mosaic virus [J].JournalofVirologicalMethods,2008,148(1-2):17.
[21]TAMURA K,STECHER G,PETERSON D,etal.MEGA6: Molecular evolutionary genetics analysis version 6.0 [J].MolecularBiologyandEvolution,2013,30(12):2725.
[22]ROCHOW W F.Identification of barley yellow dwarf viruses: comparison of biological and serolo gical methods [J].PlantDisease,1982,66(5):381.
[23]ZHOU C J,XIANG H Y,ZHUO T,etal.Nucleotide sequence of a chickpea chlorotic stunt virus relative that infects pea and faba bean in China [J].ArchivesofVirology,2012,157(7):1393.
[24]ABRAHAM A D,MENZEL W,LESEMANN D E,etal.Chickpea chlorotic stunt virus: a new polerovirus infecting cool-season food legumes in Ethiopia [J].Phytopathology,2006,96(5):437.
[25]ZHANG W,CHENG Z,XU L,etal.The complete nucleotide sequence of the barley yellow dwarf GPV isolate from China shows that it is a new member of the genusPolerovirus[J].ArchivesofVirology,2009,154(7):1125.
[26]WU B,BLANCHARD-LETORT A,LIU Y,etal.Dynamics of molecular evolution and phylogeography of barley yellow dwarf virus-PAV [J].ThePublicLibraryofScienceOne,2011,6(2):96.
[27]翟 浩,刘 艳.青海省大麦黄矮病毒的种类鉴定及基于CP基因的分子进化研究[J].中国农学通报,2011,27(24):253.
QU H,LIU Y.The specie identification of barley yellow dwarf viruses in Qinghai province and molecular evolution study based onCPgenes [J].ChineseAgriculturalScienceBulletin,2011,27(24):253.
Genomic Cloning and Structural Characterization of an Isolate of BYDV-GAV Infecting Hulless Barley in Qinghai Province
LI Tingfang1,2,3,WU Shuhua2,JI Yinghua2,ZHAO Wenhao2,ZHOU Yijun2,GUO Qingyun1,3
(1.College of Agriculture and Animal Husbandry,Qinghai University,Xining,Qinghai 810016,China; 2.Institute of Plant Protection, Jiangsu Academy of Agricultural Sciences/Key Lab of Food Quality and Safety of Jiangsu Province -State Key Laboratory Breeding Base,Nanjing,Jiangsu 210014,China; 3.Qinghai Academy of Agricultural Sciences/Scientific Observing and Experimental Station of Crop Pest in Xining,Ministry of Agriculture/Qinghai Key Laboratory of Agricultural Integrated Pest Management,Xining,Qinghai 810016,China)
Barley yellow dwarf viruses (BYDVs) cause substantial losses throughout the world wherever their hosts, mainly wheat, barley, and oats, occasionally rice and maize, are grown.In 2004, an investigation on crop virus was carried out in Qinghai province and a kind of yellow dwarf disease on hulless barley was found.Based on the symptom and the vector occurred in the fields, nine samples with typical symptom were collected and detected by RT-PCR using BYDVs primers.The result of RT-PCR showed there was a kind of BYDVs associated with the disease.The sequencing and BLAST analysis indicated the virus shared the highest similarity with BYDV-GAV (about 99.2%) and might be an isolate of BYDV-GAV.To further clarify the taxonomic status of the isolate and the characterization of its genome, three pairs of PCR primers were designed and the full genome of the isolate was cloned using three fragments strategy.The sequences were assembled and the complete genome sequence was determined (5 683 bp), which was composed of 6 ORFs and 4 UTRs, showing typical characters ofLuteovirus.Phylogenetic analysis with other members ofLuteovirusshowed the isolate clustered with BYDV-GAV in the same branch and shared the highest similarity with the isolates from Shaanxi, China.These results indicated that the virus associated with hulless barley yellow dwarf disease in Qinghai province is an isolate of BYDV-GAV.To our knowledge, this is the first report on complete genome of BYDV-GAV infecting hulless barley in Qinghai province.
Barley yellow dwarf viruses; Hulless barley; BYDV-GAV; Qinghai
时间:2017-07-07
网络出版地址:http://kns.cnki.net/kcms/detail/61.1359.S.20170707.1815.014.html
2017-02-21
2017-06-16
国家自然科学基金项目(31572074);江苏省“333”高层次人才培养工程科研项目(BRA2013262);江苏省科技基础设施建设计划项目(4911406);公益性行业(农业)科研专项经费项目(201303021)
E-mail:916716296@qq.com(李廷芳);Wushuhau@jaas.ac.cn(吴淑华,与第一作者同等贡献)
郭青云(E-mail:guoqingyunqh@163.com);周益军(E-mail:yjzhou@jaas.ac.cn)
S512.3;S330
A
1009-1041(2017)07-0900-07
