利用微卫星标记技术对红鳍东方鲀(Takifugu rubripes)家系系谱认证的研究
2017-08-12孙建华马爱军崔文晓王广宁孙志宾王新安孟雪松刘圣聪
孙建华,马爱军,崔文晓,王广宁,孙志宾,王新安,孟雪松,刘圣聪,张 涛
(1.上海海洋大学水产与生命学院,上海201306;2.中国水产科学研究院黄海水产研究所青岛市海水鱼类种子工程与生物技术重点实验室,山东青岛266071;3.青岛海洋科学与技术国家实验室海洋生物学与生物技术功能实验室,山东青岛266071;4.大连天正实业有限公司,辽宁大连116000)
利用微卫星标记技术对红鳍东方鲀(Takifugu rubripes)家系系谱认证的研究
孙建华1,2,3,马爱军2,3,崔文晓1,2,3,王广宁2,3,孙志宾2,3,王新安2,3,孟雪松4,刘圣聪4,张 涛4
(1.上海海洋大学水产与生命学院,上海201306;2.中国水产科学研究院黄海水产研究所青岛市海水鱼类种子工程与生物技术重点实验室,山东青岛266071;3.青岛海洋科学与技术国家实验室海洋生物学与生物技术功能实验室,山东青岛266071;4.大连天正实业有限公司,辽宁大连116000)
红鳍东方鲀(Takifugu rubripes)是我国北方地区的一种经济型海水鱼类,但目前对其群体遗传多样性和系谱信息研究成果较少。本文研究了目前红鳍东方鲀养殖群体的遗传多样性情况和系谱信息,为其提供遗传改良的理论基础。从2015年所建的12个红鳍东方鲀家系中随机选取5个家系,采用8个微卫星标记对其遗传多样性进行分析和系谱认证。8个位点共检测得到32个等位基因,每个位点等位基因数为3~6个,平均值为4个。平均观测杂合度(A)范围为0.453 3~0.953 3,平均期望杂合度(He)范围为0.490 8~0.790 1,平均多态信息含量(PIC)范围为0.455 5~0.754 6。不同位点共发现8个特异性等位基因,7#家系发现3个,3#和11#家系各发现2个,12#家系发现1个,10#家系未发现特异性等位基因。根据子代基因型成功推断出5个家系的亲本基因型,据此鉴别各个家系。在fms15位点,可将3#和11#家系与其他家系相区别;TOG01位点可将7#家系与其他家系相区别; f1497位点可将12#家系相区别。因此,fms15,TOG01,f1497这3个标记可以作为鉴别5个家系的特异性标记。结果说明,所选的8个微卫星标记在这5个家系中多态信息含量较高,且在已选用的8个微卫星标记中,最少用3个标记可鉴别5个红鳍东方鲀家系。综合分析表明微卫星标记能有效地为红鳍东方鲀群体的系谱分析提供技术支持,为今后红鳍东方鲀开展分子标记辅助育种提供了可靠的理论依据。
红鳍东方鲀;微卫星;遗传多样性;系谱认证
系谱认证作为一种有效的水产动物遗传育种技术,广泛应用于鱼类[1-8]、甲壳类[9-13]和贝类[14-17]。系谱认证可在选育过程中衡量家系间的遗传参数,分析家系信息,指导亲本的筛选,避免因近亲繁殖而导致种质衰退。特别是在混养条件下,可快速有效地区分各个家系,保持各家系的遗传信息,作进一步地选育工作。为充分有效地利用选育群体,则需找到一种标记可快速准确地鉴别出不同家系。目前水产动物育种采用的标记方法大体分为外挂式标记和体内标记[18],外挂式标记主要以剪鳍和颜色标记为主,以此方法标记鱼体,在一定程度上对鱼体有损伤,且对鱼的生长有负面影响。体内标记主要为被动整合雷达标记、荧光标记和染料标记等,这种方法对鱼体规格有一定要求,且随着鱼体增长,标记有可能出现缺失现象,影响后期观察。随着现代分子技术的发展,分子标记有分辨率高、准确度高、识别快等优点,在家系选育过程中发挥着重要的作用,微卫星标记作为分子标记的一种,被广泛应用于家系的鉴别之中。微卫星标记是一种共显性标记,其具有多态信息含量丰富、保守性高、通用性好、分布广等特点,作为辅助分子育种手段广泛应用于群体多样性分析,系谱认证等方面,利用微卫星标记技术对家系进行系谱分析,可有效地评估各家系的遗传参数,掌握家系信息,在种质资源保护[19]、生态修复和提高育种效率[20]上被广泛应用,并取得一定成效。
(王佳实 编辑)
红鳍东方鲀(Takifugu rubripes),隶属于鲀形目(Telraodontiformes)、鲀科(Tetradontidae)、东方鲀属(Takifugu),是暖温性、广盐性底栖肉食性鱼类。主要分布地区为朝鲜半岛、日本和中国沿海地区[21]。其口感鲜美,肉质细嫩,营养丰富,有较高的经济价值,在我国北方已经形成一定的养殖规模,产品主要出口日本、韩国。选择育种作为培育优良经济性状的手段之一,在选育前要构建遗传信息丰富的群体,群体的遗传多样性决定了遗传信息含量的丰富性,刘永新等[22]利用微卫星标记辅助构建红鳍东方鲀家系,通过34对微卫星引物对红鳍东方鲀两个群体进行遗传评估,从而制定育种计划。
国内的选育方式主要以群体选育为主,但经选育的后代个体之间亲缘关系模糊,遗传信息不明确,有可能在进一步的繁育过程中出现近交的现象,造成红鳍东方鲀种质资源退化,遗传多样性降低,养殖群体成活率下降等现象[23]。为提高种质品质,保持选育群体较高的遗传多样性,作者认为有必要在选育前期对亲鱼进行系谱认证,掌握群体选育的家系背景和遗传信息,使亲鱼保持较高水平的遗传距离,避免近亲繁殖,本研究以微卫星标记作为鉴别家系的方法为目的,展开以下研究。
1 材料与方法
1.1 样品选取
实验所用的红鳍东方鲀样品均于2015年取自大连天正实业有限公司,为当年4月所建的12个全同胞家系,挑选亲鱼为体格健壮、无外伤、色泽好、活力强、腹部性腺轮廓明显的3龄雌雄鱼12对,各家系编号分池饲养,编号从1到12为1#家系到12#家系,从中随机选取5个家系,分别为3#家系,7#家系,10#家系, 11#家系,12#家系,体重范围在80~120 g,体长范围在13~16 cm,各家系选完整健康的鱼取样30尾,共计150尾,取其尾鳍置于-80℃备用。
1.2 引物与试剂
参照Ma等[24]、邹杰等[25],Segawa[26]、郝君等[27]和Kai等[28]使用引物序列共计35对,由上海生工生物科技有限公司合成。微卫星引物序列、片段长度等详情见表1。基因组DNA提取试剂盒、d NTP、taq酶、胰RNA酶等购自生工生物工程(上海)股份有限公司。
1.3 基因组DNA的提取
实验所用红鳍东方鲀基因组DNA是用天根海洋动物组织基因组DNA提取试剂盒提取,提取方法参照使用说明书,之后用琼脂糖凝胶检测目的条带完整性,再用NANO DPOP1000分光光度计测量DNA纯度和浓度,最后将符合条件的个体DNA稀释至40 ng/μL,置于-20℃备用。
1.4 PCR反应及电泳
PCR扩增总体系15μL:Taq酶0.2μL(5 U/μL),d NTP 1.3μL(2.5 mmo L/μL),buffer 1.5μL,上下游引物各0.6μL(10μmo L),模板1μL(50 ng/μL),补充H2O 9.8μL。PCR扩增反应程序为:95℃预变性5 min;94℃变性30 s,退火30 s(各引物的退火温度见表1),72℃复性30 s,30个循环;72℃延伸10 min。对PCR扩增产物进行8%SDS-PAGE电泳,用硝酸银染色检测产物条带,筛选得到目的条带。

表1 35对微卫星引物信息及在本实验中的退火温度Table 1 Information and specific annealing temperatures of 35 pairs of microsatellite marker primers used in this study
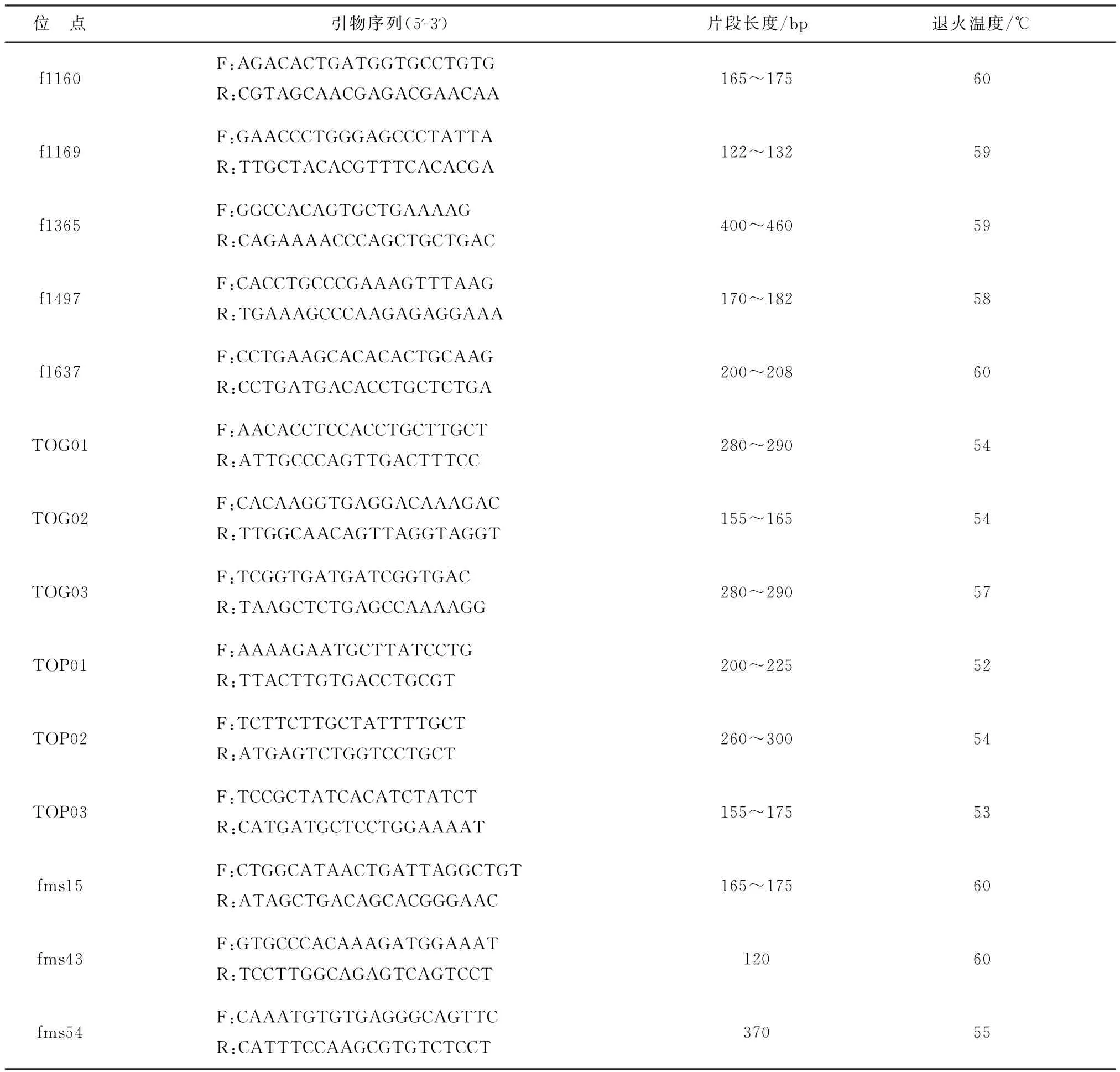
续表
1.5 数据分析
根据每个个体的条带位置确定基因型,按照从大到小的顺序对等位基因分子量大小进行排序,把各位点后两位的数字放在最前面,例如011表示TOG01位点分子量最大的等位基因1。用Popgene32软件处理数据,计算各微卫星位点的等位基因数(A)、有效等位基因数(Ne),平均观察杂合度(Ho)、平均期望杂合度(He),并计算5个家系Hardy-Weinberg遗传偏离指数(d)、平衡检验概率值(P)、遗传相似性系数(I)和遗传距离(D)。根据Botstein等[29]的方法,计算多态信息含量(PIC)。采用MEGA4.1软件根据遗传距离进行聚类分析。
2 结 果
2.1 遗传多样性分析
随机抽取30个个体,对35个微卫星标记进行多态性筛选(图略),得到8个多态性好的微卫星标记,分别为f362,f1497,f1637,fms15,fms89,f169,f1372和TOG01。将8个微卫星标记的数据用Popgene32软件分析,数据内容见表2。分析结果表明,8个微卫星位点的多态信息含量在0.455 5~0.754 6,平均多态信息含量为0.597 3,具有较为丰富的多态性。150个样本数(N)经8个微卫星位点检验共得到32个等位基因,各位点的等位基因数在3~6个,等位基因出现的最高频率(S)在0.266 7~0.690 0,每个微卫星座位平均含4个等位基因;观测杂合度在0.453 3~0.953 3,平均观测杂合度为0.709 2;期望杂合度在0.490 8~0.790 1,平均期望杂合度为0.655 6。对8个微卫星位点进行哈迪温伯格平衡(HWE)检测,结果显示,f1497位点符合哈迪温伯格平衡,其余7个微卫星位点都极显著偏离哈迪温伯格平衡(P<0.001)。8个微卫星位点在5个红鳍东方鲀家系中的遗传分化指数(Fst)均大于0。

表2 8个微卫星位点在红鳍东方鲀群体中的遗传多样性指数Table 2 Genetic diversity parameters of 8 microsatellite loci among cultured Takifugu rubripes populations
根据所得遗传距离,经MEGA4.1软件聚类分析,得到5个红鳍东方鲀家系之间的遗传距离和相似性指数(表3),并构建UPGMA(图1)。从结果可知,3#家系和12#家系之间的遗传距离最小,相似性最高,聚合在一起,然后与7#家系聚合在一起,最后再与10#家系和11#家系聚合在一起。
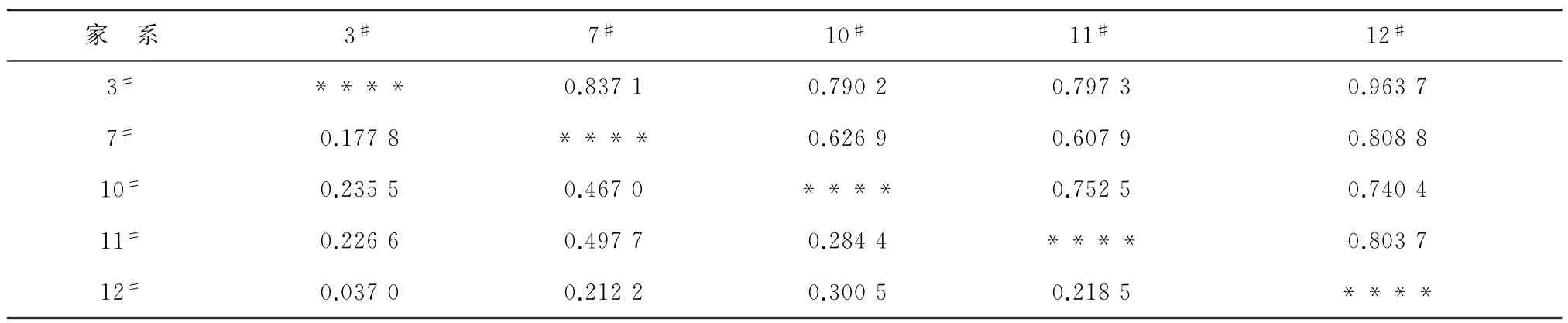
表3 红鳍东方鲀5个家系的遗传距离和相似指数Table 3 Genetic similarity index and distance in 5 Takifugu rubripes families

图1 红鳍东方鲀5个家系的UPGMA图Fig.1 UPGMA dendrogram among 5 Takifugu rubripes families
2.2 家系特异性鉴别
用8个微卫星位点在5个红鳍东方鲀家系中检测,共计得到32个等位基因。每个位点的等位基因数为3~6个。fms89位点有6个等位基因;fms15位点有5个等位基因;TOG01,f362和f1372位点有4个等位基因;f1497,f169和f1637位点有3个等位基因。5个家系中检测到8个家系特异性等位基因,在3#家系和11#家系中各检测到2个,在12#家系中检测到1个,在7#家系中检测到3个,在10#家系中没有未发现家系特异性等位基因。fms15位点的特异性基因a(图2)可用于鉴别3#家系与其他4个家系;TOG01位点的特异性基因b(图3)可用于鉴别7#家系与其他4个家系;fms15位点的特异性基因c(图4)可用于鉴别11#家系与其他4个家系;f1497位点的特异性基因d(图5)可用于鉴别12#家系与其他4个家系。因此,fms15, TOG01和f1497位点可作为区分5个家系的特异性标记。

图2 3#家系在fms15位点的微卫星DNA扩增图谱Fig.2 Demonstration of microsatellite loci amplified by fms15 primer pairs in 3#family

图3 7#家系在TOG01位点的微卫星DNA扩增图谱Fig.3 Demonstration of microsatellite loci amplified by TOG01 primer pairs in 7#family
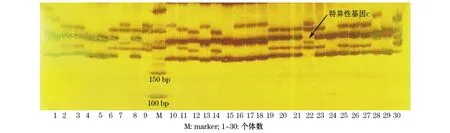
图4 11#家系在fms15位点的微卫星DNA扩增图谱Fig.4 Demonstration of microsatellite loci amplified by fms15 primer pairs in 11#family

图5 12#家系在f1497位点的微卫星DNA扩增图谱Fig.5 Demonstration of microsatellite loci amplified by f1497 primer pairs in 12#family
2.3 推断缺失亲本基因型
微卫星标记属于共显性分子标记,严格遵循孟德尔遗传定律,子代的等位基因一半来自母本,一半来自父本,因此可根据子代的等位基因来推测缺失亲本的基因型。每个家系的等位基因信息列于表4。在TOG01位点,7#家系有4种基因型(图3),分别为014/014,013/014,011/013和011/014,基因型中存在014/014,由此推测其父母本的基因型中各包含有等位基因014,又因为基因型中存在013/014和011/014,推测得知其亲本基因型为(011/014)×(013/014)。运用同样的方法可推断出5个家系在其他位点的亲本基因型,具体信息见表4。
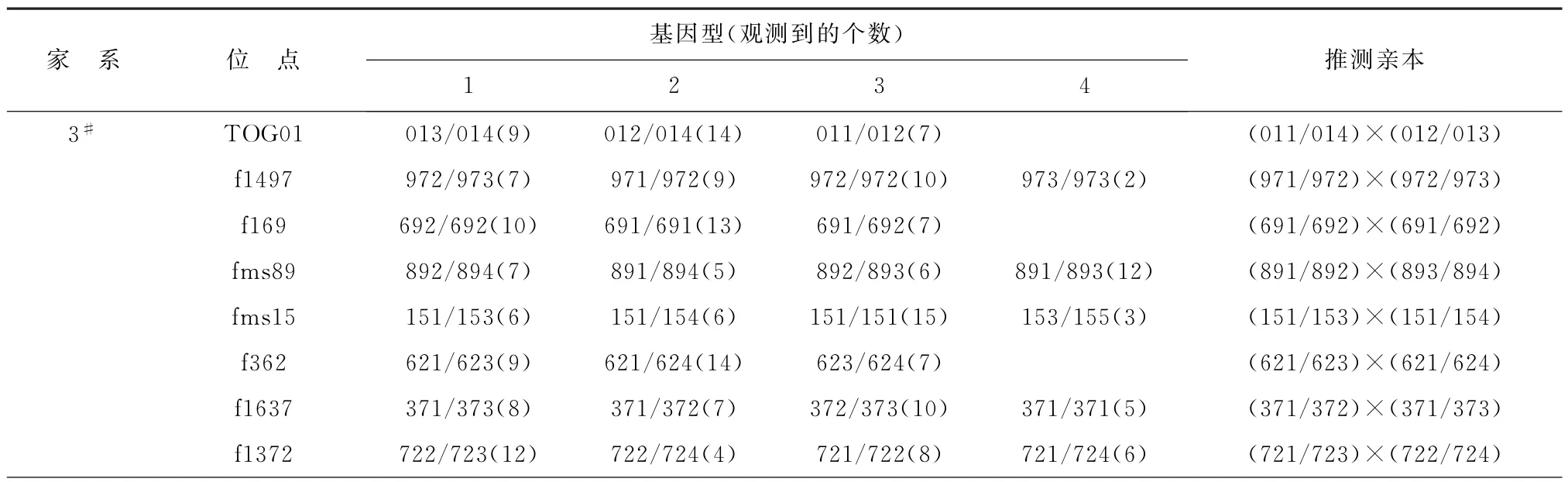
表4 各家系观察到和推断出的基因型Table 4 Observed and conferred genotypes of each family

续表
3 讨 论
明确家系系谱之间的关系是家系选育的前提,通过家系选育可提高有利基因在某一群体内出现的频率,降低育种不需要的基因频率,建立正确的系谱,掌握育种群体的系谱信息。传统的外部物理标记如颜色、电子标记等有明显的不足之处[10],与其相比,微卫星标记的优点有多态性高、遵循孟德尔分离定律、共显性遗传等特点,已成为多种水产动物系谱分析的重要工具[30]。
目前已有微卫星标记应用于海洋生物家系系谱认证的报道,董世瑞等[31]根据子代基因型成功地推断出4个中国明对虾(Penaeus orientalis)家系的父本基因型,通过基因型找到父母本,鉴别出4个家系;刘磊等[32]根据已知的亲本基因型和子代基因型,推断出6个三疣梭子蟹(Portunus trituberculatus)家系全部亲本基因型,鉴别了各个家系;杜晓东等[33]根据子代基因型,推断出4个马氏珠母贝(Pinctada martensi)家系的亲本基因型,以此鉴别不同家系;于飞等[2]根据已知亲本基因型和子代基因型,推断出7个大菱鲆(Scophthalmus maximus)家系中缺失的其他亲本基因型,以此鉴别各个家系;马群群[34]根据已知子代基因型和母本基因型,推断出4个拟穴青蟹(Scylla Paramamosain)家系中缺失的其他亲本基因型,以此鉴别各个家系。红鳍东方鲀的家系鉴别尚未报道,本研究应用8个微卫星标记,推断出5个红鳍东方鲀家系缺失的亲本基因型,其中仅用3个微卫星标记fms15,TOG01和f1497便可鉴别出5个家系。
种群进化和遗传改良离不开物种的遗传多样性,遗传多样性丰富的物种,具有更大的遗传进展的空间[35]。目前,已有相关报道通过利用微卫星标记手段对红鳍东方鲀亲本进行选配,万玉美等[36]利用21个微卫星标记对从日本引进的红鳍东方鲀群体和在秦皇岛经过群体选育的岛红鳍东方鲀群体进行遗传参数的评估。其中从日本引进的红鳍东方鲀群体的平均观测杂合度(Ho)为0.657 2,平均期望杂合度(He)为0.651 6,选育的秦皇岛红鳍东方鲀群体的平均观测杂合度(Ho)为0.523 9,平均期望杂合度(He)为0.607 3。本实验的5个红鳍东方鲀家系的平均观测杂合度(Ho)为0.709 2,平均期望杂合度(He)为0.655 6。杂合度是评估群体遗传多样性的重要指标,杂合度越高,群体的遗传多样性就越高[37],与上述实验结果相比较,本实验的红鳍东方鲀家系的遗传多样性较高,这表明家系内含有丰富的遗传信息,适应环境变化能力强,是进行选育的良好群体。
本研究结果表明,多态信息含量(PIC)在8个微卫星位点上分别为TOG01位点0.701 0,f1497位点0.507 7,f169位点0.511 9,fms89位点0.754 6,fms15位点0.455 5,f362位点0.631 2,f1637位点0.547 0, f1372位点0.669 2。根据相应的评价标准:当PIC<0.25时,此位点为低度多态性位点;当0.25<PIC<0.50时,此位点为中度多态性位点;当PIC>0.50时,此位点为高度多态位点[32]。本研究仅fms15位点多态信息含量为0.455 5,为中度多态位点,其余7个位点的多态信息含量都比0.5大,为高度多态位点。由此可知,本研究所用的8个微卫星标记在5个红鳍东方鲀家系中具有较高的多态信息含量,说明这5个群体内的遗传变异大,有着相对较高的遗传多样水平。除f1497位点外,其他7个位点在5个红鳍东方鲀家系中的数据表明,7个位点都显著偏离Hardy-Weinberg平衡(P<0.01)。这种现象出现的主要原因:1)由于样本数较多,取样时有少数混养造成误差;2)人为读取基因型时判断错误,导致出现无效等位基因;3)某些杂合过剩的位点突变,导致产生不同的等位基因。以上原因需要进一步研究解决,以免对实验结果带来影响。
通过对5个红鳍东方鲀家系进行系谱分析,发现了8个家系特异性等位基因,但这8个家系特异性等位基因是否单独属于某个家系,还需要用大量的样本进行研究。本文用8个微卫星位点中的fms15,TOG01, f1497三个微卫星位点即可区分出5个红鳍东方鲀家系。这充分证明了微卫星标记可作为追踪系谱的有效工具,通过特异性标记来区分不同家系,为家系选育提供科学的理论参考依据。
[1] FROST L A,EVANS B S,JERRY D R.Loss of genetic diversity due to hatchery culture practices in barramundi(Lates calcarifer)[J]. Aquaculture,2006,261(3):1056-1064.
[2] YU F,WANG W J,KONG J,et al.The application of microsatellite markers for genealogical identification in selective breeding program of turbot(Scophthal musmaximus L.)[J].Haiyang Xuebao,2009,31(3):127-136.于飞,王伟继,孔杰,等.微卫星标记在大菱鲆(Scophthal musmaximus L.)家系系谱印证中的应用研究[J].海洋学报,2009,31(3):127-136.
[3] MCDONALD G J,DANZMANN R G,FERGUSON M M.Relatedness determination in the absence of pedigree information in three cul-tured strains of rainbow trout(Oncorhynchus mykiss)[J].Aquaculture,2004,233(1):65-78.
[4] MIAO G D.Development of SSR Markers and construction of genetic linkage maps of half-smooth tongue sole(Cynoglossus semilaevis) [D].Qingdao:Ocean University of China,2010.苗贵东.半滑舌鳎微卫星标记开发、应用及遗传连锁图谱的构建[D].青岛:中国海洋大学,2010.
[5] JEONG D S,GONZALEZ E B,MORISHIMA K,et al.Parentage assignment of stocked black sea bream Acanthopagrus schlegelii in Hiroshima Bay using microsatellite DNA markers[J].Fisheries Science,2007,73(4):823-830.
[6] NAVARRO A,BADILLA R,ZAMORANO M J,et al.Development of two new microsatellite multiplex PCRs for three sparid species: Gilthead seabream(Sparus auratus L.),red porgy(Pagrus pagrus L.)and redbandedseabream(P.auriga,Valenciennes,1843)and their application to paternity studies[J].Aquaculture,2008,285(1):30-37.
[7] LOU J F,LEI S Y,ZHU J L,et al.AFLP analysis of genetic diversity in two cultured communities of Pseudosciaena crocea[J].Advances in Marine Science,2015,33(3):361-366.娄剑锋,雷世勇,竺俊全,等.岱衢洋与官井洋大黄鱼养殖群体遗传多样性的AFLP分析[J].海洋科学进展,2015,33(3):361-366.
[8] SEKINO M,SUGAYA T,HARA M,et al.Relatedness inferred from microsatellite genotypes as a tool for broodstock management of Japanese flounder Paralichthys olivaceus[J].Aquaculture,2004,233(1):163-172.
[9] LI Y,WONGPRASERT K,SHEKHAR M,et al.Development of two microsatellite multiplex systems for black tiger shrimp Penaeus monodon and its application in genetic diversity study for two populations[J].Aquaculture,2007,266(1):279-288.
[10] JERRY D R,PRESTON N P,CROCOS P J,et al.Parentage determination of Kuruma shrimp Penaeus(Marsupenaeus)japonicus using microsatellite markers(Bate)[J].Aquaculture,2004,235(1):237-247.
[11] CUI H Y,MA H Y,MA C Y,et al.Genetic diversity among different families of mud crab Scylla paramamosain by microsatellite markers[J].Marine Fisheries,2011,33(3):274-281.崔海玉,马洪雨,马春艳,等.利用微卫星标记比较分析拟穴青蟹不同家系的遗传多样性[J].海洋渔业,2011,33(3):274-281.
[12] MA C Y,MA H Y,MA L B,et al.Microsatellite analysis on genetic variation of imported Litopenaeus vannamei population and cultured stocks[J].Marine Fisheries,2011,33(1):1-8.马春艳,马洪雨,马凌波,等.凡纳滨对虾引进群体和2个养殖群体遗传变异的微卫星分析[J].海洋渔业,2011,33(1):1-8.
[13] LIU L,LI J,LIU P,et al.Applicability of microsatellite DNA markers in parentage determination of Portunus trituberculatus[J].Progress in Fishery Sciences,2010,31(5):76-82.刘磊,李健,刘萍,等.微卫星DNA标记用于三疣梭子蟹家系亲子关系的鉴定[J].渔业科学进展,2010,31(5):76-82.
[14] ZHANG H B,LIU X,ZHANG G F,et al.Effects of effective population size on the Fl growth and survival of bay scallop Argopecten irradians[J].Haiyang Xuebao,2005,27(2):177-180.张海滨,刘晓,张国范,等.不同有效繁殖群体数对海湾扇贝F1生长和存活的影响[J].海洋学报,2005,27(2):177-180.
[15] LI Z B.Multiplex PCR application in microsatellite of Haliotis rubra[J].Oceanologia et Limnologia Sinica,2005,36(4):319-325.黎中宝.多元PCR在黑鲍(Haliotis rubra)微卫星遗传研究中的应用[J].海洋与湖沼,2005,36(4):319-325.
[16] PORTA J,PORTA J M,MARTíNEZ-RODRíGUEZ G,et al.Development of a microsatellite multiplex PCR for Senegalese sole(Solea senegalensis)and its application to broodstockmanagement[J].Aquaculture,2006,256(1):159-166.
[17] CHENG P,YANG A G,WU B,et al.The applicability analysis on microsatellite markers for parentage determination of different shell color lines of Japanese scallop(Patinopecten yessoensis)[J].Acta Hydrobiologica Sinica,2011,35(5):768-775.程鹏,杨爱国,吴彪,等.微卫星标记在不同壳色虾夷扇贝家系亲权鉴定的适用性[J].水生生物学报,2011,35(5):768-775.
[18] LI X J,HU C,WANG L,Current research progress of fish family selective breeding[J].Fisheries Science,2016,35(1):81-86.李学军,胡灿灿,王磊,等.鱼类家系选育的研究进展[J].水产科学,2016,35(1):81-86.
[19] HU J,HOU X Y,YIN S W,et al.Genetic diversity of different geographical populations of Cheilinus undulatus revealed by microsatellite analysis[J].Advances in Marine Science,2013,31(4):538-545.胡静,侯新远,尹绍武,等.波纹唇鱼(Cheillinus undulatus)不同地理种群遗传多样性的微卫星分析[J].海洋科学进展,2013,31(4):538-545.
[20] ZHANG B,SONG W P.Recent progresses on microsatellite multiplex PCRs for parentage analysis of aquatic animals[J].Marine Fisheries,2012,34(3):350-356.张博,宋文平.微卫星多重PCR在水生动物亲权分析中的研究进展[J].海洋渔业,34(3):350-356.
[21] WANG K Q,CHEN M,GAO T X.Study on taxonomy and fauna of Takifugu[J].Journal of Ocean University of Qingdao(Natural Science),2001,31(6):855-860.王奎旗,陈梅,高天翔.东方鲀属鱼类的分类与区系分布研究[J].青岛海洋大学学报(自然科学版), 2001,31(6):855-860.
[22] LIU Y X,LIU Y,ZHOU Q,et al.Application of microsatellite markers in parental apolegamy of redfin puffer Takifugu rubripes[J]. Journal of Dalian Ocean University,2015,30(2):113-119.刘永新,刘奕,周勤,等.利用微卫星标记指导红鳍东方鲀亲本选配[J].大连海洋大学学报,2015,30(2):113-119.
[23] HE X Y,BAI J J,LIU X L,et al.Application of microsatellite makers for parentage determination in Micropterus salmoides[J].Journal of Northwest A&F University(Natural Science Edition),2009(8):55-62.何小燕,白俊杰,刘小林,等.微卫星DNA标记在大口黑鲈亲权鉴定中的应用[J].西北农林科技大学学报(自然科学版),2009(8):55-62.
[24] MA H Y,CHEN S L,LIAO X L,et al.Isolation and characterization of polymorphic microsatellite loci from a dinucleotide-enriched genomic library of obscure puffer(Takifugu obscurus)and cross-species amplification[J].Conserv Genet,2009,10(4):955-957.
[25] ZOU J,MA A J,ZHAO Y F,et al.Study on the genetic structure of three cultured populations of Takifugu obscurus with microsatellite markers[J].Progress in Fishery Sciences,2015,36(2):55-62.邹杰,马爱军,赵艳飞,等.利用微卫星标记分析3个暗纹东方鲀(Takifugu obscurus)养殖群体的遗传多样性[J].渔业科学进展,2015,36(2):55-62.
[26] SEGAWA S.Takifugu obscurus'studies in high growth genetic and molecular biology[D].Tokyo:Tokyo University,2009.
[27] HAO J,SUN X W,MENG X S.Analyzing the polymorphisms of Takifugu rubripes with microsatellite[J].Journal of Shanghai Fisheries University,2006,15(1):21-24.郝君,孙效文,孟雪松.红鳍东方鲀微卫星DNA多态性初步分析[J].上海水产大学学报,2006, 15(1):21-24.
[28] KAI W,KIKUCHI K,TOHARI S,et al.Integration of the genetic map and genome assembly of fugu facilitates insights into distinct features of genome evolution in teleosts and mammals[J].Genme Biology&Evolution,2011,3(3):424-442.
[29] BOTSTEIN D,WHITE R L,SKOLNICK M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[30] SUN N,LI Q,YU H,et al.Parentage determination of Scapharca broughtonii based on microsatellite analysis[J].Periodical of Ocean University of China,2015(9):42-48.孙楠,李琪,于红,等.利用微卫星标记的魁蚶混交家系鉴定[J].中国海洋大学学报(自然科学版),2015(9):42-48.
[31] DONG S R,KONG J,ZHANG Q W,et al.Pedigree tracing of Fenneropenaeus chinensis by microsatellite DNA markers genotyping [J].Haiyang Xuebao,2006,28(5):157-161.董世瑞,孔杰,张庆文,等.微卫星分型方法进行中国明对虾家系系谱鉴定[J].海洋学报,2006,28(5):157-161.
[32] LIU L,LI J,LIU P.Parentage determination of Portunus trituberculatus in family establishment based on microsatellite DNA markers [J].Periodical of Ocean University of China,2012,42(7-8):38-44.刘磊,李健,刘萍.基于微卫星标记的三疣梭子蟹家系系谱认证[J].中国海洋大学学报(自然科学版),2012,42(7-8):38-44.
[33] DU X D,GAO Y Z,DENG Y W,et al.Genetic structure and genealogical identification of four families of pearl oyster Pinctada martensii,as inferred by microsatellite markers[J].Journal of Fisheries of China,2011,35(12):1795-1804.杜晓东,高远镇,邓岳文,等.利用微卫星标记进行马氏珠母贝家系遗传结构分析与系谱认证[J].水产学报,2011,35(12):1795-1804.
[34] MA Q Q.Development of SNP and SSR markers and establishment of parentage analysis technique in Scylla paramamosain[D].Shanghai:Shanghai Ocean University,2012.马群群.拟穴青蟹SNP和SSR标记开发及系谱认证技术的建立研究[D].上海:上海海洋大学,2012.
[35] CHEN L Z,WANG X F,WANG S.China's biodiversity and its protection countermeasures[M].Beijing:Science Press,1993.陈灵芝,王献溥,汪松.中国的生物多样性:现状及其保护对策[M].北京:科学出版社,1993.
[36] WAN Y M,WANG L,TAN Z J,et al.Genetic analysis of two Takifugu rubripes populations and correlation of microsatellite markers and economic traits[J].Freshwater Fisheries,2011,41(5):9-16.万玉美,王蕾,谭照君,等.红鳍东方鲀两个群体的遗传结构及与经济性状的相关性分析[J].淡水渔业,2011,41(5):9-16.
[37] XU X,WANG J Y,JIANG J Z,et al.Studies on the genetic structure of Haliotis diversicolor reeve in coastal area of Guangdong Province by microsatellite markers[J].Advances in Marine Science,2012,30(2):284-294.许新,王江勇,姜敬哲,等.广东沿海杂色鲍养殖群体遗传多样性的微卫星分析[J].海洋科学进展,2012,30(2):284-294.
Parentage Determination of the Takifugu rubripes Families by Microsatellite Markers
SUN Jian-hua1,2,3,MA Ai-jun2,3,CUI Wen-xiao1,2,3,WANG Guang-ning2,3,SUN Zhi-bin2,3, WANG Xin-an2,3,MENG Xue-song4,LIU Sheng-cong4,ZHANG Tao4
(1.Fisheries and Life Science,Shanghai Ocean University,Shanghai 201306,China; 2.Qingdao Key Laboratory for Marine Fish Breeding and Biotechnology,Yellow Sea Fisheries Research Institute,Chinese Academy of Fishery Sciences,Qingdao 266071,China; 3.Laboratory for Marine Biology and Biotechnology,Qingdao National Laboratory for Marine Science and Technology,Qingdao 266071,China; 4.Dalian Tianzheng Industrial Co.Ltd.,Dalian 116000,China)
Takifugu rubripes is one of the ecnomic sea fish in northern coast of China.In recent years, there are few studies on genetic diversity and genealogical information of Takifugu rubripes.In this study the genetic diversity and genealogical information of Takifugu rubripes were investigated,providing the basis of theory for genetic improvement.Five families were randomly sampled from twelve families which have been established in 2015.Eight pairs of microsatellite primers were used to identify the genealogy and analyze the genetic diversity among five cultured populations of Takifugu rubripes.Thirty-two alleles were detected at eight microsatellite loci.The number of alleles(A)at each locus ranged from three to six with an average of four.The value of average observed heterozygosities was 0.453 3~0.953 3.The expected heterozygosities(He)was 0.490 8~0.790 1 and the mean polymorphic information content(PIC) ranged from 0.455 5 to 0.754 6.Eight family unique alleles were found out:three in 7#family,two in 3#family,two in 11#family,one in 12#family.Family unique allele was not found in family 10#.Based on the genotypes of offspring,all parental genotypes of the five families were successfully deduced.3#family and 11#family were identified from the other families at locus fms15.7#family was separated from the other families at locus TOG01.12#family was distinguished from the other families at locus f1497.The three microsatellite markers(fms15,TOG01,f1497)could be used to identify five families.Results show that there exists high genetic diversity among the five families.The five families could be identified using at least three microsatellite markers selected from Eight microsatellite markers.Microsatellite marker is an useful tool for genealogical identification of Takifugu rubripes.And it is a reliable basis for molecular marker-assisted breeding in the future.
Takifugu rubripes;microsatellite;genetic diversity;genealogical identification
June 30,2016
Q953
A
1671-6647(2017)03-0392-12
10.3969/j.issn.1671-6647.2017.03.009
2016-06-30
中央级公益性科研院所基本科研业务费专项——红鳍东方鲀育种群体的遗传差异和生长性状的遗传分析(20603022012005);大连金州新区科技计划项目资助——红鳍东方鲀(Takifugu rubripes)大规模家系选育技术研究(2012-B1-012)
孙建华(1991-),男,甘肃金昌人,硕士研究生,主要从事海水鱼类增养殖方面研究.E-mail:sjhxy91@outlook.com
*通讯作者:马爱军(1971-),女,山东荣成人,研究员,博士,博士生导师,主要从事海水鱼类增养殖方面研究.E-mail:maaj@ysfri.ac.cn
