百合S-RNase基因cDNA全长克隆及表达分析
2017-08-09丁榕王杰赵和文张克中崔金腾
丁榕,王杰,赵和文,张克中,2,3*,崔金腾,2,3
(1.北京农学院 园林学院,北京 102206; 2.城乡生态环境北京实验室,北京 102206;3.北京市乡村景观规划设计工程技术研究中心,北京 102206)
百合S-RNase基因cDNA全长克隆及表达分析
丁榕1,王杰1,赵和文1,张克中1,2,3*,崔金腾1,2,3
(1.北京农学院 园林学院,北京 102206; 2.城乡生态环境北京实验室,北京 102206;3.北京市乡村景观规划设计工程技术研究中心,北京 102206)
[目的]克隆百合S-RNase基因cDNA全长序列并进行表达信息分析,为百合不亲和机制的研究奠定基础。[方法]以东方百合(Liliumoriental)品种‘Justina’为材料,通过RACE技术克隆百合S-RNase基因的cDNA全长序列;采用半定量RT-PCR技术对百合同一时期不同器官的S-RNase基因的表达量进行定性分析;并通过NCBI在线工具以及ProtParam、SignalP、MEGA5对其序列进行生物信息学分析。[结果]克隆基因全长为766 bp,开放阅读框为672 bp,推导编码223个氨基酸;其半定量分析显示表达量由高到低的器官依次为花瓣、叶、茎、大量分泌液时的花柱、无分泌液时的花柱、鳞茎。通过生物信息学分析发现,该蛋白为一个不稳定的亲水蛋白、存在信号肽序列,属于分泌型蛋白。具有典型的RNase T2家族蛋白保守结构域特征,二级结构富含环形结构。在已知的S-RNase基因中,与野茶树的亲缘关系最近。[结论]获得百合S-RNase基因cDNA全长序列及在同一时期不同器官的S-RNase基因的表达量。
百合;S-RNase基因; 克隆; 半定量
S-RNase(S-glycoprotein RNase)是一种N-糖基化的核酸酶,主要在花柱的柱头和引导组织中表达,是茄科、蔷薇科以及玄参科等植物自交不亲和性的雌性决定因子[1]。早在1986年,首次在花烟草(Nicotianaalata)中得到一种与自交不亲和有关的花柱糖蛋白的cDNA克隆[2],随后发现其是一种与真菌RNase Rh和RNase T2具有相似的结构的核酸酶,具有能够分解RNA的核酸酶活性,称为S-RNase[3,4]。其酶活性能够选择性的降解自花花粉管中的RNA,而异花花粉管中能够正常生长[5],并且其酶活性对于自交不亲和反应是必需的,缺失后无法产生对相同S基因型花粉的抑制作用[6]。其作用原理是S-RNase能够进入花粉管发挥自身的细胞毒素作用降解RNA,从而阻止花粉管生长[7]。S-RNase基因位于S-locus,具有S 等位基因的多态性[8]。随着研究的不断深入,在茄科、蔷薇科以及玄参科等植物中已克隆到上百个S-RNase基因,其作用原理也逐渐清晰[9,10]。
百合(Lilium)是在世界范围内广泛应用的鲜切花和园林花卉,兼具食用、药用等用途。目前,关于百合品种S 基因的研究极少,亟待突破[11]。在本课题组对于东方百合‘Justina’未分泌粘液的雌蕊与分泌大量粘液的雌蕊的转录组测序结果中,S-RNase基因在分泌大量粘液的雌蕊中相对表达量较大,表明对于百合这一类具有不亲和性的物种来说,S-RNase基因对于百合不亲和机制具有重要作用。关于该基因对自交不亲和性与杂交不亲和性的机理是否存在联系还尚未明确,需要进一步研究。本研究的主要目的在于克隆该基因,并进行表达量分析,为后续的工作打下基础。
1 材料与方法
1.1 试验材料与试剂
植物材料为北京农学院东大地球根花卉大棚常规栽培管理的东方百合(Liliumoriental)品种‘Justina’。
3′-RACE-Full Length cDNA Kit试剂盒、反转录试剂盒、琼脂糖凝胶切胶回收试剂盒、pMD19-T Vector连接试剂盒、DH5α感受态细胞等均购自TaKaRa公司。
1.2 百合S-RNase基因cDNA序列全长克隆
在‘Justina’开花前一天去雄、套袋;有大量分泌液时,快速摘取花柱;液氮速冻后置于-80 ℃超低温冰箱保存备用。采用Trizol法分别提取RNA,并根据转录组测序所得的S-RNase基因序列,设计3′内、外引物(表1),进行3′cDNA末端扩增反应。通过 DNAMAN软件将3′RACE测序序列与转录组测序所得序列进行拼接,利用ORF Finder对拼接结果进行分析,得到一个包括起始密码子和终止密码子在内的672 bp的最大开放阅读框架,与NCBI数据库其他物种的S-RNase基因比对长度相符且相似性很高,说明这段核苷酸序列为该基因的全长序列。设计扩增完整ORF框的特异性引物F1、R1(表1)进行进行全长验证的PCR扩增反应。

表1 PCR 扩增所用引物及其序列
1.3 百合S-RNase基因的半定量表达分析
‘Justina’开花前一天去雄、套袋;在无分泌液时快速摘取花柱(BL1)、花瓣、叶、茎、鳞茎;在有大量分泌液时,快速摘取花柱(BL2),均液氮速冻后置于-80 ℃超低温冰箱保存备用。采用Trizol法分别提取RNA,以18S rRNA作为内参基因,由引物F1、R1(表1),进行半定量表达分析。
1.4 百合S-RNase基因的生物信息学分析
通过ORF Finder(http://www.ncbi. nlm.nih.gov/gorf/orFig.cgi)寻找S-RNase基因的开放阅读框架、将核酸序列翻译成氨基酸序列;BLAST 工具进行同源性分析(http://blast.ncbi.nlm.nih.gov/);Protein在线数据库搜索S-RNase氨基酸序列(https://www.ncbi.nlm.nih.gov/protein/?term=);CDD在线工具进行结构域分析(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi);Expasy在线系统Protparam(http://web.expasy.org/cgi-bin/protparam/protparam)预测S-RNase基因的基本理化性质在线程序预测二级结构:PredictProtein(http://www.predictprotein.org/);运用Signalp4.1(http: //www.cbs.dtu.dk/services/SignalP)对S-RNase基因氨基酸序列进行信号肽预测分析;运用MAGA 5.2对S-RNase基因进行氨基酸序列比对分析和系统进化树构建。
2 结果与分析
2.1 百合S-RNase基因cDNA序列克隆
以东方百合‘Justina’有大量分泌液时的花柱为试验材料,提取总RNA;以转录组测序所得的S-RNase基因序列为基础,进行3′RACE,得到400 bp左右的扩增产物(图1,A),测序结果显示该片段实际长度为362 bp;转录组测得序列和3′RACE所得序列拼接结果通过ORF Finder分析找到672 bp 的完整开放阅读框,进行全长克隆验证,得到约750 bp左右的扩增产物(图1,B),测序结果显示该片段实际长度为766 bp,符合预期结果。

A: 3′-RACE扩增结果;B:cDNA PCR 扩增结果图1 百合S-RNase基因扩增结果A. Amplification results of3′-RACE, B.Amplification results of cDNAFig.1 Amplification results of Lilium S-RNase注: M: 2000 DNA markerNote: M: 2000 DNA marker
2.2 百合S-RNase基因的半定量表达分析
以‘Justina’无分泌液时快速摘取花柱(BL1)、花瓣、叶、茎、鳞茎及有大量分泌液时的花柱(BL2)为材料,各取材1 g,18S rRNA作为内参基因,进行半定量表达分析。结果表明(图2),S-RNase在BL1、BL2、花瓣、叶、茎中均有不同程度的表达,而在鳞茎中没有表达。其中,花瓣表达水平最高,叶次之,BL1最低。S-RNase基因在不同器官的表达水平具有空间差异性。S-RNase基因在BL2表达量较BL1高,说明该基因对于雌蕊的授粉可能有重要作用。
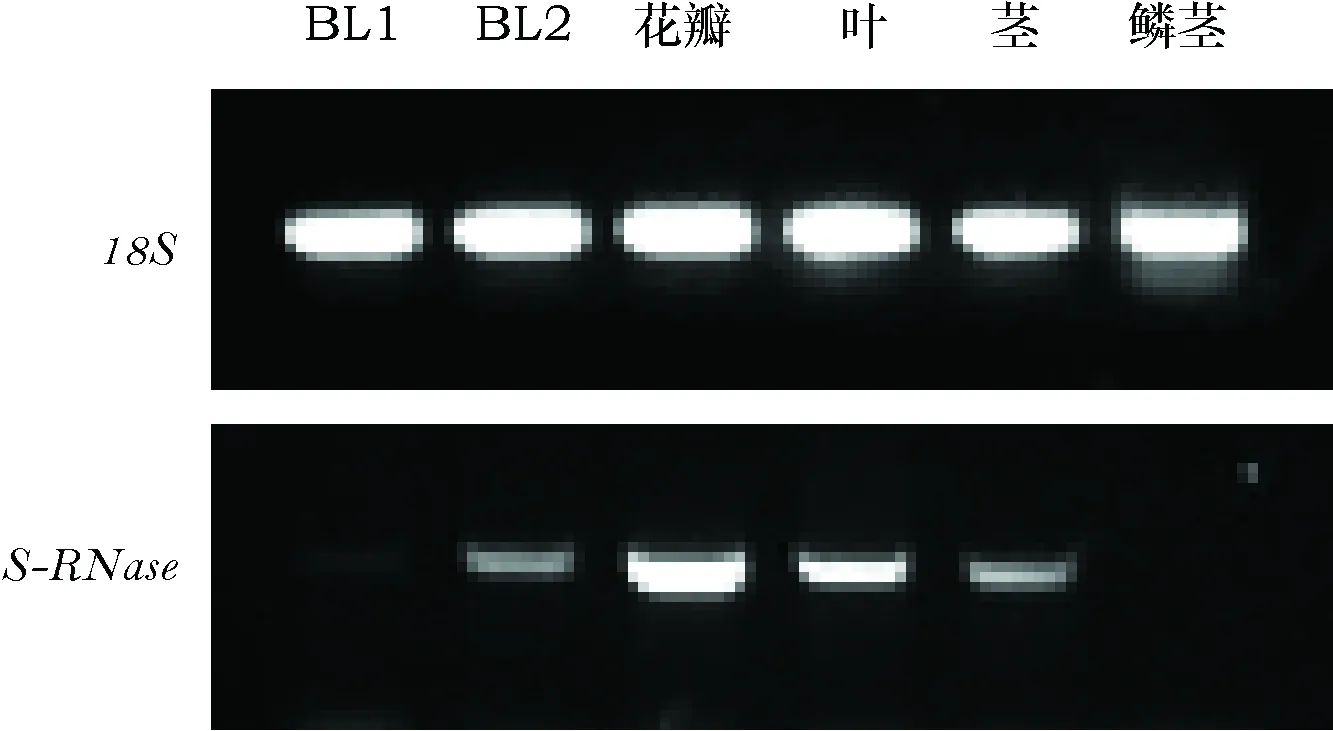
图2 半定量RT-PCR电泳结果Fig.2 The test of semi-quantitative RT-PCR
2.3 百合S-RNase基因及其编码蛋白的生物信息学分析
通过ORF Finder在线工具推导百合S-RNase基因共编码223个氨基酸(图3),其中起始密码子ATG位于31~33 bp处,终止密码子TAA位于699~702 bp处。通过CDD在线工具进行保守结构域分析,发现其具有两种RNase T2家族蛋白的特征结构域CAS (conserved amino acid)模体。通过SignalP 4.1进行信号肽预测分析,其为分泌蛋白,可能在跨膜运输中起到信号识别作用;剪切位点位于20~21位氨基酸,成熟肽始于第20位氨基酸。ProtParam预测蛋白基本质理化性质,相对分子质量为24 870.8 Da,理论等电点pI为4.87,是一个不稳定蛋白;Protscale预测整个蛋白为亲水蛋白;通过PredictProtein在线程序进行百合S-RNase蛋白二级结构预测表明,螺旋(H)比例为28.70%;折叠(E)比例为13%;环形结构(L)比例为58.30%。

图3 百合S-RNase cDNA序列及其氨基酸序列Fig.3 S-RNase gene cDNA sequence of Lilium and the deduced amino acid 注:启示密码子(ATG)、终止密码子(TAA)用红色边框标注,黄色区域为CAS模体,蓝色字体为其信号肽序列Note:The start codon (ATG) and the stop codon (TAA) were marked by red box.The CAS motifs were marked by yellow colors.The signal peptide were marked by blue font
因为S-RNase是茄科、蔷薇科以及玄参科等植物自交不亲和性的雌性决定因子,所以在NCBI数据库中分别找到了茄科植物8条S-RNase基因序列、蔷薇科植物14条S-RNase基因序列、玄参科金鱼草仅有的1条S-RNase基因序列,以及除茄科、蔷薇科以及玄参科以外的仅有的罗汉果、野茶树各一条序列,共选取了S-RNase基因的氨基酸序列25条。利用MEGA 5.2 软件,对百合S-RNase基因的推导氨基酸序列与选取的25条氨基酸序列进行比对并构建系统进化树。
根据 Kubo 等研究[12,13]以及S-RNase基因氨基酸序列多重比对结果显示(图4),茄科植物可依次分为C1、C2、HVa、HVb、C3、C4、C5,5 个保守区(C1~C5)和 2 个高变区(HVa、HVb);蔷薇科植物可依次分为C1、C2、HV、C3、C4、C5,五个保守区(C1~C5)和一个高变区(HV);东方百合以及金鱼草、罗汉果、野茶树等有明显的C1、C2、C3区以及高变区,无明显的C4及C5区域,并且茄科植物与蔷薇科植物的C4区明显不同。
采用NJ法构建系统进化树,基于1 000次重复以Bootstrap验证进行分析。结果显示(图5),所有样本被分为A和B 2个大区域,蔷薇科均植物被分到A区域,茄科植物均被分到B区域,与植物科属分类相同。蔷薇科李属植物均被分在A-1区,梨属植物与苹果属植物没有完全分开,被共同分在A-2区,其中,梨属西洋梨与苹果属植物亲缘关系较近,被分在同一分支。在双子叶植物中,东方百合与野茶树亲缘最近,与蔷薇科、茄科植物较远。玄参科金鱼草与茄科植物亲缘关系较近。
3 讨论与结论
本试验以东方百合‘Justina’的有大量分泌液时的花柱为材料,结合转录组得到的序列,克隆到一个具有完整编码区的S-RNase基因,并对基因序列及编码蛋白进行分析,发现其具有典型的RNase T2家族蛋白保守结构域特征,属于RNase T2家族[14]。经信号肽预测分析,其为分泌蛋白,与授粉过程中从花柱传导细胞中分泌至胞外基质并能够进入花粉管细胞的功能有关[15]。 S-RNase 的高变区是花柱特异性识别花粉SFB的区域,与自交不亲和反应的特异性有关[16]。S-RNase的保守区可分为五个区域(C1~C5),其中C1、C4和C5区是疏水区,与蛋白结构的稳定性有关;C2和C3区是亲水区,与蛋白的催化活性有关[17],与RNase T2保守区相同,是S-RNase 的活性区域。在比对结果中,茄科植物与蔷薇科植物的C4区不同,并且东方百合等也无明显的C4区域,这说明C4区域具有明显的科间特征。因目前东方百合等植物的S-RNase基因克隆结果太少,其C4、C5区域还需更多序列进行比对分析。可利用基因突变[18]或基因沉默[19]等技术,可以对信号肽区域的核酸序列进行干扰,使其无法进入花粉管细胞,或者对活性区域进行干扰,使其丧失核糖核酸酶活性,也应用于高变区,从而进行自交不亲和性的分子调控。

图4 百合 S-RNase 与其它物种的氨基酸序列比对图Fig.4 Sequence alignment of amino acid of the Lilium S-RNase and and other species

图5 百合S-RNase基因与其它物种的氨基酸序列系统进化树Fig.4 Sequence alignment of amino acid of the Lilium S-RNaseand and other species注:支持率低于50的隐去Note:approval rating below 50 hides
百合S-RNase基因的半定量表达分析结果显示,S-RNase基因在BL2(大量分泌液花柱)表达量较BL1(无分泌液的花柱)高,说明该基因对于雌蕊的授粉可能有重要作用。而在除花柱以外的其它不同器官组织中也有不同程度的表达,这说明S-RNase基因在百合中除参与雌蕊的授粉外可能还起到其它功能。RNase T2家族蛋白广泛存在于生物体内,能够在植物落叶、损伤和侵染反应过程中表达。已有研究表明,RNase T2家族还具有与核酸酶活性无关的其他功能[20]。
本试验以东方百合‘Justina’的有大量分泌液
时的花柱为材料,成功克隆了S-RNase基因cDNA全长序列,并分析了S-RNase基因在同一时期不同器官的表达量,为今后深入探讨百合的不亲和机制奠定基础。
[1]Higashiyama T. Peptide signaling in pollen-pistil interactions[J].Plant and Cell Physiology,2010, 51(2):177-189.
[2]Anderson M A, Cornish E C, Mau S L, et al. Cloning of cDNA for a stylar glycoprotein associated with expression of self-incompatibility in Naicotiana alata[J].Nature,1986,321(6065):38-44.
[3]McClure B A, Haring V, Ebert P R, et al. Style self-incompatibility gene products of Nicotiana alata are ribonucleases[J].Nature,1989, 342(6252):955-957.
[4]Sassa H, Nishio T, Kowyama Y, et al. Self-incompatibility (S) alleles of the Rosaceae encode members of a distinct class of the T2/S ribonuclease superfamily[J]. Molecular Genetics and Genomics, 1996, 250(5):547-557.
[5]McClure B A, Gray J E, Anderson M A, et al. Self-incompatibility in Nicotiana alata involves degradation of pollen rRNA[J].Nature, 1990,347(6295):757-760.
[6]Lee H S, Huang S, Kao T. S proteins control rejection of incompatible pollen in Petunia inflata.[J]. Nature, 1994, 367(6463):560.
[7]孟冬. 苹果MdABCF转运S-RNase至花粉管影响自交不亲和反应[D].北京:中国农业大学,2014.
[8]Hua Z H, Fields A, Kao T. Biochemical Models for S-RNase-Based Self-Incompatibility[J]. Molecular Plant, 2008, 1(4):575-585.
[9]Chen G, Zhang B, Zhao Z, et al. ‘A life or death decision’ for pollen tubes in S-RNase-based self-incompatibility[J]. Journal of Experimental Botany, 2010, 61(7):2027-2037.
[10]Chen G, Zhang B, Liu L, et al. Identification of a Ubiquitin-Binding Structure in the S-Locus F-Box Protein Controlling S-RNase-Based Self-Incompatibility[J]. Journal of Genetics and Genomics, 2012, 39(2):93-102.
[11]黄洁, 刘晓华, 管洁,等. 百合分子育种研究进展[J]. 园艺学报, 2012, 39(9):1793-1808.
[12]Kubo K, Entani T, Takara A,et al. Collaborative non-self recognition system in S-RNase-based self-incompatibility[J]. Science, 2010, 330(6005):796-799.
[13]关鹏, 曾斌, 李疆,等. 2个野扁桃自交不亲和S-RNase基因全长的克隆与序列分析[J]. 果树学报, 2016,33(8):905-916.
[14]Deshpande R A, Shankar V. Ribonucleases from T2 family[J]. Critical Reviews in Microbiology, 2002, 28(2):79-122.
[15]Meng X, Sun P, Kao T. S-RNase-based self-incompatibility in Petunia inflata[J]. Annals of Botany, 2011, 108(4):637-646.
[16]Ishimizu T, Endo T, Yamaguchi-Kabata Y, et al. Identification of regions in which positive selection may operate in S-RNase of Rosaceae: implication for S-allele-specific recognition sites in S-RNase[M].International conventions on protection of humanity and environment. W. de Gruyter, 1998:337-342.
[17]Ioerger T R,Gohlke J R,Xu B,Kao T. Primary structural features of the self-incompatibility protein in olanaceae [J].Sexual Plant Reproduction,1991,4(2):81-87.
[18]Poloznikov A A, Zakharova G S, Chubar T A, et al. Site-directed mutagenesis of tobacco anionic peroxidase: Effect of additional aromatic amino acids on stability and activity[J]. Biochimie, 2015(115):71-77.
[19]Aras S, Aydn S S, Fazllu A, et al. RNA interference in plants[J]. Türk Hijiyen Ve Deneysel Biyoloji Dergisi Turkish Bulletin of Hygiene and Experimental Biology, 2015, 72(3):255-262.
[20]Luhtala N,Parker R. T2 Family ribonucleases: ancient enzymes with diverse roles [J]. Trends in Biochemical Sciences,2010,35(5):253-259.
(编辑:马荣博)
Clone and expression analysis of the full-length cdna sequences ofS-RNaseinLilium
Ding Rong1, Wang Jie1, Zhao Hewen1, Zhang Kezhong1,2,3*, Cui Jinteng1,2,3
(1.CollegeofLandscape,BeijingUniversityofAgriculture,Beijing102206,China; 2.BeijinglaboratoryofUrbanandruralecologicalenvironment,Beijing102206,China; 3.BeijingEngineeringResearchCenterofrurallandscapeplanninganddesign,Beijing102206,China)
[Objective]The clone and the expression analysis of the full length sequence of LiliumS-RNasegene which would made a foundation for discovering the molecular regulation of incompatibility in Lilium.[Methods]The full cDNA sequence ofS-RNasewas isolated from a style of Lilium oriental ‘Justina’ by RACE. Semi-quantitative RT-PCR technology was used to analyse the expression of different organs of theS-RNasegene in the same period. Bioinformatic analysis was used by the online tools of National Center for Biotechnology Information and ProtParam, SignalP, MEGA5.[Results]The result showed that the full length of theS-RNasegene was 766 bp, with an open reading frame of 672bp, which encoded a protein with 223 amino acids. The semi-quantitative expression showed that its expression order from higher to lower were petals, leaves, stems, BL2, BL1, bulbs. It was predicted that thisS-RNaseprotein was a hydrophilic proteins with a signal peptide,which belonging to the secrete protein. This protein had a typical conserved domains of RNase T2 family. The secondary structure was rich in loop structure. The relationship analysis showed that Camellia sinensis was closest with Lilium among the knownS-RNasegene.[Conclusion]The full length sequence ofLiliumS-RNasegene was obtained and the expression of different organs of theS-RNasegene in same period were analysed by semi-quantitative RT-PCR technology.
Lilium,S-RNasegene, Cloning, Semi quantitative RT PCR
2017-03-07
2017-06-10
丁榕(1992-),女(汉),山东潍坊人,硕士研究生,研究方向:园林植物与观赏园艺
*通信作者:张克中,教授,硕士生导师,Tel:01080797210; E-mail: zkzzxd@vip.sina.com
北京市教育委员会2015年科技计划面上项目(KM201510020011);北京市属高等学校创新团队建设项目(IDHT20150503);科技创新服务能力建设-协同创新中心-林果业生态环境功能提升协同创新中心(2011协同创新中心)(市级)(PXM2017_014207_000043);北京农学院学位与研究生教育改革与发展项目(5056516004/026)
S682.29
A
1671-8151(2017)09-0649-07
