海藻糖合成酶结构和功能的研究进展
2017-08-01王东生周家海
王东生,耿 强,周家海,江 凌*,黄 和
(1.南京工业大学生物与制药工程学院,江苏 南京 210009;2.南京工业大学食品与轻工学院,江苏 南京 210009;3.中国科学院上海有机化学研究所 生命有机化学国家重点实验室,上海 200032;4.南京工业大学药学院,江苏 南京 210009)
海藻糖合成酶结构和功能的研究进展
王东生1,耿 强2,周家海3,江 凌2*,黄 和4
(1.南京工业大学生物与制药工程学院,江苏 南京 210009;2.南京工业大学食品与轻工学院,江苏 南京 210009;3.中国科学院上海有机化学研究所 生命有机化学国家重点实验室,上海 200032;4.南京工业大学药学院,江苏 南京 210009)
海藻糖合成酶能够以麦芽糖为底物,通过分子内转糖基作用,一步催化反应生成海藻糖。综述了国内外海藻糖合成酶结构和功能的最新研究进展,列举了已经被克隆和表达的海藻糖合成酶及其酶学性质,总结和比较了几种已被解析的海藻糖合成酶的结构和催化机理,指出了海藻糖合成酶的深入研究对海藻糖工业化生产的重要意义。
海藻糖合成酶;晶体结构;催化机制
海藻糖合成酶(trehalose synthase,简称TreS)能够以麦芽糖为底物,催化分子内转糖基作用生成海藻糖,即将麦芽糖的α,α-1,4-糖苷键转变为海藻糖的α,α-1,1-糖苷键。通过对不同来源TreS序列同源性分析发现,TreS是糖基水解酶13家族(glycoside hydrolase family 13,简称GH13)成员之一[1]。Tsusaki等[2]将其列为GH13的16亚族(GH13-16 subfamily,麦芽糖葡萄糖基转移酶亚族)和33亚族(GH13-33 subfamily,TreS亚族)。
由于海藻糖的特殊性质,一般不能通过化学方法合成,大多采用生物法合成。目前,工业上常用酶催化、微生物抽提和发酵这3种方法大规模生产海藻糖[3]。但由于微生物抽提法工艺复杂、生产成本高,发酵法转化率低、发酵副产物多、提取精制困难等,因此这两种方法都不适于海藻糖的大规模工业化生产。通过酶催化生产海藻糖,产物成分明确,分离工艺相对简单,可有效提高海藻糖得率。目前,酶催化生产海藻糖的途径主要有5种[4],其中TreS法生产路线最为简单,TreS以麦芽糖为底物一步催化生成海藻糖。这种方法的关键在于TreS需具有高转化率和强的温度、pH值耐受性。但是TreS法也存在转化率低,温度、pH耐受性差,副产物得率高等缺点[1]。
通过对酶催化机理的深入了解可对酶分子的改造做出理性指导。因此,对TreS催化机理的深入研究十分重要。作者综述了近年来关于TreS结构和功能的研究进展,总结了TreS的结构特点和部分已知的催化机理。
1 海藻糖合成酶的酶学性质
1996年,Nishimoto等[5-6]率先从脂肪杆菌(Pimelo-bactersp.R48)和水生栖热菌(ThermusaquaticusATCC33923)中分离得到了TreS,并初步研究了其酶学特性。此后,更多不同来源的TreS被异源表达。来源于不同菌的TreS的基本性质见表1。
表1不同来源的TreS信息统计
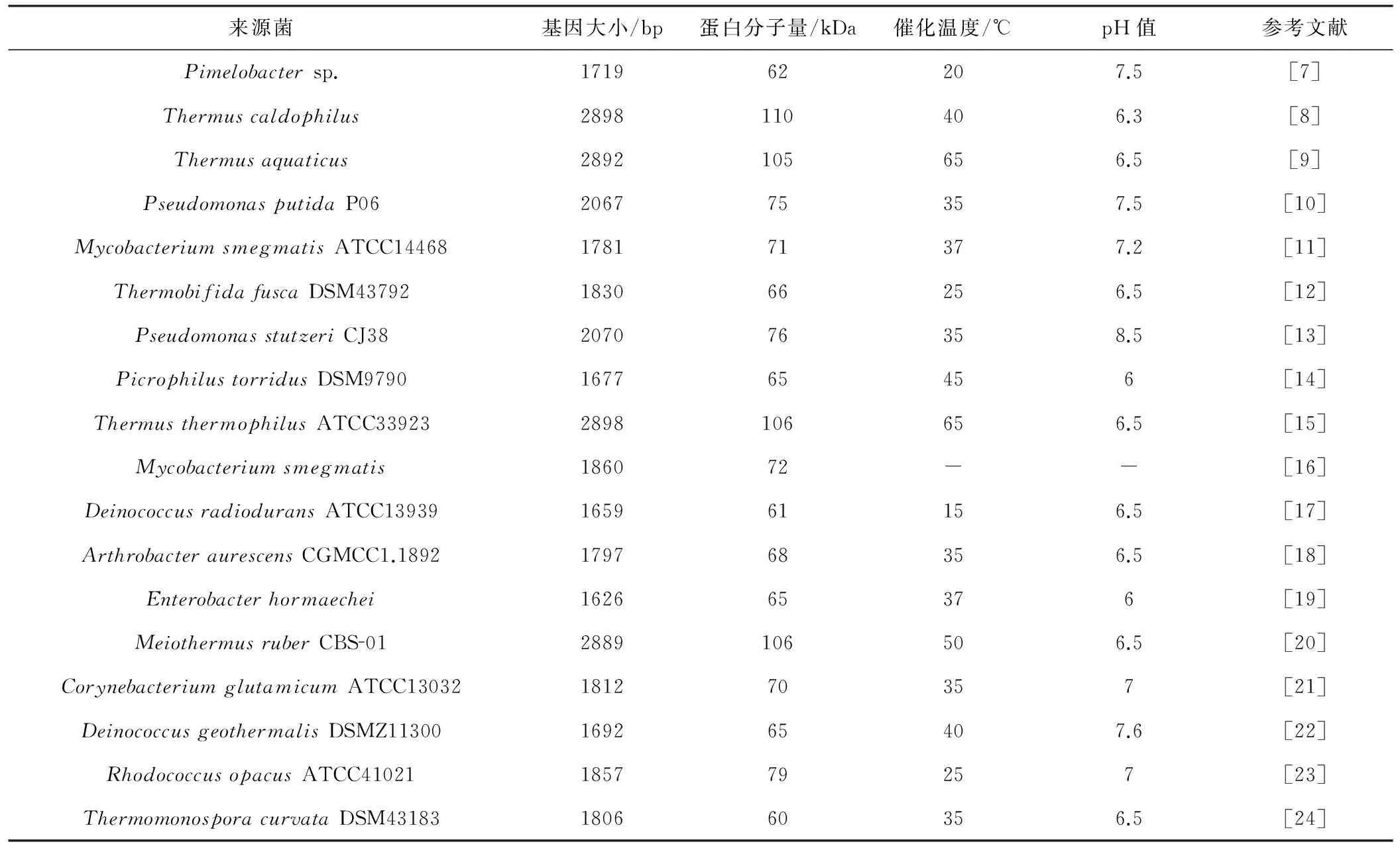
Tab.1 The information statistics of trehalose synthase from various bacteria
由表1可以发现,从基因的大小来看,来自嗜热菌的TreS基因的核苷酸数在2 800个左右,而来自常温菌的TreS基因的核苷酸只有1 800个左右。而且分子量大小与TreS的性质有直接的关系。一般而言,分子量相对较大的TreS最适反应温度都在45 ℃以上,分子量小的TreS通常在20~35 ℃之间。
TreS在溶液中呈活性状态时,通常都是以多聚体的形式存在。来源于弯曲高温单孢菌(Thermomonosporacurvata)的TreS在活性状态下是由3个大小为60 kDa的单体组成的同源三聚体蛋白[4];来源于嗜热放线菌(Thermobifidafusca)的TreS在活性状态下是由4个大小为61 kDa的单体组成的同源四聚体蛋白[25];来源于耻垢分枝杆菌(Mycobacteriumsmegmatis)的TreS在活性状态下是由6个大小约为400 kDa的单体组成的同源六聚体蛋白[11];Wang等[26]发现来源于抗辐射奇异球菌(Deinococcusradiodurans)的TreS在活性状态下是分子量约为120 kDa的同源二聚体蛋白;Roy等[27]发现来源于结核杆菌(Mycobacteriumtuberculosis)的TreS在活性状态下是同源四聚体蛋白。虽然不同TreS在活性状态下的聚合形式、单体分子量大小都不一样,但是它们的生物学功能都是一样的。
2 海藻糖合成酶结构研究进展
2.1 TreS已解析结构统计
目前,已经有来源于3种不同菌株分别包括母体、突变体和复合物的TreS结构,通过X-射线衍射法被解析出来,已解析出的TreS结构的基本信息统计见表2。
表2 TreS结构信息统计
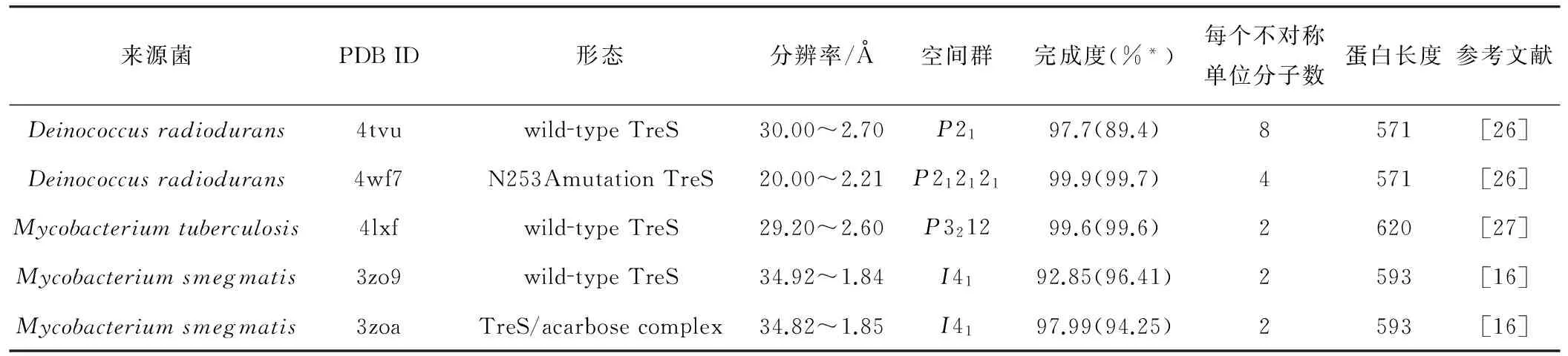
Tab.2 The information statistics of trehalose synthase structure
注:*括号内数值为最高分辨率壳层的数据。
2.2 TreS的结构特点
已解析出的TreS结构,从宏观上主要被分为4个部分,分别命名为结构域A、B、C、D(图1)。正如GH13家族其它的酶一样,每个TreS单体主要包括一个catalytic(β/α)8barrel组成的结构域D,一个C-terminalsandwich (大约有90个氨基酸残基)组成的结构域C[16,26]。结构域D是由(β/α)8barrel组成的核心区域,TreS的活性中心就在这块区域中[16,26];结构域 C是由2个反向平行的β-sheets和2个β-strands组成的,这个区域通过一个由氢键和疏水作用力形成的广泛的相互作用网络紧紧地与(β/α)8barrel连接。结构域C相对来说有其特色,例如:DrTS和MtTS有50%的序列相似性,但它们在结构域C仅有25%的序列相似性[26]。
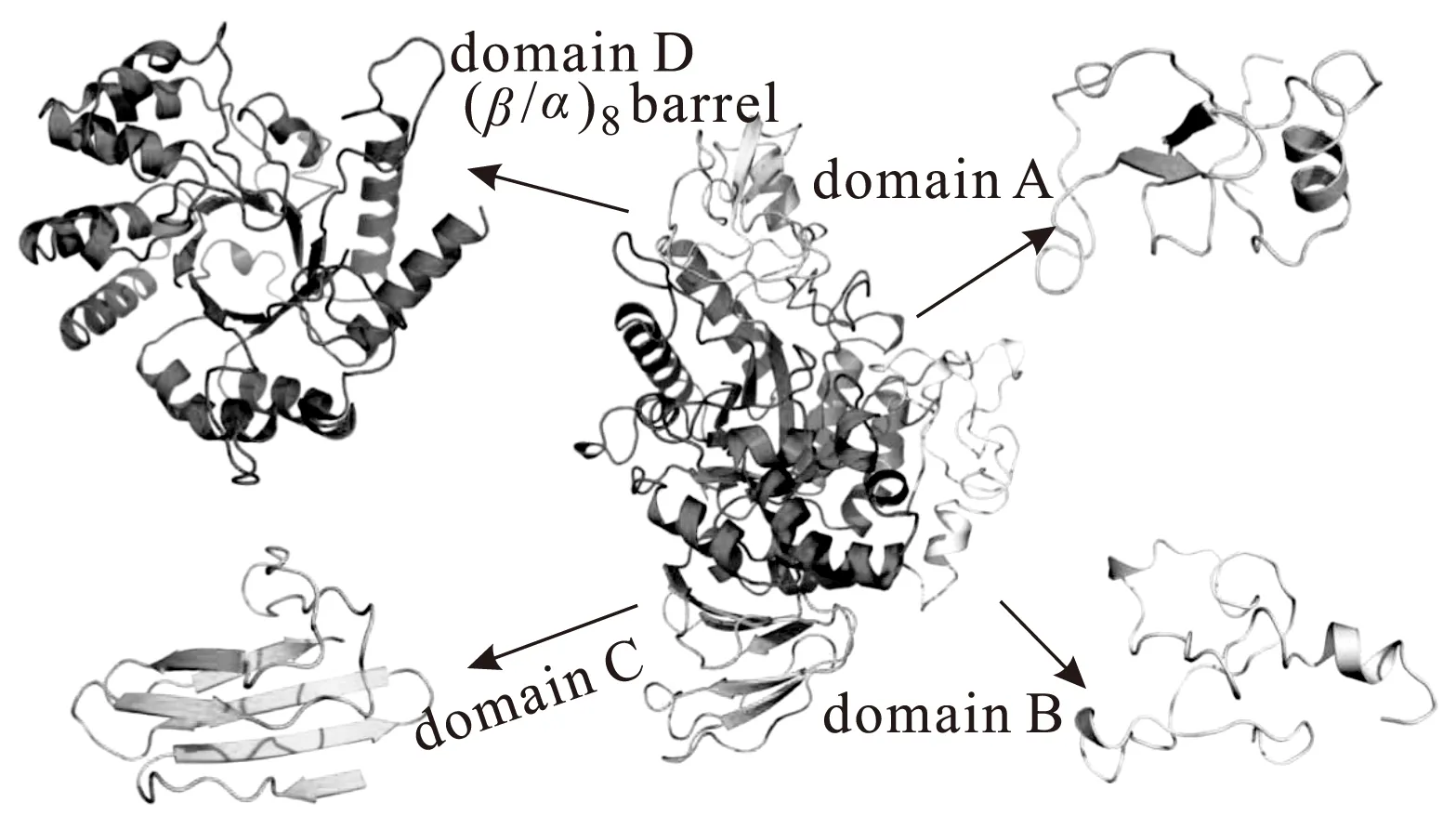
图1 TreS的晶体结构Fig.1 Crystal structure of trehalose synthase
另外两个结构域的形成是通过TreS结构内部α螺旋、β折叠等和(β/α)8barrel之间的挤压盘绕形成的,它们分别被称为结构域A和B。这两个结构域都是loop-rich结构,因此结构柔性相对较大。结构域A(大约含有80个氨基酸残基)与GH13家族的酶一样,由一个短的α螺旋和2个或3个反向平行的β-strands[16,26]组成。结构域A与(β/α)8barrel有着强烈的相互作用,它们之间能够形成10~20个氢键和大量的疏水作用力。另一方面,结构域B在TreS中是比较特别的,其中包括一个短的α螺旋,除α螺旋外其它部分没有表现出明显的二级结构,呈无规则的loop状态。结构域D不但包括(β/α)8barrel还包括一个催化区域,催化区域的组成包括催化三联体Asp-Glu-Asp 和许多保守的底物结合残基[26,28]。结构域B和C与二聚体的形成有关。结构域A中的一部分loop区域对于活性部位的敞开和闭合充当了一个可开关的“门”的作用[26]。
另外,GH13家族的许多酶都包含金属离子,Kobayashi等[29]发现金属离子的参与可能涉及到维持酶的结构稳定,而且已经发现来源于人类肠道菌Bacteroidesthetaiotaomicron的α-淀粉酶和MsTS都存在一个镁离子和一个钙离子[30]。例如:Wang等[26]在DrTS中发现有两个相似的可以结合镁离子的位点,镁离子的结合位点包括Asp24、Asp26、Asp28、Asp32的侧链和Lys30的主链O原子,形成了这样一个序列DXNXDGXGD。在一些其它的GH13家族成员中同样发现了这样一个位置,例如在Rhizobiumsp.中的MX-5Sucrose isomerase和酵母中的isomaltase就有相似的钙离子结合部位[31-32]。另一方面,钙离子的结合部位会被结合位点上的部分氨基酸残基(Asn、Asp、Glu、Tyr、Leu)调整。许多α-淀粉酶都有一个相似的钙离子结合位点,这个位点都会有一个保守的Asp残基[33]。
2.3 TreS底物结合及构象的变化
根据部分已解析的GH13家族酶的晶体结构发现:底物结合模块插入到(β/α)8barrel中,其移动可以覆盖甚至封闭活性部位的入口[26]。虽然NpAS的结构与XaSH的序列一致性只有36%,但当它们结合Tris、葡萄糖、蔗糖和麦芽七糖时,却能够表现出一个相似的构象[34]。然而,Skov等[35]发现apoD.radioduransAS(DrAS)表现出了一个显著不同的构象,它有着一个更加开放的活性位点。此外,Ravaud等[31]发现RhSI结合Tris、葡萄糖、蔗糖和抑制剂时,表现出了一个相似的封闭结构,ScIM的晶体结构也表现出了一个几乎封闭的构象,其底物与活性位点的结合机制目前还不清楚[32]。由于ScIM与RhSI有38%的序列一致性和高的结构同源性,推测它们形成这样的封闭构象有可能跟结构域A和底物结合模块有关,特别是结构域A和(β/α)8barrel中靠近活性中心的两段loop,这两段loop可能涉及到活性位点的开与关[26]。
Wang等[26]通过结构比对发现,DrTS、MsTS和MtTS的晶体结构显示出不同酶的结构差异,主要是结构域之间的相对方向不同。但是在XaS-sucrose、NpAS-maltoheptaose和DrTS-Tris的复合物结构中结构域A的相对方向是一致的,特别是在活性中心正上方的一段loop都有着一个相似的空间位置,而且apo XcSH、apo DrAS、MsTS和MtTS的结构域A也有一个相似的走向,可能是因为TreS与这些酶有较高的结构同源性,特别是相似的活性部位和结构域A。
在GH13家族的很多酶中已经证实,Tris作为一个竞争性抑制剂能够抑制蔗糖与TreS的结合[31,34],其抑制效果也已经被证实[1,17]。例如Wang等[26]在DrTS-Tris复合物中发现,活性中心附近的残基之间形成盐桥和氢键封闭了活性中心的入口,结构域A中的3个氨基酸(Ile150、Phe151、Phe173)和S7中的4个氨基酸(His318、Asp319、Glu320、Glu324)都构成活性口袋的一部分[26],这意味着底物与这些残基的相互作用将会使这两个子域相互靠近形成一个关闭的构象。Wang等还证明了DrTS野生型和N253A突变体的晶体结构除了Glu324的侧链走向外基本上是完全相同的。在N253A突变体中,Asn253和Glu324相互作用的破裂导致Glu324侧链的移动,其侧链的移动是为了给活性中心创造一个小的溶剂进入的孔洞。这就是N253A突变体有约11%的异构酶活性和约180%的水解活性的原因。与N253A相似,R148A突变体表现为约12%的异构酶活性和约150%的水解活性,这表明当148位的Arg被Ala代替时,也有可能形成了一个小的可以让水分子进入的孔洞[26]。
2.4 TreS与小分子复合物结构
Roy等[27]解析出了M.tuberculosisTreS的晶体结构。通过超速离心分析方法得知此酶在溶液中是形成四聚体,通过X-ray小角散射发现,TreS连同Pep2形成了一个异八聚体的络合物,而且Roy等证明了这个络合物的形成能够显著地提高Pep2的麦芽激酶的活性,此络合物的生成可能在糖基转移酶GlgE途径中充当一个调控机制,它的存在能够避免代谢途径中中间产物毒素的积累。
Pan等[36]发现TreS表现出α-淀粉酶的活性,能被α-葡糖苷酶的抑制剂阿卡波糖有效地竞争性抑制。Cancer等[16]获得了分辨率为1.84 Å的TreS-acarbose复合物结构,出人意料的是,阿卡波糖并没有结合在TreS的活性部位上,而是结合在了离TreS活性部位大约40 Å远的一个界限清晰的表面口袋上(图2a),阿卡波糖形成一个弯曲的构象(图2b)。在结合部位内有一个由Trp476、 Met480、 Phe577、Trp579这几个氨基酸构成的一个疏水区域负责与阿卡波糖的结合。Laskowski等[37]分析表明,结合口袋是由位于C端结构域与(β/α)8barrel中的1个α螺旋之间相邻的13个氨基酸残基组成的,而且配体在连接时形成了6个氢键。另外,蛋白结合界面覆盖面积为505 Å2,或者说是覆盖面积占了阿卡波糖分子总面积的63%。通过这个复合物结构的获得也说明了以前的一些猜测:许多GH13家族成员都有这样一个区域作为碳水化合物结合模块。事实上,在糖苷水解酶中这种辅助的模块对于碳水化合物的连接是非常常见的[38]。
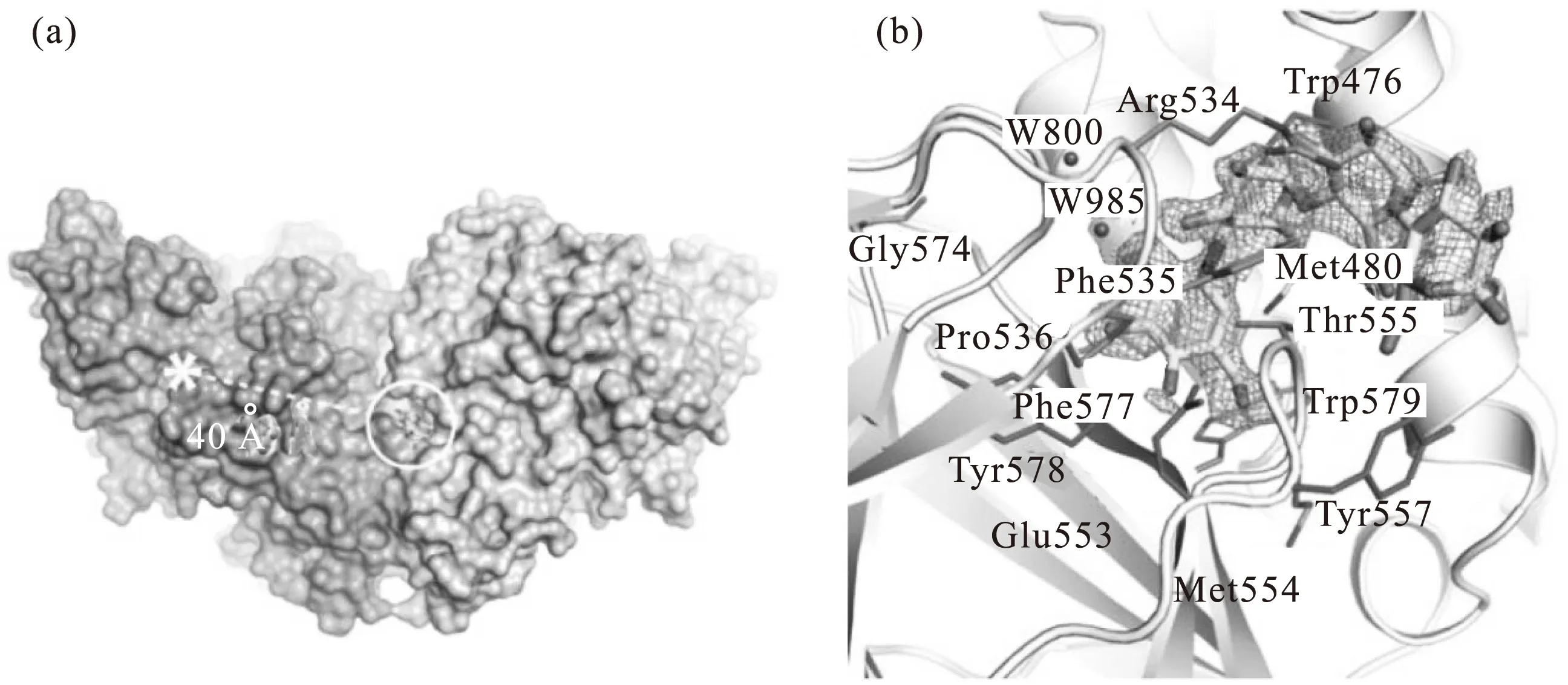
a.阿卡波糖与TreS的结合位点离活性中心有40 Å的距离 b.阿卡波糖与TreS结合部位的详细结构
3 海藻糖合成酶催化作用机理研究进展
TreS属于糖基水解酶GH13家族,一个典型的(β/α)8桶状催化结构域是该家族酶共有的结构特征[39],研究者几乎一致认为它们的催化机制是相似的。
Nishimoto等[9]证明TreS以麦芽糖为底物催化生成海藻糖的过程不是分子间的反应,而是一个分子内的两个单糖的重排过程。Koh[40]用放射性同位素标记的方法,通过分析反应物和不同产物的组成,进一步说明了TreS催化的反应是一种分子内的转糖基反应。在酶分子内先是麦芽糖的α-1,4-糖苷键断裂生成葡萄糖和葡萄糖基,然后再形成α-1,1-糖苷键,从而生成海藻糖,而在第二步水分子作为亲和试剂攻击葡萄糖基时会生成副产物葡萄糖。Wang等[26]在研究DrTS时发现,此酶狭窄且封闭的活性中心区域能够在水中保护葡萄糖基酶中间物不被水解,而且为了完成分子内异构化反应会在活性位点把双糖分解为葡萄糖或果糖,最后重新攻击共价的中间物。
Chen等[14]研究来自P.torridus的TreS时发现His106、Asp203、Glu245、His310、Asp311等5个氨基酸残基与酶的催化活性紧密相关。通过氨基酸序列比对发现,这5个氨基酸在TreS和部分GH13家族酶中是高度保守的,如分别来自T.curvata、M.smegmatis、D.radiodurans、Pimelobactersp.R48、T.Aquaticus等的TreS。当把这5个氨基酸分别突变后,突变体酶活几乎完全丧失。Zhang等[28]通过分析研究来自M.smegmatis的TreS机制,发现其与糖基水解酶家族有着一个相似的两步双取代机制(two-step,double-displacement mechanism),并发现在M.smegmatis中与上述相同的5个保守氨基酸中,Asp230是作为催化亲核试剂(catalytic nucleophile),Glu272是常规的酸/碱催化(general acid/base catalyst),His341和Asp342是保守的氨基酸(conserved carboxylic acid)。
4 展望
通过TreS生产海藻糖是当前海藻糖工业化生产的有效且廉价的途径。由于TreS生产海藻糖所体现出来的优势,以及TreS本身性质的部分缺陷,最近几年对该酶的结构和功能的研究逐渐深入。目前,TreS的结构和功能的研究已经取得了一定的进展和成果,但还有诸多问题有待于解决,如在酶的反应过程中糖苷键的断裂和生成的具体机制还有待确定等。我们相信,随着对TreS结构的了解和研究的逐渐深入,对此酶作用机制的详尽阐明,将为该酶的后续改造提供理论基础,以期达到提高酶的热稳定性、提高海藻糖的转化率和降低副产物葡萄糖得率的目的,从而为TreS的工业化应用创造更好的条件。
[1] JIANG L,LIN M,ZHANG Y,et al.Identification and characterization of a novel trehalose synthase gene derived from saline-alkali soil metagenomes[J].PLoS One,2013,8(10):e77437.
[2] TSUSAKI K,NISHIMOTO T,NAKADA T,et al.Cloning and sequencing of trehalose synthase gene fromThermusaquaticusATCC33923[J].Biochimica et Biophysica Acta-General Subjects,1997,1334:28-32.
[3] 余浪.海藻糖的研究进展及其在食品中的应用[J].农产食品科技,2007,1(4):54-58.
[4] LIANG J Y,HUANG R B,HUANG Y,et al.Cloning,expression,properties,and functional amino acid residues of new trehalose synthase fromThermomonosporacurvataDSM43183[J].Journal of Molecular Catalysis B-Enzymatic,2013,90:26-32.
[5] NISHIMOTO T,NAKANO M,NAKADA T,et al.Purification and properties of a novel enzyme,trehalose synthase fromPimelobactersp.R48[J].Bioscience Biotechnology and Biochemistry,1996,60(4):640-644.
[6] NISHIMOTO T,NAKADA T,CHAEN H,et al.Purification and characterization of a thermostable trehalose synthase fromThermusaquaticus[J].Bioscience Biotechnology and Biochemistry,1996,60(5):835-839.
[7] NISHIMOTO T,NAKANO M,IKEGAMI S,et al.Existence of a novel enzyme converting maltose into trehalose[J].Bioscience Biotechnology and Biochemistry,1995,59:2189-2190.
[8] KOH S,SHIN H J,KIM J S,et al.Trehalose synthesis from maltose by a thermostable trehalose synthase fromThermuscaldophilus[J].Biotechnology Letters,1998,20:757-761.
[9] NISHIMOTO T, NAKADA T, CHAEN H, et al. Action of a thermostable trehalose synthase from Thermus aquaticus on sucros[J]. Bioscience Biotechnology and Biochemistry, 1997, 61(5): 898-899.
[10] SU J,WANG T,MA C,et al.Homology modeling and function of trehalose synthase fromPseudomonasputidaP06[J].Biotechnology Letters,2014,36:1009-1013.
[11] PAN Y T,EDAVANA V K,JOURDIAN W J,et al.Trehalose synthase ofMycobacteriumsmegmatis:purification,cloning,expression,and properties of the enzyme[J].European Journal of Biochemistry,2004,271:4259-4269.
[12] WEI Y T,ZHU Q X,LUO Z F,et al.Cloning,expression and identification of a new trehalose synthase gene fromThermobifidafuscagenome[J].Acta Biochimica et Biophysica Sinica,2004,36:477-484.
[13] LEE J H,LEE K H,KIM C G,et al.Cloning and expression of a trehalose synthase fromPseudomonasstutzeriCJ38 inEscherichiacolifor the production of trehalose[J].Applied Microbiology and Biotechnology,2005,68:213-219.
[14] CHEN Y S,LEE G C,SHAW J F,et al.Gene cloning,expression,and biochemical characterization of a recombinant trehalose synthase fromPicrophilustorridusinEscherichiacoli[J].Journal of Agricultural and Food Chemistry,2006,54:7098-7104.
[15] WANG J H,TSAI M Y,CHEN J J,et al.Role of the C-terminal domain ofThermusthermophilustrehalose synthase in the thermophilicity,thermostability,and efficient production of trehalose[J].Journal of Agricultural and Food Chemistry,2007,55:3435-3443.
[16] CANCER S,NGUYEN N,AGUDA A,et al.The structure of theMycobacteriumsmegmatistrehalose synthase reveals an unusual active site configuration and acarbose-binding mode[J].Glycobiology,2013,23(9):1075-1083.
[17] FILIPKOWSKI P,PIETROW O,PANEK A,et al.Properties of recombinant trehalose synthase fromDeinococcusradioduransexpressed inEscherichiacoli[J].Acta Biochimica Polonica,2012,59:425-431.
[18] WU X L,DING H B,YUE M.Gene cloning,expression,and characterization of a novel trehalose synthase fromArthrobacteraurescens[J].Applied Microbiology and Biotechnology,2009,83:477-482.
[19] YUE M,WU X L,GONG W N,et al.Molecular cloning and expression of a novel trehalose synthase gene fromEnterobacterhormaechei[J].Microbial Cell Factories,2009,8:34-41.
[20] ZHU Y M,WEI D S,ZHANG J,et al.Overexpression and characterization of a thermostable trehalose synthase fromMeiothermusruber[J].Extremophiles,2010,14:1-8.
[21] KIM T K,JANG J H,CHO H Y,et al.Gene cloning and characterization of a trehalose synthase fromCorynebacteriumglutamicumATCC13032[J].Food Science and Biotechnology,2010,19:565-569.
[22] PAWEL F,ANNA P,FELCZYKOWSKA A,et al.Expression ofDeinococcusgeothermalistrehalose synthase gene inEscherichiacoliand its enzymatic properties[J].African Journal of Biotechnology,2012,11(67):13131-13139.
[23] YAN J,QIAO Y,HU J,et al.Cloning,expression and characterization of a trehalose synthase gene fromRhodococcusopacus[J].The Protein Journal,2013,32:223-229.
[24] STAM M R,DANCHIN E G J.Dividing the large glycoside hydrolase family 13 into subfamilies:towards improved functional annotations ofα-amylase-related proteins[J].Protein Eng Des Select,2006,19(12):555-562.
[25] 韦传东.海藻糖合成酶基因(TreS)的融合表达、纯化以及酶学性质研究[D].南宁:广西大学,2006.
[26] WANG Y L,CHOW S Y,LIN Y T,et al.Structures of trehalose synthase fromDeinococcusradioduransreveal that a closed conformation is involved in catalysis of the intramolecular isomerization[J].Acta Crystallogr D Biol Crystallogr,2014,70:3144-3154.
[27] ROY R,USHA V,KERMANI A,et al.Synthesis ofα-glucan inMycobacteriainvolves a hetero-octameric complex of trehalose synthase TreS and maltokinase Pep2[J].ACS Chem Biol,2013,8(10):2245-2255.
[28] ZHANG R,PAN Y T,HE S,et al.Mechanistic analysis of trehalose synthase fromMycobacteriumsmegmatis[J].Journal of Biological Chemistry,2011,286:35601-35609.
[29] KOBAYASHI M,HONDOH H,MORI H,et al.Calcium ion-dependent increase in thermostability of dextran glucosidase fromStreptococcusmutans[J].Bioscience Biotechnology and Biochemistry 2011,75(8):1557-1563.
[30] KOROPATKIN N M,SMITHM T J.SusG:a unique cell-membrane-associated alpha-amylase from a prominent human gut symbiont targets complex starch molecules[J].Structure,2010,18(2):200-215.
[31] RAVAUD S,ROBERT X,WATZLAWICK H,et al.Trehalulose synthase native and carbohydrate complexed structures provide insights into sucrose isomerization[J].Journal of Biological Chemistry,2007,282(38):28126-28136.
[32] YAMAMOTO K,MIYAKE H,KUSUNOKI M,et al.Crystal structures of isomaltase fromSaccharomycescerevisiaeand in complex with its competitive inhibitor maltose[J].FEBS Journal,2010,277(20):4205-4214.
[33] JANECEK S,SVENSSON B,MACGREGOR E A.α-Amylase:an enzyme specificity found in various families of glycoside hydrolases[J].Cellular and Molecular Life Science,2014,71(7):1149-1170.
[34] SKOV L K,MIRZA O,SPROGØE D,et al.Oligosaccharide and sucrose complexes of amylosucrase.structural implications for the polymerase activity[J].Journal of Biological Chemistry,2002,277(49):47741-47747.
[35] SKOV L K,PIZZUT-SERIN S,REMAUD-SIMEON,et al.The structure of amylosucrase fromDeinococcusradioduranshas an unusual open active-site topology[J].Acta Crystallographica,2013,69(Pt9):973-978.
[36] PAN Y T,CARROLL J D,ASANO N,et al.Trehalose synthase converts glycogen to trehalose[J].FEBS Journal,2008,275(13):3408-3420.
[37] LASKOWSKI R A,SWINDELLS M B.LigPlot+:multiple ligand-protein interaction diagrams for drug discovery[J].Journal of Chemical Information and Modeling,2011,51(10):2778-2786.
[38] JANECEK S,SVENSSON B,MACGREGOR E A.Relation between domain evolution,specificity,and taxonomy of the alpha-amylase family members containing a C-terminal starch-binding domain[J].European Journal of Biochemistry,2003,270(4):635-645.
[39] MACGREGOR E A,JANECEK S,SVENSSON B.Relationship of sequence and structure to specificity in the α-amylase family of enzymes[J].Biochimica et Biophysica Acta-Protein Structure and Molecular Enzymology,2001,1546:1-20.
[40] KOH S.Mechanistic study of the intramolccular conversion of maltose to trehalose byThermuscaldophilusGk24 trehalose synthase[J].Carbohydrate Research,2003,338(12):1339-1343.
Research Progress in Structure and Function of Trehalose Synthase
WANG Dong-sheng1,GENG Qiang2,ZHOU Jia-hai3,JIANG Ling2*,HUANG He4
(1.CollegeofBiotechnologyandPharmaceuticalEngineering,NanjingTechUniversity,Nanjing210009,China;2.CollegeofFoodScienceandLightIndustry,NanjingTechUniversity,Nanjing210009,China;3.StateKeyLaboratoryofBioorganicandNaturalProductsChemistry,ShanghaiInstituteofOrganicChemistry,ChineseAcademyofSciences,Shanghai200032,China;4.CollegeofPharmacy,NanjingTechUniversity,Nanjing210009,China)
Trehalose synthase can catalyze maltose into trehalose in one step by intermolecular transglycosylation.We summarized the latest research progress of structure and function of trehalose synthase,and enumerated trehalose synthase which had been cloned and expressed,and their enzymatic properties.We also summarized and compared the structures and catalytic mechanisms of several trehalose synthases which had been resolved,and pointed out the importance of in-depth study of trehalose synthase to the industrial production of trehalose.
trehalose synthase;crystal structure;catalytic mechanism
中国科学院上海生命有机化学国家重点实验室开放基金项目(SKLBNPC15429),国家自然科学基金青年基金项目(21506101),国家自然科学基金联合基金项目(U1603112),江苏省第十二批六大人才高峰计划项目(2015-JY-009),2016年度省级环保科研课题(2015053)
2017-03-08
王东生(1996-),男,安徽亳州人,硕士研究生,研究方向:发酵工程和酶工程;通讯作者:江凌,博士,副教授,E-mail:jiangling@njtech.edu.cn。
10.3969/j.issn.1672-5425.2017.07.002
Q784
A
1672-5425(2017)07-0006-06
王东生,耿强,周家海,等.海藻糖合成酶结构和功能的研究进展[J].化学与生物工程,2017,34(7):6-11,16.
