2010—2012年广州地区人冠状病毒分子进化分析
2017-07-24庹玖玲黄旭斌杨权罗洪娇周凯张素粉张甜曹开源徐霖
庹玖玲 黄旭斌 杨权 罗洪娇 周凯 张素粉 张甜 曹开源 徐霖
510080 广州,中山大学热带病防治研究教育部重点实验室,中山大学香港大学粤港传染病监测联合实验室(庹玖玲、罗洪娇、周凯、张素粉、张甜、曹开源、徐霖);510080 广州,中山大学附属第一医院呼吸科ICU室(黄旭斌);511436 广州,广州医科大学基础学院病原生物学与免疫学教研室(杨权)
·论著·
2010—2012年广州地区人冠状病毒分子进化分析
庹玖玲 黄旭斌 杨权 罗洪娇 周凯 张素粉 张甜 曹开源 徐霖
510080 广州,中山大学热带病防治研究教育部重点实验室,中山大学香港大学粤港传染病监测联合实验室(庹玖玲、罗洪娇、周凯、张素粉、张甜、曹开源、徐霖);510080 广州,中山大学附属第一医院呼吸科ICU室(黄旭斌);511436 广州,广州医科大学基础学院病原生物学与免疫学教研室(杨权)
目的 了解2010—2012年广州地区发热呼吸道感染患者的人冠状病毒各亚型分子进化情况。方法 采用RT-PCR法扩增HCoV-OC43、HCoV-229E和HCoV-NL63阳性标本的NP、RdRp和S基因部分片段,构建载体后进行测序,利用 Bio-edit、Mega4.0、Clustal1.83等生物信息学软件对测序成功的基因序列进行比对和分析,建立HCoV-OC43、HCoV-229E和HCoV-NL63不同基因区域的分子进化树。结果 2010—2012年期间,HCoV-OC43、HCoV-229E和HCoV-NL63亚型在广州均未检测到重大变异株或重组株。HCoV-OC43与Belgium和中国香港来源流行株亲缘关系较近(GenBank登录号 JN129834和AY903460);HCoV-229E与Amsterdam来源的病毒株亲缘关系最近(GenBank登录号JX503060);HCoV-NL63与中国北京和Amsterdam来源的流行株亲缘关系较近(GenBank登录号JX104161和DQ445911)。各亚型NP和RDRp为保守基因,S基因变异较大。结论 2010—2012年期间,广州至少存在3种HCoV-OC43、HCoV-229E和HCoV-NL63流行株,没有发现重大变异株或重组株。各亚型NP和RDRp均为保守基因,可用于病毒的检测,S基因变异较大,适用于对分子变异规律的研究。
Fund program: National Major Projects of Major Infectious Disease Control and Prevention, the Ministry of Science and Technology of the People's Republic of China(2012ZX10004-213)
冠状病毒(Coronavirus, CoV)是引起呼吸道感染的主要病原体之一[1],是目前已知的基因组最大的RNA病毒,其基因组全长约27~32 kb,为带帽的多聚寡核苷酸。所有的冠状病毒都具有相似的基因组结构:从5′到3′端依次为RNA-dependent RNA polymerase gene(RNA依赖性RNA聚合酶基因)、spike gene(S)、envelope gene(E)、membrane gene(M)和nucleocapsid gene(N)[2,3]。目前发现的可感染人的冠状病毒(Human coronavirus, HCoV)主要有6个亚型: HCoV-OC43、HCoV-229E、HCoV-NL63、SARS-CoV、HCoV-HKU1和MERS-CoV,流行分布于世界各地。冠状病毒感染能引起包括人和动物的呼吸道、胃肠系统和神经系统等多种疾病,宿主范围广,且基因突变率高,可能从动物传至人类。2002~2003年SARS-CoV引起的急性呼吸窘迫综合征(SARS)在中国广东地区暴发;2012年9月起,中东地区暴发来源不明的新型冠状病毒MERS-COV[4,5]。这两种新型HCoV均具有高致病性,可引起下呼吸道感染,死亡率高,揭示了冠状病毒可作为潜在的对人类危害性极高的传染病暴发源。由于新的突变病毒具有不可预见性,并且有可能具有高致病性,一旦传入人群,将造成严重的威胁。因此,对HCoV进行持续监测和分子进化变异分析,寻找其变异规律,对于传染病的防控和预警具有重大意义。本研究对2010年7月到2012年12月广州市人冠状病毒主要流行亚型的分子进化特征进行了分析,为广州地区人冠状病毒进化变异规律的深入研究提供重要参考依据。
1 材料与方法
1.1 临床标本 收集广州市2010年7月到2012年12月传染病重大专项华南监测实验室5家哨点医院中山大学附属第二医院、中山大学附属第三医院、广州医学院附属第一医院、广东省中医院、华侨医院符合要求的3 376例发热呼吸道感染患者咽拭子标本。患者纳入标准:发热≤3 d,体温≥38 ℃,同时伴有咳嗽,流涕或咽喉痛等其他呼吸道感染症状。采集的标本置于病毒转运液(VTM,含Earle's平衡盐溶液,4.4%碳酸氢钠,5%牛血清白蛋白,万古霉素100 μg/ml,阿米卡星30 μg/ml和制霉菌素40 U/ml)中,4 ℃~8℃条件下标本于24 h内运送至传染病监测技术平台华南监测实验室,离心取上清分装后于-80 ℃保存备用。经Real-time PCR鉴定为HCoV阳性,且相应亚型阳性的标本用于此次分子进化研究[6]。
1.2 试剂 病毒核酸提取试剂盒QIAmpMiniElute Virus Spin购于德国Qiagen公司;逆转录试剂盒Superscript Ⅲ 购自Invitrogen公司;扩增人冠状病毒各亚型基因的Premix Ex Taq® Version 2.0和pMD®18-T Vector购自宝生物工程(大连)有限公司;扩增引物由Invitrogen公司合成;用于DNA回收纯化的胶回收试剂盒购自北京百泰克生物技术有限公司。1.3 病毒核酸提取 病毒核酸提取步骤严格按照试剂盒说明书进行,提取的RNA立即用于后续实验或冻于-80 ℃冰箱保存备用。
1.4 逆转录 使用随机引物将病毒RNA逆转录成cDNA,用于后续的PCR扩增。操作步骤严格按照试剂盒说明书进行。合成的 cDNA立即用于后续实验或保存于-20 ℃冰箱备用。
1.5 各亚型基因扩增 根据GenBank中HCoV亚型OC43、229E和NL63的 NP、RDRp及S基因序列分别设计引物,引物序列见表1。采用Premix Ex Taq(TaKaRa),以病毒cDNA为模板进行PCR扩增反应,反应体系:Premix Ex Taq 10 μl,引物上游 1 μl(10 μmol/L),引物下游1 μl(10 μmol/L),cDNA 2 μl,ddH2O 6 μl。反应程序:95 ℃ 5 min,95°C 30 s,52°C 40 s,72°C 1 min,72°C 10 min,4°C forever,2~4步35个循环。

表1 冠状病毒亚型NP、RdRp及S基因部分序列扩增引物
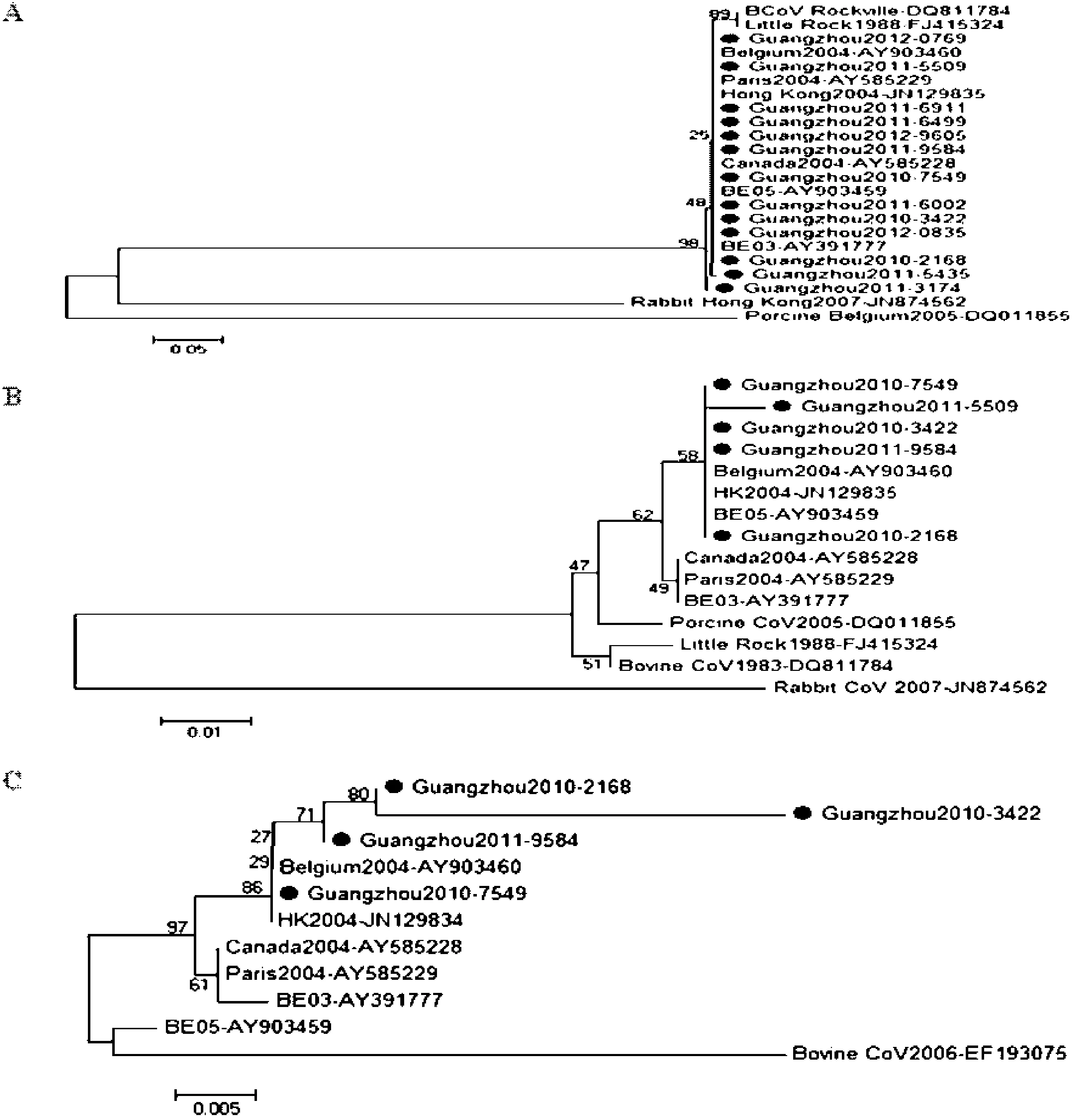
“●”表示 2010—2012年广州HCoV-OC43株图1 HCoV-OC43的NP、RdRp和S基因的系统进化树分析“●”indicates the HCoV-OC43 stains in Guangzhou, 2010—2012Fig.1 Phylogenetic analysis of HCoV-OC43 based on NP, RdRp and S gene partial sequences
1.6 质粒构建和测序 扩增反应产物经2%琼脂糖凝胶电泳检测,回收阳性条带,利用试剂盒进行回收纯化,操作步骤严格按照说明书进行。纯化的产物与pMD18-T Vector于16°C连接1 h,随后转化感受态细胞并培养过夜。挑取单个菌落,PCR鉴定,阳性菌落摇床培养过夜后,取1 ml菌液送至上海英骏公司测序。
1.7 分子进化分析 将测序得到的序列提交GenBank进行Blast相似性比较与同源性分析,并与GenBank中具有代表性的冠状病毒株序列用软件Clustal X进行比对。使用Mega 4.1软件中的邻接法,分别对HCoV-OC43、HCoV-229E和HCoV-NL63的NP、RdRp及S基因构建进化树,分析HCoV各亚型广州株的进化特征。
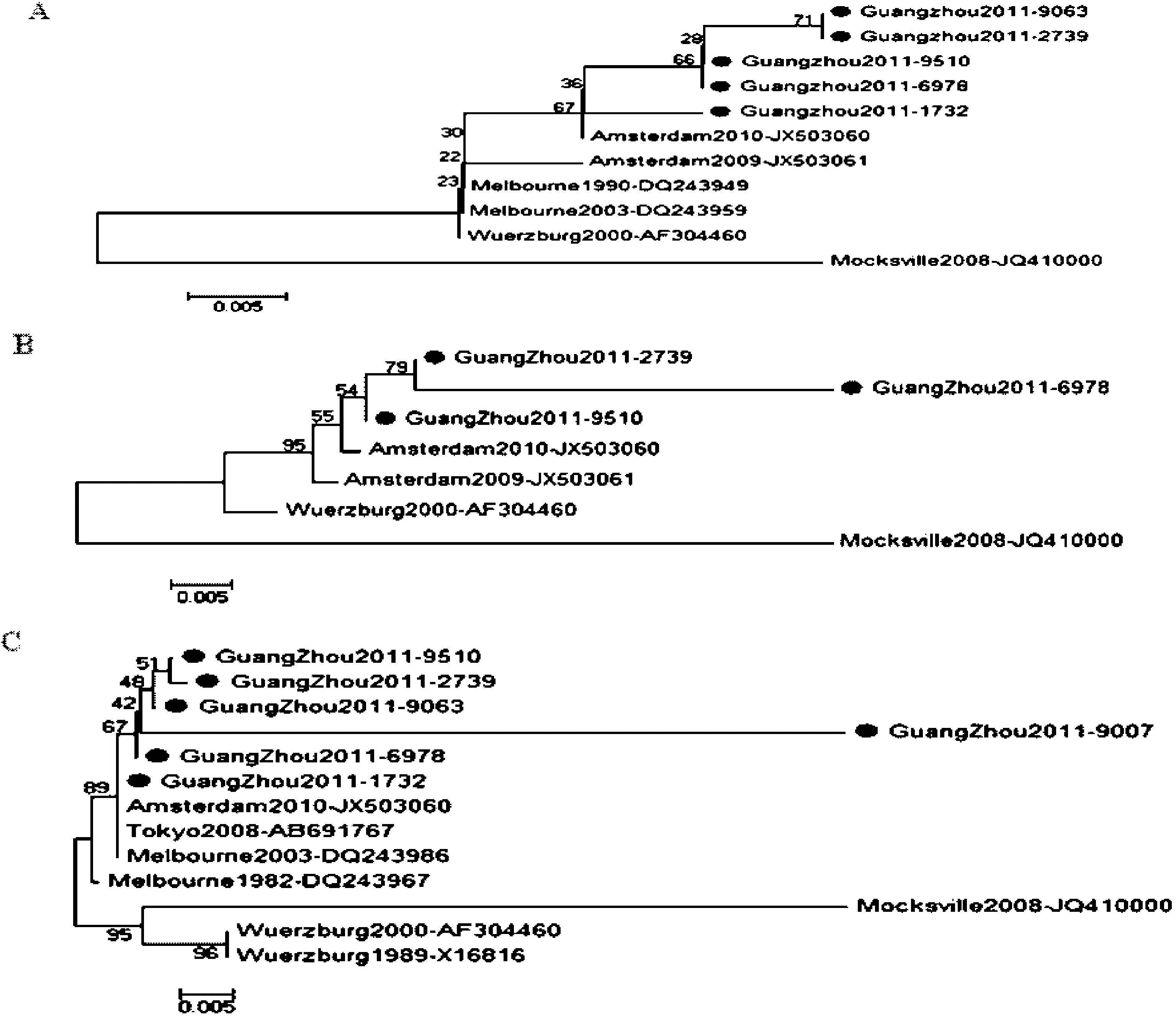
“●”表示 2010—2012年广州HCoV-229E株图2 HCoV-229E的NP、RdRp和S基因的系统进化树分析“●”indicates the HCoV-229E stains in Guangzhou, 2010—2012Fig.2 Phylogenetic analysis of HCoV-229E based on NP, RdRp and S gene partial sequences
2 结果
2.1 HCoV-OC43的NP、RdRp及S基因片段进化树分析 HCoV-OC43各基因的系统进化树分析见图1。结果表明HCoV-OC43的NP和RDRp基因为相对保守基因(图1A、图1B),与国外和中国香港等来源的序列具有很高的同源性,可根据其序列信息设计引物用于病毒的检测;S基因变异率较大(图1C),将其序列扩增进行变异分析可能会发现变异株,并且能更准确的预测病毒的变异规律。从分子进化结果来看,2010—2012年期间广州至少存在3种流行株,其中一株与中国香港Genbank登陆号为JN129834的流行株亲缘性较近,另外一株与Belgium来源Genbank登陆号为AY903460的流行株亲缘性较近,两种流行株都与Canada和Paris来源的流行株亲缘关系较远;Guangzhou2011-3174流行株可能为较大变异株,有必要对其进行深入分析。
2.2 HCoV-229E的NP、RdRp及S基因片段进化树分析 如图2所示,2010—2012年期间广州至少存在3种不同的HCoV-229E流行株,且与Amsterdam 2010年GenBank登陆号为JX503060的流行株亲缘关系最近,与Mocksville来源GenBank登陆号为JQ410000流行株亲缘关系最远。2010—2012年期间广州只有在2011年检测出HCoV-229E的感染,序列的高度相似性提示其可能来源于Amsterdam株。
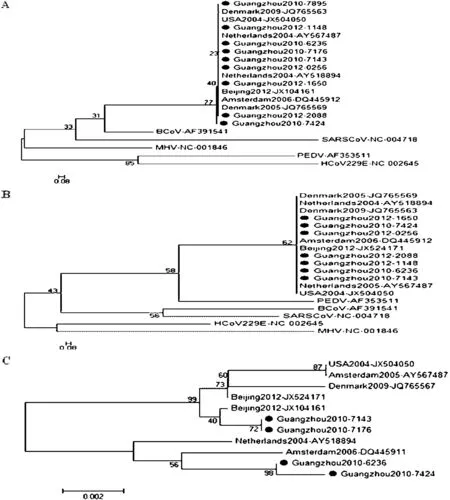
“●”表示 2010—2012年广州HCoV-NL63株图3 HCoV-NL63的NP、RDRp和S基因的系统进化树分析“●”indicates the HCoV-NL63 stains in Guangzhou, 2010—2012Fig.3 Phylogenetic analysis of HCoV-NL63 based on NP, RdRp and S gene partial sequences
2.3 HCoV-NL63的NP、RdRp及S基因片段进化树分析 HCoV-NL63各基因的系统进化树分析见图3。从S基因进化树分析的结果来看,2010—2012年期间广州至少存在3种HCoV-NL63流行株,其中一种流行株与中国北京2012年Genbank登陆号JX104161的流行株亲缘关系较近,另外两株与Amsterdam来源的Genbank陆号DQ445911流行株亲缘关系较近(图3C)。与HCoV-OC43相似,NP和RdRp基因具有高度的保守性(图3A、图3B),与其他地区来源的序列同源性较高,故其序列可用于病毒的检测,S基因则更适用于对病毒进行变异规律分析。
3 讨论
2010—2012年间广州地区HCoV主要流行亚型为OC43、229E和NL63,相应阳性标本的检出分别为23、13和19例(阳性率分别为0.68%,0.39%和0.56%)[6]。为了了解各亚型的分子变异规律,监测是否有重大突变株或重组株出现,本研究采用RT-PCR的方法分别对各型的NP、RdRp和S基因部分序列进行测序并进行系统进化树分析。结果显示,各亚型均存在real-time PCR检测阳性但是PCR扩增阴性的标本,这可能与以下原因有关:(1)标本用于扩增前于-80°C条件下保存了2~3年,期间可能导致某些标本中的病毒核酸降解;(2)标本中病毒核酸含量较少,而real-time PCR灵敏度较高,所以鉴定结果为阳性,但是RT-PCR扩增对病毒核酸的量有一定的要求,低含量的核酸易导致扩增失败;(3)某些病毒株基因序列发生了突变。
分子进化分析结果表明,2010—2012年期间,广州至少存在3种HCoV-OC43流行株、3种HCoV-229E和3种HCoV-NL63流行株。HCoV-OC43与Belgium和中国香港来源流行株亲缘关系较近;HCoV-229E与Amsterdam来源的病毒株亲缘关系最近,由于广州的人员流动性大,国际交流多,故不排除广州所流行的HCoV-229E株来源于Amsterdam株的可能性;HCoV-NL63与中国北京和Amsterdam来源的流行株亲缘关系较近。其中一株HCoV-OC43流行株与其他毒株亲缘关系较远,可能为较大变异株,可对其进行更广泛的基因测序或全基因组测序研究。
广州是我国经济发达城市,也是国际化大都市,人口流动量较大,人员的流动可能是造成广州地区多种毒株流行的原因之一。与Woo等[7]研究结果相似,本研究结果表明,2010—2012年间广州地区主要流行的3种HCoV亚型的NP和RdRp基因都比较保守,与其他来源的毒株同源性很高,而S基因变异均较大。因此,NP和RdRp基因适用于作为病毒的检测的靶基因,而对于病毒变异规律的分析则选择S基因更为合适。与HCoV-229E和HCoV-NL63相比,HCoV-OC43的NP基因和RDRp基因PCR扩增成功率基本一致,但其S基因的扩增成功率较低,这进一步说明了HCoV-OC43的基因突变率要高于HCoV-229E和HCoV-NL63[8],可能是其在2010—2012年期间检出率最高,成流行优势亚型的原因之一。同时,我们对近几年广州地区人冠状病毒的流行病学监测也发现HCoV-OC43的发病率要高于其他亚型[9],因此有必要对OC43亚型的变异进行持续跟踪和进一步的深入研究。
综上所述,2010—2012年间广州地区HCoV主要流行亚型OC43、229E和NL63阳性标本的分子进化分析结果显示,2010—2012年间广州地区HCoV的3种主要流行亚型都至少存在3种流行株,目前未发现重大变异株或重组株,但HCoV-OC43的基因变异性要高于HCoV-229E和HCoV-NL63亚型。各亚型NP和RDRp均为保守基因,可用于病毒的检测,S基因变异较大,适用于对分子变异规律的研究。但是HCoV拥有庞大的基因组,其分子变异规律的深入分析尚需进一步对编码蛋白或全基因组序列进行测定。本研究结果为2010—2012年间广州地区HCoV主要流行亚型的分子流行病学和变异规律的深入研究打下了坚实的基础,为HCoV的防控提供了重要的参考依据。
[1] Osterhaus AD. New respiratory viruses of humans[J]. Pediatr Infect Dis J, 2008,27(10 Suppl): 71-74. doi: 10.1097/INF.0b013e3181684d7c.
[2] Dijkman R, van der Hoek L. Human coronaviruses 229E and NL63: close yet still so far[J]. J Formos Med Assoc, 2009,108(4):270-279. doi: 10.1016/S0929-6646(09)60066-8.
[3] Poon LL, ChanChO KH, Wong K, et al. Detection of SARS coronavirus in patients with severe acute respiratory syndrome by conventional and real-time quantitative reverse transcription-PCR assays[J]. Clin Chem, 2004,50(1):67-72. doi:10.1373/clinchem.2003.023663
[4] Raj VS, Osterhaus AD, Fouchier RA, et al. MERS: emergence of a novel human coronavirus[J]. Curr Opin Virol, 2014,5C:58-62. doi:10.1016/j.coviro.2014.01.010.
[5] 周伯平,陈心春,王火生,等.一种新型冠状病毒基因的克隆和序列分析与传染性非典型肺炎病原学的调查[J]. 中华实验和临床病毒学杂志, 2003,(02):38-40.
[6] 庹玖玲, 徐霖, 关琳琳, 等.2010—2012年广州地区人冠状病毒及其亚型分布[J]。热带医学杂志, 2014,14(1):19-22.
[7] Woo PC, Huang Y, Lau SK, et al. Coronavirus genomics and bioinformatics analysis[J]. Viruses, 2010,2(8):1804-1820. doi: 10.3390/v2081803.
[8] Gaunt ER, Hardie A, Claas EC, et al. Epidemiology and clinical presentations of the four human coronaviruses 229E, HKU1, NL63, and OC43 detected over 3 years using a novel multiplex real-time PCR method[J]. J Clin Microbiol, 2010,48(8):2940-2947. doi: 10.1128/JCM.00636-10.
[9] 张素粉,徐霖,罗虹娇,等.2012-2015年广州地区人冠状病毒及其亚型流行病学特征[J]. 热带医学杂志,2016,04:430-433.
(本文编辑:陈培莉)
Molecular evolution of human coronavirus in Guangzhou from 2010 to 2012
Tuo Jiuling, Huang Xubin, Yang Quan, Luo Hongjiao, Zhou Kai, Zhang Sufen, Zhang Tian, Cao Kaiyuan, Xu Lin
Key Laboratory of Tropical Disease Control, Ministry of Education, Sun Yat-sen University, SunYat-sen University-University of Hong Kong Joint Laboratory of Infectious Disease Surveillance, Guangzhou 510080, China (Tuo JL, Luo HJ, Zhou K, Zhang SF, Zhang T, Cao KY, Xu L); ICU of Pneumology Department, The First Affiliated Hospital, Sun Yat-Sen University, Guangzhou 510080, China (Huang XB); Department of Pathogen Biology & Immunology, School of Basic Sciences, Guangzhou Medical University, Guangzhou 511436, China (Yang Q)
Tuo Jiuling, Email:452069764@qq.com; Xu Lin, Email:xulin@mail.sysu.edu.cn
Objective To investigate the molecular evolution characteristics of human coronavirus (HCoV) subtypes in patients with fever and respiratory tract infection in Guangzhou from 2010 to 2012. Methods Partial fragments of NP, RdRp and S genes of HCoV-OC43, HCoV-229E and HCoV-NL63 positive samples were amplified by RT-PCR and sequencing. Bioinformatics software, including Bio-edit, Mega4.0 and Clustal1.83 were used for comparison and analysis of NP, RDRp and S gene sequences. Molecular evolutionary tree of different gene regions of HCoV-OC43, HCoV-229E and HCoV-NL63 were built. Results No remarkable variation or recombinant strain of HCoV-OC43, HCoV-229E and HCoV-NL63 was found in Guangzhou during 2010—2012. The HCoV-OC43 substrains were genetically closest to the strains found in Belgium and Hong Kong (GenBank accession number JN129834 and AY903460). HCoV-229E substrains were genetically closest to those found in Amsterdam (GenBank accession number JX503060) and HCoV-NL63 most genetically close to those in Amsterdam and Beijing (GenBank accession number JX104161 and DQ445911). The NP and RDRp genes of all subtypes were highly conserved, while S gene was more variable. Conclusions There were at least 3 substrains of HCoV-OC43, HCoV-229E and HCoV-NL63 epidemic in Guangzhou during 2010—2012, and no remarkable variation or recombinant viral strain was found. The NP and RDRp genes of all subtypes were highly conserved and can be used in virus detection, while S gene was more variable and suitable for phylogenetic and variation study.
Respiratory tract infection; Human coronavirus OC43; Human coronavirus 229E; Human coronavirus NL63; Molecular evolution
庹玖玲,Email:452069764@qq.com;徐霖,Email:xulin@mail.sysu.edu.cn
10.3760/cma.j.issn.1003-9279.2017.03.007
呼吸道感染;人冠状病毒OC43;人冠状病毒NL63;人冠状病毒229E;分子进化
国家重大传染病防治科技重大项目(2012ZX10004-213)
2016-08-21)
