N端9个谷氨酸或7个天冬氨酸显著增强普鲁兰酶在大肠杆菌中表达分泌
2017-07-18栗亚美安展飞陈阿娜杨艳坤白仲虎
栗亚美, 安展飞, 陈阿娜, 杨艳坤, 白仲虎
(1. 江南大学 粮食发酵工艺与技术国家工程实验室, 无锡 214122; 2. 江南大学 工业生物技术教育部重点实验室, 无锡 214122; 3.江南大学 糖化学与生物技术教育部重点实验室, 无锡 214122)
N端9个谷氨酸或7个天冬氨酸显著增强普鲁兰酶在大肠杆菌中表达分泌
栗亚美1,2,3, 安展飞1,2,3, 陈阿娜1,2,3, 杨艳坤1,2,3, 白仲虎1,2,3
(1. 江南大学 粮食发酵工艺与技术国家工程实验室, 无锡 214122; 2. 江南大学 工业生物技术教育部重点实验室, 无锡 214122; 3.江南大学 糖化学与生物技术教育部重点实验室, 无锡 214122)
各种标签被广泛应用于大肠杆菌分泌表达异源蛋白,其中最简单有效地便是氨基酸标签,尤其是带电荷氨基酸标签。为了研究N端添加不同种类和数目的带电荷氨基酸标签对普鲁兰酶(pullulanase,BaPul13A)在大肠杆菌中表达分泌水平的影响,构建了25株BaPul13A变体。实验结果表明:N端添加9个连续的谷氨酸标签,降低了mRNA的转录水平,增加了N端mRNA的稳定性,使得普鲁兰酶总酶活达到179.60 U/mL,比不添加氨基酸标签的对照菌高278.28%。N端添加7个连续的天冬氨酸标签,增强了大肠杆菌细胞内膜通透性,大量的普鲁兰酶,48.18 U/mL,分泌到培养基中。通过蛋白质结构预测软件I-TASSER分析得知,N端添加不同带电荷氨基酸标签引起CBM41结构域发生变化,猜测该变化是导致BaPul13A变体表达分泌水平差异的主要原因。
带电荷氨基酸标签;表达分泌;mRNA;膜通透性;蛋白结构预测
大肠杆菌,因其丰富而配套的载体和菌株系统,以及内在的蛋白分泌系统,被称为一类完美的宿主[1-2]。在大肠杆菌中,蛋白质通过氨基酸末端信号序列的引导,穿过细胞质膜进入周质空间或者胞外[3],无论是在N端或是C端,序列的改变都会引起异源蛋白表达分泌水平的差异[4-7]。Kim等[1]提到N端添加5个连续的天冬氨酸标签能够促进脂肪酶B分泌,上清脂肪酶B产量达到65 U/mL,Kato 等[8]在牛胰蛋白酶抑制剂变体(BPTI-22) C端添加了5个赖氨酸标签后,可溶性产量提高了6.2倍,而Yamane 等[9]证明,在信号肽后面引入碱性氨基酸残基会抑制蛋白转运。
Bacillusacidopullulyticus酸性普鲁兰芽孢杆菌普鲁兰酶(BaPul13A),是一种专一性切割 α-1,6糖苷键的淀粉脱支酶,在工业上,与专一性切割 α-1,4糖苷键的淀粉酶共同作用,完成淀粉分解过程[10-12]。目前,工业化的BaPul13A是在地衣芽孢杆菌Bacilluslicheniformis中进行可溶性表达和分泌的[10]。由于BaPul13A蛋白分子质量大,在大肠杆菌中很难实现大量可溶性表达甚至分泌。而一些传统的可溶蛋白载体[8]如麦芽糖结合蛋白(MBP)[13]和谷胱甘肽-S-转移酶(GST)[14],分子量较大甚至会破坏目的蛋白的结构和功能。氨基酸标签(SEP-tags, 可溶性增强标签)分子质量小,同时也能起到提高目的蛋白可溶性和分泌性的效果,甚至在目的蛋白未折叠时就会发挥作用[8]。所以氨基酸标签是用来可溶性表达和分泌大分子蛋白最合适的选择。
目前国内关于氨基酸标签的研究很少,主要集中在组氨酸标签上[15],并且对不同氨基酸标签对普鲁兰酶表达和分泌的差异影响不甚了解,同时对造成这种差异的原因也没有进行过探究。同时,本实验室前期运用许多传统方法[10],都没有实现普鲁兰酶的高效表达和大量分泌。据此,本文通过构建一系列N端带有不同带电荷氨基酸标签的普鲁兰酶变体菌株,研究N端氨基酸标签对普鲁兰酶表达和分泌的影响。
1 材料和方法
1.1 菌株和质粒
大肠杆菌Escherichiacoli(E.coli) BL21(DE3)用于普鲁兰酶表达。酸性普鲁兰芽孢杆菌普鲁兰酶基因(Accession No.Ax203843.1) (pul13A)由上海Invitrogen合成。载体pET28a(+)-pelB-pul13A,含有信号肽基因pelB和普鲁兰酶基因pul13A,由本实验室构建。
1.2 基因操作
通过聚合酶链式反应(PCR)扩增合成N端带有不同带电荷氨基酸标签的普鲁兰酶基因。以pET28a(+)-pelB-pul13A为模板,正反向引物通过特别设计并且都含有特定的DNA限制性内切酶酶切位点。为了保证限制性内切酶识别序列(NcoI: CCATGG)恰恰在载体pET28a(+)-pelB-pul13A的pelB信号肽后面并且避免移码,我们在正向引物内切酶识别序列后添加了两个密码子CC变成CCATGGCC,因此正向引物编码的氨基酸都含有丙氨酸。在每个PCR反应中反向引物pul13A+XhoI (5′-CCGCTCGAGTTATTGTTTGAGAATAAGCGTACTTATAGC-3′)均相同。比如,利用正向引物(5′-CATGCCATGGCCGATGATTCTACTTCGACTAAAGTTATTGTTC-3′)和反向引物pul13A+XhoI可以扩增出N端带一个天冬氨酸标签的重组普鲁兰酶基因。然后,将PCR产物和载体用NcoI和XhoI消化,T4 DNA 连接酶连接,转化BL21(DE3)感受态。转化子进行酶切和测序验证后用于后期发酵实验。重组菌株名称,PCR引物及添加的氨基酸标签如表1。

表1 N端添加标签类型及其对应的PCR引物
( )n代表括号内的元素重复n次;丙氨酸:Ala或A;天冬氨酸:Asp或 D;谷氨酸:Glu或E;组氨酸:His或H;精氨酸:Arg或R;赖氨酸:Lys或K
1.3 培养基和培养条件
N端带有不同带电荷氨基酸标签的BaPul13A变体菌BL21(DE3)于-80℃保存在25%甘油中,用添加了50 μg/mL卡那霉素的LB培养基(5 g/L 酵母提取物, 10 g/L 蛋白胨, 10 g/L NaCl) 37℃,230 r/min过夜培养活化。活化菌以终D600 nm=0.05转接到新鲜的含有50 μg/mL卡那霉素的TB/SB培养基(12 g/L 蛋白胨, 24 g/L 酵母提取物, 2.31 g/L KH2PO4, 12.55 g/L K2HPO4;3H2O, 10 g/L glycerol, pH=7.0)中,37℃,230 r/min培养5 h后,添加0.1 mol/L IPTG,改变培养温度为20℃、230 r/min培养20 h收获,用于酶活测定和SDS-PAGE。
1.4 细胞分级处理
取发酵液,12 000 r/min离心10 min,上清代表胞外可溶组分(ESF)。周质空间组分通过渗透冲击法获得[16]。菌体沉淀经50 mmol/L Tris-HCl buffer (pH=9.0)洗涤2次,然后加入等体积的OS缓冲液(30 mmol/L Tris-HCl、 20% 蔗糖、1 mmol/L EDTA、pH=8.0),室温缓慢搅拌10 min,12 000 r/min 4℃离心10 min,上清定义为OS组分(OSIF),沉淀加入等体积冰冻的5 mmol/L MgSO4,冰上缓慢搅拌10 min,12 000 r/min 4℃离心10 min,上清即为OS组分(OSIIF)。沉淀溶解在50 mmol/L Tris-HCl buffer (pH=9.0)中,超声破碎(功率20%,超声2 s,停2 s,20 min),12 000 r/min 4℃离心10 min,上清即为胞内可溶组分(ISF)。分离的所有组分保存在-80℃。
1.5 普鲁兰酶活的测定
通过DNS法[17]测定普鲁兰酶酶活,原理是DNS和还原糖(由普鲁兰酶分解普鲁兰多糖产生)的氧化还原反应,0.1 mL粗酶液(上清组分或周质空间组分)加入到含有0.2 mL 1%普鲁兰糖的100 mmol/L醋酸-醋酸钠缓冲液的EP管内,混匀,60℃水浴10 min,迅速加入0.45 mL 3,5-二硝基水杨酸溶液(DNS),沸水浴10 min,于冰上冷却后稀释20倍测定D540 nm吸光度,并根据标准曲线,计算出酶活。酶活力单位定义为在上述条件下每分钟分解普鲁兰糖所释放的还原碳水化合物的毫克数,其还原力相当于产生1 μmol葡萄糖所需要的酶量。
1.6 生物信息学工具
为了研究N端含有不同带电荷氨基酸标签的BaPul13A的二级结构变化,我们用软件Iterative Threading ASSEmbly Refinement (I-TASSER)进行预测[18],链接是http://zhanglab.ccmb.med.umich.edu/I-TASSER/。为了计算N端mRNA最小自由能,我们用软件hybrid-ss-min program进行预测[19],链接是http://unafold.rna.albany.edu/?q=mfold/RNA-Folding-Form2.3。
1.7 荧光定量PCR
新鲜的菌体沉淀用RNA提取试剂盒(Tiangen, China)提取RNA。提取的RNA用反转录试剂盒PrimeScript RT reagent Kit with gDNA Eraser (TaKaRa,China)进行反转录。定量PCR体系按照SYBRPremixEXTaqTMII (Tli RNaseH Plus) (TaKaRa, China)试剂盒说明书配制。用来检测普鲁兰酶基因的引物(the forward primer: 5′-CGCGATTTTTCGATTGATGCTA-3′, the reverse primer: 5′-GCTGTTCCTTCTGGAGTTGTGGC-3′) ,以及内参基因16 sRNA引物(the forward primer: 5′-TCGGGAACCGTGAGACAGG-3′, the reverse primer: 5′-CGGACTACGACGCACTTTATGAG-3′)均由苏州金唯智公司合成。所用仪器是Applied Biosystems(USA)。
1.8 细胞内外膜通透性的测定
细胞内外膜通透性的测定方法根据文献[20]并作适当调整:12 000 r/min 5 min离心收集菌体,用PBS洗涤2次,固定终D600 nm=0.5。取经过上述处理的新鲜菌液200 μL,于96孔板中加入1.1 g/L邻硝基苯β-D-半乳糖苷(ONPG) 20 μL或头孢硝噻吩(Nitrocefin) 20 μL,利用酶标仪检测D420 nm或者D500 nm变化来表示内膜或外膜通透性的改变。
2 结果和讨论
2.1 N端不同带电荷氨基酸标签的普鲁兰酶变体菌株的构建
为了分析N端带电荷氨基酸标签对BaPul13A表达分泌的影响,我们构建了25株普鲁兰酶变体菌株。包含2个负电荷氨基酸(天冬氨酸、谷氨酸),3个正电荷氨基酸(组氨酸、精氨酸、赖氨酸)及其不同数目的组合。比如,L1(L6、L11、L16和L21)菌株是N端带有1个Glu (1Asp、1His、1Arg和1Lys)标签的普鲁兰酶变体菌株。L2 (L7、L12、L17和L22)菌株是N端带有3个Glu(3Asp、3His、3Arg及3Lys)标签的普鲁兰酶变体菌株。L3 (L8、L13、L18和L23)菌株是N端带有5个Glu(5Asp、5His、5Arg及5Lys)标签的普鲁兰酶变体菌株。L4 (L9、L14、L19和L24) 菌株是N端带有7个Glu (7Asp、7His、7Arg及7Lys)标签的普鲁兰酶变体菌株。L5 (L10、L15、L20和L25)菌株是N端带有9个Glu (9Asp、9His、9Arg及9Lys)标签的普鲁兰酶变体菌株。由于丙氨酸是生物化学和结构上的中性氨基酸[21],为了避免移码,在起始密码子ATG后面添加了CC,所以GCC恰好在pelB信号肽和pul13A普鲁兰酶基因起始位点之间。为了实现普鲁兰酶更好地表达分泌并且能够通过T7 promoter和Lac operator控制本底表达,PCR所用模板是来自于载体pET28a(+) 和pET22b(+)-pelB-Pul13A 经BglII和XhoI双酶切连接后构建的pET28a(+)-pelB-pul13A。
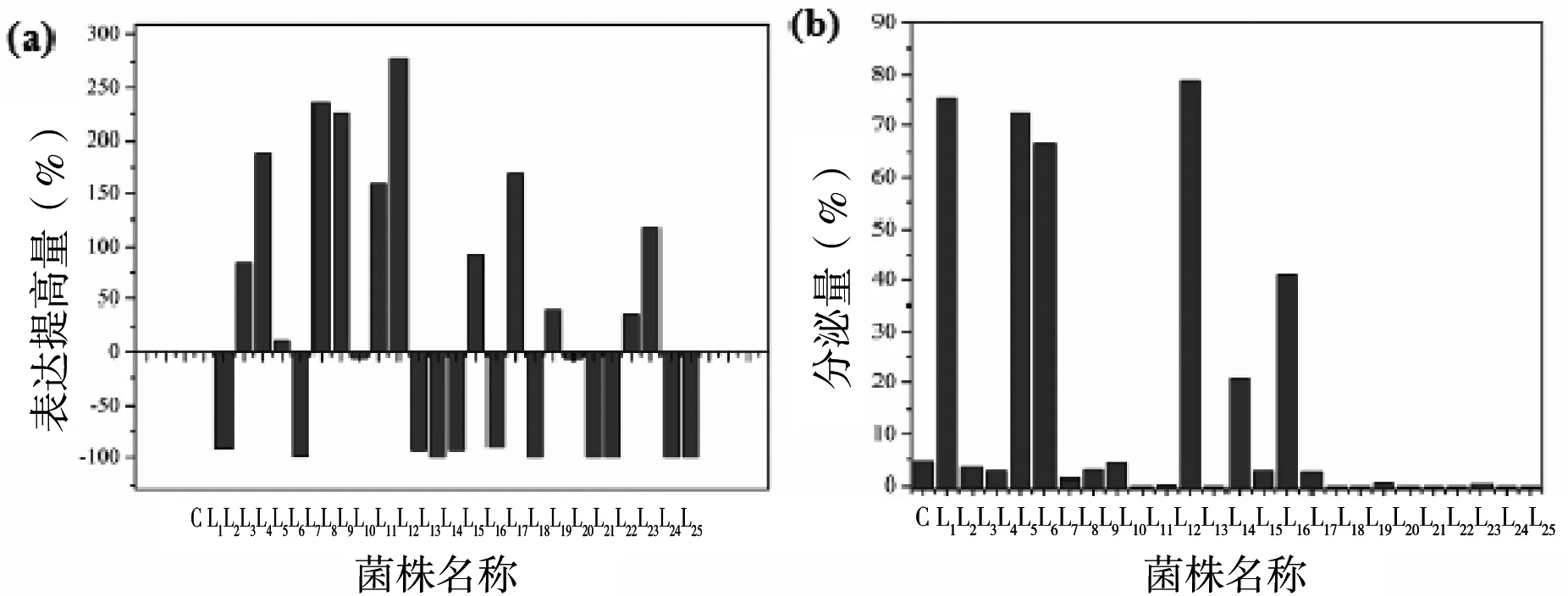
图1 25株变体菌及对照菌发酵后表达提高量(a)和分泌量(b)
2.2 带电荷氨基酸标签对普鲁兰酶BaPul13A表达分泌的影响
为了系统地研究N端不同带电荷氨基酸的添加对普鲁兰酶表达和分泌水平的影响,我们采用相同的培养和操作方法处理25株普鲁兰酶变体菌株及不添加氨基酸标签的对照菌株。各组分酶活(表2)和SDS-PAGE(图2)如下:
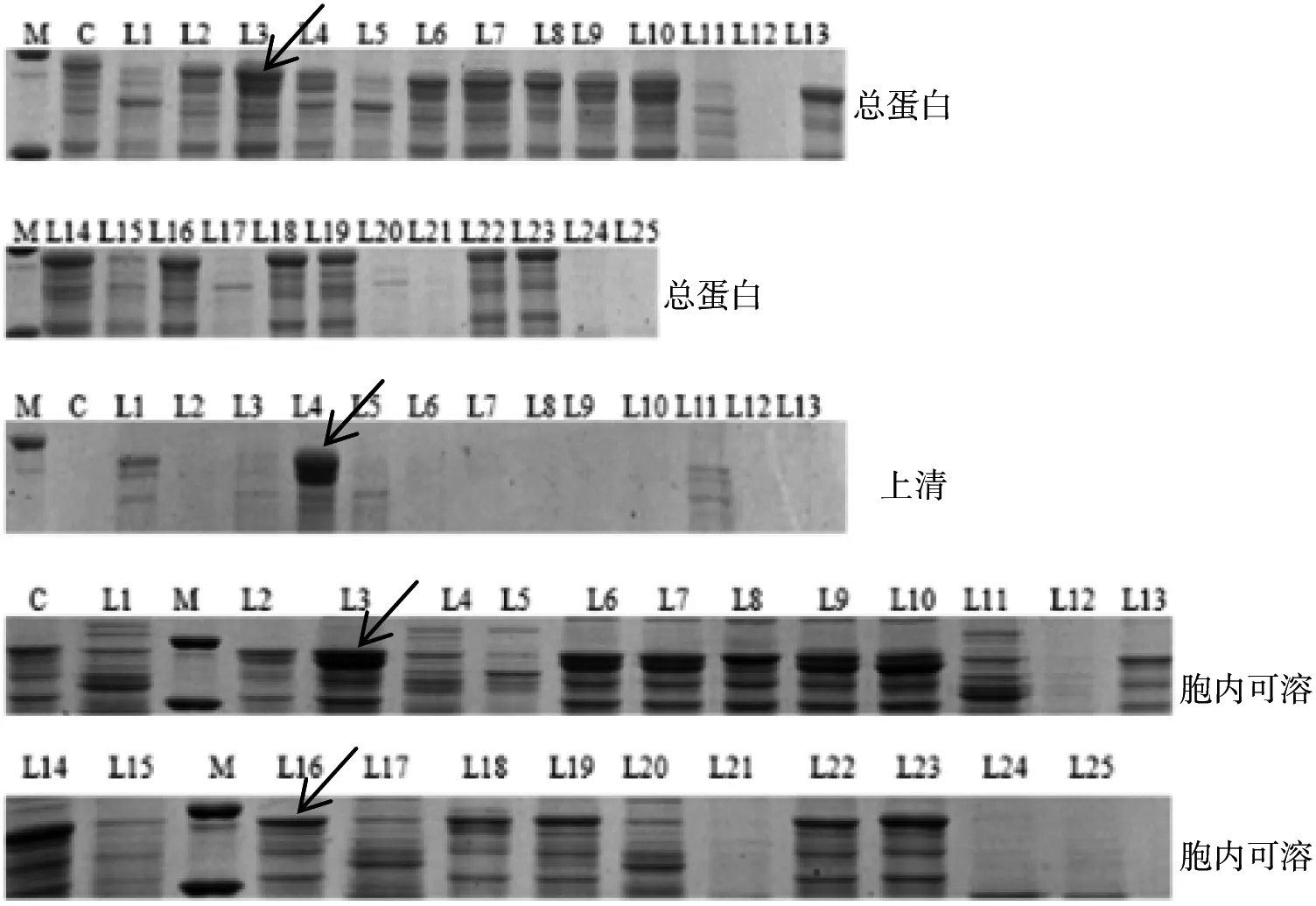
图2 不同带电荷氨基端重组普鲁兰酶SDS-PAGE分析
所有数据均重复3次且平均值的标准差小于5%。ESF代表胞外组分酶活,OSIF代表周质空间OSI酶活,OSIIF代表周质空间OSII酶活,ISF代表胞内酶活,TEA代表总酶,TAE/D600 nm代表比酶活。TAE等于ESF、OSIF、OSIIF、ISF之和;表达提高量等于该菌比酶活与对照菌比酶活之差再除以对照比酶活的值;分泌量等于胞外酶活(ESF、OSIF、OSIIF三者之和)与总酶活的比值由表2可知,普鲁兰酶N端添加不同类型和长度的带电荷氨基酸标签会对普鲁兰酶变体菌株的生长,可溶性表达以及分泌表达都会产生不同程度的影响,促进或抑制。以D600 nm为例,除了少数几个菌株(L2、L6、L7、L9、L10和L13)的生长受到抑制,大部分都有促进作用。
对比表2和图2可知,目的蛋白分子质量约为105.4 ku,各组分酶活的检测数据与SDS-PAGE一致。
就表达量而言(表2),对照菌带有pelB信号肽但是N端没有带电荷氨基酸标签,普鲁兰酶表达量为53.82 U/mL。并且,只要添加合适数量的不同带电荷氨基酸都会对普鲁兰酶表达量的提高有所帮助。N端带有5个天冬氨酸标签(5D-pul13A)的L3菌株表达量较对照提高了188.46%,N端带有7个组氨酸标签(7H-pul13A)的L14菌株表达量较对照提高了92.60%,N端带有1个精氨酸标签(1R-pul13A)的L16菌株表达量较对照提高了169.15%,N端带有5个赖氨酸标签(5K-pul13A)的L23菌株表达量较对照提高了118.53%,此外N端带有9个谷氨酸标签(9E-pul13A)的L10菌株表达量较对照提高了278.78%,并且表达量为最高,179.60 U/mL。在所有的变体菌株中,N端添加负电荷氨基酸标签(酸性氨基酸标签)的BaPul13A变体,除了L8表达量与对照相近以及L1、L5明显低于对照外,其他菌株,包括N端带有E、3E、7E、3D、5D和7D,都对普鲁兰酶可溶性表达起到一定的促进作用,分别提高235.41%、225.18%、158.89%、84.25%、188.46%和10.77%。添加正电荷氨基酸标签(碱性氨基酸标签)的BaPul13A变体中,7H-BaPul13A的表达量提高到125.30 U/mL,比酶活是8.84 U/mL/D600 nm,较BaPul13A的表达量提高92.60%;1R-BaPul13A、5R-BaPul13A 的表达量分别提高到142.28 U/mL、110.89 U/mL,比酶活是12.35 U/mL/D600 nm、6.46 U/mL/D600 nm,较BaPul13A的表达量高169.15%、40.76%;3K-BaPul13A和5K-BaPul13A的表达量分别提高到110.82 U/mL和120.54 U/mL,比酶活是6.25 U/mL/D600 nm和10.03 U/mL/D600 nm,较BaPul13A的表达量高36.7%和118.35%,其他的BaPul13A变体表达量均下降,甚至有些菌株检测不到酶活。
就分泌量而言,由图1、图2和表2可知,对照菌表达的普鲁兰酶基本上无分泌。很明显,N端7D的L4实现了普鲁兰酶胞外大量分泌,胞外和周质空间总酶活达到50.07 U/mL,其中上清酶活为48.18 U/mL,分泌蛋白占总蛋白72.30%。其他菌株对普鲁兰酶分泌没有产生明显的影响。有研究表明,正电荷氨基酸残基,在信号肽疏水结构域之前,对蛋白通过细胞质膜的转运起促进作用,在信号肽疏水结构域之后,对蛋白通过细胞质膜的转运起抑制作用[9]。并且有人提出,多聚赖氨酸会影响蛋白通过SecYEG的转运时间和效率,随着多聚赖氨酸数目增多,相应的转运时间会增加,转运效率会降低[23-24]。有一种猜想认为,磷脂双分子层会抑制N端带正电荷氨基酸的蛋白通过,因为信号肽通过电化学梯度引导蛋白过膜,N端正电荷氨基酸影响电化学梯度,进而抑制转运[25-26]。
2.3 N端氨基酸标签对pul13A mRNA 和BaPul13A变体菌株细胞膜通透性影响
2.3.1 N端氨基酸标签对pul13A mRNA 转录水平和N端mRNA稳定性影响
在所有的变体中,选取了表达量相对较高的5个菌株(L3、L10、L14、L16和L23)进行了荧光定量PCR测定如表3。通过两步法测定pul13A转录水平,重复3次。标准曲线用来检测引物可用,以及决定样品的稀释倍数。
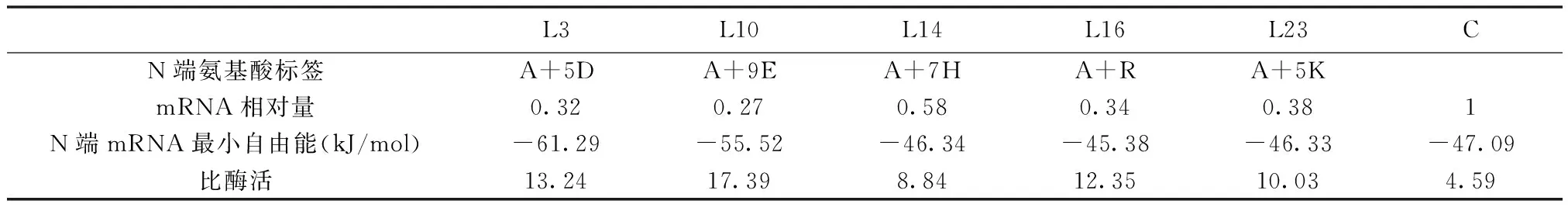
表3 不同带电荷氨基端重组普鲁兰酶mRNA分析
其中C作为对照,mRNA相对量为1,其他菌株mRNA相对量均较对照组小。在pul13A mRNA相对量和Pul13A变体比酶活之间存在相反的变化趋势,pul13A mRNA相对量越低,Pul13A变体比酶活越高。L10,pul13A mRNA相对量最低为0.27,比酶活最高为17.39。同时,L14,pul13A mRNA相对量最高为0.58,比酶活为8.84,比L10比酶活低2倍。产生这种现象的原因可能是,转录水平的降低有助于mRNA有足够的时间转运和正确折叠,避免形成大量包涵体。
N端mRNA的稳定性也会影响转录和mRNA的量,进而影响表达翻译。我们采用在线预测软件,hybrid-ss-min program,预测N端最小自由能。N端预测序列包含pelB序列,添加的带电荷氨基酸标签序列以及从起始密码子开始的前20个氨基酸序列。预测值越负代表N端mRNA越稳定性。对比得知,带负电荷氨基酸标签的L3和L10,最小自由能较负,N端mRNA较其他菌株稳定。带正电荷氨基酸标签的L14、L16和L10,最小自由能与不带氨基酸标签的C菌基本一致。
2.3.2 N端氨基酸标签对BaPul13A变体菌株细胞内外膜通透性影响
带电荷氨基酸标签会影响异源蛋白在大肠杆菌中的可溶性表达和分泌[2]。一方面,在成熟序列前端两个正电荷氨基酸的存在会显著影响蛋白转运[9];另一方面,N端高密度的负电荷能诱导更多的D5-CalB蛋白从细胞质到周质空间的转运,并使外膜无序[1]。为了揭示L4菌株中7D-BaPul13A如何高效分泌到培养基中,我们检测了L4菌株和对照菌细胞内外膜通透性。如图3,L4内膜通透性较对照呈现出明显地增长趋势,但是外膜通透性较对照没有明显的变化。因此,我们认为N端负电荷氨基酸标签通过增加内膜通透性能有效帮助蛋白转运。但是,在周质空间折叠好的7D-Pul13A通过一些未知的与外膜的相互作用帮助其有效分泌,而非仅仅通过增加外膜通透性。

图3 大肠杆菌细胞内(a)、外膜(b)通透性
■ 代表L4;●代表对照菌
3 讨论
很明显,N端或C端不同类型或长度的氨基酸对表达和分泌产生不同的影响[27-28],但是,我们发现,在氨基酸的选择上还是存在一定的规律可循。比如,N端7个天冬氨酸能有效增强7D-BaPul13A的分泌,同时Kim等[1]也提到,N端5个天冬氨酸标签保证脂肪酶B胞外产量达到65 U/mL。因此可以通过生物信息学工具设计一个系统的实验进行相应的数据分析,从而发现内在规律,指导我们选择合适的氨基酸实现高效表达和分泌。
为了进一步阐明25株变体表达分泌差异机制,我们用I-TASSER(Iterative Threading ASSEmbly Refinement),一种通过按等级划分的方法预测蛋白结构和功能的原件,对BaPul13A变体蛋白结构进行预测。Turkenburg等已经得到了Pul13A的晶体结构,由于CBM41结构域处于无序状态,所以晶体结构上并没有CBM41结构域[12]。而我们的氨基酸标签添加在N端,因此我们采用蛋白预测软件I-TASSER方法,预测了包含CBM41在内的所有结构域(CBM41-X45a-X25-X45b-CBM48-GH48);采用Discovery Studio Client进行蛋白预测结构分析。
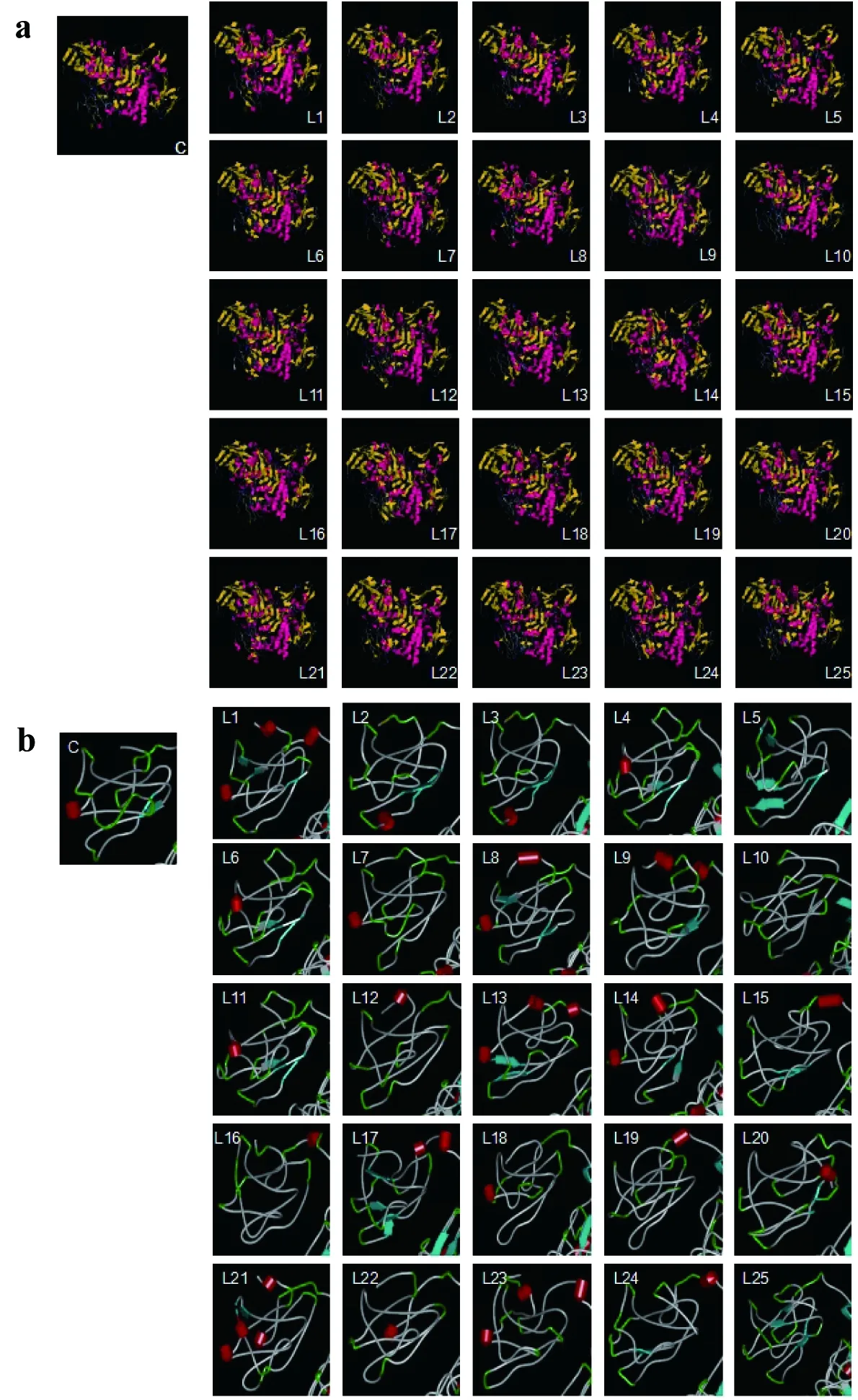
图4 Pul13A变体蛋白结构(a)及CBM41结构域(b)预测结果
对照菌及25株变体菌普鲁兰酶3D二级结构图见图4-a,显示除了CBM41结构域外,BaPul13A变体其他结构域与晶体结构域一致;从对照菌及25株变体菌普鲁兰酶放大后的CBM41结构域可以看出,变体菌中α-螺旋、β-折叠数目以及无规则卷曲均发生变化(图4-b)。不同的氨基酸标签可以与CBM41结构域相互作用,进而造成CBM41结构域的改变。这和本实验室以前的研究结果相一致:CBM41结构域的缺失使得普鲁兰酶酶活大约提高了50倍。因此,CBM41的缺失或改变均会引起分泌表达差异。
N端氨基酸标签不仅仅会增强表达和分泌,还有很多其他的用途。氨基酸标签,一种可溶性增强标签(SETs),通过克服可溶性和稳定性问题,使得蛋白符合核磁共振技术要求[29]。而且通过氨基酸标签,而不单是组氨酸标签,可以开发一种新型的过量表达和分泌的载体[30]。
[1]KIM S K, PARK Y C, LEE H H, et al. Simple amino acid tags improve both expression and secretion of Candida antarctica lipase B in recombinantEscherichiacoli[J]. Biotechnology & Bioengineering, 2015, 112(2) : 346-355.
[2]JUNG H J, KIM S K, MIN W K, et al. Polycationic amino acid tags enhance soluble expression of Candida antarctica lipase B in recombinantEscherichiacoli[J]. Bioprocess & Biosystems Engineering, 2011, 34(7) : 833-839.
[3]KAJAVA A V, SERGEY N Z, AMDREY E K, et al. The net charge of the first 18 residues of the mature sequence affects protein translocation across the cytoplasmic membrane of gram-negative bacteria [J]. Journal of Bacteriology, 2000, 182(8) : 2163-2169.
[4]KOBAYASHI T, MIWA S, HIROFUMI N, et al. Short peptide tags increase the yield of C-terminally labeled protein [J]. Biotechnol Lett, 2007, 29(7) : 1065-1073.
[5]XU C G, FAN X J, FU Y J, et al. Effect of location of the His-tag on the production of soluble and functionalButhusmartensiiKarschinsect toxin [J]. Protein Expr Purif, 2008, 59(1) : 103-109.
[7]WOESTENENK E A, HAMMARSTRÖM M, VAN DEN B S, et al. His tag effect on solubility of human proteins produced inEscherichiacoli: a comparison between four expression vectors[J]. J Struct Funct Genomics, 2004, 5(3): 217-229.
[8]KATO A, MAKI K, EBINA T, et al. Mutational analysis of protein solubility enhancement using short peptide tags [J]. Biopolymers, 2007, 85(1) : 12-18.
[9]YAMANE K, MIZUSHIMA S. Introduction of basic amino acid residues after the signal peptide inhibits protein translocation across the cytoplasmic membrane ofEscherichiacoli[J]. J Biol Chem, 1988, 263(36) : 19690-19696.
[10]CHEN A, LI Y, LIU X, et al. Soluble expression of pullulanase fromBacillusacidopullulyticusinEscherichiacoliby tightly controlling basal expression[J]. J Ind Microbiol Biotechnol, 2014, 41(12) :1803-1810.
[11]CHEN A, LI Y, NIE J, et al. Protein engineering ofBacillusacidopullulyticuspullulanase for enhanced thermostability using in silico data driven rational design methods [J]. Enzyme Microb Technol, 2015, 78 : 74-83.
[12]TURKENBURG J P, BRZOZOWSKI A M, SVENDSEN A, et al. Structure of a pullulanase fromBacillusacidopullulyticus[J]. Proteins, 2009, 76(2) : 516-519.
[13]DI GUAN C, LI P, RIGGS P D, et al. Vectors that facilitate the expression and purification of foreign peptides inEscherichiacoliby fusion to maltose-binding protein [J]. Gene, 1998, 67(1): 21-30.
[14]SMITH D B, JOHNSON K S. Single-step purification of polypeptides expressed inEscherichiacolias fusions with glutathione S-transferase [J]. Gene, 1988, 67(1) : 31-40.
[15]刘英丽, 刘骏明, 王 静, 等. 栓菌漆酶的异源表达及重组酶碳端和氮端组氨酸标签修饰对酶学特性的影响 [J]. 食品科学, 2014, 35(21) : 143-148.
[16]NEU H C, HEPPEL L A. Some observations on the "latent” ribonuclease ofEscherichiacoli[J]. Proc Natl Acad Sci U S A, 1964, 51 : 1267-1274.
[17]KANG J, PARK K M, CHOI K H, et al. Molecular cloning and biochemical characterization of a heat-stable type I pullulanase fromThermotoganeapolitana[J]. Enzyme & Microbial Technology, 2011, 48(3): 260 266.
[18]YANG J, YAN R, ROY A, et al. The I-TASSER suite: protein structure and function prediction [J]. Nat Methods, 2015, 12(1): 7-8.
[19]KUDLA G, MURRAY A W, TOLLERVEY D, et al. Coding-sequence determinants of gene expression inEscherichiacoli[J]. Science, 2009, 324(5924): 255-258.
[20]ERIKSSON M, NIELSEN P E, GOOD L. Cell permeabilization and uptake of antisense peptide-peptide nucleic acid (PNA) intoEscherichiacoli[J]. The Journal of Biological Chemistry, 2002, 277(9): 7144-7147.
[21]ISLAM M M, KHAN M A, KURODA Y, et al. Analysis of amino acid contributions to protein solubility using short peptide tags fused to a simplified BPTI variant [J]. Biochim Biophys Acta, 2012, 1824(10) : 1144-1150.
[22]LIANG F C, BAGESHWAR U K, MUSSER S M, et al. Position-dependent effects of polylysine on Sec protein transport [J]. J Biol Chem, 2012, 287(16) : 12703-12714.
[23]DU PLESSIS D J, NOUWEN N, DRIESSEN A J. The Sec translocase [J]. Biochimica et Biophysica Acta, 2011, 1808(3): 851-865.
[24]PAOLO N, THOMAS B, ARNOLD J M D. Sec-and Tat-mediated protein secretion across the bacterial cytoplasmic membrane-distinct translocases and mechanisms [J]. Biochimica et Biophysica Acta, 2008, 1778(9): 1735-1756.
[25]Mickael D, Michel H, Regine D, et al. Secretion and subcellular localizations of bacterical proteins: a semantic awareness issue [J]. Trends in Microbiology, 2009, 17(4) : 139-145.
[26]LORRAINE S M, REMBRANDT JF H, KATRINA T F. Structural insights into the type II secretion nanomachine [J]. Current Opinion in Structural Biology, 2012, 22(2) : 208-216.
[27]BLEVE G, LEZZI C, SPAGNOLO S, et al. Role of the C-terminus ofPleurotuseryngiiEry4 laccase in determining enzyme structure, catalytic properties and stability [J]. Protein Engineering Design & Selection, 2013, 26(1) : 1-13.
[28]TREVINO S R, SCHOLTZ J M, PACE C N. Amino acid contribution to protein solubility: Asp, Glu, and Ser contribute more favorably than the other hydrophilic amino acid in RNasa Sa [J]. J Mol Biol, 2007, 366(2) : 449-460.
[29]ZHOU P, WAGNER G. Overcoming the solubility limit with solubility-enhancement tags: successful applications in biomolecular NMR studies [J]. J Biomol NMR, 2010, 46(1) : 23-31.
[30]ASHRAF S S, BENSON R E, PAYNE E S, et al. A novel muti-affinity tag system to produce high levels of soluble and biotinylated proteins inEscherichiacoli[J]. Protein Expression and Purification, 2004, 33 (2): 238-245.
N-terminal 9-glutamine and 7-aspartate tags significantly increase the expression and secretion of pullulanase from
BacillusacidopullulyticusinEscherichiacoli
LI Ya-mei1,2,3, AN Zhan-fei1,2,3, CHEN A-na1,2,3, YANG Yan-kun1,2,3, BAI Zhong-hu1,2,3
(1. National Engineering Laboratory for Cereal Fermentation Technology; 2. The Key Laboratory of Industrial Biotechnology, Ministry of Education; 3. The Key Laboratory of Carbohydrate Chemistry and Biotechnology, Ministry of Education, School of Biotechnology, Jiangnan University, Wuxi 214122, China)
Various tags, especially charged amino acids, were widely applied in soluble expression and secretion of heterogenous proteins inEscherichiacolirecent years. To know how the charged amino acids types and numbers influent the Pul13A expression and secretion, 25 Pul13A mutants were constructed, which had different N-terminal amino acid tags. Among them, the nine-glutamine tag could decrease the transcription level and increase the N-terminal mRNA stability, and Pul13A′s activity changed to 179.60 U/mL that was 278.28% higher than the control without any amino acid tag. Meanwhile, the N-terminal seven-aspartate tag enhanced the inner membrane permeability, and secreted massive pullulanse into the culture medium ( 48.18 U/mL ). Furthermore, it was found that the CBM41 structural domain was different in different mutants, which maybe mainly result in the differences of the expression and secretion levels.
charged-amino-acid tags; expression and secretory; mRNA; membrane permeability; protein structure prediction
2016-03-30;
2016-04-14
中央高校基本科研业务费专项资金资助(JUSRP51401A);863项目(2015AA020802);973项目(2013CB733602); 国家自然科学基金项目(31570034)
栗亚美,硕士研究生,主要从事发酵工程研究,E-mail: 1198066405@qq.com
白仲虎,教授,主要从事发酵工程领域科研工作,E-mail: baizhonghu@jiangnan.edu.cn;杨艳坤,副教授,主要从事发酵工程领域科研工作,E-mail: yangyankun@jiangnan.edu.cn
Q78;TQ925
A
2095-1736(2017)03-0011-07
doi∶10.3969/j.issn.2095-1736.2017.03.011
