黄陂牛mtDNA D-loop区遗传多样性研究
2017-07-18党瑞华黄永震蓝贤勇梁小军雷初朝
孙 婷,党瑞华,黄永震,蓝贤勇,梁小军,陈 宏,雷初朝﹡
(1.西北农林科技大学动物科技学院,陕西 杨凌 712100;2.宁夏农林科学院动物科学研究所,宁夏 银川 750002)
科学试验
黄陂牛mtDNA D-loop区遗传多样性研究
孙 婷1,党瑞华1,黄永震1,蓝贤勇1,梁小军2,陈 宏1,雷初朝1﹡
(1.西北农林科技大学动物科技学院,陕西 杨凌 712100;2.宁夏农林科学院动物科学研究所,宁夏 银川 750002)
【目的】探究黄陂牛的遗传多样性与母系起源。【方法】采用PCR扩增、测序及生物信息学方法。【结果】在21头黄陂牛mtDNA D-loop区共检测到55个变异位点,定义了10个mtDNA单倍型,平均单倍型多样度为0.7760,平均核苷酸多样度为0.0234,表明黄陂牛具有丰富的遗传多样性。构建的NJ系统发育树表明黄陂牛具有瘤牛和普通牛两个母系起源。【结论】黄陂牛有丰富的遗传多样性,有瘤牛和普通牛两个母系起源,且受瘤牛的影响较大。
黄陂牛;mtDNA D-loop;遗传多样性;母系起源
中国黄牛的品种数量多且分布范围广泛,是世界牛属遗传资源的重要组成部分。目前,我国有53个地方黄牛品种[1]。按地理分布区域的不同,将中国黄牛分为北方黄牛、中原黄牛和南方黄牛三大类[2]。黄陂牛属于南方黄牛,是一个役肉兼用型优良地方品种,主要分布在武汉市黄陂区,故被命名为黄陂牛[3]。1982年,全国品种志编委会将分布在大别山及其周边地区的黄牛统一命名为大别山黄牛,黄陂牛也属于此类[3]。
哺乳动物线粒体DNA(mitochondrial DNA,mtDNA)是一个双链闭合的环状DNA分子,具有结构简单、遵循母系遗传、无重组、进化速度快等特点,在动物的系统发育、演化及遗传多样性方面得到广泛应用[4]。和其他哺乳动物一样,牛的mtDNA由37个蛋白编码基因和一段910 bp的D-loop区组成[5]。Yu等[6]对12个中国南方牛品种154个个体进行mtDNA RFLP分析,结果表明普通牛和瘤牛是中国南方牛的主要起源。Lai等[7]通过对84头中国黄牛mtDNA D-loop区的研究表明中国黄牛可以分为普通牛和瘤牛两大来源,且瘤牛对中国南方牛的影响更大。Lei等[8]对中国黄牛mtDNA D-loop全序列的研究也表明中国黄牛是瘤牛与普通牛的混合母系起源。本研究对黄陂牛的mtDNA D-loop全序列进行测定,分析黄陂牛的遗传多样性及母系起源,为黄陂牛的保种与利用提供遗传基础。
1 材料与方法
1.1 样品采集
采集18头黄陂牛耳组织样,用75%酒精保存,于-20℃冰箱保存备用。从GenBank上下载了3条黄陂牛序列,其登录号为:AY521096-AY521098,并分别下载了一条印度瘤牛序列(L27733)和一条德国普通牛序列(AF492351)作为对照。
1.2 基因组DNA提取及PCR扩增测序
采用酚-氯仿法提取基因组DNA。采用雷初朝等[9]设计的引物扩增线粒体DNA D-loop全序列,正链引物和反链引物分别为:F:5′-CTGCAGTCTCACCATCAACC-3′; R:5′-GGGGTGTAGATGCTTGC-3′。PCR反应体系总体积为20 μL,扩增程序为:95℃预变性4 min;94℃变性30 s,59℃退火60 s,72℃延伸90 s,共40个循环;最后72℃延伸10 min,4℃保存。PCR产物用1.0%的琼脂糖凝胶电泳检测,将符合测序要求的PCR产物送至西安擎科泽西生物科技有限责任公司测序。
1.3 数据处理与统计分析方法
利用DNASTAR软件包中的SeqMan程序对序列进行人工校对,确保所测序列的准确性。用DNASP 5.0计算单倍型多样度、核苷酸多样度、Tajima's D值、Fu's Fs 值等。用分子进化遗传分析软件MEGA5.0构建Neighbor-Joining系统发育树。
2 结果与分析
2.1 黄陂牛mtDNA D-loop区遗传多样性
本文分析的21条黄陂牛mtDNA D-loop全序列长度在909-911 bp之间,其中,有8个是909 bp,有10个是910 bp,有3个是911 bp。mtDNA D-loop区的长度变化主要是由于存在插入缺失而导致的。黄陂牛mtDNA D-loop全序列由A、T、C、G四种碱基组成,其中A碱基含量最高(33.20%),其次为T(28.30%)和C(25.00%),G碱基含量最低,仅为13.50%。A+T的平均含量为61.50%,G+C的平均含量为38.50%,A+T的平均含量明显高于G+C,表现出碱基的偏好性。
对21条黄陂牛mtDNA D-loop全序列进行比对,共检测到55个变异位点,包括54个转换,1个颠换。这55个变异位点定义了10种单倍型,其平均单倍型多样度(Hd)为0.7760,平均核苷酸多样度(Pi)为0.0234,平均核苷酸差异(k)为21.2190。
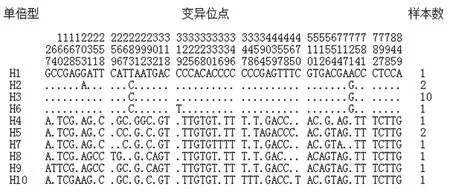
图1 21个黄陂牛个体 mtDNA D-loop区10个单倍型的多态位点“.”代表与第一条序列相同。
对黄陂牛进行错配分布分析(图2),分布图呈现多峰,表明黄陂黄牛过去没有经历过强烈的群体扩张。且对黄陂牛进行中性检验,Tajima's D值(1.4533)和Fu's Fs(0.8913)值均差异不显著(P>0.10),也表明黄陂牛在过去没有经历过群体扩张,群体数量保持相对稳定。
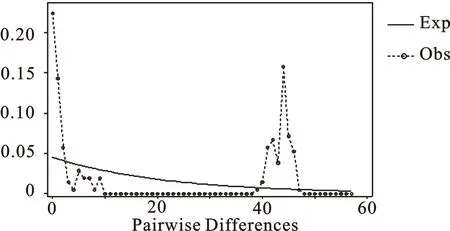
图2 黄陂牛不配对分布图。实线代表稳定种群的期望错配分布;虚线代表错配分布的观察值。
2.2 黄陂牛NJ系统发育树
利用MEGA5.0软件对黄陂牛进行系统发育分析,以印度瘤牛(L27733)和德国普通牛(AF492351)作为参考序列,构建NJ系统发育树。从图3可以看出,黄陂牛被分为瘤牛和普通牛两大支系,说明黄陂牛是瘤牛与普通牛的混合母系起源。
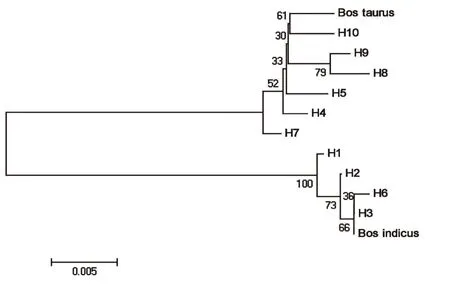
图3 黄陂牛10个mtDNA D-loop单倍型构建的NJ系统树
3 讨论
本文分析了21头黄陂牛的mtDNA D-loop区全序列,其四种碱基的含量分别为:A(33.20%)、T(28.30%)、C(25.00%)、G(13.50%),表现出碱基的偏好性,这是一般多细胞动物mtDNA的显著特征[10]。A+T(61.50%)的平均含量明显高于G+C(38.50%),而在绵羊[11]、山羊[12]、牦牛[13]、猪[14]、鸡[15]等的研究结果也表明,mtDNA D-loop区的A+T 含量高于G+C含量,说明动物mtDNA D-loop区的核苷酸含量在物种间有一定的相似性。
单倍型多样度(Hd值)和核苷酸多样度(Pi值)是衡量一个群体 mtDNA 变异程度的重要指标[16],单倍型多样度和核苷酸多样度的值越大,说明群体的遗传多样性越丰富,反之,则说明群体的遗传多样性越缺乏。周艳等[17]对8个中国南方牛品种的mtDNA D-loop区研究表明,其单倍型多样度为0.2860~0.9000,核苷酸多样度位0.0003~0.2840,表现出丰富的遗传多样性。黄陂牛在地理分布上属于南方牛,本研究中的21头黄陂牛的mtDNA D-loop区的单倍型多样度(Hd)为0.7760,平均核苷酸多样度(Pi)为0.0234,说明黄陂牛也具有丰富的遗传多样性。构建NJ系统发育树将黄陂牛分为瘤牛和普通牛两大支系,在21个黄陂牛个体中,13个瘤牛起源的个体(占61.90%)共享4个单倍型,8个普通牛起源的个体(占38.10%)共享6个单倍型,表明黄陂牛受瘤牛的影响更大。Lai等[7]、周艳等[17]的研究结果也表明中国南方黄牛受瘤牛的影响更大。总之,本研究结果表明,黄陂牛mtDNA D-loop区的遗传多样性比较丰富,具有普通牛和瘤牛两大母系起源,且受瘤牛的影响更大。
[1] 国家畜禽遗传资源委员会.中国畜禽遗传资源志—牛志 [M].北京:中国农业出版社,2011.
[2] 《中国牛品种种志》编写组.中国牛品种志[M].上海:上海科学技术出版社,1988.
[3] 李晓锋,索效军,熊琪,等.黄陂黄牛调查报告及发展建议[J].中国牛业科学,2013,39(3):53-56.
[4] Bruford MW,Bradley DG,Luikart G.DNA markers reveal the complexity of livestock domestication[J].Nature Reviews Genetics,2003,4(11):900-910.
[5] Anderson S,de Bruijn MH,Coulson AR,etal.Complete sequence of bovine mitochondrial DNA.Conserved features of the mammalian mitochondrial genome[J].Journal of Molecular Biology,1982,156(4):683-717.
[6] Yu Y,Nie L,He Z,etal.Mitochondrial DNA variation in cattle of South China:origin and introgression[J].Animal Genetics,1999,30(4):245-250.
[7] Lai SJ,Liu YP,Liu YX,etal.Genetic diversity and origin of Chinese cattle revealed by mtDNA D-loop sequence variation.[J].Molecular Phylogenetics & Evolution,2006,38(1):146-154.
[8] Lei CZ,Chen H,Zhang HC,etal.Origin and phylogeographical structure of Chinese cattle[J].Animal Genetics,2006,37(6):579-582.
[9] 雷初朝,陈宏,杨公社,等.中国部分黄牛品种mtDNA遗传多态性研究[J].遗传学报,2004,31(1):57-62.
[10] Gach MH,Brown WM.Characteristics and distribution of large tandem duplications in brook stickleback (Culaea inconstans) mitochondrial DNA[J].Genetics,1997,145(2):383-394.
[11] Wang X,Ma YH,Chen H,etal.Genetic and phylogenetic studies of Chinese native sheep breeds (Ovis aries) based on mtDNA D-loop sequences[J].Small Ruminant Research,2007,72(2-3):232-236.
[12] Liu RY,Yang GS,Lei CZ.The Genetic Diversity of mtDNA D-loop and the Origin of Chinese Goats[J].Acta Genetica Sinica,2006,33(5):420-428.
[13] 张成福,徐利娟,姬秋梅,等.西藏牦牛mtDNA D-loop区的遗传多样性及其遗传分化[J].生态学报,2012,32(5):1387-1395.
[14] 綦文晶,何剑雄,郭晓萍,等.广西3个猪种线粒体DNA D-loop序列遗传多样性及系统进化研究[J].基因组学与应用生物学,2016,35(9):2367-2374.
[15] 吴婵,李辉,李敬瑞,等.基于线粒体DNA控制区(mtDNA D-loop)序列分析贵妃鸡的遗传多样性[J].中国畜牧兽医,2012,39(12):137-140.
[16] Nagarajan M,Nimisha K,Kumar S.Mitochondrial DNA variability of domestic river buffalo (Bubalus bubalis) populations:Genetic evidence for domestication of river buffalo in Indian subcontinent[J].Genome Biology Evolution,2015,7(5):496-503.
[17] 周艳,陈宏,贾善刚,等.中国南方部分黄牛品种mtDNA D-loop区的遗传变异与分类分析[J].西北农林科技大学学报(自然科学版),2008,36(5):7-11.
Genetic Diversity of Mitochondrial DNA D-loop Region in Huangpi Cattle
SUN Ting1,DANG Rui-hua1,HUANG Yong-zhen1,LAN Xian-yong1,LIANG Xiao-jun2,CHEN Hong1,LEI Chu-zhao1*
(1.CollegeofAnimalScienceandTechnology,NorthwestA&FUniversity,Yangling,Shaanxi712100,China; 2.InstituteofAnimalScience,NingxiaAcademyofAgricultureandForestryScience,Yinchuan,Ningxia,750002,China)
【Objective】The aim of the study was to analyze the genetic diversity and maternal origin of Huangpi cattle.【Method】 The complete mitochondrial DNA (mtDNA) D-loop regions of 21 Huangpi cattle were analyzed using PCR,sequencing and bioinformatics methods.【Result】There were 55 polymorphic sites were detected in mtDNA D-loop of Huangpi cattle,which constituted10 haplotypes.The average haplotype diversity and nucleotide diversity were 0.7760 and 0.0234,respectively,which indicated that the genetic diversity of Huangpi cattle was abundant.The NJ tree indicated that Huangpi cattle had two types of maternal origins from Bos taurus and Bosindicus.【Conclusion】The genetic diversity of Huangpi cattle was abundant.Huangpi cattle had two types of maternal origins from Bos taurus and Bosindicus,and Bosindicus contributed more.
Huangpi cattle;mt DNA D-loop;genetic diversity;maternal origin
7-02-10
2017-02-15
国家肉牛牦牛产业技术体系资助(CARS-38)
孙 婷(1989-),女,陕西凤翔人,硕士生,主要从事动物遗传资源研究。
*通讯作者:雷初朝(1968-),男,湖南常宁人,教授,博导,主要从事牛遗传资源研究。
S823
A
1001-9111(2017)03-0001-03
