循环长链非编码RNA作为生物标志物在肿瘤分子诊断中的应用
2017-07-01江楠田海华潘金昌龚朝辉
江楠,田海华,2,潘金昌,龚朝辉
循环长链非编码RNA作为生物标志物在肿瘤分子诊断中的应用
江楠1,田海华1,2,潘金昌1,龚朝辉1
1 宁波大学医学院生物化学与分子生物学研究所,浙江宁波 315211 2 宁波市康宁医院检验科,浙江宁波 315201

江楠, 田海华, 潘金昌, 等. 循环长链非编码RNA作为生物标志物在肿瘤分子诊断中的应用. 生物工程学报, 2017, 33(6): 910–922.Jiang N, Tian HH, Pan JC, et al. Circulating long noncoding RNAs as biomarkers in tumor diagnosis. Chin J Biotech, 2017, 33(6): 910–922.
长链非编码RNA (Long noncoding RNA,lncRNA) 被发现广泛参与基因表达、表观遗传调控和X染色体失活等重要生命过程,还与肿瘤发生和发展密切相关。lncRNA可能以微泡、外泌体或蛋白质复合物形式进入人体循环系统中,形成循环lncRNA稳定而广泛存在于血液、尿液等体液中。文中简要回顾了近来关于循环lncRNA的来源,以及作为生物标志物的检测方法,着重总结分析了循环lncRNA作为潜在肿瘤生物标志物在肺癌、乳腺癌、胃癌、肝癌、结直肠癌和前列腺癌等常见恶性肿瘤中的早期诊断价值。与传统生物标志物相比,循环lncRNA具有作为新型生物标志物的独特优势和临床应用价值。
循环长链非编码RNA,肿瘤,生物标志物,分子诊断
据美国2016年癌症统计报告,癌症已经成为全美以及全球主要的公共健康问题,在未来几年内很可能会超过心脏病成为排列第一的致死原因[1]。癌症致死的原因是多方面的,而早期诊断是提前发现并进行干预的关键,目前临床上使用的X射线检查、超声检查、计算机断层扫描 (Computed tomography,CT)、核磁共振成像 (Magnetic resonance imaging,MRI) 和内腔镜检查等手段能从不同角度对肿瘤进行早期的诊断,在一定程度上提高了肿瘤的早期诊断率,但仍存在许多不同程度的局限性。自1978年Herberman提出肿瘤标志物的概念后,目前已发现了100多种肿瘤标志物,为肿瘤的早期诊断提供了新的途径[2]。随着肿瘤分子生物学和组学技术的不断进展,肿瘤标志物的研究已成为癌症诊断领域的热点。近几年,研究发现长链非编码RNA (Long noncoding RNA,lncRNA) 在许多生物进程中起到重要作用,包括基因调控[3]、细胞周期检验点[4]、细胞迁移[5]等,也在癌症的发生和发展中发挥着重要的作用。同时,利用基于肿瘤患者外周血的循环核酸检测逐步引起人们的重视,循环lncRNA在肿瘤早期临床诊断和治疗中的应用越来越广泛。本文就近年来循环lncRNA作为生物标志物在肿瘤诊断中的应用进展作简要综述。
1 循环lncRNA的来源
lncRNA是一类转录本长度超过200个核苷酸且自身不编码蛋白的RNA分子,它起初被认为是RNA聚合酶Ⅱ转录的副产物,是基因组转录的“噪音”,并不具有生物学功能[6-7]。然而在近几年的研究中发现,lncRNA可以通过多种机制在多种层面上影响基因的表达水平,且越来越多的研究表明lncRNA在肿瘤发生、发展和转移中扮演着重要角色。在20世纪八九十年代就有研究报道了循环RNA的存在[8-9],为后来的循环lncRNA研究奠定了基础。由于RNA不稳定,容易被血液中的核糖核酸酶降解,人们对lncRNA是如何分泌入血液并稳定存在的机制目前还未完全了解。综合已有的研究分析,循环lncRNA的来源可能通过如下途径 (图1):1) 来源于活细胞,即循环lncRNA可能来自于活细胞的主动分泌过程,其中比较多的是以外泌体 (Exosomes) 和微泡 (Microvesicles) 形式存在。虽然血液中存在核糖核酸酶,但是lncRNA仍能够稳定存在,主要是由于外泌体或者微泡等的保护。Dong等[10]检测了血液中外泌体、凋亡小体和微泡中的RNA含量,发现血液中lncRNA主要分布在外泌体之中,这也说明lncRNA可能是通过这种细胞外囊泡的形式分泌入血。同时,循环lncRNA也可能是通过类似其他非编码RNA入血的方式,比如微RNA (MicroRNA,miRNA) 可以通过与Ago2蛋白结合形成复合物,而稳定存在于血液循环中[11],这表明lncRNA可通过与其他分泌物一同结合而进入循环系统。2) 来源于凋亡或者坏死的细胞。肿瘤原发灶或者转移灶中存在一类具有转移倾向的肿瘤细胞,通过上皮-间叶转换过程,而使其更容易侵入血管内皮而进入血液循环,称之为循环肿瘤细胞 (Circulating tumor cell,CTC)[12]。CTCs入血之后存活时间一般不会超过24 h。进入血液的肿瘤细胞往往保留有起源组织的特异性标志物,即上皮标志物。通过对癌症患者及对照组的外周血进行分析,发现在癌症患者血液中上皮特异性蛋白的mRNA存在的百分比为36%−100%,由此表明循环RNA可能来自于这些侵入血管内的循环肿瘤细胞[13]。同样可能来自细胞凋亡或者其他形式的细胞死亡的血浆游离DNA (Cell-free DNA,cfDNA) 也为循环lncRNA来自于凋亡或者坏死细胞提供可能[14]。
2 循环lncRNA作为肿瘤标志物
目前临床上常用的肿瘤标志物主要是肿瘤抗原和异位激素等。比如,常见的癌胚抗原 (CEA) 用于预警结直肠癌和乳腺癌[15],甲胎蛋白 (AFP)用于预警胃癌、肠癌等,这些传统生物标志物在肿瘤早期诊断中应用广泛[16]。然而,传统标志物存在诸多不足,其中包括特异性和灵敏度不高,同一种标志物可以预示多种癌症风险的可能,也存在较大的漏诊和误诊的几率。自lncRNA被发现以来,人们通过定量RT-PCR技术在血浆、血清、尿液等人体体液中检测到lncRNA[17-19]。因此,循环lncRNA成为新的肿瘤标志物的关注焦点[20]。较早发现的lncRNA H19与多种肿瘤有密切关系,在32对相互匹配的胃癌患者和健康人群的血液样本中检测发现,H19显著地高表达于胃癌癌症患者中,且和患者生存率等病理因素存在着较为明显的关系[21]。作为一种生物标志物,循环lncRNA可以稳定存在于循环系统中,具有免受核酸酶降解的特性[22]。同时,循环lncRNA可在血液、尿液等体液中检测出来。因此,循环lncRNA的来源简单丰富,取材方便,适用于早期检查诊断,具有较高的诊断价值。
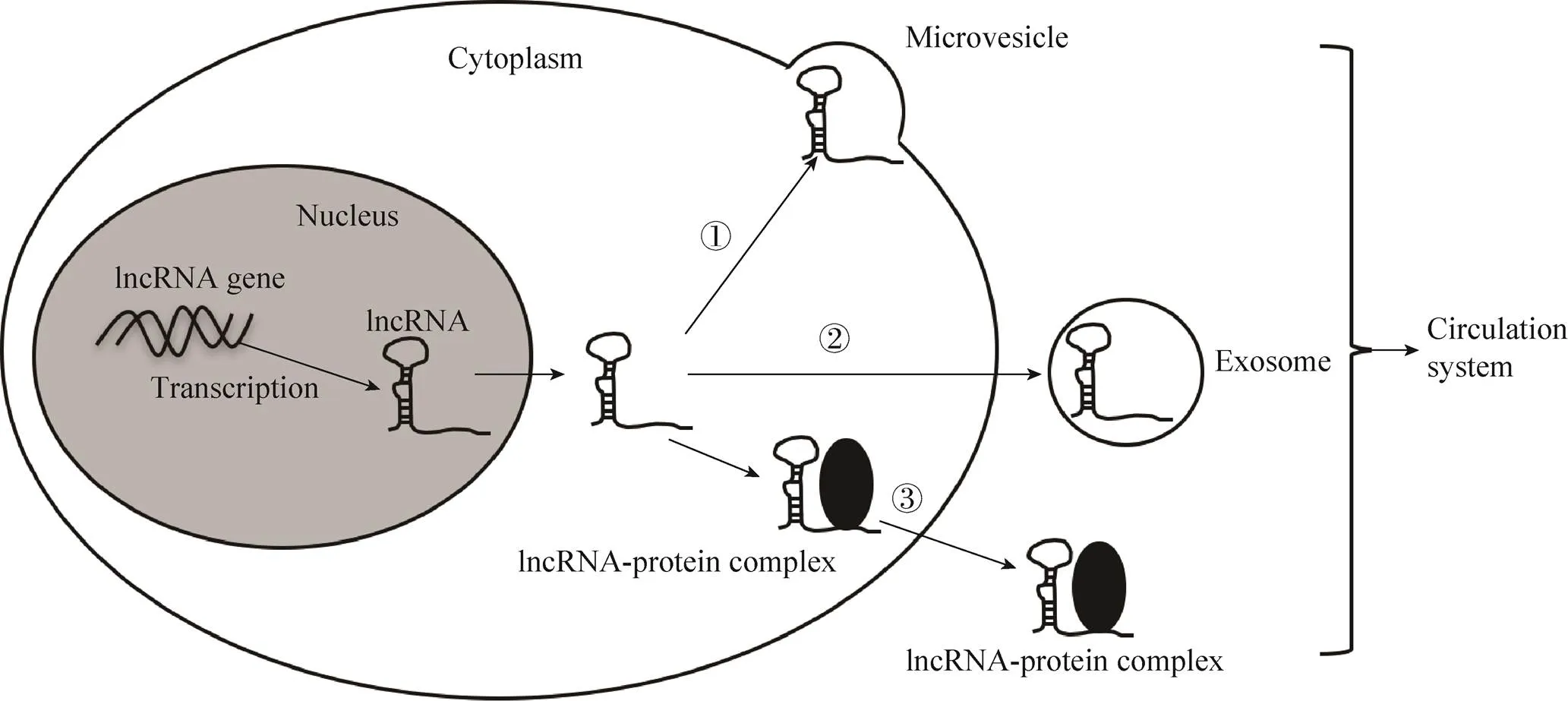
图1 循环长链非编码RNA的来源
3 循环lncRNA的检测
检测循环lncRNA的常用方法有微阵列芯片 (Microarray)、RNA 测序 (RNA sequencing)、Northern印迹杂交 (Northern blotting) 和定量RT-PCR (Quantitative RT-PCR) 等。lncRNA微阵列芯片是将已知序列的lncRNA利用微处理技术,固定在玻璃或薄膜芯片上,对患者和健康对照样本进行lncRNA表达谱分析,通过生物信息学的方法筛选出具有表达差异的lncRNA,并可通过定量RT-PCR或Northern blotting技术对候选lncRNA进行验证,确定其表达水平。利用芯片检测的优势是可以平行地、大量地、全面地分析,具有高度自动化、规模化及微型化的特性,但是微阵列分析的成本费用较高,同时生成的数据量非常庞大且数据形态比一般实验数据更加复杂,后续需要进行大量的数据分析来提高其检测的准确性[23]。lncRNA测序 (lncRNA-Seq) 是一种使用特定方法降低样本中rRNA的丰度,然后对富集到的RNA进行文库构建、测序及分析,从而快速、全面、准确地获得与特定生物学过程 (例如发育、疾病等) 相关lncRNA信息的研究方法。该方法可以更加高效地获取lncRNA序列,且突破了常规lncRNA芯片检测技术的使用范围限制[24-25]。但RNA-Seq成本较高,对RNA纯度要求高、文库构建步骤繁琐。Northern 印记杂交 (Northern blotting) 是一种将RNA从琼脂糖凝胶中转印到硝酸纤维素膜上,利用RNA探针进行检测的方法。它具有灵敏性高的特点,不仅可用于检测lncRNA在血样中的表达水平,还可结合使用RNA marker检测lncRNA的分子大小,这对于排除其他小分子RNA的污染有重要意义。Northern 印迹杂交也可作为lncRNA定量的方法,其缺点是对RNase污染敏感,任一步操作不当都会严重影响实验结果。另外,Northern印迹杂交对样品的需求量较高,需要微克级样品才能避免假阴性,有时样品量达到40 μg以上才会出现明显杂交信号[26-27]。定量RT-PCR也是现在用来检测lncRNA的一种常用检测手段,同时也是验证芯片技术等的准确性的金标准。因其较为简便的步骤和低廉的成本从而得到了大量的应用[28]。总之,以上各种常见检测方法各有优缺点,通常在实践中根据不同的目的选择不同的方法来检测lncRNA表达情况 (表1)。
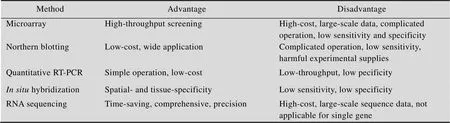
表1 循环lncRNA主要研究技术及优缺点
4 循环lncRNA与肿瘤诊断
由于循环lncRNA可被精确检测,因此循环系统lncRNA可作为一种新型生物标志物用于肿瘤诊断。目前通过检测循环lncRNA表达情况,已在多种肿瘤的临床诊断中得到应用。
4.1 肺癌
肺癌的发病率和致死率居各恶性肿瘤之首。相当一部分患者被确诊时已是晚期,预后较差,5年生存率不足15%。目前肺癌的早期诊断技术正在不断取得进展,其中发掘新型肿瘤标志物刻不容缓。循环lncRNA作为一种微创的生物标志物,已在肺癌中得到验证。Weber等[29]在45例非小细胞肺癌 (Non-small cell lung cancer,NSCLC) 患者和25例健康志愿者血液样本中检测lncRNA MALAT 1的表达来确定其作为循环标志物的价值,结果发现其在NSCLC患者中显著高表达,受试者工作特征曲线 (Receiver operating characteristic curve,ROC) 分析ROC曲线下的面积 (Area under ROC curve,AUC) 值为0.79,这表明循环MALAT1作为NSCLC诊断的价值较高,其微创性和较高的特异性都表明MALAT1作为肿瘤标志物的应用价值。Hu等[30]通过检测NSCLC患者血浆中异常表达的lncRNA来筛选新的标志物。首先通过在20例肺癌患者和20例健康志愿者的样本中筛选出了6个与组织中特异性表达一致的候选lncRNA,在扩大样本库中再一次进行检测验证,其中3个仍有显著意义,分别为lncRNA SPRY4-IT1、ANRIL和NEAT1。进一步分析对其诊断的价值,分别得到其AUC值分别为0.603、0.798和0.693,同时联合检测上述3种lncRNA呈现出更高的敏感性和特异性,其AUC为0.876。这些结果表明上述3种lncRNA有望成为诊断NSCLC的新型标志物。Tang等[31]首先通过基因芯片技术筛选出8个在NSCLC患者中特异性表达的lncRNA,同时设置训练集 (Training set) 包括20例肺癌患者和20例健康志愿者样本,利用定量RT-PCR技术进行验证,获得3个有意义的lncRNA (RP11-397D12.4、AC007403.1和ERICH1-AS1)。进一步在包括232例肺癌患者和135例健康志愿者血液样本的验证集 (Validation set) 中进行表达水平检测,均为显著高表达。随后为了检验这3种lncRNA的诊断价值,分别进行了ROC曲线和AUC值的分析,均显示其有较好的特异性和敏感性。Tang等还对这3种lncRNA进行了双盲检测,在50例癌症样本和50例健康志愿者中准确率高达89%。这样避免了主观意识对客观结果的干扰,提高了准确率。Wang等[32]发现lncRNA UCA1在NSCLC患者癌组织中显著高表达于癌旁组织,进一步检测该基因在癌症患者血浆中的表达,发现在患者中显著性地高表达,并且通过对比发现60例癌症患者的血浆中lncRNA表达和癌组织中的表达关系十分紧密 (=0.881),AUC值为0.886,这些结果表明UCA1有作为循环标志物的价值。Liang等[33]通过前期研究发现lncRNA GAS5在NSCLC组织当中出现差异性表达,之后通过检测lnRNA GAS5在90例NSCLC患者和30例健康志愿者血浆样本中的表达发现出现显著下调,并且lncRNA GAS5与NSCLC肿瘤分期存在非常大的关系 (=0.024),Ⅲ和Ⅳ期的表达量要明显低于Ⅰ和Ⅱ期。GAS5的AUC值为0.832 (<0.000 1;敏感性为82.2%;特异性为72%),表明其可以用于区别NSCLC患者和普通人群,同时将其和目前常用的癌胚抗原 (CEA) 结合使用后,AUC值高达0.909。这些都说明GAS5具有成为NSCLC生物标志物的潜在价值。综上所述,循环系统中发现的差异表达lncRNA与肺癌患者的各种病理因素相关,有望作为肺癌诊断的新型生物标志物。
4.2 乳腺癌
乳腺癌是发生在乳腺腺上皮组织的恶性肿瘤,其发病率和死亡率近年来急剧上升[34]。循环lncRNA在乳腺癌诊断中的应用已有探索。Xu等[35]通过在68例乳腺癌患者和68例健康正常人血清样本中使用定量RT-PCR方法检测lncRNA RP11-445H22.4的表达,结果发现该lncRNA在乳腺癌患者的血清中显著高表达。为了更好地探究该lncRNA的临床价值,研究人员进一步比较了lncRNA与常见肿瘤血清标志物AFP、CEA、CA125和CA153等的ROC和AUC值,结果表明lncRNA RP11-445H22.4的敏感性和特异性最高。除此之外,通过与传统超声波的诊断方法进行比较,lncRNA也有更为显著的敏感性。这些结果表明lncRNA RP11-445H22.4有可能成为新的潜在的乳腺癌生物标志物。Miao等[36]在78对乳腺癌患者的癌组织和癌旁组织中检测lncRNA MALAT1的表达,发现在癌组织中显著高表达,而且lncRNA MALAT1在乳腺癌组织中的表达与淋巴结转移有关,但和患者的年龄、肿瘤大小、细胞分化、组织学分型、雌激素受体 (ER)和雄激素受体 (PR) 等无关。同时,在乳腺癌患者的血清样本中,lncRNA MALAT1的表达显著高于良性乳腺疾病,ROC曲线分析AUC值为0.833,分析表明其敏感性和特异性可以将乳腺癌和其他良性疾病区分开,这表明lncRNA MALAT1可作为诊断用标志物筛选乳腺癌患者。Zhang等[37]用同样的方法先在癌组织和癌旁组织中确定lncRNA H19的差异性表达后,再在102例乳腺癌患者血清样本和96例健康对照当中检测其表达,发现其显著高表达 (<0.05),其AUC为0.81,高于CEA和CA153两个传统乳腺癌循环标志。除此之外,实验还发现lncRNA H19在手术后的血清样本中的表达要显著低于术前水平 (=0.000 6),表明H19除了具有成为乳腺癌标志物外还能用于预后检测。以上研究表明,循环lncRNA已在乳腺癌中得到初步应用,显示了较大的应用潜力。
4.3 胃癌
胃癌发病率和致死率占恶性肿瘤的第二位,诊断延误和有限的治疗手段让胃癌一直成为临床上的一个重要挑战。Hashad等[21]通过研究发现lncRNA H19在胃癌中有特异性的表达。经过检测32例胃癌患者和32例年龄和性别都相匹配的健康志愿者血液样本之后,发现lncRNA H19在胃癌患者中显著高表达,并且与性别和年龄都无关。结合目前常用的肿瘤血清标志物CEA分析,发现lncRNA H19的表达水平与CEA的水平有关,同时经过ROC曲线分析发现lncRNA H19作为生物标志物要优于CEA。联合分析CEA和lncRNA H19用于胃癌诊断,其AUC值进一步提高到80.4%。这些结果表明lncRNA H19是一个非常有价值的潜在的新型胃癌标志物,并且和CEA进行联合诊断将取得更好的效果。Jin等[38]通过对173例胃癌患者 (其中包括100例早期胃癌患者,62例接受过外科手术患者和11例复发患者的血液样本检测),以及30例肠息肉、30例非典型增生或肠上皮化生病人和110例年龄和性别都相匹配的健康志愿者血液样本检测lncRNA HULC表达,发现相较于正常健康人和患有肠息肉等普通胃部疾病的患者,胃癌患者HULC呈显著高表达。在血清中,HULC的表达从早期癌症状态到癌症阶段出现了动态改变。进一步地将HULC (AUC=0.888) 和CEA (AUC=0.694)、CA72-4 (AUC=0.514) 等其他常用血清标志物相比较,发现HULC具有更有效的诊断能力。Dong等[39]通过以“癌症”为关键词在LncRNA-Disease关联数据库中筛选出65个癌症相关lncRNA,再通过NCBI网站中的Refseq database进一步筛选了其中的39个lncRNA。然后筛选出在40对癌组织和癌旁组织中出现有统计学意义的差异性表达的lncRNA,再将这些lncRNA在10例胃癌患者血液样本和10例健康志愿者血液样本中进行检测,最终得到lncRNA CUDR、LSINCT-5和PTENP1三个显著下调且联合使用时有最好诊断能力 (AUC=0.92) 的lncRNA。与常用血液胃癌标志物CEA和CA19-9相比有较好的诊断价值,说明血清中这3种lncRNA有希望成为比CEA和CA19-9更好的胃癌诊断标志物。综上所述,在胃癌诊断当中,循环lncRNA在大量研究中已展现其应用价值,并且在多指标联合诊断中可以发挥更大的功效,为未来胃癌的创新诊断方式研究提供了新的手段。
4.4 肝癌
肝细胞癌 (Hepatocellular carcinoma,HCC) 是世界范围内的常见癌症,也是致死率位居第二的癌症。Wang等[40]通过分析血液中特异性表达的lncRNA,发现在肝癌患者中显著高表达于乙肝患者和健康人群,通过对其诊断价值分析得出lncRNA uc003wbd和AF085935可以用于区分肝癌与乙肝和健康人,这表明上述两个lncRNA有较大诊断价值。同时,Lu等[41]通过在137例HCC患者、104例乙肝患者和138例健康志愿者总共379例血清样本中检测lncRNA uc003wbd和lncRNA-AF085935的表达,发现HCC患者和乙肝患者的血清样本中lncRNA都是显著高表达于健康志愿者组,同时HCC组中的表达也显著高于乙肝患者组。进一步ROC曲线分析,表明在HCC患者和乙肝患者的区别中,lncRNA uc003wbd的AUC值为0.70,lncRN AF085935的AUC值为0.86。而在区别HCC患者和健康对照组时,上述两个lncRNA的AUC值分别为0.86和0.96。这些结果说明这两种lncRNA在HCC和乙肝患者中都显著地高表达于正常人群,同时它们在HCC患者中的高表达又可用于区分HCC和乙肝。因此,这两种lncRNA都有可能成为HCC新的血清生物标志物。Kamel等[42]通过在Database of Cancer Gene Network from Public Gene Expression Data筛选出UCA1和WRAP53两个和HCC相关的lncRNA,然后分别在82例早期HCC患者、34例丙型肝炎患者、44例健康志愿者的血样样本中和20对HCC患者癌组织和癌旁组织中检测了这两个lncRNA的表达,结果表明上述两个lncRNA在HCC患者血液样本中显著高表达,而且在癌组织中同样也是显著高表达,同时发现了lncRNA在血清样本和癌组织中的表达存在较高的相关性 (UCA1,<0.01;WRAP53,<0.02)。Li等[43]通过在8个候选lncRNA中先进行小样本量筛选出3个具有差异表达的lncRNA HULC和Linc00152,在肝细胞癌血清样本中显著高表达。之后放大样本量再次进行表达量检测,lncRNA HULC和Linc00152仍具有差异性,且值均小于0.000 1。两者通过ROC分析之后得到的AUC值分别为0.78和0.85,联合诊断后达到0.87。两个lncRNA更是在双盲检测中达到81.9%的准确率。Yu等[44]的研究认为标志物如果只建立在单一的与肿瘤相关的lncRNA上,其特异性与敏感性都不是特别好。若将多种肿瘤相关lncRNA结合使用后可大大提高标志物的诊断价值,其发现PVT1和uc002mbe可以用于区分HCC患者和健康组,AUC值为0.764 (95%CI:0.648−0.833),效果优于传统标志物AFP,因而具有非常高的应用价值。以上结果表明,多种循环lncRNA均具有成为肝细胞癌新型标志物的潜力,将得到广泛应用。
4.5 结直肠癌
结肠直肠癌是消化道常见的恶性肿瘤,其发病率和病死率在消化系统恶性肿瘤中仅次于胃癌、食管癌和原发性肝癌。Yang等[45]检测10对结肠癌及癌旁组织中lncRNA HOTAIR的表达发现HOTAIR在癌组织中显著高表达,进一步检测lncRNA HOTAIR在47例结肠癌患者血清和40例健康志愿者血清样本之中的相对表达,同样发现lncRNA HOTAIR在结肠癌患者血清样本中显著高表达于健康志愿者。通过ROC曲线分析发现当血清中lncRNA HOTAIR的表达值大于13.30时,诊断结肠癌的敏感性为65.96%,特异性为85%,其AUC值为0.741。这些结果表明血清中的HOTAIR的表达有可能用于结肠癌的诊断。该研究组还进一步检测了lncRNA HOTAIR在血清中表达的稳定性,通过放置在室温数天和反复冻融数次之后检测相对表达后发现HOTAIR稳定地存在于结肠癌患者血清中。这表明lncRNA在血清中有较高稳定性,具备作为肿瘤标志物的重要特征。Kun等[46]在80对结肠癌患者的癌和癌旁组织中检测lncRNA UCA1的相对表达,结果发现在癌组织中lncRNA UCA1呈显著高表达。进一步在20例结肠癌患者血清样本和20例健康志愿者血清样本中检测发现在结肠癌患者中UCA1显著高表达,组织中的UCA1表达水平和血清中表达水平有明显的相关性,并且血清中UCA1的表达在切除性手术14 d之后显著下降,显示其还可作为结肠癌预后的标志物。以上研究结果表明,血清中lncRNA的可检测性和稳定性都表明其具有成为结肠癌诊断用标志物的潜力。
4.6 前列腺癌
前列腺癌是一种男性十分常见的癌症类型,在全球范围内发生呈上升趋势,尤其是在亚洲国家。目前早期诊断前列腺癌主要依靠血液中前列腺特异性抗原 (PSA) 检测。lncRNA PCA3是一个目前在前列腺癌中被较好研究过的lncRNA,在前列腺癌组织中显著高表达,并且可以在尿液中检测到,是较早的可以在尿液中检测的分子诊断标志物[47]。Feibus等[48]研究发现lncRNA PCA3在前列腺癌患者尿液中显著高表达。同时Lee等[49]利用lncRNA PCA3作为阳性对照,GAPDH作为内参对照,有6种lncRNA (AK024556、XLOC_07697、LOC100287482、XLOC_005327、XLOC_008559和XLOC_009911)已经被证明在前列腺癌组织中显著高表达,进一步研究发现在患者尿液样本中同样发现它们呈显著高表达。Isin等[50]研究发现lincRNA-p21可以用来区分前列腺癌和良性前列腺增生 (BPH)。他们检测了lincRNA-p21在30例前列腺癌患者和49例BPH患者的尿液样本中的表达,发现lincRNA-p21显著高表达于前列腺癌症患者的尿液中,进一步比较lincRNA-p21和其与常规血清标志物PSA联合使用的ROC曲线,发现联合检测lincRNA-p21和PSA后特异性显著增加,但敏感性没有显著改变。Wang等[51]同样对尿液样本中的lncRNA MALAT-1进行检测,利用MALAT-1分数 (MALAT-1分数= 2Ct(PSA)-Ct(MALAT-1)×1 000) 来判断其价值,结果发现在前列腺穿刺阳性的患者中MALAT-1分数显著高于穿刺阴性患者。同时ROC曲线结果也显示lncRNA MALAT-1有着比PSA更高的AUC值,这些都说明lncRNA MALAT-1有望成为新型的前列腺癌潜在标志物。
综上所述,循环lncRNA在常见恶性肿瘤的早期诊断中具有广泛的应用价值,有可能发展成为一种新型的生物标志物 (表2)。
5 小结与展望
目前癌症仍旧是困扰人类的重大问题,而在癌症治疗当中早期诊断更是起到了非常大的作用,如果患者能够在早期诊断中发现癌症,许多病例完全可以被治愈。如今随着分子生物学技术的不断进步和高通量测序技术的发展,人们越来越关注以前被称为是基因组“垃圾”的lncRNA。同时大量研究表明lncRNA在基因表达调控以及细胞的生长、增殖、凋亡和细胞通讯之间都起到了重要的作用[52-53],目前研究也发现lncRNA在多种肿瘤中有着不同程度的表达失调,并且与肿瘤发生发展、侵袭迁移等生物学过程存在密切关系[54]。同时进入循环系统的lncRNA被发现能够稳定地存在于血液或其他体液 (尿液、唾液、乳汁等) 中,这样使得lncRNA能够方便地被检测出来。循环lncRNA作为一种创伤性小、特异性和敏感性都较传统肿瘤标志物高的一种分子标志物,越来越引起人们的重视。
然而目前循环lncRNA的检测仍然存在诸多不足,尚不可直接在临床实践中用于各种癌症的早期诊断。比如循环lncRNA的内参基因没有较为统一的说法,无法确定选取何种基因作为内参是稳定的,以及如何用合适的内参基因来计算循环lncRNA的表达,如何提高检测的准确性等技术问题还有待进一步研究。同时,许多循环lncRNA尽管通过前置放大之后,由于其表达水平相对于其他循环核酸较低,在循环系统中仍难以检测出。因此,如何提高循环lncRNA的检测,在方法的选择和建立上都有待改进[55]。再者,目前发现存在差异性表达的循环lncRNA缺少针对特定肿瘤的特异性,如H19被发现在胃癌、肝癌、结直肠癌、乳腺癌等多种癌症中存在差异性表达。而癌症的发生发展是多基因联合作用的结果,仅检测一种循环lncRNA,其特异性和灵敏性均有限,研究发现基于多个lncRNA的联合检测,以及与传统血清标志物的联合诊断应用可以大大提高诊断价值,这将是今后重要的发展方向。随着lncRNA的基础理论和应用研究的不断深入和发展,循环lncRNA用于癌症辅助诊断的价值将得到进一步的提高。
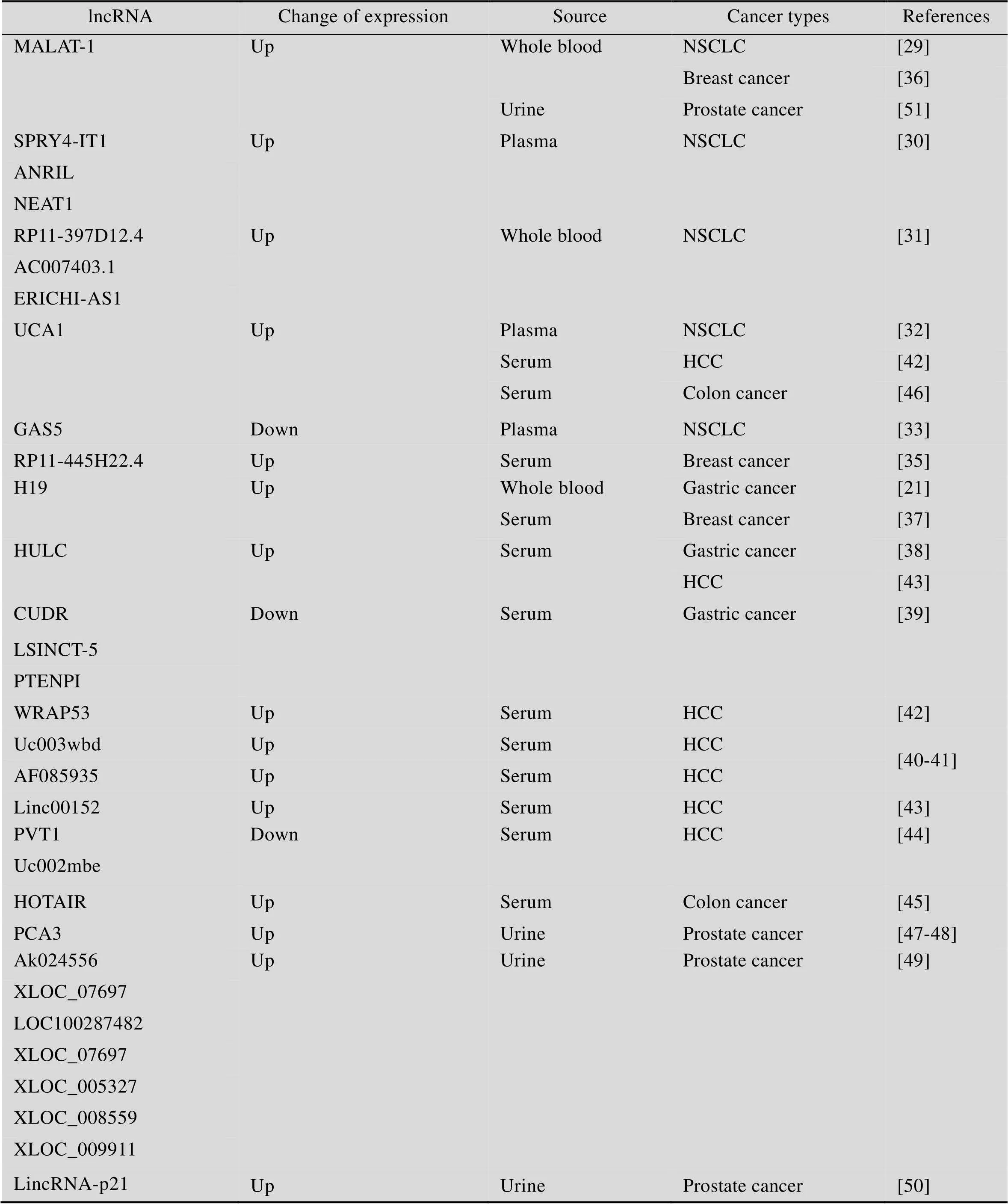
表2 循环lncRNA在肿瘤诊断中的应用
[1] Siegel RL, Miller KD, Jemal A. Cancer statistics. CA Cancer J Clin, 2016, 66(1): 10–29.
[2] Sawyers CL. The cancer biomarker problem. Nature, 2008, 452(7187): 548–552.
[3] Batista PJ, Chang HY. Long noncoding RNAs: cellular address codes in development and disease. Cell, 2013, 152(6): 1298–1307.
[4] Liu XW, Xiao ZD, Han L, et al. LncRNAengages a metabolic checkpoint by regulating AMPK under energy stress. Nat Cell Biol, 2016, 18(4): 431–442.
[5] Gupta RA, Shah N, Wang KC, et al. Long non-coding RNAreprograms chromatin state to promote cancer metastasis. Nature, 2010, 464(7291): 1071–1076.
[6] Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs. Cell, 2009, 136(4): 629–641.
[7] Zhang H, Chen ZH, Wang XX, et al. Long non-coding RNA: a new player in cancer. J Hematol Oncol, 2012, 6: 37.
[8] Bazanova NV, Seĭts IF. Can the presence of an RNA-lipoprotein complex in human blood serum give evidence of a cancerous disease?. Eksp Onkol, 1989, 11(2): 37–39.
[9] Kopreski MS, Benko FA, Kwak LW, et al. Detection of tumor messenger RNA in the serum of patients with malignant melanoma. Clin Cancer Res, 1999, 5(8): 1961–1965.
[10] Dong L, Lin WR, Qi P, et al. Circulating long RNAs in serum extracellular vesicles: their characterization and potential application as biomarkers for diagnosis of colorectal cancer. Cancer Epidemiol Biomarkers Prev, 2016, 25(7): 1158–1166.
[11] Arroyo JD, Chevillet JR, Kroh EM, et al. Argonaute2 complexes carry a population of circulating microRNAs independent of vesicles in human plasma. Proc Natl Acad Sci USA, 2011, 108(12): 5003–5008.
[12] Pantel K, Speicher MR. The biology of circulating tumor cells. Oncogene, 2016, 35: 1216–1224.
[13] Wang JM, Huang PL. Research progress of circulating RNA detection and micrometastasis of the peripheral blood in tumors. J Southeast Univ Med Sci Ed, 2002, 21(3): 265–268 (in Chinese). 王景美, 黄培林. 循环RNA测定与肿瘤外周血微转移的研究进展. 东南大学学报: 医学版, 2002, 21(3): 265–268.
[14] Stroun M, Lyautey J, Lederrey C, et al. About the possible origin and mechanism of circulating DNA: apoptosis and active DNA release. Clin Chim Acta, 2001, 313(1–2): 139–142.
[15] Lee YN. Carcinoembryonic antigen in patients with breast or colon cancer. West J Med, 1978, 129(5): 374–380.
[16] Li C, Nie CH, Zhang LJ, et al. Advances in research on application of tumor markers and their screening technique. Prog Pharm Sci, 2014, 38(1): 1–13 (in Chinese).李策, 聂彩辉, 张力君, 等. 肿瘤标志物的应用及其筛选技术研究进展. 药学进展, 2014, 38(1): 1–13.
[17] Tinzl M, Marberger M, Horvath S, et al. DD3PCA3 RNA analysis in urine-a new perspective for detecting prostate cancer. Eur Urol, 2004, 46(2): 182–186.
[18] Arita T, Ichikawa D, Konishi H, et al. Circulating long non-coding RNAs in plasma of patients with gastric cancer. Anticancer Res, 2013, 33(8): 3185–3193.
[19] Isin M, Ozgur E, Cetin G, et al. Investigation of circulating lncRNAs in B-cell neoplasms. Clin Chim Acta, 2014, 431: 255–259.
[20] Su YJ, Yu J, Huang YQ, et al. Circulating long noncoding RNA as a potential target for prostate cancer. Int J Mol Sci, 2015, 16(6): 13322–13338.
[21] Hashad D, Elbanna A, Ibrahim A, et al. Evaluation of the role of circulating long non-coding RNAas a promising novel biomarker in plasma of patients with gastric cancer. J Clin Lab Anal, 2016, 30(6): 1100–1105.
[22] Silva A, Bullock M, Calin G. The clinical relevance of long non-coding RNAs in cancer. Cancers (Basel), 2015, 7(4): 2169–2182.
[23] Zhang C, Gao W, Wen S, et al. Potential key molecular correlations in laryngeal squamous cell carcinoma revealed by integrated analysis of mRNA, miRNA and lncRNA microarray profiles. Neoplasma, 2016, 63(6): 888–900.
[24] Guo YB. Sequencing technology in gene diagnosis and its application. Hereditas (Beijing), 2014, 36(11): 1121–1130 (in Chinese). 郭奕斌. 基因诊断中测序技术的应用及优缺点. 遗传, 2014, 36(11): 1121–1130.
[25] Agrawal V, Varadan V, Banerjee N, et al. Abstract P6–03–08: novel recurrent lncRNA fusions detected in breast cancer using RNA-Seq technology in a neoadjuvant setting. Cancer Res, 2016, 76(S4): P6–03–08.
[26] Huang Q, Mao ZN, Li SQ, et al. A non-radioactive method for small RNA detection by northern blotting. Rice, 2014, 7: 26.
[27] Qiu JJ, Yan JB. Genomic imprinting and long noncoding RNA. Chin Biotechnol, 2014, 34(7): 63–68 (in Chinese). 邱家俊, 颜景斌. 基因印记与lncRNA. 中国生物工程杂志, 2014, 34(7): 63–68.
[28] Nolan T, Hands RE, Bustin SA. Quantification of mRNA using real-time RT-PCR. Nat Protoc, 2006, 1(3): 1559–1582.
[29] Weber DG, Johnen G, Casjens S, et al. Evaluation of long noncoding RNA MALAT1 as a candidate blood-based biomarker for the diagnosis of non-small cell lung cancer. BMC Res Notes, 2013, 6: 158.
[30] Hu XD, Bao JT, Wang Z, et al. The plasma lncRNA acting as fingerprint in non-small-cell lung cancer. Tumour Biol, 2016, 37(3): 3497–3504.
[31] Tang Q, Ni Z, Cheng Z, et al. Three circulating long non-coding RNAs act as biomarkers for predicting NSCLC. Cell Physiol Biochem, 2015, 37(3): 1002–1009.
[32] Wang HM, Lu JH, Chen WY, et al. Upregulated lncRNA-UCA1 contributes to progression of lung cancer and is closely related to clinical diagnosis as a predictive biomarker in plasma. Int J Clin Exp Med, 2015, 8(7): 11824–11830.
[33] Liang WJ, Lv T, Shi XF, et al. Circulating long noncoding RNA GAS5 is a novel biomarker for the diagnosis of nonsmall cell lung cancer. Medicine, 2016, 95(37): e4608.
[34] Jemal A, Center MM, Desantis C, et al. Global patterns of cancer incidence and mortality rates and trends. Cancer Epidemiol Biomarkers Prev, 2010, 19(8): 1893–1907.
[35] Xu N, Chen F, Wang FL, et al. Clinical significance of high expression of circulating serum lncRNA RP11–445H22.4 in breast cancer patients: a Chinese population-based study. Tumor Biol, 2015, 36(10): 7659–7665.
[36] Miao Y, Fan R, Chen L, et al. Clinical significance of long non-coding RNA MALAT1 expression in tissue and serum of breast cancer. Ann Clin Lab Sci, 2016, 46(4): 418–424.
[37] Zhang KJ, Luo ZL, Zhang Y, et al. Circulating lncRNA H19 in plasma as a novel biomarker for breast cancer. Cancer Biomarkers, 2016, 17(2): 187–194.
[38] Jin CJ, Shi W, Wang F, et al. Long non-coding RNA HULC as a novel serum biomarker for diagnosis and prognosis prediction of gastric cancer. Oncotarget, 2016, 7(32): 51763–51772.
[39] Dong L, Qi P, Xu MD, et al. Circulating CUDR, LSINCT-5 and PTENP1 long noncoding RNAs in sera distinguish patients with gastric cancer from healthy controls. Int J Cancer, 2015, 137(5): 1128–1135.
[40] Wang K. Serum incRNAs profiles serve as novel biomarkers for early diagnosis of HBV-positive hepatocarcinoma. Shanghai: The Second Military Medicine University, 2013. 王康. 血清长链非编码RNAs作为伴乙肝肝癌早期诊断标记物的研究[D]. 上海: 第二军医大学, 2013.
[41] Lu JJ, Xie F, Geng L, et al. Investigation of serum lncRNA-uc003wbd and lncRNA-AF085935 expression profile in patients with hepatocellular carcinoma and HBV. Tumor Biol, 2014, 36(5): 3231–3236.
[42] Kamel MM, Matboli M, Sallam M, et al. Investigation of long noncoding RNAs expression profile as potential serum biomarkers in patients with hepatocellular carcinoma. Transl Res, 2016, 168: 134–145.
[43] Li J, Wang XC, Tang JW, et al. HULC and Linc00152 act as novel biomarkers in predicting diagnosis of hepatocellular carcinoma. Cell Physiol Biochem, 2015, 37(2): 687–696.
[44] Yu JY, Han JQ, Zhang J, et al. The long noncoding RNAs PVT1 and uc002mbe.2 in sera provide a new supplementary method for hepatocellular carcinoma diagnosis. Medicine, 2016, 95(31): e4436.
[45] Yang X, Li BS, Hu CJ, et al. Establishment of detection method of serum lncRNA HOTAIR level and its significance in diagnosis of colon cancer. J Third Mil Med Univ, 2016, 38(10): 1148–1153.杨歆, 黎伯胜, 胡长江, 等. 血清中lncRNA HOTAIR检测方法的建立及其在结肠癌诊断中的应用价值. 第三军医大学学报, 2016, 38(10): 1148–1153.
[46] Tao K, Yang J, Hu Y, et al. Clinical significance of urothelial carcinoma associated 1 in colon cancer. Int J Clin Exp Med, 2015, 8(11): 21854–21860.
[47] Lee GL, Dobi A, Srivastava S. Prostate cancer: diagnostic performance of theurine test. Nat Rev Urol, 2011, 8(3): 123–124.
[48] Feibus AH, Sartor O, Moparty K, et al. Clinical use of PCA3 and TMPRSS2: ERG urinary biomarkers in African-American men undergoing prostate biopsy. J Urol, 2016, 196(4): 1053–1060.
[49] Lee B, Mazar J, Aftab MN, et al. Long noncoding RNAs as putative biomarkers for prostate cancer detection. J Mol Diagn, 2014, 16(6): 615–626.
[50] Işın M, Uysaler E, Özgür E, et al. Exosomal lncRNA-p21 levels may help to distinguish prostate cancer from benign disease. Front Genet, 2015, 6: 168.
[51] Wang FB, Ren SC, Chen R, et al. Development and prospective multicenter evaluation of the long noncoding RNA MALAT-1 as a diagnostic urinary biomarker for prostate cancer. Oncotarget, 2014, 5(22): 11091–11102.
[52] Ganepola GAP, Nizin J, Rutledge JR, et al. Use of blood-based biomarkers for early diagnosis and surveillance of colorectal cancer. World J Gastrointest Oncol, 2014, 6(4): 83–97.
[53] Li JQ, Tian HH, Yang J, et al. Long noncoding RNAs regulate cell growth, proliferation, and apoptosis. DNA Cell Biol, 2016, 35(9): 459–470.
[54] Li H, Yu BQ, Li JF, et al. Overexpression of lncRNA H19 enhances carcinogenesis and metastasis of gastric cancer. Oncotarget, 2014, 5(8): 2318–2329.
[55] Schlosser K, Hanson J, Villeneuve PJ, et al. Assessment of circulating lncRNAs under physiologic and pathologic conditions in humans reveals potential limitations as biomarkers. Sci Rep, 2016, 6: 36596.
(本文责编 陈宏宇)
Circulating long noncoding RNAs as biomarkers in tumor diagnosis
Nan Jiang1, Haihua Tian1,2, Jinchang Pan1, and Zhaohui Gong1
1 Institute of Biochemistry and Molecular Biology, Medical School of Ningbo University, Ningbo 315211, Zhejiang, China 2 Department of Laboratory Medicine, Ningbo Kangning Hospital, Ningbo 315201, Zhejiang, China
Long noncoding RNAs (lncRNAs) are involved in vital life processes of gene expression, epigenetic regulation and X-chromosome inactivation. lncRNAs are also closely associated with tumor initiation and progression. Moreover, lncRNAs may enter human circulation system in the form of microvesicle or exosome, or in combination with RNA binding protein. Interestingly, the circulating lncRNAs are widely existed in body fluids, such as blood and urine. We review the origin of circulating lncRNAs, and the detection methods as potential biomarkers. We focus on the early diagnosis value of circulating lncRNAs as tumor biomarkers in lung, breast, gastric, liver, colorectal and prostate cancers. Compared with the traditional biomarkers, the circulating lncRNAs show the unique advantages and clinical values as novel biomarkers.
circulating lncRNA, tumor, biomarker, molecular diagnosis
10.13345/j.cjb.160463
November 30, 2016; Accepted: January 24, 2017
Zhaohui Gong. Tel: +86-574-87600740; Fax: +86-574-87608638; E-mail:zhaohui@ncri.org.cn
Supported by:Natural Science Foundation of Zhejiang Province (No. LY15C060003), Natural Science Foundation of Ningbo City (No. 2015A610220), Sci-Tech Project of Ningbo City (No. 2014C50058), K.C.Wong Magna Fund in Ningbo University and the Key Project of Student Research Innovation Plan in Ningbo University (No. 2016002).
浙江省自然科学基金(No. LY15C060003),宁波市自然科学基金(No. 2015A610220),宁波市社发科技攻关项目(No. 2014C50058),宁波大学王宽诚教育基金,宁波大学大学生科研创新计划重点项目 (No. 2016002) 资助。
网络出版时间:2017-02-13
http://www.cnki.net/kcms/detail/11.1998.Q.20170213.1503.004.html
