蓝细菌乙醇光合细胞工厂的发展与展望
2017-07-01齐允晶王家林栾国栋谈晓明吕雪峰
齐允晶,王家林,栾国栋,谈晓明,吕雪峰
蓝细菌乙醇光合细胞工厂的发展与展望
齐允晶1,2,王家林1,栾国栋2,谈晓明2,吕雪峰2
1 青岛科技大学化工学院,山东青岛 266042 2 中国科学院青岛生物能源与过程研究所中国科学院生物燃料重点实验室,山东青岛 266101

齐允晶, 王家林, 栾国栋, 等. 蓝细菌乙醇光合细胞工厂的发展与展望. 生物工程学报, 2017, 33(6): 891–909.Qi YJ, Wang JL, Luan GD, et al. Cyanobacteria cell factories for ethanol photosynthetic production: development and prospect. Chin J Biotech, 2017, 33(6): 891–909.
生物乙醇是极具应用潜力和代表性的生物能源产品之一。以蓝细菌为光合平台,利用二氧化碳和太阳能直接进行乙醇合成可以同时起到降低二氧化碳排放和提供可再生能源的效果,具有重要的研究与应用价值。本文回顾了蓝细菌乙醇光合细胞工厂相关技术的发展历程和现状,从途径优化、底盘选择和代谢工程策略等层面对其最新进展和所遇到的问题进行了总结介绍,并对该技术未来发展方向进行了展望。
蓝细菌,生物乙醇,代谢工程,细胞工厂,乙醇耐受性
全球性的环境污染问题和潜在的能源危机正推动着用于合成新一代绿色生物燃料的可持续性技术路线和生产模式的发展,以期能够有效补充和替代正在被大量消耗的石化燃料[1-2]。作为最早实现商业化推广和应用的生物燃料产品,乙醇已经被广泛接受和应用为燃油添加剂甚至替代品[3-5]。目前绝大部分的生物乙醇来源于生物炼制过程,其生产技术可以根据原料和底物来源划分为3代。最初的生物乙醇合成以含糖量丰富的农作物生物质为原料,其中以“玉米乙醇”最具代表性,但是其“与粮争地、与人争粮”以及原料供应不足的问题引发了极大的社会争议,进而直接限制了该技术的实际可推广性[6-7]。第二代生物乙醇以非粮生物质为主要原料,通过对以木质纤维素为代表的农业废弃物、林业废弃物等的收集、处理和发酵进行乙醇合成,该技术体系缓解了“粮食乙醇”在原料供应上的天然不足,但是纤维素原料预处理过程中对能量、水和纤维素酶等的需求极大地提高了二代生物乙醇的生产成本,束缚了该技术的应用潜力[8]。以真核和原核藻类等易于培养、生长迅速的光合自养生物的生物质提供底物进行生物炼制,可以极大地降低原材料预处理的难度和耗费进而节省生产成本,被称之为第三代生物燃料技术,表现出良好的发展前景[9-11]。
代谢工程与合成生物学相关理论和技术的发展,使得通过基因工程改造,利用光合生物平台将太阳能和二氧化碳直接转化为生物燃料产品的技术逐渐成熟。与传统的生物炼制过程相比,该路线减少了原材料预处理、底物提炼过程的损耗,降低了对培养体系中各种营养成分的需求,也大大节省了化学品合成全过程中对淡水和用地的需求,这些优势使其逐渐成为科研与产业界关注的焦点[12-14]。蓝细菌 (Cyanobacteria),又称蓝藻 (Blue-green algae),是一类能够进行光合作用的原核、自养微生物。与真核微藻相比,蓝细菌具有结构简单、生长迅速、遗传操作便捷等优势,因此成为极具潜力的生物基化学品光合平台[13,15]。在涵盖集胞藻、聚球藻、鱼腥藻等不同种属的模式蓝细菌藻株中,都已经发展起了成熟的遗传操作体系;而转录组学、蛋白质组学和代谢物组学等各种系统生物技术的广泛应用则促进了对蓝细菌在不同环境、不同培养条件下,生理、生化和代谢层面各种响应机制的理解与认识。上述研究和技术基础成为助推蓝细菌光合平台基础上各种生物基化学品和生物燃料产品合成路线成功开发的强大动力。
现阶段通过外源基因引入和天然代谢网络的修饰,已经成功在蓝细菌中实现了氢[16]、醇[17-18]、酮[19]、酸[20-21]、醛[22]、烃[23-24]、糖[25-26]等数十种天然和非天然代谢产物的光合合成。乙醇是最早报道的也是现阶段最具代表性的蓝细菌光合生物燃料产品,开发高效的蓝细菌乙醇光合细胞工厂,既可以促进新的乙醇合成技术体系的发展,又对其他化学品光合平台的构建具有充分的示范意义。本文将对蓝细菌光合生物乙醇相关技术的发展现状进行回顾和总结,并对其前景和方向进行展望。
1 蓝细菌乙醇光合合成的核心途径
在对蓝细菌野生型藻株的研究中发现,其中少数藻株具有极为微弱的乙醇合成能力。1991年,Heyer等对随机挑选的37株野生型蓝细菌藻株进行发酵培养并分析了其代谢产物,结果在其中 16株野生型蓝细菌的发酵产物中检测到了乙醇,最高产量达到0.46 mg/L[27];2010年Carrieri等发现盐胁迫可以使螺旋藻发酵产乙醇的产量提高120倍,达到0.75 mmol/g干重[28]。但总的来说,天然蓝细菌藻株的乙醇合成能力弱而且合成条件苛刻,只能在发酵条件下产生,而且代谢途径不清楚,缺乏实际改造和应用潜力。真正具备改造和应用潜力的蓝细菌乙醇光合合成技术是随着代谢工程和合成生物学技术的发展而逐步成熟的,通过将异源的、高效的乙醇合成途径通过遗传操作导入蓝细菌底盘细胞,并辅之以对天然代谢背景的改造,真正实现了蓝细菌胞内代谢流重构,将其光合作用中固定的二氧化碳导向乙醇合成。
现阶段,所有的蓝细菌乙醇光合细胞工厂的开发都是通过丙酮酸脱羧酶-Ⅱ型乙醇脱氢酶 (Pyruvate decarboxylase-alcohol dehydrogenase Ⅱ,Pdc-AdhII) 途径的引入、重构和调控而实现的(如图1所示)。最初的蓝细菌乙醇光合细胞工厂采用的是来自运动发酵单胞菌中的PdcZM-AdhIIZM途径[17],该途径也普遍应用于其他微生物乙醇细胞工厂的构建中[29-31]。
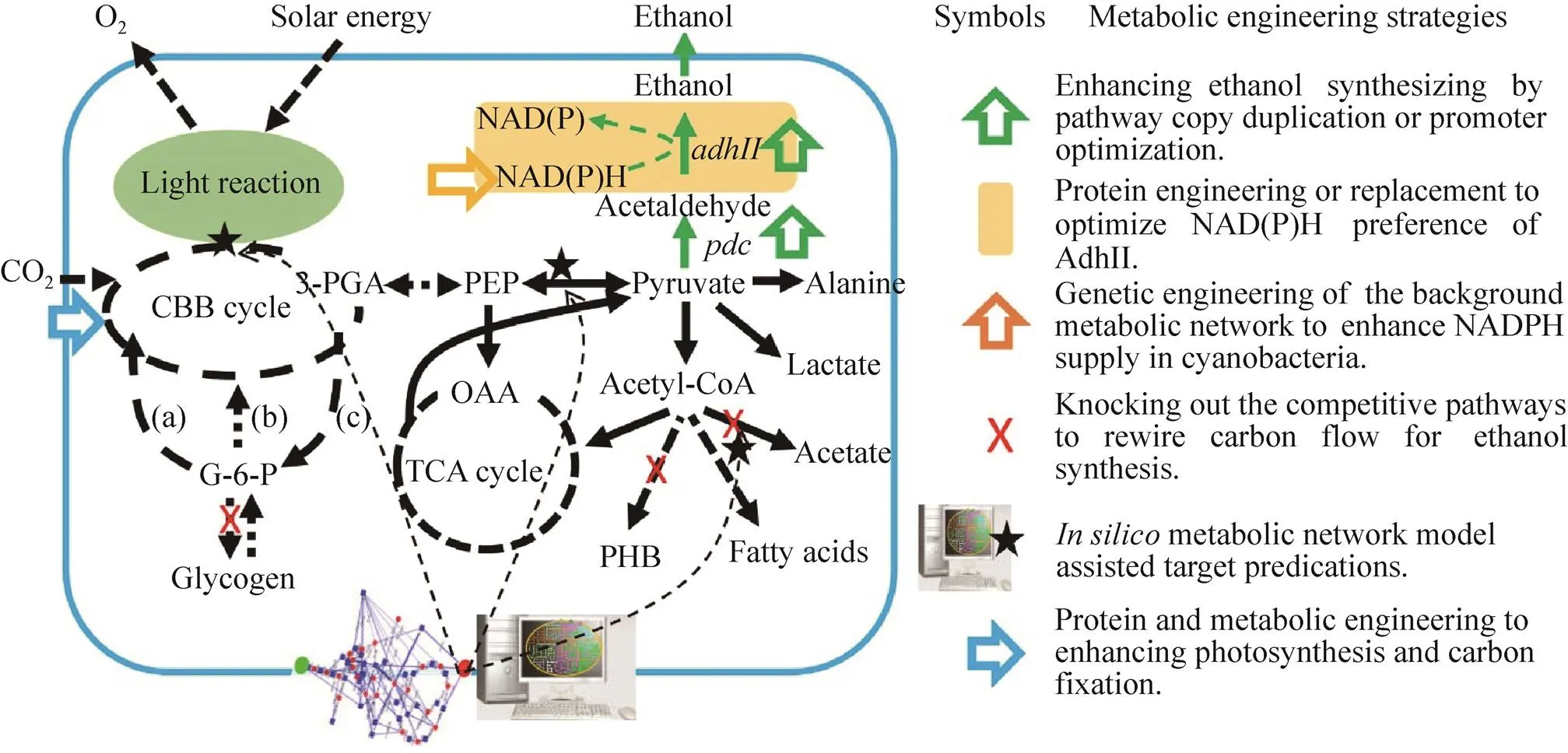
图1 蓝细菌乙醇光合细胞工厂的设计和代谢工程改造策略
1.1 丙酮酸脱羧酶
丙酮酸脱羧酶 (Pyruvate decarboxylase) 是一种焦磷酸硫胺素 (Thiamine pyrophosphate,TPP) 依赖型的羧化酶,其全酶状态由辅酶TPP、金属离子Mg2+(或Mn2+) 以及蛋白自身构成,作用于α-酮酸,经脱羧反应形成二氧化碳和相应的醛分子。丙酮酸在脱羧过程中羰基碳原子上会产生电负性,导致分子结构不稳定,而辅因子TPP的存在可以与其结合,避免此种情况。丙酮酸脱羧酶广泛存在于豆质植物、麻黄等植物 (大豆、麻黄等)、真菌 (酿酒酵母、曲霉等) 以及细菌 (运动发酵单胞菌、醋杆菌等) 中,在动物细胞中不存在该酶,不同来源的丙酮酸脱羧酶性质略有不同。
丙酮酸脱羧酶一直被认为是同型乙醇发酵中的关键酶,其表达与活性极大地影响着乙醇合成的效率。现阶段,蓝细菌乙醇光合细胞工厂中普遍采用的基因来自于运动发酵单胞菌。2002年Raj等从棕榈发酵细菌中克隆、鉴定了一种更高效的丙酮酸脱羧酶PdcZP,相比较于来自运动发酵单胞菌的PdcZM,该酶在pH 6.0和7.0两种条件下都具有更低的m、更高的比活[32]。2012年Algenol公司使用该基因pdc替换pdc,使工程藻株的乙醇产量提高了10%–20%[33-34]。在未来,通过对微生物资源和酶资源的充分挖掘或者通过对现有丙酮酸脱羧酶基因进行定向进化改造[35]以获得活性、稳定性更高的丙酮酸脱羧酶基因,将有希望进一步提高乙醇合成效率。
1.2 Ⅱ型乙醇脱氢酶
醇脱氢酶大量存在于植物、动物和微生物细胞中[36],Ⅱ型醇脱氢酶是一种含锌酶类,以烟酰胺腺嘌呤二核苷酸 (NADH) 或烟酰胺腺嘌呤二核苷酸磷酸 (NADPH) 为辅酶,其全酶形态为含4个亚基的四聚体,每个亚基结合 1个NADH/NADPH和1个Zn2+,催化伯醇和相应醛之间的可逆反应。最初,用于改造蓝细菌进行乙醇合成的Ⅱ型醇脱氢酶基因同样来自运动发酵单胞菌[17,37]。为了提高蓝细菌乙醇合成效率,对乙醇脱氢酶进行的探索和研究则更为广泛和深入。Algenol公司的研究人员对来自运动发酵单胞菌的adhI和adhII、来自集胞藻sp. PCC6803 (以下简称为集胞藻PCC6803) 的,以及来自大肠杆菌K12和嗜热型蓝细菌BP-1的两种双功能醛/醇脱氢酶分别与pdc串联后由铜离子诱导型启动子控制,在集胞藻PCC6803中表达并测定乙醇合成情况。结果证明来自PCC6803自身的基因藻株最适合用于乙醇合成,相应工程藻株获得了最高的乙醇产量[33]。
本实验室此前的研究中获得了类似的结果,通过将对来自集胞藻、鱼腥藻 (sp. PCC7120,以下简称为鱼腥藻PCC7120)、聚球藻 (s sp. PCC7942,以下简称为聚球藻PCC7942) 的9种醇脱氢酶基因 (集胞藻PCC6803的、和,鱼腥藻PCC7120的、、、和,聚球藻PCC7942的) 以及adhII基因分别和pdc串联后在.中表达后,通过工程菌株生长和产醇能力进行初筛,发现pdc-slr1192的组合实现了最高的乙醇合成速率,在集胞藻PCC6803工程藻株中将运动发酵单胞菌来源的adhII替换为,经过20 d培养后的乙醇产量从0.4 g/L提高至0.6 g/L,约为50%的提高幅度[38]。
相比较于异源的adhII,的优势可能来自于两方面:首先,作为催化可逆反应的酶,在乙醛和乙醇两种底物之间亲和性的差异要大于运动发酵单胞菌中的AdhIZM和AdhIIZM[33, 38],后者的可逆性导致了光照培养后期一部分积累的乙醇被重新转化为乙醛进而降低了乙醇产量,而使用的工程藻株则无此现象。另外,更倾向于以NADPH而非NADH为辅酶[39],而集胞藻PCC6803中NADPH的丰度远远超过NADH[40]。上述因素意味着,使用为AdhII的集胞藻PCC6803工程藻株在乙醇光合合成过程中可以获得更稳定的底物和辅因子供应,因而具有更好的产醇潜力。
1.3 Pdc-AdhII途径的系统分析
Pdc-AdhII途径在蓝细菌中将丙酮酸转化为乙醛进而转化为乙醇,在此过程中既存在两种酶之间的承接协作又存在外源途径与底盘细胞之间的适配互作。长远角度看,解析、优化Pdc-AdhII途径,明确其与底盘细胞之间的适配关系对进一步提高乙醇光合效率有重要意义。2015年,本实验室采用体外重构动态分析的策略,对pdc-slr1192途径进行了系统分析。Luan等首先脱离复杂的胞内环境在胞外重新构建该途径,以纯化后的酶 (PdcZM/slr1192)、底物 (丙酮酸,pyruvate)、辅因子 (NADPH/ NADH/TPP)、金属离子 (Mg2+) 以及中间产物 (乙醛,acetaldehyde) 对整个途径的催化效率影响分别进行定量的滴定分析,结果发现PdcZM的K为0.326 μmol/L,而最大反应速率V是 2.074 μmol/(L·s),相应数值分别为 0.109 μmol/L和1.722 μmol/(L·s),表明影响乙醇合成的限制因素是PdcZM而非;而在总蛋白浓度设置恒定的分析中,PdcZM-slr1192的浓度比为4∶6时全途径具有最大的乙醇合成催化活性。我们进而在集胞藻PCC6803中构建了PdcZM-slr1192浓度配比不同的工程菌株,通过代谢工程结合酶活与蛋白含量分析实验,证明pdc而非的表达量和活性是现阶段蓝细菌工程藻株乙醇合成的限制性因素,而提高PdcZM活性和表达强度应该是进一步代谢工程改造的方向。此外,在体外重构分析中,NADPH、丙酮酸、TPP、Mg2+和乙醛等组分的m值分别为0.136、6.496、0.011、0.104和0.393 mmol/L,结合蓝细菌胞内NADPH和丙酮酸等成分的实际含量分析,提高NADPH和丙酮酸的供应量应该是提高乙醇合成效率的重要选择[41]。
2 蓝细菌乙醇光合细胞工厂的底盘细胞选择
蓝细菌是最古老的生命体之一,其种类繁多、分布广泛,在海洋、淡水以及陆域等多种生态系统中都有分布;而不同藻属中有单细胞、丝状体或丝状不分化等多种细胞形式;不同藻株在光合固碳速率、碳汇途径、固氮能力等生理代谢特性上也有差异[42]。系统考虑代谢改造潜力和未来工程化应用的需要而合理地选择底盘细胞,对构建高效的乙醇光合细胞工厂有着重要意义。表1对不同底盘细胞基础上构建的一系列蓝细菌乙醇光合工程藻株进行了总结。
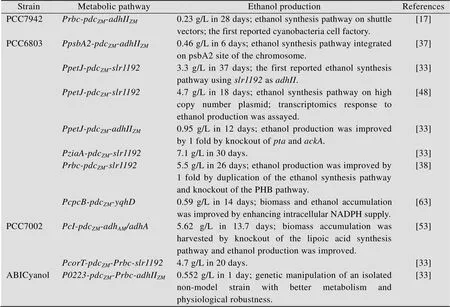
表1 代表性的蓝细菌乙醇光合细胞工厂
2.1 聚球藻sp. PCC7942和集胞藻PCC6803
利用蓝细菌进行乙醇光合合成最初是在聚球藻PCC7942实现的。1999年,Deng等将pdc-adhII途径克隆在穿梭载体pCB4上导入集胞藻PCC7942,采用启动子 (编码蓝细菌自身核酮糖-1,5-二磷酸羧化酶基因的启动子) 控制表达,在培养4周后工程藻株的乙醇产量达到0.23 g/L,这也是最早的通过代谢工程手段实现的蓝细菌化学品光合合成的报道[17]。虽然聚球藻PCC7942后来广泛应用于异丁醛、异丁醇[22]、正丁醇[18]、2,3-丁二醇[43]、蔗糖[25]、异丙醇[44]、1,3-丙二醇[45]和丙酮[46]等多种化学品的光合合成,但蓝细菌光合合成乙醇的研究却主要集中于另一种模式藻株集胞藻PCC6803中。
相比较于PCC7942,集胞藻PCC6803细胞内染色体高拷贝的特性[47]意味着相同的表达元件在PCC6803中可能会有更高强度、更稳定的表达,而现在普遍应用的编码AdhII的基因也来自于集胞藻PCC6803,其催化反应与宿主的适配性可能也优于聚球藻PCC7942。2009年,Dexter等将pdc-adhII途径整合至PCC6803基因组上,使用光诱导型强启动子控制表达,经过6 d培养后乙醇产量即达到0.46 g/L,约为此前聚球藻PCC7942工程藻株产量的2倍,而平均合成速率提高8倍,表明PCC6803可能比PCC7942更适于作为乙醇光合工程藻株构建的底盘细胞[37]。同年,Algenol公司采用集胞藻PCC6803内源的基因代替adhII,采用铜离子诱导型的启动子控制整条途径表达,工程藻株培养37 d后乙醇产量达到3.6 g/L[33]。2014年Dienst等采用高拷贝质粒将同样的PpetJ-pdc-slr1192途径导入集胞藻PCC6803,模拟光暗12 h交替的培养条件,工程藻株产率达到至287 mg/(L·d),经过18 d培养后积累了4.7 g/L的乙醇[48]。Algenol公司以集胞藻PCC6803为底盘细胞还尝试了大量不同启动子对pdc-slr1192途径的表达效果及对乙醇合成能力的影响,其中采用铜离子诱导型时,工程藻株经过30 d培养产量达到 7.1 g/L[33],实现了长时间内稳定的乙醇光合合成。本实验室在2012年的研究中发现,通过在集胞藻PCC6803基因组上将启动子控制的pdc-途径增加一个拷贝(Prbc-pdc-),可以使乙醇产量大幅提高,在26 d培养后从2 g/L的水平提高至5.5 g/L,产率达到212 mg/(L·d)[38];在后续的工作中我们发现通过在基因组上引入第3个-pdc-拷贝,乙醇产量可以进一步提高至8.8 g/L (未发表 数据)。
2.2 聚球藻PCC7002
聚球藻PCC7942和集胞藻PCC6803的生理和代谢背景相对清晰,遗传操作工具发展成熟,但是这两种藻株存在共同的问题是其对温度、高光、高盐、酸碱变化等环境胁迫的耐受性较差,生长速度也较慢,上述因素限制了已有的蓝细菌乙醇光合细胞工厂的规模化和产业化推广应用的前景。相比较而言,聚球藻s sp. PCC7002 (以下简称为聚球藻PCC7002) 可能是一种更好的选择[49],该藻株最适生长温度为38 ℃ (PCC6803为30 ℃),可以耐受500 μmol photons/(m2·s) 的光照强度(PCC6803和PCC7942最适光强为300 μmol photons/(m2·s));在38 ℃、500 μmol photons/(m2·s)的光照强度下,该藻株细胞复制代时为4.1 h,而PCC7942为8.5 h[50];而且PCC7002是海洋蓝细菌,其耐盐性更好,进行规模化生产时可以直接使用海水进行培养,因此在工程化应用和推广上具有显著的优势;此外,聚球藻PCC7002细胞中携带有拷贝数很高的内源性质粒,以内源性质粒序列为整合靶点可以有效地提高整合途径的拷贝数,提高表达强度[51-52],而这一点对现阶段提高化学品光合合成通量来说具有重要意义。2012年,Algenol公司的研究人员在PCC7002中以启动子 (钴离子诱导型) 和启动子分别控制pdc和表达,培养20 d后,乙醇产量可达到4.7 g/L;同年,美国Joule公司申请专利介绍的研究策略中,通过向PCC7002中引入1个pdc和2个不同来源的基因,同时敲除PCC7002自身的硫辛酸合成途径 (,),以限制工程藻株生物量积累而最大化乙醇合成强度,经过329 h培养即可生产5.62 g/L的乙醇,为PCC7002底盘细胞的最高乙醇产量,同时也实现了已知的最高乙醇光合生产强度[53-54]。
2.3 非模式藻株
传统的模式藻株虽然在遗传操作工具的开发、生理和代谢特性的解析上有着更丰富的积累,但其在环境胁迫适应性、生长速度等与规模扩大化培养密切相关的性质上往往难以令人满意,因此充分挖掘蓝细菌种质资源多样性与丰富性,探索更适合乙醇光合合成、更具有应用潜力的非模式藻株,对于蓝细菌乙醇光合细胞工厂的未来发展有着重要意义。Algenol公司已经报道了一种新的蓝细菌藻株,命名为ABICyanol,相比于已知的对各种环境胁迫因素耐受性较强的模式藻株聚球藻PCC7002,该藻株在乙醇耐受性、高温耐受性和过氧化耐受性等方面又有了明显的提高,对该藻株遗传改造结果表明,选用其自身的启动子控制pdc表达而启动子控制基因表达,在光照培养初期乙醇的单天生产强度可以达到0.552 g/(L·d),但这种高强度乙醇合成维持时间较短,在培养超过7 d后产醇强度逐渐降低[55]。上述研究表明通过对非模式藻株资源的充分挖掘可以为蓝细菌乙醇光合细胞工厂的发展注入新的活力,而后续对ABICyanol等藻株的生理和代谢特性有更深入的理解后,有望实现高强度、更具稳定性和持续性的乙醇光合合成,从而真正提高此技术的应用化潜力。
最近报道的一种聚球藻藻株UTEX 2973是又一种值得尝试的底盘细胞。尽管UTEX2973藻株在基因组上与聚球藻PCC7942具有高达98%的相似性 (包括55个SNP和1个188.6 kb大片段的位置翻转),该菌却表现出了显著提高的环境胁迫适应性和更快的生长速度,在高光强 (500 μmo photons/(m2·s))和高温 (41 ℃) 培养条件下其代增时间只有2.1 h,远远超过此前报道过的各种模式藻株[50]。而本实验室在前期研究中发现通过向该藻株中导入蔗糖转运蛋白编码基因,就可以改造成高效的蔗糖光合平台,且可以经过多次重悬培养而相对稳定地进行蔗糖合成与分泌[56],意味着该藻株具有改造成为高效光合平台并用于乙醇合成的潜力。
3 蓝细菌乙醇光合细胞工厂的代谢工程改造策略
3.1 提高乙醇合成途径的表达强度
在已有的蓝细菌光合细胞工厂开发过程中普遍发现,目标产物的合成效率往往受制于核心代谢途径的催化效率,强化关键代谢途径表达强度通常是改变碳流、优化目标产物合成速率的首要选择[57-58]。这一策略在蓝细菌乙醇光合细胞工厂开发中也得到了证实和应用 (如图1中绿色空框箭头所示)。如前所述,本实验室在对集胞藻PCC6803的改造中,通过逐步提高Prbc-pdc-slr1192途径在染色体上进行整合的拷贝数,极大地提高了乙醇的产量和合成速率[38]。而在聚球藻PCC7002的改造中,该藻株自身携带有数个拷贝数远高于染色体数目的内源性质粒,选择此类高拷贝内源质粒作为整合靶点以提高关键途径的拷贝数,将会成为对聚球藻PCC7002乙醇光合细胞工厂进一步改造的重要策略。
3.2 竞争性途径敲除
通过敲除碳源竞争性途径来提高乙醇合成性能的成功报道较少,只有Algenol公司在导入了pdc-adhⅡ途径的集胞藻PCC6803的同时,敲除乙酸激酶 () 基因和磷酸转乙酰酶 () 基因,使工程藻株12 d培养过程中的乙醇产量从0.47 g/L成功加倍达到0.95 g/L[33](如图1中红色X所示)。
而集胞藻PCC6803中两种主要的碳汇途径糖原和PHB的敲除对乙醇合成则没有正面的影响。2012年本实验室发现向集胞藻PCC6803中导入1拷贝的-pdc-同时敲除PHB合成途径 (--) 并不能有效提高乙醇产量;类似结果在对集胞藻PCC6803乳酸合成工程藻株的改造中也得到证实[59],意味着PHB途径的敲除并不能有效影响或促进工程藻株乙醇的产量。本实验室对乙醇高产藻株Syn-HZ24中的糖原合成途径 (,) 进行了敲除,结果在无胁迫培养条件下乙醇产量没有任何提高;虽然对工程藻株进行缺氮处理时,糖原合成缺失的藻株中乙醇合成的碳源分配会得到加强,但其产量仍低于在无胁迫培养条件下的水平,意味着这种非正常培养状态下提升并无法真正实现乙醇光合产量的有效提高 (未发表数据)。作为蓝细菌中最重要的碳汇机制,糖原代谢所扮演的角色并不是简单的碳流竞争途径,而是起到了全局性缓冲与“稳压器”的作用。糖原的敲除往往给细胞造成生理、代谢层面的负面影响,降低细胞对环境胁迫的适切性[60-61],进而造成化学品光合合成整体效能的降低,这一现象在其他蓝细菌化学品光合平台的改造中也得到了证实[59,62]。
3.3 辅因子工程
以集胞藻PCC6803为例,促进蓝细菌乙醇光合性能提升的一个重要进展是通过将NADH偏好性的AdhIIZM替换为NADPH偏好性的slr1192,极大地改善了代谢途径与宿主背景代谢之间的适配性,也证明了在辅因子供给层面途径对乙醇光合细胞工厂进行优化的重要性。如前所述,本实验室在此前对PdcZM-slr1192途径的体外重构和动态分析时发现,NADPH对该途径催化乙醇合成的m值为0.136 mmol/L,而饱和值超过0.5 mmol/L,而实际上集胞藻PCC6803中实际NADPH浓度在0.05–0.10 mmol/L左右[40-41],因此增加NADPH的供应可能成为提高乙醇产量的有效策略。2016年,Choi等在集胞藻PCC6803野生型过量表达其内源的葡萄糖-6-磷酸脱氢酶 (基因编码),有效提高了胞内NADPH含量,可以使PCC6803在自养和混养体系中的生物量均有增加;而进一步实验中,在引入了乙醇合成途径 (pdc-yqhD,为来源于大肠杆菌的乙醇脱氢酶编码基因,以NADPH为辅酶) 的工程藻株中过量表达时,培养14 d后乙醇产量增加33%,由0.44 g/L提高到0.59 g/L[63]。通过辅因子工程来提高蓝细菌乙醇光合性能,另一种潜在策略是改善乙醇脱氢酶辅因子的偏好性。Meng等采用理性设计策略成功优化了来自德式乳杆菌的D-乳酸脱氢酶的辅因子偏好性,使之可以同样利用NADH和NADPH作为催化辅因子,而且采用两种辅因子的时候催化活性均有所提高[64]。如果类似策略能够成功应用于AdhII (slr1192) 的改造则等同于增加了该酶在蓝细菌胞内的辅因子供应量 (从NADPH到NADPH+NADH),从而进一步提高乙醇合成的稳定性与效率。
3.4 基于代谢网络模型的改造靶点预测
虽然基于对局部代谢途径的分析和传统异养微生物代谢工程改造经验的指导,已经在蓝细菌乙醇光合细胞工厂和其他化学品光合平台的开发中进行了大量遗传操作来实现碳流的重新分配,但此类直观、简单的基因敲除缺乏对整个蓝细菌代谢网络全局调控和表达调控网络的充分理解,尤其缺乏对蓝细菌特有的光合固碳系统本身特性以及其与中心代谢互作关系的考量。蓝细菌乙醇光合细胞工厂的深度优化需要更具针对性的代谢工程改造靶点预测和改造策略设计。基于系统生物学海量数据的积累和挖掘,在基因组测序基础上的各种基因组尺度代谢网络模型的发展,已成为指导代谢工程改造不可或缺的有力工具,并广泛应用于预测改造靶点,提高代谢通量。利用代谢网络模型进行敲除靶点和敲除策略的设计以优化碳流、提高蓝细菌乙醇光合合成效率的研究已经有了初步进展 (图1)。
2013年,Sengupta等以集胞藻PCC6803基因组规模代谢模型为基础,结合通量平衡分析 (Flux balance analysis,FBA)、最小化代谢调整 (Minimization of metabolic adjustment,MOMA) 以及调控开关最小化 (Regulatory on/off minimization,ROOM) 等多种代谢建模技术进行基因敲除效果分析。结果显示,集胞藻PCC6803代谢网络中通过敲除腺苷酸激酶基因 (Adenylate kinase,,)、磷酸转乙酰酶基因(Phosphotransacetylase,,) 和乙酸激酶基因 (Acetate kinase,,) 可以显著提高乙醇合成通量[65]。而前文中已经提到的,Algenol公司在代谢工程改造实验中已经证实同时敲除集胞藻PCC6803中和基因确实提高了工程藻株中乙醇的产量,充分证明采用代谢网络模型辅助预测敲除靶点的策略是有效的,有望提高蓝细菌乙醇光合细胞工厂的改造效率和效果。
2014年,Erdrich等基于集胞藻PCC6803的基因组规模的化学计量模型,结合CASOP (Computational approach for strain optimization aiming at high productivity,应对高生产率变化的优化计算方法) 和cMCS (Minimal cut set,最小切集) 系统分析并设计了集胞藻中乙醇合成工程藻株的改造和优化方案,对大量基因敲除模拟的分析结果显示,降低胞内ATP和NADPH的比率可以促进乙醇的合成,而二者比率的改变可以通过以下方式实现:1) 降低ATP合成;2) 提高ATP消耗;3) 提高NADPH合成;4) 降低NADPH消耗;具体到实际的代谢工程改造,包括阻断环式电子传递流、引入ATP代谢空循环机制等策略[66]。而上述研究结果在2015年Knoop等的后续研究中得到进一步证实[67]。通过代谢网络重构,研究人员分析了集胞藻PCC6803中代谢产物合成过程中的化学计量特性和平衡关系,监测了细胞生长模式到产物合成模式中代谢系统特性的转变,结果显示PCC6803的线性电子传递链中ATP/NADPH比率 (1.28∶1) 与细胞生长和乙醇合成的适配关系不同,对生物量积累而言最优的ATP/NADPH比率为1.51,而乙醇合成最适比率则为1.17。这一结果与Endrich等研究工作中对ATP/NADPH比率调整需求的预测是相符的。
2016年,Yoshikawa团队开发了钝顶节旋藻NIES-39的基因组规模代谢模型 (包含746组代谢反应、673种代谢产物),并结合FBA方法对藻株代谢改造进行模拟和预测。钝顶节旋藻本身就可以进行乙醇合 成[28,68],而基因和途径敲除模拟分析发现,铵盐比硝酸盐更适合作为钝顶节旋藻产醇的氮源;敲除NAD(P)H脱氢酶、细胞色素C氧化酶等呼吸链功能相关基因以促进NADPH供应可能有助于促进乙醇合成,将藻株固定的碳源向乙醇的分配比提高到43%;而通过敲除磷酸烯醇式丙酮酸合酶、烯醇酶或磷酸甘油酸变位酶等基因以阻断酵解途径可以将乙醇合成占碳源的分配比率提高至50%以上[69]。
虽然现阶段代谢模型基础上所进行的敲除靶点预测、敲除策略设计等还普遍缺乏实验验证,但是相比于传统的直观层面上单纯对竞争旁路的敲除策略相比,此种技术体系下所提出的针对蓝细菌固有生理功能和光合特性的改造策略,无疑会在未来成为提高乙醇光合合成效率的首要选择。
3.5 蓝细菌新型基因组编辑技术的发展
一直以来,蓝细菌的遗传操作主要是通过同源重组将外源DNA向基因组上特点位点进行整合,以实现外源基因的表达或内源基因的敲除。外源DNA可以通过直接吸收 (集胞藻PCC6803、聚球藻PCC7932以及聚球藻PCC7002等) 或接合转移 (鱼腥藻PCC7120和聚球藻UTEX2973等) 的方式导入宿主细胞,其与基因组的整合则依靠抗性筛选和PCR-测序来进行鉴定。而蓝细菌细胞的染色体往往具有多倍性 (即单个细胞中含有多条相同染色体)[70-71],在通过抗性筛选和PCR初筛,获得基因组上携带有正确整合元件的转化子后,通常还需要进行一系列的传代,以提高整合程度,获得基因型纯合的突变藻株。上述过程效率低、耗时长,难以满足蓝细菌高效遗传操作,特别是基因组多靶点改造的需要。
以CRISPR-CAS9为代表的新型基因组编辑技术的出现为蓝细菌代谢工程领域提供了新的精确、高效的遗传操作工具[72]。现在基于CRISPR系统的蓝细菌基因组编辑技术主要可以分为基因沉默和基因敲除两类。2016年Yao等[73]证明应用核酸酶活性缺失的Ⅱ型CRISPR- CAS9系统 (来源于酿脓链球菌) 可以在聚球藻PCC7942中对目标基因的表达进行抑制,并成功实现了对4个醛还原酶和醛脱氢酶基因表达的同时沉默 (抑制效率50%−95%)。Gordon等利用相同系统和类似策略在聚球藻PCC7002中也实现了目标基因的表达调控,并应用于代谢工程改造[74]。华盛顿大学Pakrasi教授的研究团队开展了利用CRISPR系统直接进行基因敲除的研究,在聚球藻UTEX2973中通过CAS9蛋白的瞬时表达和另一种核酸酶Cpf1基因的稳定表达分别实现了目标基因的无痕敲除和外源基因的整合[75-76],其中Cpf1系统在集胞藻PCC6803和鱼腥藻PCC7120中也证实有效。而针对聚球藻PCC7942,Li等证明CRISPR-CAS9系统可以在基因组的靶点上引发双链断裂,进而通过双链断裂修复机制极大地提高了外源DNA同源重组的效率、并降低了对同源臂长度的要求[77]。现阶段,基于CRSIPR技术的各种蓝细菌基因组编辑和代谢调控工具正在不断涌现,对代谢背景中多个靶点的同时改造也成为可能,结合3.4部分中所述的改造靶点预测技术,无疑将在未来为蓝细菌乙醇光合合成能力的提升提供新的动力。
4 蓝细菌底盘细胞对乙醇胁迫的响应与适应机制解析
对大多数蓝细菌,尤其是应用于代谢工程改造的几种模式藻株而言,乙醇并不是其主要的天然代谢产物,乙醇在胞内检测不到存在或者含量极低,因此其对细胞生理和代谢的平衡与稳定影响非常微弱。而引入了Pdc-AdhII途径的工程藻株中,碳流、能量以及还原力的分配会发生显著变化,乙醇甚至成为主要代谢产物,乙醇合成可以占到工程藻株固碳量的50%以 上[34,38],这就意味着工程藻株从光合作用到细胞生长的各种生理和代谢活动都可能受到显著影响;另一方面,当乙醇合成能力进一步加强,乙醇含量在培养体系中达到1%甚至1.5%时,细胞生理和代谢活动会受到胁迫,从细胞结构到蛋白功能等各个层面都会受到扰动和抑制[78-79],进而影响工程藻株生长和生产的性能。而现阶段,蓝细菌乙醇光合工程藻株的最高产量可达到 8 g/L左右,接近于乙醇1%的胁迫临界浓度。因此,理解蓝细菌对乙醇胁迫、乙醇合成的响应与适应机制,挖掘乙醇耐受性改造靶点,对于蓝细菌乙醇光合细胞工厂的继续发展和优化有着重要意义。
4.1 蓝细菌对外源添加乙醇胁迫的响应与适应机制
天津大学合成微生物学实验室以集胞藻PCC6803为对象,以蓝细菌对外源添加的高浓度乙醇的响应和适应机制为切入点,在转录组、蛋白质组、代谢组等各个层面上进行了较为系统的解析。2012年,研究人员以1.25%、1.5%和2%三种浓度的乙醇对集胞藻PCC6803细胞进行胁迫处理,进而通过iTRAQ LC-MS/MS技术分析到了细胞中1 509个蛋白表达情况的变化,其中乙醇胁迫处理24 h后有135个蛋白的表达有了显著变化 (以1.5倍差异为阈值),而48 h后则达到293个。进一步的分析显示,面临高浓度外源乙醇时集胞藻PCC6803采取了启动压力应激系统 (General stress response)、调整细胞被膜组分与结构、加强转运蛋白合成等多种策略来应对胁迫;此外,乙醇胁迫还诱导了PCC6803中与光合作用相关的多种蛋白的上调表达,同时胞内叶绿素a含量也有所提高[80],意味着细胞可能需要通过加强光合作用为胁迫应激提供更多的物质和能量。上述蛋白质组分析的结果在后续的转录组分析中得到了进一步的验证;此外,转录组分析还进一步发现,PCC6803会通过强化PHB合成和乙二醛途径来缓解乙醇毒性[81]。
针对全局性分析的结果,研究人员又结合基因敲除和回补策略,对其中转运蛋白和转录调控因子系统的响应进行针对性分析。2014年,Song等证实转录因子编码基因直接与集胞藻PCC6803的乙醇耐受能力相关,该基因的缺陷型藻株在1.5%浓度的乙醇胁迫下,生长受到显著抑制;而相应的蛋白质组分析显示,该基因的缺失造成了54个蛋白表达的下调和87个基因的表达上调。该基因编码蛋白可以直接与 1个16.6 kDa的小热激蛋白 ()、1个钠离子依赖型的碳酸氢根转运蛋白 (,) 以及1个二氧化碳浓缩机制相关蛋白CcmK () 编码基因的上游序列直接结合[82]。2015年,Zhu等对集胞藻PCC6803转录因子系统对乙醇胁迫的响应进行了更大范围的研究,系统敲除了集胞藻PCC6803中34个假定转录调节基因,在乙醇胁迫条件下比较其与野生型的生长差异。结果发现、和的3个基因的敲除突变株与野生型相比生长变差,而代谢组学分析发现上述3种转录因子的缺失在代谢物和代谢模块层面引发的扰动存在显著的重叠现象,意味着其调控机制存在一定的交互性[83]。2015年Zhang等对PCC6803中转运蛋白系统与乙醇耐受性的相关性进行了系统研究,通过对58个转运蛋白编码基因的敲除和相应的乙醇耐受性分析发现,基因的突变体对1.5%的乙醇胁迫敏感,生长受到明显抑制。通过对缺失突变株代谢组学层面的分析发现,集胞藻PCC6803中色氨酸合成、不饱和脂肪酸合成以及次级代谢产物合成等代谢模块,以及乙酰CoA、ADP-葡萄糖、ATP、ADP、葡萄糖-6-磷酸、果糖-6-磷酸、核糖-5-磷酸等代谢物与细胞的乙醇耐受性密切相关[84]。上述研究针对集胞藻PCC6803对外源添加的乙醇胁迫的相应机制,系统采用了基因敲除-表型分析、转录组、代谢组和蛋白质组层面的分析手段,对乙醇的胁迫机制和细胞响应策略获得了全面的认知,也为进一步采用代谢工程策略改造细胞本身的乙醇耐受性提供了丰富的 靶点。
4.2 蓝细菌乙醇光合工程藻株对内源性乙醇合成的响应与适应机制
德国弗莱堡大学的研究人员和荷兰阿姆斯特丹大学的研究人员从另一种角度进行了研究,有针对性地解析蓝细菌乙醇光合合成工程藻株对内源性合成的乙醇的响应机制。2014年Dienst等将PpetJ-pdc-slr1192途径通过穿梭载体导入PCC6803,获得了乙醇高产藻株,18 d内可以合成4.7 g/L的乙醇,在整个培养过程中选取了4个时间点,对乙醇合成株和野生型藻株在生理和转录水平上的差异进行了分析。很有意思的是,在乙醇合成藻株中,在转录水平上藻蓝蛋白β亚基编码基因CpcB和两组铁离子转运蛋白的表达也都持续降低,而表型水平上整个过程中叶绿素和藻蓝蛋白含量显著降 低[48],这一点与此前Qiao等[80]在外源乙醇胁迫处理时观察到的叶绿素含量升高的现象恰恰相反。2015年Borirak等在集胞藻PCC6803中,采用PpsbA2-pdc-adhII构建乙醇合成藻株,进而通过蛋白组学定量分析发现工程藻株中有77种蛋白上调,64种蛋白下调。功能聚类分析表明,乙醇合成工程藻株中与二氧化碳浓缩、固定相关的很多蛋白,如卡尔文循环关键蛋白核酮糖1,5-二磷酸脱羧酶和碳浓缩机制核心功能蛋白CcmK1等,其含量普遍上调,意味着乙醇合成导致的碳流分散促进了工程细胞加快碳的固定以维持生长和其他生理代谢所需 (工程藻株中碳源到乙醇的流向分配比高达65%);与Dienst等结果类似的是发现蛋白质合成机器组分以及藻胆体组分 (CpcA、CpcB、CpcC1、CpcC2、CpcG2) 含量受到抑制和下调;此外乙醇的合成还在胞内引发了氧化应激反应(GshB含量升高)。
值得注意的是,乙醇外源添加或内源合成都会影响集胞藻PCC6803细胞中节律性蛋白的表达。在Borirak等构建的工程藻中节律性蛋白 (Circadian clock protein,生物钟蛋白,使蓝细菌适应昼夜节律的调节功能需要KaiA、KaiB和KaiC三种组分来完成) KaiA和KaiC显著上调,而此前Qiao等的蛋白组分析中发现外源乙醇胁迫会引发KaiB的表达提高,Wang等的转录组分析则发现KaiC的上调。上述研究表明,无论是内源产生的乙醇还是外源添加的乙醇,都会引发蓝细菌Kai系统组分的表达变化。在现阶段蓝细菌乙醇光合工程藻株的实验室培养中普遍采用了持续光照条件,而未来推向规模化培养时在户外昼夜交替条件下,该系统的扰动所造成的潜在影响则值得引起重视[85]。
上述的响应机制在图2中进行了具体展示和归纳。
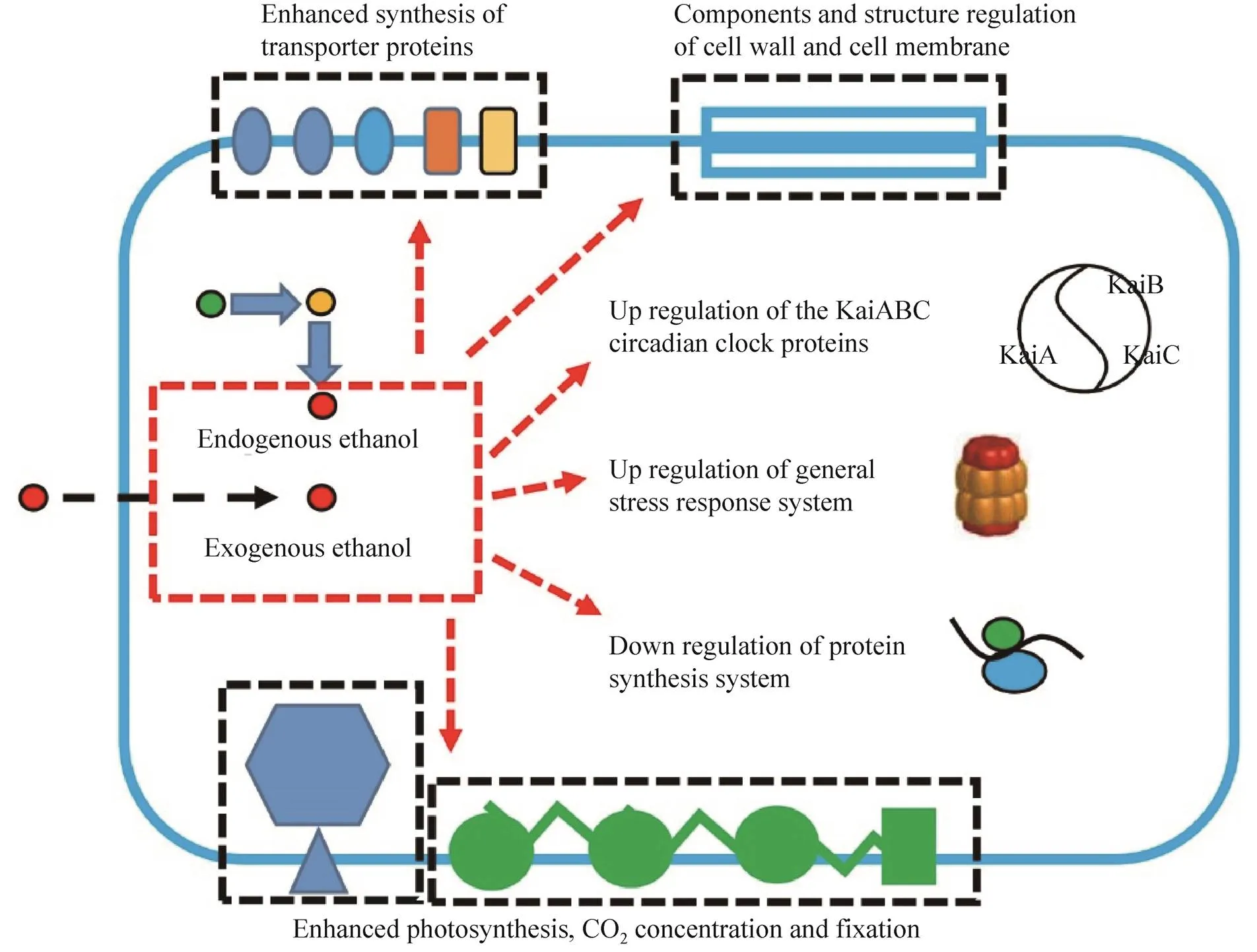
图2 集胞藻PCC6803对乙醇的动态响应机制示意图
5 展望
乙醇是最早报道的、通过代谢工程手段改造蓝细菌实现光合生物合成的化学品,也是最有代表性的蓝细菌光合化学品。到目前为止,蓝细菌乙醇合成技术还处于初级阶段,乙醇产量较低 (小于10 g/L),合成强度低(不超过 0.5 g/(L·d)),相比较于已经成熟的酿酒酵母乙醇合成等生物技术体系,其距离规模化培养和产业化应用还有很长的路要走。但是,如上文中已经展示的,蓬勃发展和广泛应用的系统生物技术体系已经对蓝细菌对乙醇的响应和适应机制进行了全面、立体的诠释;各种层面的代谢网络模型对潜在的藻株改造靶点和改造策略进行了预测和设计;以CRISPR-CAS等技术为代表的先进、高效的遗传操作体系的发展将使得多靶点、多层次的精细改造和途径调控成为可能,在可见的未来,蓝细菌乙醇光合藻株的性能有望迎来新的突破。同时,发展高效的蓝细菌乙醇光合藻株对其他化学品光合合成平台的开发具有很好的示范与启示意义。蓝细菌乙醇光合藻株核心代谢途径简单、清楚,藻株培养和产物分析技术成熟、便捷,各种代谢工程和合成生物学策略可以很快实施和验证,从而为其他高值化学品的光合合成的实现和优化提供概念和方向性的指引。
光合固碳效率的提升将成为其中最具吸引力的发展方向。光合作用是蓝细菌细胞生长和化学品合成的基础,光合固碳效率从根本上决定着蓝细菌化学品光合平台的效能和潜力。Atsumi等的研究证实在聚球藻PCC7942中过量表达卡尔文循环关键蛋白1.5-二磷酸核酮糖羧化酶 (Rubisco),可以提高工程藻株中异丁醛的产量[22],表明通过改善光合作用效率来提高代谢物合成能力的策略是可行、有效的。在未来,通过系统采用改造Rubisco等关键蛋白[86-87]、强化碳浓缩机制 (Carbon concentrating mechanism)[88]以及调控光呼吸系统[89]等策略,有望促使蓝细菌工程藻株的光合效率得到进一步提高。中国科学院微生物研究所李寅研究员团队最近发现,通过引入新的NADPH消耗途径可以有效促进光合固碳效率,加快工程细胞生长速度[90],表明在代谢途径设计的过程中将蓝细菌光合固碳系统的特点纳入考虑,有望实现平台生长性能与合成性能的协同提高。对于蓝细菌乙醇光合细胞工厂而言,此前的主要设计和改造策略集中于胞内固有碳流的重新分配上,而对于从源头上强化光合固碳、为乙醇合成提供更强的物质-能量动力将是未来进一步发展的必然选择。随着对蓝细菌光合固碳、中心代谢以及生理功能等生理和代谢机制认识的不断加深,以及合成生物学和代谢工程手段的丰富和完善,更为高效、稳定的蓝细菌光合平台必将成功构建,结合乙醇合成途径的优化和代谢互作背景的适配调控,蓝细菌平台的乙醇光合合成能力将有望迎来质的飞跃。
[1] Kennes D, Abubackar HN, Diaz M, et al. Bioethanol production from biomass: carbohydrate vs syngas fermentation. J Chem Technol Biotechnol, 2016, 91(2): 304–317.
[2] Keasling JD, Chou H. Metabolic engineering delivers next-generation biofuels. Nat Biotechnol, 2008, 26(3): 298–299.
[3] Thangavelu SK, Ahmed A, Ani FN. Review on bioethanol as alternative fuel for spark ignition engines. Renew SustEnerg Rev, 2016, 56: 820–835.
[4] Tabah B, Pulidindi IN, Chitturi VR, et al. Utilization of solar energy for continuous bioethanol production for energy applications. RSC Adv, 2016, 6(29): 24203–24209.
[5] Agarwal AK. Biofuels (alcohols and biodiesel) applications as fuels for internal combustion engines. Prog Energ Combust Sci, 2007, 33(3): 233–271.
[6] Weber C, Farwick A, Benisch F, et al. Trends and challenges in the microbial production of lignocellulosic bioalcohol fuels. Appl Microbiol Biotechnol, 2010, 87(4): 1303–1315.
[7] Rude MA, Schirmer A. New microbial fuels: a biotech perspective. Curr Opin Microbiol, 2009, 12(3): 274–281.
[8] Himmel ME, Ding SY, Johnson DK, et al. Biomass recalcitrance: engineering plants and enzymes for biofuels production. Science, 2007, 315(5813): 804–807.
[9] Ji SQ, Wang B, Lu M, et al. Direct bioconversion of brown algae into ethanol by thermophilic bacterium. Biotechnol Biofuels, 2016, 9: 81.
[10] Murphy JD, Thamsiriroj T. What will fuel transport systems of the future? Mater Today, 2011, 14(11): 518–524.
[11] Xia A, Jacob A, Tabassum MR, et al. Production of hydrogen, ethanol and volatile fatty acids through co-fermentation of macro-and micro-algae. Bioresour Technol, 2016, 205: 118–125.
[12] Zhou J, Li Y. Engineering cyanobacteria for fuels and chemicals production. Protein Cell, 2010, 1(3): 207–210.
[13] Lu XF. A perspective: photosynthetic production of fatty acid-based biofuels in genetically engineered cyanobacteria. Biotechnol Adv, 2010, 28(6): 742–746.
[14] Melis A. Solar energy conversion efficiencies in photosynthesis: minimizing the chlorophyll antennae to maximize efficiency. Plant Sci, 2009, 177(4): 272–280.
[15] Zhou J, Zhu TC, Cai Z, et al. From cyanochemicals to cyanofactories: a review and perspective. Microb Cell Fact, 2016, 15: 2.
[16] McNeely K, Xu Y, Bennette N, et al. Redirecting reductant flux into hydrogen production via metabolic engineering of fermentative carbon metabolism in a cyanobacterium. Appl Environ Microbiol, 2010, 76(15): 5032–5038.
[17] Deng MD, Coleman JR. Ethanol synthesis by genetic engineering in cyanobacteria. Appl Environ Microbiol, 1999, 65(2): 523–528.
[18] Lan EI, Liao JC. ATP drives direct photosynthetic production of 1-butanol in cyanobacteria. Proc Natl Acad Sci USA, 2012, 109(16): 6018–6023.
[19] Zhou J, Zhang HF, Zhang YP, et al. Designing and creating a modularized synthetic pathway in cyanobacteriumenables production of acetone from carbon dioxide. Metab Eng, 2012, 14(4): 394–400.
[20] Liu XY, Sheng J, Curtiss R III. Fatty acid production in genetically modified cyanobacteria. Proc Natl Acad Sci USA, 2011, 108(17): 6899–6904.
[21] Angermayr SA, Paszota M, Hellingwerf KJ. Engineering a cyanobacterial cell factory for production of lactic acid. Appl Environ Microbiol, 2012, 78(19): 7098–7106.
[22] Atsumi S, Higashide W, Liao JC. Direct photosynthetic recycling of carbon dioxide to isobutyraldehyde. Nat Biotechnol, 2009, 27(12): 1177–1180.
[23] Gao X, Gao F, Liu D, et al. Engineering the methylerythritol phosphate pathway in cyanobacteria for photosynthetic isoprene production from CO2. Energ Environ Sci, 2016, 9(4): 1400–1411.
[24] Wang WH, Liu XF, Lu XF. Engineering cyanobacteria to improve photosynthetic production of alka(e)nes. Biotechnol Biofuels, 2013, 6(1): 69.
[25] Ducat DC, Avelar-Rivas JA, Way JC, et al. Rerouting carbon flux to enhance photosynthetic productivity. Appl Environ Microbiol, 2012, 78(8): 2660–2668.
[26] Du W, Liang FY, Duan YK, et al. Exploring the photosynthetic production capacity of sucrose by cyanobacteria. Metab Eng, 2013, 19: 17–25.
[27] Heyer H, Krumbein WE. Excretion of fermentation products in dark and anaerobically incubated cyanobacteria. Arch Microbiol, 1991, 155(3): 284–287.
[28] Carrieri D, Momot D, Brasg IA, et al. Boosting autofermentation rates and product yields with sodium stress cycling: application to production of renewable fuels by cyanobacteria. Appl Environ Microbiol, 2010, 76(19): 6455–6462.
[29] He MX, Wu B, Qin H, et al.: a novel platform for future biorefineries. Biotechnol Biofuels, 2014, 7: 101.
[30] Yomano LP, York SW, Zhou S, et al. Re-engineeringfor ethanol production. Biotechnol Lett, 2008, 30(12): 2097–2103.
[31] Nikel PI, De Lorenzo V. Robustness ofKT2440 as a host for ethanol biosynthesis. New Biotechnol, 2014, 31(6): 562–571.
[32] Raj KC, Talarico LA, Ingram LO, et al. Cloning and characterization of the Zymobacter palmae pyruvate decarboxylase gene (pdc) and comparison to bacterial homologues. Appl Environ Microbiol, 2002, 68(6): 2869–2876.
[33] Coleman JR, Duehring U, Enke H, et al, Genetically modified photoautotrophic, host cell, useful to produce ethanol, comprises genetic modification changing enzymatic activity or affinity of endogenous host cell enzyme and genetic modification comprising overexpressed enzyme: WO, 2009098089, 2009-08-13.
[34] Dexter J, Armshaw P, Sheahan C, et al. The state of autotrophic ethanol production in cyanobacteria. J Appl Microbiol, 2015, 119(1): 11–24.
[35] Stevenson BJ, Liu JW, Ollis DL. Directed evolution of yeast pyruvate decarboxylase 1 for attenuated regulation and increased stability. Biochemistry, 2008, 47(9): 3013–3025.
[36] Sun HW, Plapp BV. Progressive sequence alignment and molecular evolution of the Zn-containing alcohol dehydrogenase family. J Mol Evol, 1992, 34(6): 522–535.
[37] Dexter J, Fu PC. Metabolic engineering of cyanobacteria for ethanol production. Energy Environ Sci, 2009, 2(8): 857–864.
[38] Gao ZX, Zhao H, Li ZM, et al. Photosynthetic production of ethanol from carbon dioxide in genetically engineered cyanobacteria. Energy Environ Sci, 2012, 5(12): 9857–9865.
[39] Vidal R, López-Maury L, Guerrero MG, et al. Characterization of an alcohol dehydrogenase from the cyanobacteriumsp. strain PCC 6803 that responds to environmental stress conditions via the Hik34-Rre1 two-component system. J Bacteriol, 2009, 191(13): 4383–4391.
[40] Takahashi H, Uchimiya H, Hihara Y. Difference in metabolite levels between photoautotrophic and photomixotrophic cultures ofsp. PCC 6803 examined by capillary electrophoresis electrospray ionization mass spectrometry. J Exp Bot, 2008, 59(11): 3009–3018.
[41] Luan GD, Qi YJ, Wang M, et al. Combinatory strategy for characterizing and understanding the ethanol synthesis pathway in cyanobacteria cell factories. Biotechnol Biofuels, 2015, 8: 184.
[42] Waterbury JB, Watson W, Guillard RRL, et al. Widespread occurrence of a unicellular, marine, planktonic, cyanobacterium. Nature, 1979, 277(5694): 293–294.
[43] Oliver JWK, Machado IMP, Yoneda H, et al. Cyanobacterial conversion of carbon dioxide to 2,3-butanediol. Proc Natl Acad Sci USA, 2013, 110(4): 1249–1254.
[44] Kusakabe T, Tatsuke T, Tsuruno K, et al. Engineering a synthetic pathway in cyanobacteria for isopropanol production directly from carbon dioxide and light. Metab Eng, 2013, 20: 101–108.
[45] Hirokawa Y, Maki Y, Tatsuke T, et al. Cyanobacterial production of 1,3-propanediol directly from carbon dioxide using a synthetic metabolic pathway. Metab Eng, 2016, 34: 97–103.
[46] Chwa JW, Kim WJ, Sim SJ, et al. Engineering of a modular and synthetic phosphoketolase pathway for photosynthetic production of acetone from CO2inPCC 7942 under light and aerobic condition. Plant Biotechnol J, 2016, 14(8): 1768–1776.
[47] Griese M, Lange C, Soppa J. Ploidy in cyanobacteria. FEMS Microbiol Lett, 2011, 323(2): 124–131.
[48] Dienst D, Georg J, Abts T, et al. Transcriptomic response to prolonged ethanol production in the cyanobacteriumsp. PCC6803. Biotechnol Biofuels, 2014, 7: 21.
[49] Ludwig M, Bryant DA. Acclimation of the global transcriptome of the cyanobacteriumsp. strain PCC 7002 to nutrient limitations and different nitrogen sources. Front Microbiol, 2012, 3: 145.
[50] Yu JJ, Liberton M, Cliften PF, et al.UTEX 2973, a fast growing cyanobacterial chassis for biosynthesis using light and CO2. Sci Rep, 2015, 5: 8132.
[51] Xu Y, Alvey RM, Byrne PO, et al. Expression of genes in cyanobacteria: adaptation of endogenous plasmids as platforms for high-level gene expression insp. PCC 7002. Methods Mol Biol, 2011, 684: 273–293.
[52] Akiyama H, Kanai S, Hirano M, et al. A novel plasmid recombination mechanism of the marine cyanobacteriumsp. PCC7002. DNA Res, 1998, 5(6): 327–334.
[53] Reppas NB. Metabolic switch: WO, 2012/071547, 2012-05-31.
[54] Green BD, Reppas NB, Robertson DE. Ethanol production in microorganisms: US, 8048666 2011-11-01.
[55] Piven I, Friedrich A, Dühring U, et al.sp. for production of compounds: US, 9157101. 2015-10-13.
[56] Song K, Tan XM, Liang YJ, et al. The potential ofUTEX 2973 for sugar feedstock production. Appl Microbiol Biotechnol, 2016, 100(18): 7865–7875.
[57] Angermayr SA, Hellingwerf KJ. On the use of metabolic control analysis in the optimization of cyanobacterial biosolar cell factories. J Phys Chem B, 2013, 117(38): 11169–11175.
[58] Angermayr SA, Van Der Woude AD, Correddu D, et al. Exploring metabolic engineering design principles for the photosynthetic production of lactic acid bysp. PCC6803. Biotechnol Biofuels, 2014, 7: 99.
[59] Van Der Woude AD, Angermayr SA, Veetil VP, et al. Carbon sink removal: increased photosynthetic production of lactic acid bysp. PCC6803 in a glycogen storage mutant. J Biotechnol, 2014, 184: 100–102.
[60] Gründel M, Scheunemann R, Lockau W, et al. Impaired glycogen synthesis causes metabolic overflow reactions and affects stress responses in the cyanobacteriumsp. PCC 6803. Microbiol, 2012, 158: 3032–3043.
[61] Suzuki E, Ohkawa H, Moriya K, et al. Carbohydrate metabolism in mutants of the cyanobacteriumPCC 7942 defective in glycogen synthesis. Appl Environ Microbiol, 2010, 76(10): 3153–3159.
[62] Davies FK, Work VH, Beliaev AS, et al. Engineering limonene and bisabolene production in wild type and a glycogen-deficient mutant ofsp. PCC 7002. Front Bioeng Biotechnol, 2014, 2: 21.
[63] Choi YN, Park JM. Enhancing biomass and ethanol production by increasing NADPH production insp. PCC 6803. Bioresour Technol, 2016, 213: 54–57.
[64] Meng HK, Liu P, Sun HB, et al. Engineering a d-lactate dehydrogenase that can super-efficiently utilize NADPH and NADH as cofactors. Sci Rep, 2016, 6: 24887.
[65] Sengupta T, Bhushan M, Wangikar PP. Metabolic modeling for multi-objective optimization of ethanol production in amutant. Photosynth Res, 2013, 118(1/2): 155–165.
[66] Erdrich P, Knoop H, Steuer R, et al. Cyanobacterial biofuels: new insights and strain design strategies revealed by computational modeling. Microb Cell Fact, 2014, 13(1): 128.
[67] Knoop H, Steuer R. A computational analysis of stoichiometric constraints and trade-offs in cyanobacterial biofuel production. Front Bioeng Biotechnol, 2015, 3: 47.
[68] Aoyama K, Uemura I, Miyake J, et al. Fermentative metabolism to produce hydrogen gas and organic compounds in a cyanobacterium,. J Ferment Bioeng, 1997, 83(1): 17–20.
[69] Yoshikawa K, Aikawa S, Kojima Y, et al. Construction of a genome-scale metabolic model ofNIES-39 and metabolic design for cyanobacterial bioproduction. PLoS ONE, 2015, 10(12): e0144430.
[70] Simon R. DNA content of heterocysts and spores of the filamentous cyanobacterium Anabaena variabilis. FEMS Microbiol Lett, 1980, 8: 241-245.
[71] JainI H, Vijayan V, O’Shea EK. Spatial ordering of chromosomes enhances the fidelity of chromosome partitioning in cyanobacteria. Proc Natl Acad Sci USA, 2012, 109(34): 13638–13643.
[72] Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819–823.
[73] Yao L, Cengic I, Anfelt J, et al. Multiple gene repression in cyanobacteria using CRISPRi. ACS Synth Biol, 2016, 5(3): 207–212.
[74] Gordon GC, Korosh TC, Cameron JC, et al. CRISPR interference as a titratable,regulatory tool for metabolic engineering in the cyanobacteriumsp. strain PCC 7002. Metab Eng, 2016, 38: 170–179.
[75] Wendt KE, Ungerer J, Cobb RE, et al. CRISPR/Cas9 mediated targeted mutagenesis of the fast growing cyanobacteriumUTEX 2973. Microbial Cell Factories, 2016, 15: 115.
[76] Ungerer J, Pakrasi HB. Cpf1 is aversatile tool for CRISPR genome editing across diverse species of cyanobacteria. Sci Rep, 2016, 6: 39681.
[77] Li H, Shen CR, Huang CH, et al. CRISPR-Cas9 for the genome engineering of cyanobacteria and succinate production. Metab Eng, 2016, 38: 293–302.
[78] Ding JM, Huang XW, Zhang LM, et al. Tolerance and stress response to ethanol in the yeast. Appl Microbiol Biotechnol, 2009, 85(2): 253–263.
[79] Chen TJ, Wang JQ, Yang R, et al. Laboratory-evolved mutants of an exogenous global regulator, IrrE from, enhance stress tolerances of. PloS ONE, 2011, 6(1): e16228.
[80] Qiao JJ, Wang JX, Chen L, et al. Quantitative iTRAQ LC-MS/MS proteomics reveals metabolic responses to biofuel ethanol in cyanobacterialsp. PCC 6803. J Proteome Res, 2012, 11(11): 5286–5300.
[81] Wang JX, Chen L, Huang SQ, et al. RNA-seq based identification and mutant validation of gene targets related to ethanol resistance in cyanobacterialsp. PCC 6803. Biotechnol Biofuels, 2012, 5: 89.
[82] Song ZD, Chen L, Wang JX, et al. A transcriptional regulator Sll0794 regulates tolerance to biofuel ethanol in photosyntheticsp. PCC 6803. Mol Cell Proteomics, 2014, 13(12): 3519–3532.
[83] Zhu Y, Pei GS, Niu XF, et al. Metabolomic analysis reveals functional overlapping of three signal transduction proteins in regulating ethanol tolerance in cyanobacteriumsp. PCC 6803. Mol Biosyst, 2015, 11(3): 770–782.
[84] Zhang YN, Niu XF, Shi ML, et al. Identification of a transporter Slr0982 involved in ethanol tolerance in cyanobacteriumsp. PCC 6803. Front Microbiol, 2015, 6: 487.
[85] Borirak O, De Koning LJ, Van Der Woude AD, et al. Quantitative proteomics analysis of an ethanol-and a lactate-producing mutant strain ofsp. PCC6803. Biotechnol Biofuels, 2015, 8: 111.
[86] Cai Z, Liu GX, Zhang JL, et al. Development of an activity-directed selection system enabled significant improvement of the carboxylation efficiency of Rubisco. Protein Cell, 2014, 5(7): 552–562.
[87] Whitney SM, Houtz RL, Alonso H. Advancing our understanding and capacity to engineer nature's CO2-sequestering enzyme, Rubisco. Plant Physiol, 2011, 155(1): 27–35.
[88] Kamennaya NA, Ahn S, Park H, et al. Installing extra bicarbonate transporters in the cyanobacteriumsp. PCC6803 enhances biomass production. Metab Eng, 2015, 29: 76–85.
[89] Shih PM, Zarzycki J, Niyogi KK, et al. Introduction of a synthetic CO2fixing photorespiratory bypass into a cyanobacterium. J Biol Chem, 2014, 289(14): 9493–9500.
[90] Zhou J, Zhang FL, Meng HK, et al. Introducing extra NADPH consumption ability significantly increases the photosynthetic efficiency and biomass production of cyanobacteria. Metab Eng, 2016, 38: 217–227.
(本文责编 陈宏宇)
Cyanobacteria cell factories for ethanol photosynthetic production: development and prospect
Yunjing Qi1,2, Jialin Wang1, Guodong Luan2, Xiaoming Tan2, and Xuefeng Lü2
1 College of Chemical Engineering, Qingdao University of Science and Technology, Qingdao 266042, Shandong, China 2 Key Laboratory of Biofuels, Qingdao Institute of Bioenergy and Bioprocess Technology, Chinese Academy of Sciences, Qingdao 266101, Shandong, China
Bioethanol is one of the most promising and representative biofuel products. Photosynthetic production of ethanol using CO2and solar energy based on cyanobacteria is of great significance for research and application, due to the potential to reduce CO2emission and to provide renewable energy simultaneously. Here we review the history and updated development of cyanobacteria cell factories for ethanol photosynthetic production, the progress and problems in pathway optimization, chassis selection, and metabolic engineering strategies, and finally indicate the future development in this area.
cyanobacteria, bioethanol, metabolic engineering, cell factory, ethanol tolerance
10.13345/j.cjb.160457
November 24, 2016; Accepted: January 24, 2017
Guodong Luan. Tel: +86-532-80662711; E-mail: luangd@qibebt.ac.cn
Supported by: National Natural Science Foundation of China (No. 31600034).
国家自然科学基金(No. 31600034)资助。
