蒙古扁桃编码Ca2+结合蛋白基因的系统性鉴定和表达分析
2017-06-28毛惠平
刘 洋,铁 英,杨 佳,祁 智,毛惠平,
(1.内蒙古和盛生态科技研究院有限公司,内蒙古 呼和浩特 010010;2.内蒙古大学 生命科学院,内蒙古 呼和浩特 010010)
蒙古扁桃编码Ca2+结合蛋白基因的系统性鉴定和表达分析
刘 洋1,铁 英1,杨 佳2,祁 智2,毛惠平1,2
(1.内蒙古和盛生态科技研究院有限公司,内蒙古 呼和浩特 010010;2.内蒙古大学 生命科学院,内蒙古 呼和浩特 010010)
为进一步解析蒙古扁桃抗逆境的遗传机理,采用高通量转录组测序技术,结合生物信息学和基因表达分析,在蒙古扁桃体内鉴定到16个编码Ca2+结合蛋白的全长转录本,包括4个Ca2+ATPase(ECA/ACA),3个具有多个跨膜域的新型Ca2+通道蛋白(ERD),5个Ca2+/H+反向转运蛋白(CAX),2个Ca2+依赖的蛋白激酶(CPK)和2个钙调素蛋白(CAM),它们的结构域同拟南芥同源蛋白基本相同。ERD、CAX、CPK和CAM各有1个基因在蒙古扁桃根和叶中有明显表达,其中的ERD、CPK和CAM类的基因表达在不同程度上受干旱和盐胁迫调节,说明有可能调节蒙古扁桃的抗逆能力。
蒙古扁桃;转录组测序;钙结合蛋白;钙调素
通过分子育种手段培育抗逆境作物的核心是掌握有明确抗逆境功能的基因。钙离子(Ca2+)和钙结合蛋白在植物细胞感应和抵抗逆境中发挥重要作用,因此,寻找具有抗逆境功能的编码钙结合蛋白基因一直是分子育种的一个方向。目前研究主要集中在以拟南芥为代表的模式植物中[1-2]。在细胞层面,渗透胁迫、干旱相关激素脱落酸(ABA)、盐碱、低温和高温等非生物胁迫,以及细菌、真菌等生物胁迫都可以诱导细胞质和细胞器内的Ca2+瞬时升高,调节位于细胞内不同部位对Ca2+具有不同亲和性的蛋白活性,进一步调节下游逆境相关基因表达[3-4]。在模式植物拟南芥基因组内存在多种Ca2+结合蛋白和Ca2+转运蛋白,例如CDPK、CIPK、CBL、CML、CaM、CNGC、GLR等,其中一些蛋白在拟南芥逆境生理方面发挥核心功能[5]。
生长在不同自然环境的野生植物是天然的功能基因库,尤其是天然抗逆境的荒漠植物体内可能存在模式植物不具有的,具有抗逆境功能的编码钙结合蛋白基因。但是由于野生植物生长缓慢、基因组结构复杂,对野生植物基因的研究远远落后于模式植物。这些年随着基因测序技术的快速发展[6],测序需要的时间和成本都大幅度下降,这使得一个普通的实验室也可以对感兴趣的野生植物的遗传信息进行基本分析。最近几年,一些长期适应极端环境的野生植物完成了转录组或基因组测序,例如胡杨[7]、沙冬青[8]、双花扁豆[9]、牛心朴子草[10]、西藏砂生槐[11]、籽蒿[12]。
本研究选取的蒙古扁桃属于蔷薇科,是第三纪孑遗、濒危国家二级保护植物。主要分布在蒙古国戈壁省及东戈壁省,我国内蒙古西部以及宁夏回族自治区北部,陕西省和甘肃省北部荒漠地区[13-16]。基于分子标记的遗传多样性分析表明,内蒙古西部包头萨拉齐地区的蒙古扁桃种群多样性最丰富[17]。蒙古扁桃具有天然抗干旱、盐碱和低温的能力[18],可以用于荒漠地区生态林建设和植被恢复工程,目前蒙古扁桃的苗木繁殖和基于组织培养的快繁技术体系已经建立[19-22]。蒙古扁桃的抗逆境能力,一方面同其栽培条件相关[23],另一方面同其内在解剖结构以及其根部的共生丛枝菌根有关[24-25]。
2015年蒙古扁桃完成了干旱处理样本的转录组测序工作[26],为进一步解析蒙古扁桃抗逆境的遗传机理提供了基础信息。到目前为止,基于转录组数据,针对蒙古扁桃基因的深入研究还是空白。本研究在转录组测序的基础上,在蒙古扁桃体内鉴定到多种Ca2+结合蛋白,并进一步对其中的CML家族在干旱和盐碱条件下的转录表达进行了研究。
1 材料和方法
1.1 蒙古扁桃叶转录组测序
选取健康饱满的野生蒙古扁桃种子(采自内蒙古阿拉善地区)用自来水浸泡,放在4 ℃冰箱7 d以后,在封闭的光照培养室内进行催芽,催芽条件:12 h光照,12 h黑暗,120 μmol/(m2·s)光强,温度维持在23 ℃左右。发芽的种子播种在营养土 ∶蛭石为1∶1的花盆中,在内蒙古大学南区玻璃日光温室内培养。每盆3~5株植物。每7 d给300 mL自来水,时间固定在9:00时。选取长势一致高度在20 cm左右的蒙古扁桃幼苗,在统一给水7 d以后,剪下长势良好的植物的部分叶子,利用RNAiso for Polysaccharide-Rich Plant Tissue(TaKaRa)试剂盒提取RNA。将储存在异丙醇中的RNA通过干冰保存并委托北京博奥晶典生物技术有限公司进行数据量4 G左右的转录组测序。测序平台是Illumina HiSeq2500。测序基本流程见图1。

图1 转录组测序基本流程
1.2 蒙古扁桃编码Ca2+结合蛋白基因序列的鉴定
对于公司提供的含有英文基因注释信息的Excel表格,利用Calcium或Ca2+进行搜索。在Notepad++环境下,打开根据转录组序列预测的蛋白氨基酸序列文件,利用基因序列代号汇总每一条注释带有Calcium或Ca2+的蛋白序列信息。利用在线蛋白数据库www.uniprot.org预测蛋白的基本性质。利用InterPro-Scan(http://www.ebi.ac.uk/Tools/pfa/iprscan)对这些蛋白的氨基酸序列进行保守域检测。利用在线ClustalW进行多序列比对和系统进化树构建(http://www.genome.jp/tools/clustalw/)。根据起始密码子、终止密码子和保守结构域的完整性进一步去除非全长片段,最终得到最有可能编码Ca2+结合蛋白的全长序列。
1.3 蒙古扁桃基因表达分析
选取高度在20 cm左右的蒙古扁桃幼苗,在统一给水7 d以后,选择长势良好的植物,收集叶和根组织速冻,用于基因组织特异性表达研究。对于要进行胁迫处理的植物,分成3组,每组3盆苗,在统一给水7 d以后,将时间设为0 d,然后分别给予每组植物(Ⅰ)300 mL自来水(对照),(Ⅱ)300 mL含有200 mmol/L NaCl的溶液(盐处理)和(Ⅲ)干旱不浇水处理,在第7天和第14天的时候收集长势一致的叶子进行速冻。3次生物学重复相隔7 d。试验在2015年5-9月进行。
速冻的叶子在7 d以内,经过液氮研磨以后,利用RNAiso for Polysaccharide-Rich Plant Tissue(Takara)试剂盒提取RNA,进一步用DNA酶消耗RNA中残留的基因组DNA污染。RNA准确含量用QUBIT RNA BR ASSAY KIT(Invitrogen)试剂盒和QUBIT 2.0 FLUOROMETER(Invitrogen)确定。以3 g RNA为模板,利用TransScript-Uni One-Step gDNA Removal 和 cDNA Synthesis SuperMix反转录成cDNA(Transgene)。
实时定量PCR(Real time quantitative PCR)使用qTOWER 2.2 设备(Analytik Jena,Germany)和TransStart Tip Green qPCRSuperMix试剂(Transgene)。参比基因GAPDH的引物来自内蒙古农业大学白老师实验室[26]。基因表达水平分析分别在对照和盐处理、对照和干旱处理间进行。对于每一组分析,基因表达水平首先利用参比基因在对照和处理间的表达差异进行校对,然后将同一种处理条件下,同一个基因3个生物学重复中最低表达量设定为1,对同一基因在不同生物学重复和不同时间点处理的表达量进行归一化处理。显著性差异利用Excel中的T-TEST、等方差、成对(Pair)方法进行分析。
2 结果与分析
2.1 蒙古扁桃叶RNA提取和转录组测序的基本情况
蒙古扁桃叶子相对较厚,肉质。利用TRIplant Reagent多糖多酚植物总RNA抽提试剂(BioTeke)和RNAiso for Polysaccharide-Rich Plant Tissue(Takara),得到清晰高质量RNA。利用Illumina HiSeq2500测序平台,对蒙古扁桃叶RNA进行转录组测序,得到原始测序片段(Raw reads)42 681 238,去除低质量测序数据,最终得到有效片段40 890 134(Clean reads),占整个测序片段的94%,总共碱基数量5 G,说明测序质量可靠。对测到的有效片段进行进一步的组装,得到88 435条可能编码功能基因的序列(Unigene),其中65%的长度在1 kb以下,35%的长度大于1 kb,其中12.8%的序列长度大于2 kb(表1)。

表1 转录本组装结果
利用COG数据库(Clusters of orthologous groups)对Unigene进行基本的功能预测(图2),500多个Unigene注释为未知功能(Function unknown),其他Unigene都与已知功能基因有一定的同源性,其中300个基因参与细胞信号转导(Signal transduction),200个基因参与生物胁迫反应(Defense mechanisms),它们有可能是蒙古扁桃耐受逆境的分子基础。
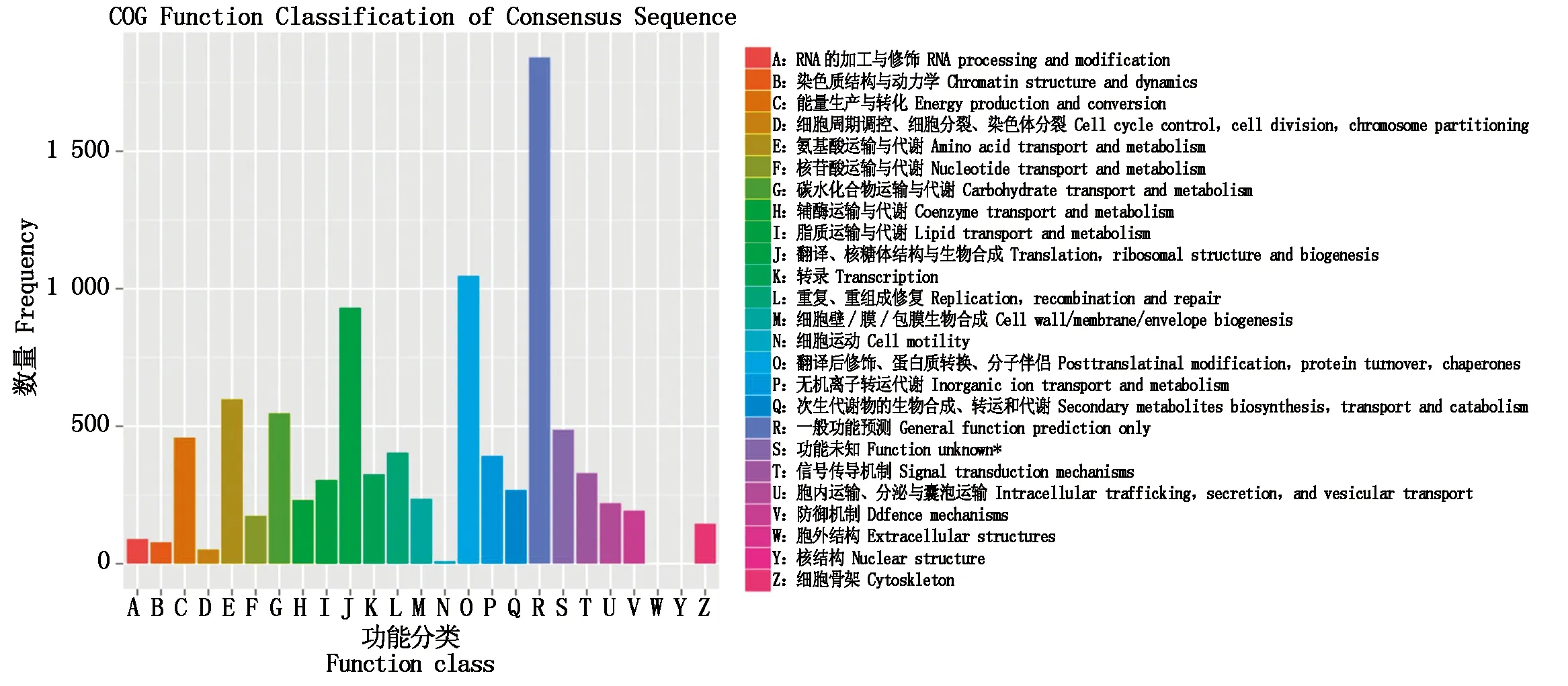
图2 蒙古扁桃转录本COG注释归类
2.2 蒙古扁桃编码Ca2+结合蛋白基因的系统性鉴定和序列分析
通过对基因注释信息进行关键字搜索,对基因序列以及对应的氨基酸序列进行系统分析,最终确定16条编码Ca2+结合蛋白的全长转录本,包括4个Ca2+ATPase(ECA/ACA)(表2),3个具有多个跨膜域的新型Ca2+通道蛋白(表3),5个Ca2+/H+反向转运蛋白(CAX)(表4),2个Ca2+依赖的蛋白激酶(CPK)和2个钙调素蛋白(CAM)(表5)。这些转录本对应的氨基酸序列同拟南芥同源序列长度基本相当,同时有50%~89%的相同性,说明蒙古扁桃的Ca2+结合蛋白序列同模式植物拟南芥有较高的同源性。

表2 蒙古扁桃编码Ca2+-ATPase基因全长转录本同拟南芥同源序列的比对

表3 蒙古扁桃编码多个跨膜域新型Ca2+通道蛋白基因全长转录本同拟南芥同源序列的比对
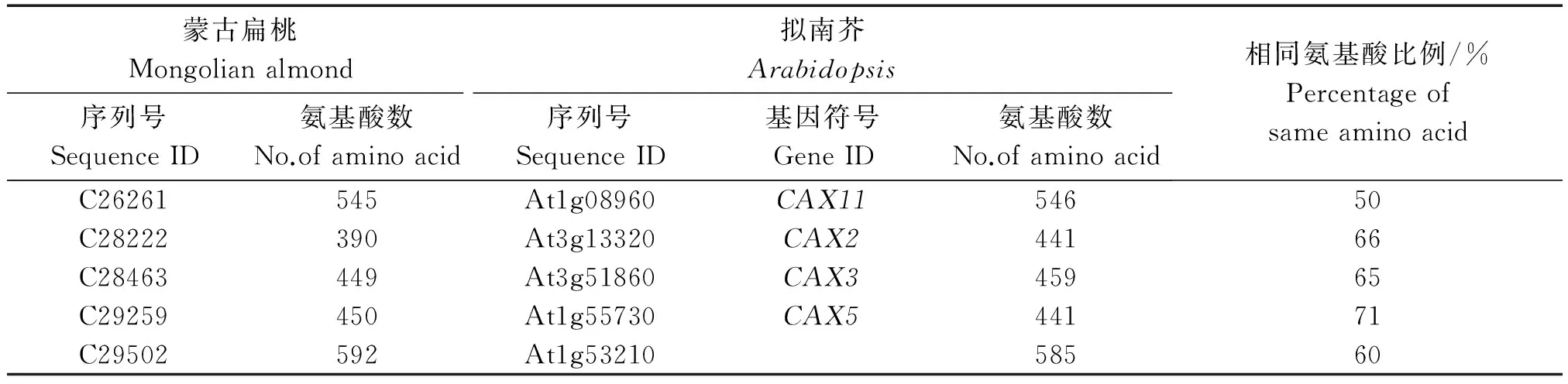
表4 蒙古扁桃编码Ca2+/H+转运蛋白基因全长转录本同拟南芥同源序列的比对
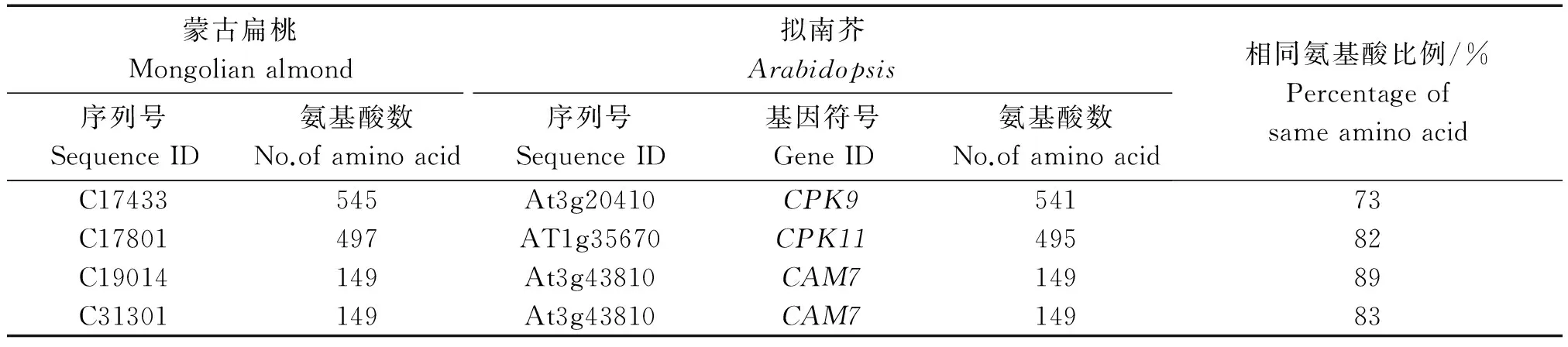
表5 蒙古扁桃编码Ca调蛋白基因全长转录本同拟南芥同源序列的比对
进一步对这16条蒙古扁桃编码Ca2+结合蛋白的序列进行了保守结构域分析,并同拟南芥同源序列的保守结构域进行了比对。发现2个植物的ECA基因编码蛋白都含有8个预测跨膜域,同时相对分布位置也一致(图3)。2个植物的CPK序列在N端都具有保守的ATP结合位点和蛋白激酶结构域,在C端都含有4个典型的EF手型(EF-Hand)Ca2+结合位点(图3)。2个植物的CAM序列都具有均匀分布的4个典型的EF手型(EF-Hand)Ca2+结合位点(图3)。对于新型Ca2+通道蛋白,尽管蒙古扁桃C23875基因和对应的拟南芥ERD4都编码有9个预测跨膜域的蛋白,但第7和第8个跨膜域的相对位置是不同的(图4)。

在ECA3、CAX11中的黑色区域代表跨膜域;CPK9和CAM7中的EF代表EF手型Ca2+结合域;数字代表氨基酸位点;拟南芥同源基因编码蛋白预测的结构域与蒙古扁桃相同。In the ECA3,CAX11,the black region indicates the transmembrane-domain;The EF in the CPK9 and CAM7 stands for the EF-hand Ca2+ binding structure;The number in the figure indicates the amino acid position;The Arabidopsis homology gene encoding protein′s predicated structure is same as the Mongolian almond′s.

黑色区域.跨膜域;数字.氨基酸位点。The black region.The transmembrane-domain;The number.The amino acid position.
2.3 蒙古扁桃编码Ca2+结合蛋白基因的表达分析
为了对这16条编码Ca2+结合蛋白转录本的生理功能有一个基本的了解,首先利用定量PCR研究了它们在根和叶组织中的特异性表达。大多数基因在根和叶中都没有检测到表达。只有C23875_ERD4、C28463_CAX3、C17801_CPK11和C19014_CAM7在叶和根中都有明显表达(图5),其中,C23875_ERD4在根中表达最高,C19014_CAM7在叶中表达最高(图5)。

图5 蒙古扁桃编码钙结合蛋白基因在叶和根中的相对表达
进一步研究了干旱和盐胁迫对这4个基因在叶中表达的影响。在7 d或14 d 200 mmol/L NaCl或干旱处理的植物叶中,相对于对照植物,C23875_ERD4、C17801_CPK11和C19014_CAM7的表达量升高较大,C28463_CAX3的表达没有明显变化。其中,C23875_ERD4和C19014_CAM7对NaCl处理反应最强烈,表达量随着NaCl处理时间的延长进一步升高(图6)。

图6 蒙古扁桃编码钙结合蛋白基因的表达对盐和干旱胁迫的反应
3 结论与讨论
生长在特殊生境的野生植物是天然的功能基因库。现代低成本高通量的测序技术使开发野生植物基因库成为可能。本研究利用高通量转录组测序技术,结合生物信息学和基因表达分析,在野生荒漠植物蒙古扁桃体内鉴定到16个编码Ca2+结合和转运蛋白的全长转录本,包括2015年在拟南芥体内鉴定到的具有9个跨膜域的新型Ca2+通道蛋白EDR4。生物信息学分析证明蒙古扁桃的Ca2+ATPase(ACA/ECA)、Ca2+/H+(CAX)、CPK以及CAM蛋白具有和拟南芥同源蛋白高度类似的蛋白结构域,间接说明它们很有可能具有类似的功能。蒙古扁桃EDR4的第7个跨膜域的相对位置和拟南芥完全不同,说明它们的Ca2+转运特性可能是不一样的。
拟南芥体内和EDR4在蛋白结构上类似的蛋白OSCA1被证明是跨膜Ca2+通道,感知细胞外的渗透势,介导Ca2+从叶保卫细胞外进入细胞内[24]。拟南芥中的CPK11是一个典型的Ca2+依赖蛋白激酶,其表达受干旱和高盐迅速诱导,通过磷酸化ABF类转录因子,正向调控ABA介导的信号转导[25]。拟南芥的CAM7直接作用bZIP转录因子HY5的启动子,进一步调节和植物光形态建成相关的基因表达。把这些基因编码蛋白的功能进行总结[26],我们可以假设,干旱和盐胁迫有可能首先通过开启位于蒙古扁桃细胞质膜的EDR4 Ca2+通道蛋白,介导细胞外Ca2+进入细胞质,使蒙古扁桃细胞质Ca2+浓度升高,激活位于细胞质的Ca2+依赖蛋白激酶CPK11;进入细胞核的Ca2+通过直接激活CAM7启动胁迫相关基因的表达。
[1] Kudla J,Batistic O,Hashimoto K.Calcium signals:the Lead currency of plant information processing[J].The Plant Cell,2010,22(3):541-563.
[2] 高 天,铁 英,王 妍,等.拟南芥低钙诱导cDNA文库的构建[J].北方农业学报,2016,44(5):8-14.
[3] Reddy A S,Ali G S,Celesnik H,et al.Coping with stresses:roles of Calcium-and Calcium/Calmodulin-regulated gene expression[J].Plant Cell,2011,23(6):2010-2032.
[4] 张胜博,刘景辉,李立军,等.燕麦AsNAC1耐盐基因的克隆及初步验证[J].北方农业学报,2016,44(5):1-7.
[5] Robertson D.Modulating plant calcium for better nutrition and stress tolerance[J].ISRN Botany,2013,2013(5):1-22.
[6] 呼斯乐,白 晨,张惠忠,等.转基因作物检测技术研究进展[J].内蒙古农业科技,2014(5):98-101.
[7] Ma T,Wang J,Zhou G,et al.Genomic insights into salt adaptation in a desert poplar [J].Nat Commun,2013,4(7):2797.
[8] Pang T,Ye C Y,Xia X,et al.De novo sequencing and transcriptome analysis of the desert shrub,Ammopiptanthus mongolicus,during cold acclimation using Illumina/Solexa [J].BMC Genomics,2013,14(1):488.
[9] Bhardwaj J,Chauhan R,Swarnkar M K,et al.Comprehensive transcriptomic study on horse gram(Macrotylomauniflorum):De novo assembly,functional characterization and comparative analysis in relation to drought stress[J].BMC Genomics,2013,14(1):647.
[10] Ma X,Wang P,Zhou S,et al.De novo transcriptome sequencing and comprehensive analysis of the drought-responsive genes in the desert plantCynanchumkomarovii[J].BMC Genomics,2015,16(1):753.
[11] Li H E,Yao W J,Fu Y R,et al.De Novo assembly and discovery of genes that are involved in drought tolerance in tibetansophoramoorcroftiana[J].PLoS One,2015,10(1):e111054.
[12] Zhang L,Hu X,Miao X,et al.Genome-Scale transcriptome analysis of the Desert ShrubArtemisiasphaerocephala[J].PLoS One,2016,11(4):e0154300.
[13] 黄小鹏,斯琴巴特尔,吴 榕.阿拉善荒漠区蒙古扁桃的种群龄级结构与空间分布格局[J].内蒙古林业科技,2014,40(1):7-11.
[14] 马松梅,聂迎彬,段 霞.等蒙古扁桃植物的潜在地理分布及居群保护优先性[J].生态学报,2015,35(9):2960-2966.
[15] 李 刚,王 科.东大山林区蒙古扁桃的调查与保护对策研究[J].甘肃科技,2015,31(7):138-140.
[16] 杨跃文,季 蒙,刘新前,等.内蒙古贺兰山自然保护区不同生境下蒙古扁桃种群结构与空间分布[J].山地学报,2015,33(1):42-47.
[17] 张 杰,王 佳,李浩宇,等.濒危植物蒙古扁桃不同地理种群遗传多样性的ISSR分析[J].生态学报,2012,32(14):4443-4452.
[18] 杜巧珍,红 雨.包贺喜图.珍稀濒危植物蒙古扁桃研究进展[J].内蒙古师范大学学报:自然科学汉文版,2010,39(3):308-312.
[19] 梅曙光,张国强,朱玉安,等.蒙古扁桃育苗技术研究[J].宁夏农林科技,2011,52(9):29-30.
[20] 哈伟贵.蒙古扁桃在海原南华山的人工繁殖育苗及栽培技术探讨[J].现代园艺,2014(20):24-24.
[21] 耿 军,朱立新,贾克功.蒙古扁桃单芽茎段培养体系的建立[J].西北农业学报,2007,16(5):218-221.
[22] 吴建华,王立英,李 健.蒙古扁桃的组织培养与快速繁殖技术研究[J].安徽农业科学,2010,38(4):1733-1734.
[23] 王 进,张 勇,赵 刚,等.蒙古扁桃种子萌发和幼苗生长对渗透胁迫的响应[J].中国园艺文摘,2015,32(8):236-236.
[24] 金丽萍,崔世茂,杜金伟,等.干旱胁迫对不同生态条件下蒙古扁桃叶片PAL和C_4H活性的影响[J].华北农学报,2009,24(5):118-122.
[25] 王琚钢,高晓敏,白淑兰,等.丛枝菌根对蒙古扁桃抗旱性影响研究[J].干旱区资源与环境,2014,28(12):138-142.
[26] Wang J G,Zheng R,Bai S L,et al.Mongolian almond(Prunusmongolicamaxim):the Morpho-physiological,biochemical and transcriptomic response to drought stress[J].PLoS One,2015,10(4):e0124442.
Mongolian Almond Ca2+Binding Protein Encoding Gene Systemic Identification and Expression Analysis
LIU Yang1,TIE Ying1,YANG Jia2,QI Zhi2,MAO Huiping1,2
(1.Inner Mongolia Hesheng Ecology Science and Technology Research Limited,Huhhot 010010,China;2.College of Life Sciences,Inner Mongolia University,Huhhot 010010,China)
Mongolian Almond(PrunusmongolicaMaxim)belongs to Rosaceae,under national protection of second class,primarily distributes at the Northwestern Dessert Region of China and is tolerant to drought and poor soil.Calcium ion is the most important signaling molecule for plant sensing and resistance to environment stresses.In this study,based on transcriptome analysis of the leaf,we identified sixteen strings of full length transcripts,including four Ca2+ATPase(ECA/ACA),three novel Ca2+channel protein with several transmembrane domains(ERD),five Ca2+/H+antiporter(CAX),two Ca2+dependent protein kinase(CPK)and two calmodulins(CAM).Their predicated structures are similar to those homologies inArabidopsis.Four single genes from theERD,CAX,CPKandCAMcategories,respectively,had obvious expression in the leaves and roots,among which theERD,CPKandCAMgenes expression had been regulated by drought and salt at different extent.It indicated that the genes might modulate the plant′s environmental stress-tolerant ability.
Mongolian almond;Transcriptome;Ca2+binding protein;Calmodulin
2016-11-12
内蒙古自治区科技重大专项(内财教 [2014]2020号)
刘 洋(1983-),女,内蒙古包头人,工程师,硕士,主要从事旱生灌木生理及分子生物学研究。
毛惠平(1973-),女,内蒙古呼和浩特人,讲师,博士,主要从事植物逆境胁迫生理及相关基因功能研究。
S662.9
A
1000-7091(2017)02-0081-06
10.7668/hbnxb.2017.02.013
