PCR-DGGE分析资中冬尖发酵过程中细菌多样性
2017-06-22汪淼,李张,孙群*
汪 淼,李 张,孙 群*
(四川大学生命科学院,生物资源与生态环境教育部重点实验室,四川 成都 610064)
PCR-DGGE分析资中冬尖发酵过程中细菌多样性
汪 淼,李 张,孙 群*
(四川大学生命科学院,生物资源与生态环境教育部重点实验室,四川 成都 610064)
目的:采用聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denatured gradient gel electrophoresis,PCR-DGGE)技术分析资中冬尖发酵过程中微生物群落结构及其动态变化。方法:从资中采集4 份不同发酵年份冬尖样品,提取样品总DNA,采用巢式PCR法扩增细菌16S rDNA V3区,扩增产物采用DGGE分离,对细菌主要优势条带进行克隆、测序、构建系统发育树。结果:冬尖在发酵过程中细菌多样性较丰富,且群落结构发生了较大变化。不同发酵时间样品间,细菌群落结构相似性为9%~67%,其中第2年与第3年样品相似性最高,达67%。资中冬尖发酵过程中,所涉及的细菌主要分为3 类,分别为厚壁菌门(Firmicutes)、变形杆菌门(Proteobacteria)和非培养菌,其中厚壁菌门为优势菌群,占53%;变形杆菌门占37%;非培养菌仅占10%。结论:采用PCR-DGGE技术能更全面、更真实地反映资中冬尖在发酵过程中微生物群落的多样性及其动态变化,为冬尖的生产工艺和风味物质的形成提供理论支撑。
冬尖;聚合酶链式反应-变性梯度凝胶电泳;细菌多样性
冬菜是我国传统的蔬菜腌制品,在国内外都颇有盛名,根据地域与加工方法的不同,主要分为川冬菜、京冬菜、津冬菜和上海五香冬菜等,其中四川冬菜主要以资中冬尖和南充冬菜最为著名[1]。资中冬尖选用荠菜为原料,其生产工艺特征是采用风脱水生产法,即先在风中晾晒,使之脱水,然后再进行盐渍,采用此加工方法可以减少蔬菜中所含可溶性物质的流出。资中冬尖发酵周期较长,一般1~4 a,随着发酵时间的延长,其风味及香气越浓郁,质量越好。成品冬尖呈褐黑色,味鲜细嫩,咸淡适口,回味带甜,深受消费者喜爱[2-4]。
冬尖发酵过程中,微生物群落结构的变化以及菌群间的相互作用对冬尖风味的形成起着重要作用,目前对冬尖的研究主要集中在其加工工艺、病害防治、卤水发酵、副产物等方面[5-7],而对冬尖发酵过程中微生物群落结构的变化研究甚少。早在1995年,万波等[8]采用传统培养法对资中冬尖发酵工艺中的微生物学问题进行了研究,对冬尖发酵过程中微生物群落结构变化有了初步了解,但由于采用的是传统培养法,严重降低了样品的微生物多样性,不能准确、全面地描绘发酵过程中微生物的概貌,且在此次研究中,分离培养出的微生物仅归纳到类,并未鉴定到种,因此有必要对资中冬尖发酵过程中微生物群落结构的变化做进一步研究。2012年,董玲等[9]采用16S rDNA-RFLP方法对腌制4 a的南充冬菜进行了细菌群落组成及多样性分析,但并未对发酵过程中微生物群落结构的变化进行研究。
1993年,Muyzer等[10]首次将聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denatured gradient gel electrophoresis,PCR-DGGE)引入微生物生态领域,该技术可通过免培养法再现原始群落结构,避免分离培养法造成的分析误差[11-12],目前该技术已成为研究微生物遗传多样性、种群结构和菌群差异的一种先进手段,被广泛应用于食品领域,如监测食品中微生物在时间或空间上的动态变化[13-15]、鉴定功能微生物和病原菌[16-18]、评价和控制食品质量[19-20]等。近年来,国内外许多学者采用PCRDGGE技术对腌制蔬菜中的微生物多样性进行研究,但主要涉及酸菜[21]、泡菜[22-23]、榨菜[24-25]、芽菜[26]、韩国泡菜[27-29]、麻竹笋[30]等,对冬尖的研究鲜见报道。本研究以不同发酵周期的资中冬尖为对象,采用PCR-DGGE技术,分析冬尖发酵过程中微生物群落结构及其动态变化,为冬尖的生产工艺和风味物质的形成提供理论基础。
1 材料与方法
1.1 材料与试剂
发酵0(入池之前冬尖)、1、2、3 a的冬尖样品,由汇源集团资中佳酿食品厂提供。
PCR master mix、PMD19-T Vector 日本Takara公司;去离子甲酰胺 上海酶联生物科技有限公司;四甲基二乙胺、双丙烯酰胺 美国Sigma公司;过硫酸铵 生工生物工程股份有限公司;凝胶回收试剂盒美国O m e g a公司;D H5 α感受态细胞、蛋白酶K(proteinase K,PK) 天根生化科技有限公司;聚乙烯吡咯烷酮(polyvinyl pyrrolidone,PVP)成都贝斯特试剂有限公司;十二烷基硫酸钠(sodium dodecyl sulfonate,SDS)、溴化十六烷基三甲基铵(cetyltrimethyl ammonium bromide,CTAB) 北京索莱宝科技有限公司。
1.2 仪器与设备
冷冻高速离心机 德国Eppendorf公司;PCR仪、凝胶成像仪、电泳仪、变性梯度凝胶电泳仪 美国Bio-Rad公司。
1.3 方法
1.3.1 总DNA提取
[31-32],在无菌条件下,将样品剪碎后加入20 μL TENP缓冲液,180 r/min摇床20 min。取1 mL样液加入EP管中,10 000 r/min离心2 min,弃上清液收集菌体,此步骤重复3 次;向菌体中加入1 mL PBS液,吹打均匀,10 000 r/min离心1 min,弃上清液,此步骤重复2 次;向EP管中加入少量小玻珠和适量石英砂,然后加入1 mL DNA提取液(100 mmol/L Tris、100 mmol/L EDTA、200 mmol/L NaCl、3% PVP,pH 9.0),旋涡振荡2 min,吹散菌团,再加入20 μL溶菌酶溶液充分混匀,37 ℃水浴30 min;向EP管中加入100 μL SDS和10 μL PK混匀,先37 ℃水浴30 min,再65 ℃水浴30 min,6 000 r/min离心15 min,收集上清液转入新EP管;向上清液中加入100 μL5 mol/L NaCl和80 μL CTAB-NaCl混匀,65 ℃水浴10 min,再加入780 μL氯仿-异戊醇(24∶1,V/V)混匀,4 ℃、15 000 r/min离心5 min,转上清液入新EP管;向EP管中加入等体积酚-氯仿-异戊醇(25∶24∶1,V/V)混匀,4 ℃、15 000 r/min离心5 min,取上清液转入新EP管中,再加入0.6 倍体积的异丙醇混匀,-20 ℃条件下静置1 h;EP管中加入500 μL 70%乙醇洗涤,4 ℃、15 000 r/min离心10 min,弃上清液,室温干燥10 min,加入50 μL 1×TE buffer,-20 ℃条件下保存备用。
1.3.2 PCR扩增
采用巢式PCR与降落PCR相结合进行扩增,引物见表1。
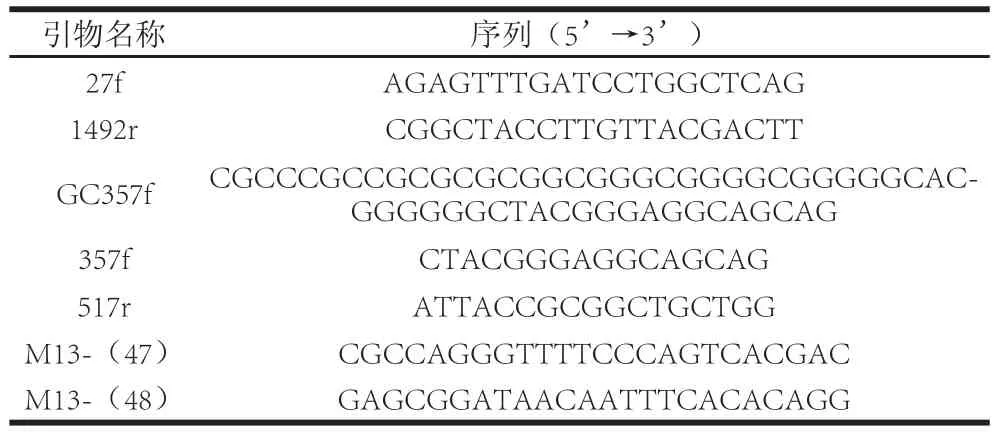
表1 PCR扩增引物Table1 PCR primers applied in this study
1.3.2.1 第1轮PCR扩增
以总DNA为模板,采用引物27f和1492r对细菌16S rDNA进行扩增。反应体系:上下游引物各0.5 μL,PCR master mix 12.5 μL,DNA模板0.5 μL,灭菌ddH2O补足至25 μL。反应程序采用降落PCR模式:94 ℃预变性4 min;94 ℃变性1 min,65 ℃退火1 min,每个循环退火温度依次降低0.5 ℃,72 ℃延伸2 min,20 个循环;当退火温度降至55 ℃,再以该温度扩增10 个循环;最后72 ℃终延伸10 min。
1.3.2.2 第2轮PCR扩增
以稀释100 倍的16S rDNA扩增产物为模板,采用引物GC357f和517r[23,33]对细菌16S rDNA V3区进行扩增。反应体系:上下游引物各1 μL、PCR master mix 25 μL、DNA模板2 μL、灭菌ddH2O补足至50 μL。反应程序:94 ℃预变性5 min;94 ℃变性1 min,58 ℃退火1 min,72 ℃延伸2 min,35 个循环;最后72 ℃终延伸10 min。
1.3.3 DGGE及条带测序
每个样品取35 μL PCR产物与7 μL 6×loading buffer混匀后加入到点样孔中。采用8%聚丙烯酰胺变性凝胶,变性梯度为45%~65%。在1×TAE缓冲液中,PCR产物在恒温60 ℃、恒压60 V条件下电泳12 h。电泳结束后取出胶放入SYBR GreenⅠ染液中浸染30 min,使用凝胶成像仪拍照。DGGE图谱中条带的亮度在一定程度上能反映菌种的数量,较亮的条带即为样品中的优势菌群,对图谱中较亮的条带进行切胶,条带回收,回收的条带进行一次无GC夹板的PCR扩增,扩增子载入PMD19-T中进行克隆,阳性克隆子菌液送美吉生物公司测序。
1.4 数据处理
将DGGE图谱用Quantity One软件进行处理,分析各样品条带的灰度值、数量及迁移率,对各样品相似性采用非加权组平均法(unweighted pair-group method with arithmetic means,UPGMA)法进行聚类分析,同时根据香龙-威纳多样性指数(H)、丰富度指数(D)和均匀度指数(EH)评价各样品的多样性[34],分别根据公式(1)~(3)计算:


式中:Pi为第i条条带的灰度值占样品总灰度值的比值;S为每一泳道中的总条带数;N为条带总数。
测序结果采用DNAstar和Cluster软件进行序列分析,下载最相似的菌株序列作为系统发育树的参考序列。然后采用MEGA软件,Neighbor-Joining法构建系统发育树,自展数(Bootstrap)为1 000。
2 结果与分析
2.1 巢式PCR扩增结果
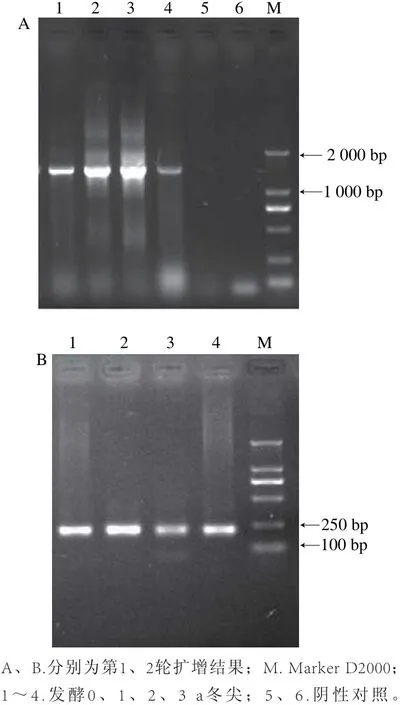
图1 细菌巢式PCR电泳图Fig.1 Electrophoresis profiles of bacterial nested PCR products
由图1A可知,第1轮PCR对细菌16S rDNA进行扩增,目的产物大小约1.6 kb,所有样品扩增条带均与目的条带一致,可用于后续实验。由图1B可知,第2轮PCR对细菌16S rDNA V3区进行扩增,目的产物大小约230 bp,所有样品扩增条带均与目的条带一致,可用于DGGE分析。
2.2 细菌DGGE指纹图谱
DGGE图谱中条带的多少能直接反映样品中微生物的多样性,条带的亮度在一定程度上能反映菌种数量的多少[35]。从图2可知,不同年份冬尖样品电泳后分离出的条带数目、迁移率及亮度均存在差异,说明冬尖在发酵过程中细菌群落结构发生了改变。从DGGE图谱可以很直观地看出发酵1 a的冬尖样品其电泳条带数目最多,丰度最大,而发酵2 a的冬尖样品其条带数目相对较少。多样性指数是研究群落物种和个体数及其分布均匀度的综合指标[34],为了更准确地比较不同样品的多样性情况,根据1.4节计算各样品的多样性指数、丰富度指数和均匀度指数。第1年冬尖样品其多样性指数和丰度最高,分别为2.540和1.675,而第2年冬尖样品其多样性指数和丰度最低,分别为1.450和0.829,这个结果与DGGE图谱直接观测结果相一致。
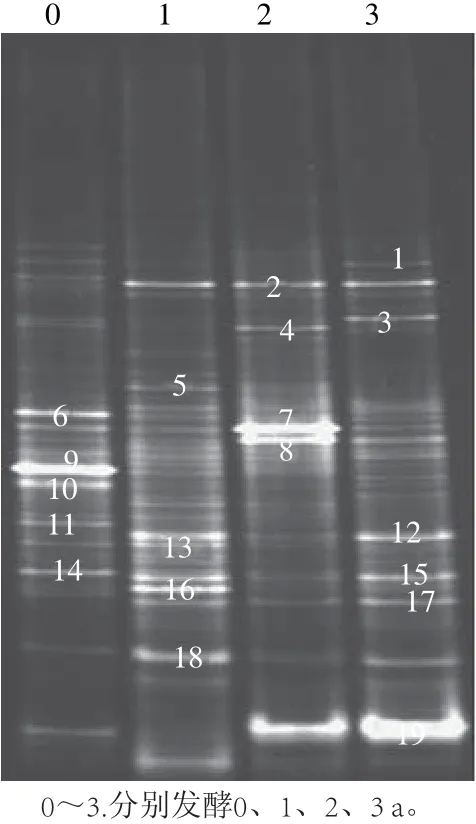
图2 冬尖发酵各阶段细菌DGGE图谱Fig.2 DGGE profiles of bacterial community in Dongjian at different fermentation stages
2.3 DGGE指纹图谱聚类分析
在DGGE指纹图谱中,不同样品共有条带数目的多少可以反映样品间微生物群落结构的相似性,一般采用UPGMA对样品间微生物群落结构的相似性进行评估。资中冬尖0~3 a样品的细菌聚类分析结果见图3。从图3可知,各样品间的相似性在9%~67%,说明冬尖在发酵过程中,随着发酵时间的延长,细菌群落结构发生了较大变化。其中第2年和第3年冬尖样品的相似性最高,为67%,而第1年与第2、3年冬尖的相似性最低,分别只有9%和17%。这可能是因为冬尖在发酵过程中,第1年为好氧向厌氧转变的阶段,此时大部分微生物还能生长繁殖,因此第1年冬尖样品中微生物群落结构最丰富,从DGGE指纹图谱中也能看出其条带数目最多。当发酵继续进行到第2、3年时,已完全转变为厌氧环境,且营养物质也被大量消耗,使大部分微生物生长繁殖受到抑制,只有一部分适应此环境的特定菌群能很好的生长,从而使第2年和第3年冬尖样品中微生物菌群结构具有一定趋向性,这也解释了为什么第2、3年样品中微生物菌群结构相似性最高。
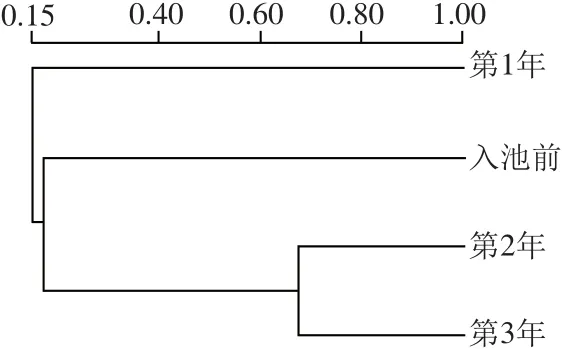
图3 冬尖细菌DGGE指纹图谱聚类分析Fig.3 Cluster analysis of DGGE profiles for bacterial communities of Dongjian
2.4 DGGE条带测序及同源性分析
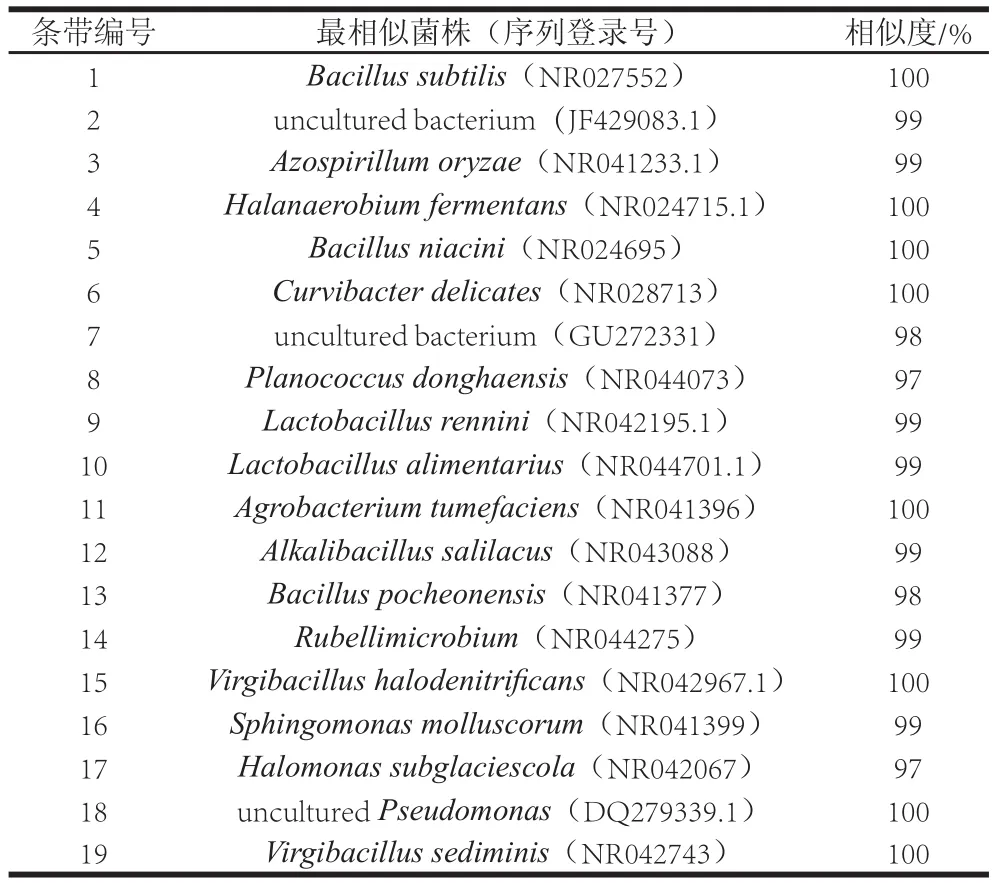
表2 细菌DGGE条带测序结果Table2 Sequencing results from the bands excised from the bacterial DGGE gels
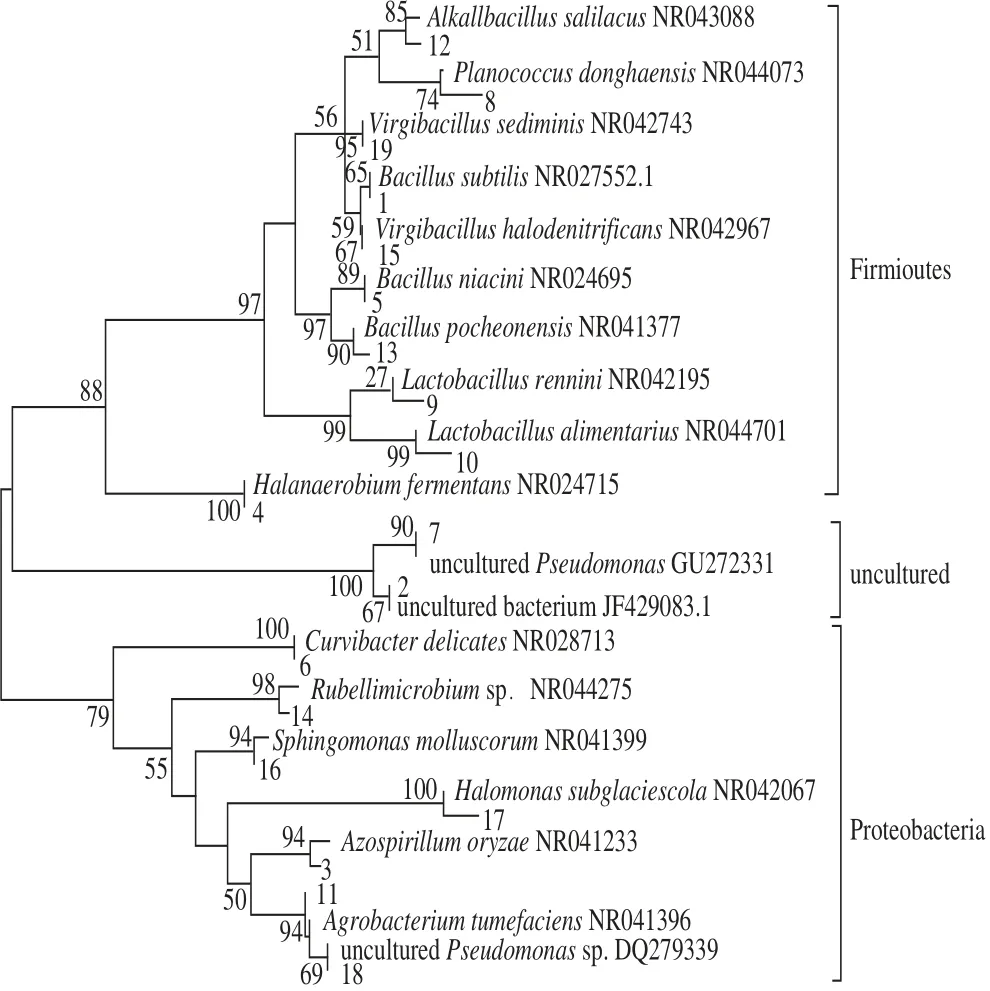
图4 冬尖细菌系统发育分析Fig.4 Phylogenetic analysis of bacterial species in Dongjian
将细菌DGGE图谱上较亮的代表条带,共19 条进行切胶、克隆、测序,并构建进化树,测序结果见表2,进化树图谱见图4。由表2结果显示,所有序列与数据库中16S rDNA序列的相似性在97%~100%之间,说明均具有鉴定意义。从细菌N-J进化图可以看出,资中冬尖在发酵过程中,所涉及的细菌主要分为3 类,分别为厚壁菌门(Firmicutes)、变形杆菌门(Proteobacteria)和非培养菌。其中厚壁菌门细菌类群为优势菌,占53%;其次是变形杆菌门,占37%;最少的为非培养菌,占10%。
3 讨论与结论
资中冬尖是以芥菜为原料采用自然发酵制成的蔬菜腌制品,其风味的形成受众多因素影响,如原料、工艺、发酵时间、温度等,其中发酵过程中微生物的菌群变化与代谢活动是影响冬尖风味的重要因素。目前关于资中冬尖发酵过程中微生物多样性的研究较少,本研究以不同发酵周期的资中冬尖为对象,采用PCR-DGGE技术,对冬尖发酵过程中微生物群落结构及其动态变化进行分析。
结果表明,不同年份的冬尖样品其电泳后分离出的条带数目、迁移率及亮度均存在差异,说明冬尖在发酵过程中细菌群落结构发生改变,且发酵1 a的冬尖样品其条带数目最多,细菌多样性较丰富,而发酵2 a的冬尖样品其条带数目相对较少。对DGGE指纹图谱进行聚类分析,结果表明,各样品间的相似性在9%~67%,说明冬尖在发酵过程中,细菌群落结果发生了较大改变,其中第2年和第3年冬尖样品的相似性最高,为67%,而第1年与第2年冬尖的相似性最低仅9%。从DGGE图谱上切割回收19 条条带,测序结果表明,资中冬尖在发酵过程中,所涉及的细菌主要分为3 类,分别为厚壁菌门、变形杆菌门和非培养菌。其中厚壁菌门细菌类群为优势菌,占53%,其次是变形杆菌门,占37%,最少的为非培养菌,占10%。董玲等[9]采用16S rDNA-RFLP方法对腌制4 a的南充冬菜进行了细菌多样性分析,结果表明发酵4 a的南充冬菜,其细菌结构也主要分为3类,分别为变形杆菌门、后壁门和放线菌门,其中以变形杆菌门细菌类群为优势菌群。从本研究分离菌株所属类别可以看出,资中冬尖在发酵过程中,存在大量中度嗜盐菌和芽孢杆菌科类群,这可能与资中冬尖发酵过程中形成的高盐厌氧环境有关。芽孢杆菌可以产生抗逆性极强的内生芽孢来度过不利的生长条件,因此即使是在高盐厌氧这种恶劣的环境中也可以生长繁殖,此外,芽孢杆菌具有较好的抑菌防病作用,有利于冬尖发酵的防腐。中度嗜盐菌分布在很多属中,主要生活在海洋、盐场、盐湖和腌制品等高盐环境中[36]。资中冬尖的盐浓度在16%左右,在这种高盐环境中,大多数微生物的生长都受到了抑制,但却非常适合中度嗜盐菌的生长,因此在冬尖发酵样品中检测出大量中度嗜盐菌,包括:Halanaerobium fermentans、Alkalibacillus salilacus、抱川芽孢杆菌(Bacillus pocheonensis)、盐脱硝枝芽孢杆菌(Virgibacillus halodenitrificans)、冰下盐单胞菌(Halomonas subglaciescola)、沉积物枝芽孢杆菌(Virgibacillus sediminis)。
根据测序结果,对DGGE指纹图谱中条带在不同样品中出现的情况进行分析。条带1和3的测序结果分别为枯草芽孢杆菌(Bacillus subtilis)(100%)和Azospirillum oryzae(99%)的近源种,结合DGGE指纹图谱可以看出,条带1和3在入池之前的冬尖样品中出现,但条带亮度均较暗,随着发酵时间的延长,条带在第1、2年冬尖样品中消失,但在第3年样品中又重新出现,且条带3为第3年冬尖样品中的优势菌。出现这种现象的原因可能是条带1和3在第1、2年样品中属于弱势菌群,而DGGE一般只能检测出在总的微生物群落中占1%以上的种群,而对弱势菌群不能检测出。万波等[8]采用传统培养法对资中冬尖发酵1、2、3 a的样品进行了微生物群落的研究,结果表明乳酸菌在资中冬尖发酵过程中受到了抑制并不是主要菌群。条带9和10测序结果分别为Lactobacillus rennini(99%)和消化乳杆菌(Lactobacillus alimentarius)(99%)的近源种。资中冬尖的盐含量在16.1%左右,而一般认为盐含量低于12%有利于乳酸菌的生长,高于12%乳酸菌生长受到抑制。从DGGE指纹图谱可以看出,条带9和10是入池之前冬尖样品中的优势菌,条带较亮,但随着发酵时间的延长,条带消失或者变暗,说明乳酸菌在冬尖发酵过程中受到了抑制,此结果与万波等[8]研究结果相一致。条带11为根癌土壤杆菌(Agrobacterium tumefaciens)(100%)的近源种,只存在于入池前冬尖样品中,在发酵过程中消失。根癌农杆菌是一种严重危害植物生长的病害,能感染双子叶植物的受伤组织,引起冠瘿瘤,资中冬尖的原料芥菜属于双子叶植物,容易受根瘤农杆菌的侵染,冬尖在发酵过程中形成了高盐厌氧环境,可能正是这种恶劣的环境抑制了根瘤农杆菌在发酵过程中的生长。综上所述,采用PCR-DGGE技术能更直观、更全面地展现冬尖发酵过程中细菌群落结构及其动态变化,为冬尖生产工艺及其风味形成原理提供理论依据。
参考文献:
[1] 叶兴乾. 果品蔬菜加工工艺学[M]. 北京: 中国农业出版社, 2009: 5-6.
[2] 陈曾三. 资中冬尖和南充冬菜[J]. 中国调味品, 2003, 28(8): 41-44. DOI:10.3969/j.issn.1000-9973.2003.08.011.
[3] 李宗斌. 资中冬尖的加工技术[J]. 重庆师专学报, 2001, 20(4): 103-104.
[4] 刘达玉, 董凯锋, 王卫, 等. 资中冬尖产业化生产工艺与技术[J]. 中国调味品, 2011, 36(9): 76-79. DOI:10.3969/j.issn.1000-9973.2011.09.020.
[5] 薛玉霞. 冬菜常见生理病害防治措施[J]. 农药市场信息, 2011(3): 45.
[6] 邓林, 刘延岭. 四川冬菜卤水发酵制取酱油的工艺研究[J].食品与发酵科技, 2012, 48(3): 26-29. DOI:10.3969/j.issn.1674-506X.2012.03-007.
[7] 杨勇, 詹永, 祝卢艺, 等. 利用冬菜副产物研制新型复合调味汁[J]. 中国调味品, 2011, 36(2): 75-77.
[8] 万波, 赵海, 李安明, 等. 冬尖发酵的微生物学研究[J]. 应用与环境生物学报, 1995, 1(2): 196-200.
[9] 董玲, 蒲彪, 敖晓琳. 四川冬菜中细菌群落组成及多样性[J]. 微生物学报, 2012, 52(4): 519-525. DOI:10.13343/j.cnki.wsxb.2012.04.012.
[10] MUYZER G, de WAAL E, UITTERLINDEN A. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700.
[11] 许文涛, 郭星, 罗云波, 等. 微生物菌群多样性分析方法的研究进展[J].食品科学, 2009, 30(7): 258-265.
[12] LÜ X C, WENG X, ZHANG W, et al. Microbial diversity of traditional fermentation starters for Hong Qu glutinous rice wine as determined by PCR-mediated DGGE[J]. Food Control, 2012, 28(2): 426-434. DOI:10.1016/j.foodcont.2012.05.025.
[13] ZOI P, NICHOLAS C, GWEN F, et al. Comparison of the bacterial species diversity of spontaneous cocoa bean fermentations carried out at selected farms in Ivory Coast and Brazil[J]. Food Microbiology, 2011, 28(5): 964-973. DOI:10.1016/j.fm.2011.01.010.
[14] GONZÁLEZ-ARENZANA L, LÓPEZ R, SANTAMARÍA P, et al. Dynamics of lactic acid bacteria populations in Rioja wines by PCR-DGGE, comparison with culture-dependent methods[J]. Applied Microbiology and Biotechnology, 2013, 97(15): 6931-6941. DOI:10.1007/s00253-013-4974-y.
[15] 袁雪林, 杨洁, 胡敏, 等. 新疆喀什地区传统发酵酸乳中乳酸菌多样性的初步分析[J]. 食品工业科技, 2015, 36(10): 202-204. DOI:10.13386/j.issn1002-0306.2015.10.033.
[16] NIKOLIC M, TERZIC-VIDOJEVIC A, JOVCIC B, et al. Characterization of lactic acid bacteria isolated from Bukuljac, a homemade goat’s milk cheese[J]. International Journal of Food Microbiology, 2008, 122(1/2): 162-170. DOI:10.1016/ j.ijfoodmicro.2007.11.075.
[17] 毕水莲, 陈妙瑞, 张志刚, 等. Fla-DGGE法对食品中空肠弯曲菌和结肠弯曲菌的检测和分型[J]. 现代食品科技, 2010, 26(10): 1148-1152. DOI:10.13982/j.mfst.1673-9078.2010.10.020.
[18] 徐晓东, 张孝卫, 杜雪, 等. 应用PCR-DGGE技术快速检测病原微生物的研究[J]. 中国卫生检验杂志, 2011, 21(5): 1058-1060.
[19] RANDAZZO C L, VAUGHAN E E, CAGGIA C. Artisanal and experimental Pecorino Siciliano cheese: microbial dynamics during manufacture assessed by culturing and PCR-DGGE analyses[J]. International Journal of Food Microbiology, 2006, 109(1/2): 1-8. DOI:10.1016/j.ijfoodmicro.2005.11.002.
[20] RANDAZZO C L, RIBBERA A, PITINO I, et al. Diversity of bacterial population of table olives assessed by PCR-DGGE analysis[J]. Food Microbiology, 2012, 32(1): 87-96. DOI:10.1016/j.fm.2012.04.013.
[21] 从敏, 李欣蔚, 武俊瑞, 等. PCR-DGGE分析东北传统发酵酸菜中乳酸菌多样性[J]. 食品科学, 2016, 37(7): 78-82. DOI:10.7506/spkx1002-6630-201607015.
[22] 张先琴, 张小平, 敖晓琳, 等. PCR-DGGE分析四川地区家庭制作泡菜中微生物多样性[J]. 食品科学, 2013, 34(12): 129-134. DOI:10.7506/ spkx1002-6630-201312027.
[23] 梁新乐, 朱扬玲, 蒋予箭, 等. PCR-DGGE法研究泡菜中微生物群落结构的多样性[J]. 中国食品学报, 2008, 8(3): 134-137. DOI:10.16429/ j.1009-7848.2008.03.013.
[24] 李正国, 付晓红, 邓伟. 传统分离培养结合DGGE法检测榨菜腌制过程的细菌多样性[J]. 微生物学通报, 2009, 36(3): 371-376. DOI:10.13344/j.microbiol.china.2009.03.017.
[25] 燕平梅, 乔宏萍, 赵文婧, 等. 基于PCR-DGGE方法分析榨菜中乳酸菌群落结构[J]. 食品科学, 2016, 37(13): 136-139. DOI:10.7506/ spkx1002-6630-201613024.
[26] WEISS A, HERTEL C, GROTHE S, et al. Characterization of the cultivable microbiota of sprouts and their potential for application as protective cultures[J]. Systematic and Applied Microbiology, 2007, 30(6):483-493. DOI:10.1016/j.syapm.2007.03.006.
[27] KIM M, CHUN J. Bacterial community structure in kimchi, a Korean fermented vegetable food, as revealed by 16S rRNA gene analysis[J]. International Journal of Food Microbiology, 2005, 103(1): 91-96. DOI:10.1016/j.ijfoodmicro.2004.11.030.
[28] LEEA J S, HEO G Y, LEE J W, et al. Analysis of kimchi microflora using denaturing gradient gel electrophoresis[J]. International Journal of Food Microbiology, 2005, 102(2): 143-150. DOI:10.1016/ j.ijfoodmicro.2004.12.010.
[29] NAM Y D, CHANG H W, KIM K H, et al. Metatranscriptome analysis of lactic acid bacteria during kimchi fermentation with genome-probing microarrays[J]. International Journal of Food Microbiology, 2009, 130(2): 140-146. DOI:10.1016/j.ijfoodmicro.2009.01.007.
[30] 郑炯, 夏雪娟, 叶秀娟, 等. PCR-DGGE技术分析腌制麻竹笋中微生物多样性[J]. 食品科学, 2014, 35(21): 170-174. DOI:10.7506/spkx1002-6630-201421033.
[31] 王丽娜, 许修宏, 宛煜嵩, 等. 三种土壤微生物总DNA提取方法的比较[J]. 基因组学与应用生物学, 2009, 28(2): 331-334. DOI:10.3969/ gab.028.000331.
[32] 李安英, 张潞生, 高微微, 等. 适于变性梯度凝胶电泳(DGGE)分析的草莓根际土壤微生物的DNA模板制备[J]. 农业生物技术学报, 2009, 17(4): 701-706. DOI:10.3969/j.issn.1674-7968.2009.04.027.
[33] DONG D, YAN A, LIU H, et al. Removal of humic substances from soil DNA using aluminium sulfate[J]. Journal of Microbiological Methods, 2006, 66(2): 217-222. DOI:10.1016/j.mimet.2005.11.010.
[34] KUANG Y, TANI K, SYNNOTT A J, et a1. Characterization of bacterial population of raw milk from bovine mastits by culture-independent PCR-DGGE method[J]. Biochemical Engineering Journal, 2009, 45(1):76-81. DOI:10.1016/j.bej.2009.02.010.
[35] 江云飞, 蔡柏岩. PCR-DGGE技术在细菌多样性研究中的条件优化[J].生物技术, 2009, 19(5): 84-87.
[36] KUSHNER D J. Life in high salt and solute concentrations: halophilic bacteria[M]//Microbial life in extreme environments. Salt Lake City:Academic Press, 1978: 317-368.
PCR-DGGE Analysis of Bacterial Diversity during Fermentation of Zizhong Dongjian, a Chinese Traditional Fermented Vegetable Product
WANG Miao, LI Zhang, SUN Qun*
(Key Laboratory of Bio-Resources and Eco-Environment, Ministry of Education, College of Life Sciences, Sichuan University, Chengdu 610064, China)
Objective: To investigate dynamic changes in the microbial community structure during the fermentation of Zizhong Dongjian, a Chinese traditional fermented vegetable product, by polymerase chain reaction-denatured gradient gel electrophoresis (PCR-DGGE). Methods: Four samples of Dongjian with different fermentation times were collected from Zizhong, Sichuan province. Total bacterial DNA was extracted from the samples, and nested PCR was applied to amplify the V3 region of 16S rDNA for identification based on DGGE fingerprints. Meanwhile, the dominant bands were cloned, selected and sequenced, and a phylogenetic tree was constructed. Results: During the fermentation process of Dongjian, rich bacterial diversity was observed and the microbial community structure was changed greatly. The similarity of bacterial community between Dongjian samples with different fermentation times ranged from 9% to 67%. Among them, samples fermented for two and three years exhibited the highest similarity to each other, up to 67%. Furthermore, the bacteria involved in the fermentation process of Dongjian were mainly divided into3 categories: Firmicutes, Proteobacteria, and unculturable bacteria. Among these, Firmicutes was the most dominant species followed by Proteobacteria, which accounted for 53% and 37% of the total population, respectively, while unculturable bacteria accounted for only 10%. Conclusions:DGGE fingerprint can provide a comprehensive and true reflection of dynamic changes in the microbial diversity of Dongjian during fermentation. Meanwhile, it also provides a theoretical basis for the production process for Dongjian and the formation of flavor substances.
Dongjian; polymerase chain reaction-denatured gradient gel electrophoresis; bacterial diversity
10.7506/spkx1002-6630-201712018
TS201.3
A
1002-6630(2017)12-0119-06
汪淼, 李张, 孙群. PCR-DGGE分析资中冬尖发酵过程中细菌多样性[J]. 食品科学, 2017, 38(12): 119-124.
10.7506/ spkx1002-6630-201712018 http://www.spkx.net.cn
WANG Miao, LI Zhang, SUN Qun. PCR-DGGE analysis of bacterial diversity during fermentation of Zizhong Dongjian, a Chinese traditional fermented vegetable product[J]. Food Science, 2017, 38(12): 119-124. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201712018. http://www.spkx.net.cn
2016-07-07
汪淼(1987—),女,硕士,研究方向为微生物检测。E-mail:wangmiaosichuan@163.com
*通信作者:孙群(1967—),女,教授,博士,研究方向为微生物技术与食品安全。E-mail:qunsun@scu.edu.cn
