蚕豆酱传统发酵微生物群落中的菌株相互作用分析
2017-06-19范子豪朱林江刘思远王佳琪李崎
范子豪,朱林江,刘思远 ,王佳琪 ,李崎*
1(江南大学,工业生物技术教育部重点实验室,江苏 无锡,214122) 2(江南大学 生物工程学院,江苏 无锡,214122)
蚕豆酱传统发酵微生物群落中的菌株相互作用分析
范子豪1,2,朱林江1,2,刘思远1,2,王佳琪1,2,李崎1,2*
1(江南大学,工业生物技术教育部重点实验室,江苏 无锡,214122) 2(江南大学 生物工程学院,江苏 无锡,214122)
蚕豆酱传统发酵过程中微生物菌群在不同批次保持相对稳定,为了分析这种微生物群落的稳定形成机制,该研究以从发酵过程中分离的不同基因型菌株为实验对象,利用杯碟法和滤纸片法分析优势好氧菌与兼性厌氧芽孢杆菌、乳酸菌及酵母菌的相互作用,同时利用PCR技术检测优势菌株基因组内可能存在的抗菌肽编码基因。研究结果表明,所有厌氧菌和酵母菌株均被1株或多株优势生长的好氧芽孢杆菌菌株抑制,而且部分优势菌株存在较强的抑制作用,同时抑制多个菌种的生长,其中以1株BacillussubtilisHYM05,1株B.amyloliquefaciensHYM24以及1株B.tequilensisHYM42为典型代表。通过PCR探测,发现HYM42和HYM05的基因组中存在编码Subtilosin以及Plipastatin的基因,而3株菌的基因组中均没有完整地编码Surfactin的3个基因。所以,蚕豆酱传统酿造过程中,好氧芽孢杆菌存在生长优势,通过多个菌株之间的协同相互作用,抑制其他微生物生长,在各个批次之间形成相对稳定的微生物群落结构。
相互作用;杯碟法;滤纸片法;芽孢杆菌;细菌素
豆瓣酱,也叫蚕豆酱,以豆瓣、谷物、面粉等植物性原料发酵而成,是我国很多地区传统的调味品。豆瓣营养丰富,经过发酵后,大分子物质经过微生物代谢和一些物理化学反应后被分解转化成多肽、氨基酸、脂肪酸、低聚糖、单糖等,加上光照、温度变化等也产生一系列非酶促化学反应。这些复杂的微生物发酵和非酶促反应,给豆瓣酱产品带来独特的风味特色[1-2]。
豆瓣酱的酿造历史可以追溯到清朝。数百年来人们都遵循着最传统的酿造技艺,大致可以分为4个部分,包括原料的处理,制曲,发酵和罐装[3]。酿造的核心是微生物发酵,豆瓣酱的发酵过程是一个天然的多菌种混合发酵过程。蒯辉等人利用基于培养的PCR指纹技术和未培养的PCR-DGGE技术,跟踪研究了豆瓣酱发酵过程中微生物群落的动态变化。研究结果表明,豆瓣酱的发酵过程有多种微生物参与,好氧芽孢杆菌为优势菌群,包括枯草芽孢杆菌、淀粉液化芽孢杆菌、特基拉芽孢杆菌等;同时检测到数量较少的厌氧菌、乳酸菌和酵母菌,其中厌氧菌以兼性的蜡状芽孢杆菌为主,而乳酸菌在发酵前期的盐水中大量存在,但发酵10天后其数量显著下降,并持续保持较低水平;酵母菌在发酵中期开始生长并被检测到,但是一直保持在较低水平。此外,通过比较多个不同时间的发酵批次的微生物群落表明,这种微生物群落结构的变化可在不同发酵批次之间保持相对稳定[4]。
不同地区的豆酱发酵过程的微生物群落结构存在一定差异,体现地域差异性,比如我国东北地区的大酱传统发酵过程含有芽孢杆菌、乳杆菌、明串珠菌等[5],而在东部地区生产豆酱的过程则以芽孢杆菌、乳杆菌、鲁氏酵母等为主[6];部分韩国豆酱的生产过程由芽孢杆菌主导,而另一些则由乳酸菌主导[7]。
所以,传统豆酱生产过程中会形成具有各自特点的微生物群落,目前的研究主要集中在酱醅微生物群落组成,对豆瓣酱发酵过程中微生物群落稳态形成机制却研究很少。一般而言,微生物相互作用是微生物群落结构稳定形成的重要基础,本次研究在前期的蚕豆酱发酵微生物群落结构研究基础上,利用分离培养的各个菌株,分析菌株之间的相互作用,并且利用PCR技术初步分析了具有抑菌能力的好氧芽孢杆菌菌株的抗菌肽编码基因,希望能为酱醅中微生物群落稳定的原因提供合理解释及证据。
1 材料与方法
1.1 实验所用菌种
实验所用菌种均为从豆瓣酱酱醅中分离所得,并且由本实验室保藏。在菌株相互作用分析实验过程中,将所用菌株分为实验菌和指示菌,实验菌是指在酱醅发酵过程中的优势好氧菌株,包括解淀粉芽孢杆菌,特基拉芽孢杆菌以及枯草芽孢杆菌;指示菌为酱醅发酵过程中,数量保持在较低水平的菌株,包括蜡状芽孢杆菌、乳酸菌和酵母菌,具体菌种名,菌种来源以及编号见表1。

表1 实验菌和指示菌菌种名称及编号
1.2 培养基及培养条件
芽孢杆菌使用LB培养基进行活化和扩培,37 ℃好氧摇床培养24 h;乳酸菌使用MRS培养基进行活化和扩培,37 ℃厌氧培养24 h;酵母菌使用YPD培养基进行活化和扩培,28 ℃好氧培养48 h。
1.3 优势好氧菌对蜡状芽孢杆菌、乳酸菌以及酵母菌的抑制作用分析
1.3.1 滤纸片法[8]
使用中速定性滤纸,将滤纸剪成直径1.6 cm的小滤纸片,灭菌备用。实验菌株好氧培养,使用LB培养基,培养至OD600达到1.2。将灭菌的琼脂培养基(芽孢杆菌用LB,乳酸菌用MRS,酵母用YPD培养基)冷却至50 ℃左右倒板,凝固后,涂布100 μL活化好的指示菌,再用滤纸片均匀地蘸取实验菌培养液,平坦地放置在平板上,以无菌水作为对照,37 ℃培养48 h后测量抑菌圈直径。
1.3.2 杯碟法(牛津杯法)[9]
牛津杯使用药典级,内径6 mm,外径8 mm,高10 mm,误差0.02 mm,灭菌备用。实验菌培养至OD600达到1.2后,12 000 r/min离心10 min,取上清液,用0.22 μm滤膜滤菌备用。将灭菌的琼脂培养基制成平板,指示菌在菌浓达到OD600为0.7的时候涂布平板。将牛津杯竖直放在涂布有指示菌的平板上,轻轻按压。在牛津杯中加入230 μL上述培养液上清液,并将平板放入4 ℃冰箱中预扩散8~10 h,然后放入恒温培养箱中,适宜温度培养48 h,观察是否出现抑菌圈。
1.4 PCR检测优势芽孢杆菌基因组的抗菌肽合成基因
1.4.1 实验菌基因组DNA的提取
各个菌株基因组的提取,采用十六烷基三甲基溴化铵法(CTAB)[10]。
1.4.2 抗菌肽基因的PCR检测
采用TouchDown-PCR (TD-PCR)进行PCR检测,可降低假阳性的现象[11-13]。
检测subtilin,subtilosin,surfactin以及plipastatin的编码基因[11-13]。编码基因以及对应的引物如表2所示。TD-PCR反应体系为50 μL,包括25 μL Premix Taq (TaKaRa),5 μL上游引物,5 μL下游引物,5 μL模板以及10 μL ddH2O。TD-PCR反应条件以编码subtilin的SpaS基因为例,95 ℃预变性3 min,首先94 ℃变性30 s,60 ℃退火50 s,72 ℃延伸1 min完成第1个循环,随后退火温度降低0.5 ℃继续上一个循环,循环20次,直至退火温度降低为50 ℃,然后进行95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸50 s,15个循环,其余基因只需要改变温度梯度的上下限和退火温度即可,最后用1%琼脂糖凝胶电泳验证。

表2 Subtilin,subtilosin,surfactin以及plipastatin的编码基因及其引物
2 结果与讨论
2.1 优势芽孢杆菌与蜡状芽孢杆菌的相互作用
在蚕豆酱的发酵过程中,优势好氧芽孢杆菌的数量维持在108CFU/g水平,而兼性厌氧的蜡状芽孢杆菌在103~104CFU/g,2者相差4个数量级。使蜡状芽孢杆菌维持在较低水平,是该蚕豆酱发酵的特点之一。为了解析这一群落结构形成的内在机制,将所有分离的好氧菌菌株与随机挑选的蜡状芽孢杆菌菌株进行相互作用分析。根据初筛结果,所有的优势芽孢杆菌中,有3株具有较为广谱的抑菌能力的细菌,分别是BacillussubtilisHYM05,B.amyloliquefaciensHYM24和B.tequilensisHYM42,后续实验均以这3株菌为实验菌。指示菌所用的蜡状芽孢杆菌均为酱醅中分离所得,随机挑选其中5株作为实验指示菌,编号分别为HYM79,HYM88,HYM89,HYM78和HYM77。实验中,3株实验菌对指示菌均表现出较好的抑制性能,抑菌实验结果如表3所示。滤纸片法和杯碟法的结果相一致(表4),而滤纸片法可以更为直观地通过抑菌圈直径来判断实验菌抑菌能力的强弱。3株实验菌对不同蜡状芽孢杆菌菌株的抑菌效果不同,HYM05不能抑制HYM78的生长,而对HYM77的抑制效果较为明显,抑菌圈直径达到了2.27cm,如图1A所示。HYM42能够抑制5株蜡状芽孢杆菌中的4株,且产生较明显的抑菌圈,如以HYM78为指示菌时,抑菌圈最大,直径为2.33cm,如图1B所示。由以上结果可知,HYM05、HYM24和HYM42之间存在不同的抑菌机制,此外,所有分离培养的蜡状芽孢杆菌菌株均被1株或多株好氧芽孢杆菌菌株所抑制。所以,蜡状芽孢杆菌在蚕豆发酵过程中维持在较低水平,可能与好氧芽孢杆菌的抑制作用相关。其实,这种芽孢杆菌之间的相互作用,也在其他传统发酵过程中被发现,如韩国豆酱[14]和西非的发酵调味品等[15]。

图1 滤纸片法分析B. subtilisHYM05,B. amyloliquefaciensHYM42对B.cereusHYM77和HYM78的抑菌作用Fig.1 The inhibitory effect of HYM05 and HYM42 on HYM77 and HYM75 respectively

BacillussubtilisBamyloliquefaciensBtequilensisHYM05HYM24HYM42蜡状芽孢杆菌BacilluscereusHYM79+--BcereusHYM88++++BcereusHYM89++++BcereusHYM78-+++BcereusHYM77++++乳酸菌WeissellaconfusaHYM106+--WconfusaHYM105---WparamesenteroidesHYM109+--WparamesenteroidesHYM157-+-StreptococcusmacedonicusHYM119--+SmacedonicusHYM136+--酵母菌Zygosaccharomycesrouxii4#--++
注:-表示没有抑菌圈,+表示有抑菌圈且直径在1.6~1.9 cm,++表示有抑菌圈且直径在2.0~2.3 cm。

表4 杯碟法相互作用结果
注:-表示没有抑菌圈,+表示有抑菌圈。
2.2 优势芽孢杆菌与乳酸菌的相互作用分析
不同于其他传统发酵过程,蚕豆酱发酵过程中乳酸菌的数量很少。虽然在发酵初期的盐水能够大量存在,多个菌株已被分离培养,但发酵10 d后往往很难被检测。这一现象与其他豆酱发酵过程存在较大差异[4],所以将分离培养的乳酸菌菌株与具有优势生长的好氧菌株之间进行相互作用分析。首先是随机选择2株乳酸菌与所有好氧芽孢杆菌菌株进行相互作用分析,结果表明同样的3株菌HYM05、HYM24和HYM42对乳酸菌表现出一定的抑制作用,如表3所示。牛津杯法得到的结果和滤纸片法不同,HTM24和HYM42在牛津杯法实验中没有表现出对乳酸菌的抑制作用,如表4所示。
3株实验菌中,HYM05对乳酸菌的抑制作用最为明显,如对HYM109的抑菌圈为1.97 cm,如图2所示。根据抑菌圈大小判断,对乳酸菌的抑制作用要小于对蜡状芽孢杆菌的抑制作用。而在蚕豆酱发酵过程中,乳酸菌的数量明显小于蜡状芽孢杆菌,所以乳酸菌在蚕豆酱发酵过程中可能受到这些因素的抑制作用,难以生长。

图2 滤纸片法分析B. subtilisHYM05对乳酸菌W. paramesenteroidesHYM109的抑菌作用Fig.2 The inhibitory effect of HYM05 on HYM109
2.3 优势芽孢杆菌与酵母的相互作用分析
蚕豆酱发酵过程的中后期,酵母开始生长,但数量维持在103CFU/g的较低水平,表明酵母菌生长也受到一定抑制。蚕豆酱发酵过程中分离的酵母,均属于鲁氏酵母,而且根据M13-PCR指纹的基因型分析,都是同1株菌,即Zygosaccharomycesrouxii。研究中考察了上述3株菌对酵母的抑制作用,以1株从酱醅中分离得到的鲁氏酵母(Zygosaccharomycesrouxii4#)为例,滤纸片法和牛津杯法的实验结果如表3和表4所示。
由表3和表4可以看出,只有HYM42表现出了对酵母的抑制作用,抑制作用相当明显,抑菌圈直径达到了2.33 cm,如图3所示。

图3 HYM05对Zygosaccharomycesrouxii4#的抑菌作用Fig.3 The inhibitory effect of HYM05 on Zygosaccharomycesrouxii 4#
2.4 Touchdown PCR检测优势芽孢杆菌基因组的抗菌肽合成基因
芽孢杆菌能够分泌多种抗菌物质,包括核糖体途径合成的细菌素subtilin、ericin、subtilosin A等和非核糖体途径合成的低分子量抗菌肽表面活性素surfactin、伊枯草菌素(iturin)和溶杆菌素(bacilysin)等[16-18]。根据以上菌株的相互作用分析,结果表明B.subtilisHYM05,B.amyloliquefaciensHYM24和B.tequilensisHYM42这3株菌具有较强的抑菌作用,而且存在不同的抑菌表型,所以采用TouchdownPCR检测技术,分析这3株基因组中可能存在的抗菌肽合成基因。这种PCR检测方法可用于初步鉴定微生物合成细菌素或抗菌肽的能力[13]。实验结果如表5所示,结果表明HYM42以及HYM05的基因组中存在可以编码抗菌肽的基因,而HYM24的基因组中,由于某些基因的缺失,并没有发现能够编码对应细菌素/脂肽的基因,需要做进一步的研究。

表5 四种细菌素/脂肽PCR探测结果
3株实验菌的基因组中,均没有编码Subtilin的spaS基因。HYM42和HYM05的基因组中,存在编码Subtilosin的全部基因以及编码Plipastatin的pps基因,电泳图谱如图4所示。Subtilosin是一种环状细菌素,分子量较小,有特殊蛋白结构,它的抗菌谱很宽,对革兰氏阳性、阴性、好氧菌和厌氧菌都有很好的抑菌作用[19]。Plipastatin是一种脂肽类抗生素,有较为广谱的抑菌谱。3株菌中,编码Surfactin的3个基因均有缺失,HYM24缺失sfp基因,而HYM42和HYM05则缺失srf/lch基因。
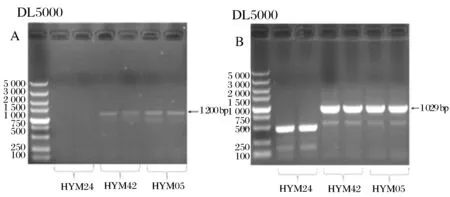
A:编码抗菌肽subtilosin基因的检测,其PCR扩增产物大小1200bp,B:编码plipastatin基因的PCR检测,大小1029bp图4 Subtilosin以及plipastatin的编码基因电泳图谱Fig.4 The electrophoretogram of genes encoding subtilosin and plipastatin
3 结论
蚕豆酱传统发酵过程中形成的微生物群落,是在特定生产环境下,由工艺条件控制,而自发形成的群落。这种群落结构能够在不同发酵批次之间保持相对稳定,比如本研究的蚕豆酱发酵过程,总是由好氧芽孢杆菌主导,乳酸菌和酵母含量少。这种群落结构稳定地自发形成,应与微生物之间的相互作用相关。本次研究结果表明,多个优势生长的好氧菌菌株能够协同抑制兼性厌氧的蜡状芽孢杆菌、乳酸菌以及酵母菌,表现出一定的生长优势。其中对蜡状芽孢杆菌的抑制作用最为显著,表明好氧芽孢杆菌的优势生长将其控制在较低水平,提高食品安全。好氧菌对乳酸菌和酵母菌的抑制作用相对较弱,但是后两者的数量在蚕豆酱发酵过程中保持在十分低的水平,表明它们的生长应该受到另一些因素的抑制,有待进一步研究。
发酵过程的优势好氧菌往往由不同菌种和多种不同基因型菌株组成,本次研究结果证实,这些菌株存在显著的表型差异,他们之间存在协同抑菌功能。其中分离出来3株菌具有不同的抑菌能力,通过PCR检测关键合成基因表明,其中两株菌即B.subtilisHYM05和B.tequilensisHYM42可能合成细菌素Subtilosin和Plipastatin;而B.amyloliquefaciensHYM24合成的抗菌肽则需要做进一步分析。总之,本次研究表明传统发酵过程微生物群落结果的形成,应与微生物相互作用相关,而且这种相互作用是各个微生物菌株个体之间协同作用的结果。
[1] 高岭.郫县豆瓣的生产工艺改进[J].中国调味品. 2006,31(5): 34-40.
[2] 孙常雁,马莺,李德海,等.自然发酵黄豆酱酱曲培养过程中蛋白酶的形成及蛋白质的分解[J].食品科技,2007,32(8):188-192.
[3] 包启安,周嘉华.中国传统工艺全集∥酿造[M].郑州:大象出版社,2007.
[4] 蒯辉.蚕豆酱发酵过程中微生物动态变化的研究[D].无锡:江南大学,2015.
[5]WUJR,ZHANGJC,SHIP,etal.BacterialcommunityinvolvedintraditionalfermentedsoybeanpastedajiangmadeinnortheastChina[J].AnnalsofMicrobiology,2013,63(4): 1 417-1 421.
[6]ZHAOJ,DAIX,LIUX,etal.ChangesinmicrobialcommunityduringChinesetraditionalsoybeanpastefermentation[J].InternationalJournalofFoodScience&Technology,2009,44(12):2 526-2 530.
[7]NAMYD,LEESY,LIMSIMicrobialcommunityanalysisofKoreansoybeanpastesbynext-generationsequencing[J].IntJFoodMicrobiol, 2012,155(1-2):36-42.
[8] 吕跃东,王勇,张文革,等.拮抗真菌菌株BOS-013对稻瘟病菌的抑制作用[J].安徽农业科学,2011,39(11):6 450-6 452.
[9] 刘冬梅,李理,杨晓泉,等.用牛津杯法测定益生菌的抑菌活力[J].食品研究与开发,2006,27(3):110-111.
[10]ARCURIEF,ELSHEIKHAAF,RYCHLIKT,etal.Determinationofcheeseoriginbyusing16SrDNAfingerprintingofbacteriacommunitiesbyPCR-DGGE:PreliminaryapplicationtotraditionalMinascheese[J].FoodControl,2013,30(1):1-6.
[11]WUNDERLICHOVAL,BUNKOVAL,KOUTNYM,etal.Noveltouchdown-PCRmethodforthedetectionofputrescineproducinggram-negativebacteriainfoodproducts[J].FoodMicrobiol,2013,34(2):268-276.
[12] 王天云,张贵星,薛乐勋.一种简便高效的改良降落PCR[J].中国生物工程杂志,2003,23(11):80-82.
[13] 王乃东,何浪,薛立群.TouchdownPCR扩增多样性小鼠Fab抗体基因[J].中国农学通报,2010,26(23):22-25.
[14]EOMJS,LEESY,CHOIHS.BacillussubtilisHJ18-4 from traditional fermented soybean food inhibitsBacilluscereusgrowth and toxin-related genes[J].J Food Sci,2014,79(11):2279-2287.
[15] KABORE D, NIELSEN D S, SAWADOGO-LINGANI H,et al.Inhibition ofBacilluscereusgrowth by bacteriocin producingBacillussubtilisisolated from fermented baobab seeds (maari) is substrate dependent[J].Int J Food Microbiol,2013,162(1):114-119.
[16] SHARMILA BANERJEE J. N. H. Structure and expression oaf gene encodingt he precursor of subtilin,a small protein antibiotic[J].The Journal of Biological Chemistry,1988,263(19):9 508-9 514.
[17] STEIN T, DUSTERHUS S, STROH A, et al.Subtilosin Production by TwoBacillussubtilisSubspecies and Variance of the sbo-alb Cluster[J].Applied and Environmental Microbiology,2004,70(4):2 349-2 353.
[18] SUTYAK K E, WIRAWAN R E, AROUTCHEVA A A, et al.Isolation of theBacillussubtilisantimicrobial peptide subtilosin from the dairy product-derivedBacillusamyloliquefaciens[J].J Appl Microbiol,2008,104(4):1 067-1 074.
[19] CHARLES E S, FLORENCE Y A, VISHNU D, et al.The spectrum of antimicrobial activity of the bacteriocin subtilosin A[J].Journal of Antimicrobial Chemotherapy (JAC),2007,59(2):297-300.
Interaction analysis of the strains isolated from traditional fermented broad bean Paste
FAN Zi-hao1,2, ZHU Lin-jiang1,2, LIU Si-yuan1,2, WANG Jia-qi1,2, LI Qi1,2*
1(The Key Laboratory of Industrial Biotechnology, Ministry of Education, Jiangnan University, Wuxi 214122, China) 2(School of Biotechnology, Jiangnan University, Wuxi 214122, China)
To analyze the mechanism of the microbial community stability during the formation of broad bean paste, filter paper method and oxford cup method were used. Touch-down PCR (TD PCR) was also performed to detect the genes encoding certain antibiotics including subtilosin, subtilin, surfactin and plipastatin. Results showed that all the anaerobic bacteria and yeast could be inhibited by one or more aerobic bacteria, such asBacillussubtilisHYM05,B.amyloliquefaciensHYM24, andB.tequilensisHYM42. TD PCR detection indicated that the genes encoding subtilosin and plipastatin existed in the genome of HYM42 and HYM05.
interaction; filter paper method; oxford cup method;Bacillussp.; antibiotics
10.13995/j.cnki.11-1802/ts.201704005
硕士研究生(李崎教授为通讯作者,E-mail:shmily1248505604@hotmail.com)。
国家自然科学基金(No. 31601445 &No. 31571942);国家高新技术研究发展计划(863计划,No.2013AA102106-03);江苏省第四期“333高层次人才培养工程”(BRA2012048)和江苏省111引智计划(No.111-2-06)
2016-10-06,改回日期:2016-11-20
