虾夷扇贝苗种培育水中可培养细菌群落分析
2017-06-19孙雪莹刘继晨李明赵学伟梁峻孙丕海马悦欣大连海洋大学农业部北方海水增养殖重点实验室辽宁大连60獐子岛集团股份有限公司辽宁大连600大连海洋大学海珍品苗种培育基地辽宁大连60
孙雪莹,刘继晨,李明,赵学伟,梁峻,孙丕海,马悦欣(.大连海洋大学农业部北方海水增养殖重点实验室,辽宁大连60;.獐子岛集团股份有限公司,辽宁大连600; .大连海洋大学海珍品苗种培育基地,辽宁大连60)
虾夷扇贝苗种培育水中可培养细菌群落分析
孙雪莹1,刘继晨1,李明2,赵学伟2,梁峻2,孙丕海3,马悦欣1
(1.大连海洋大学农业部北方海水增养殖重点实验室,辽宁大连116023;2.獐子岛集团股份有限公司,辽宁大连116001; 3.大连海洋大学海珍品苗种培育基地,辽宁大连116023)
为研究虾夷扇贝Patinopecten yessoensisis苗种培育过程中幼体发育5个时期苗种培育水中(受精卵期水样W1,担轮幼虫期水样W2,D型幼虫期水样W3,壳顶幼虫期水样W4,稚贝期水样W5)可培养细菌的群落组成,采用传统培养方法,从每个水样分离细菌30株,通过16S rRNA基因测序分析鉴定细菌。结果表明:分离的150株细菌可归为2门、3纲、6目、9科、10属、22种;在门水平,W1~W5的优势门均为变形菌门,其次为厚壁菌门;在属水平,假交替单胞菌属为所有水样第一优势属,此外,W1的优势属依次为交替单胞菌属、弧菌属和海杆菌属,W2的优势属依次为弧菌属、交替单胞菌属和Cobetia,W3的优势属依次为弧菌属、交替单胞菌属和芽孢杆菌属,W4的优势属依次为交替单胞菌属、芽孢杆菌属和弧菌属, W5的优势属依次为弧菌属、Pseudophaeobacter、芽孢杆菌属和赤杆菌属。研究表明,扇贝幼体不同发育时期苗种培育水中所含可培养细菌种类比较丰富,细菌群落结构存在差异。
虾夷扇贝;育苗培育水;16S rRNA基因;细菌群落
水产养殖过程中,尤其是在敏感的幼体饲养阶段,水质对健康动物的生长发育至关重要。水质指标包括物理、化学指标,以及所含细菌的数量和类型[1]。细菌性疾病是世界范围内水产养殖生产的最大限制因素之一,其中弧菌属细菌是最常报道的病原体[2-4]。
细菌的来源之一是苗种培育过程中使用的海水。Sandaa等[5]研究发现,大扇贝Pecten maximus苗种培育过程中,不同养殖系统(静态系统、循环水系统和经过臭氧处理的循环水系统)水中细菌群落结构高度相似。Godoy等[6]研究表明,使用抗生素处理紫扇贝Argopecten purpuratus幼体,其养殖水中可培养细菌的数量和优势属与未处理组不同,幼体存活率也不同。在虾夷扇贝Patinopecten yessoensisis苗种培育过程中,常发生苗种大量死亡的现象,已证明灿烂弧菌Vibrio splendidus和塔斯马尼亚弧菌Vibrio tasmaniensis是扇贝幼体死亡的致病菌[4],目前,尚不清楚苗种培育水中细菌是否与幼体疾病相关。本试验中,使用传统培养方法和16S rRNA基因序列分析了扇贝幼体不同发育时期苗种培育水中细菌群落组成,以期为苗种培育生态系统的调控与管理及病害防治提供参考。
1 材料与方法
1.1 材料
虾夷扇贝苗种培育在獐子岛海珍品原良种场进行,将经过沙滤和沉淀处理后的海水100 L加入塑料桶中,扇贝幼体养殖密度为5~6只/mL,每天7:00~8:00换水1次,每次换水50 L。饵料以叉鞭金藻Dicrateria inornata为主,孵化初期,每天投喂叉鞭金藻3次(8:00、15:00、23:00),每次100 mL[(7~9)伊105cells/mL],至受精后7 d,补充添加10 mL[(2~4)伊106cells/mL]新月菱形藻Nitzschia closterium,随后每天增加10 mL,至100 mL时不再增加,后续试验中每天投喂饵料3次,每次添加叉鞭金藻和新月菱形藻各100 mL。通过气石充气,水温保持在(15依1)益,盐度为(32依0.5)。
1.2 方法
1.2.1 样品的采集于2014年3—4月在虾夷扇贝幼体发育的5个时期,无菌采集苗种培育水样1 L(3个苗种培育桶各采集500 mL混合),受精卵、担轮幼虫、D型幼虫、壳顶幼虫和稚贝时期的样品分别标记为W1、W2、W3、W4和W5。
1.2.2 细菌的分离、计数和纯化将采集的水样进行梯度稀释,取0.1 mL稀释液涂布于2216E平板上,在15益下培养7 d,计数。在适当稀释度的平板上各随机挑取30个菌落,于2216E平板上分离纯化,获得纯培养菌株于冰箱中保存。
1.2.3 细菌DNA的提取、PCR扩增和序列测定将纯菌株接种到2216E液体培养基中培养过夜,使用土壤基因组DNA快速提取试剂盒[生工生物工程(上海)股份有限公司]提取细菌基因组DNA,提取方法参照试剂盒说明书。细菌16S rRNA基因片段的扩增引物为F27/R1492,由生工生物工程(上海)股份有限公司合成,引物序列参照文献[7]。PCR反应体系及反应程序参考文献[4]。用10 g/L琼脂糖凝胶电泳检测PCR产物是否符合目标条带。由生工生物工程(上海)股份有限公司完成PCR产物的测序。
1.3 数据处理
将获得的各菌株序列在EzBioCloud(http:// www.ezbiocloud.net/eztaxon)公共数据库中进行序列同源性比对,从结果中取第一个相似性最高的序列。将比对后相似性大于97%的菌株作为一个种[8],反之则为两个。为比较幼体不同发育时期苗种培育池水细菌群落差异,计算Shannon多样性指数[9]、Pielou均匀度指数[10]和S覬rensen相似性指数[11]。
2 结果与分析
2.1 扇贝苗种培育水中异养细菌计数
扇贝苗种培育水中异养细菌计数结果如表1所示,W1的细菌数量最少,W3的细菌数量最多。
2.2 扇贝苗种培育水中细菌群落组成分析
经16S rRNA基因序列比对,150株细菌归属于2门,即变形菌门Proteobacteria和厚壁菌门Fir-micutes;3纲,即酌变形菌纲酌-Proteobacteria、琢变形菌纲琢-Proteobacteria和芽孢杆菌纲Bacillus (图1);6目,即交替单胞菌目Alteromonadales、弧菌目Vibrionales、海洋螺菌目Oceanospirillales、芽孢杆菌目Bacillales、红杆菌目Rhodobacterales和鞘脂单胞菌目Sphingomonadales;9科,即假交替单胞菌科Pseudoalteromonadaceae、交替单胞菌科Alteromonadaceae、弧菌科Vibrionaceae、Marino-bacter_f、盐单胞菌科Halomonadaceae、芽孢杆菌科Bacillaceae、希瓦氏菌科Shewanellaceae、红杆菌科Rhodobacteraceae和赤杆菌科Erythrobacteraceae (图2);10属,即假交替单胞菌属Pseudoaltero-monas、交替单胞菌属Alteromonas、弧菌属Vibrio、海杆菌属Marinobacter、Cobetia、芽孢杆菌属Bacil-lus、希瓦氏菌属Shewanella、亚硫酸杆菌属Sulfito-bacter、Pseudophaeobacter和赤杆菌属Erythrobacter (图3)。

表1 扇贝苗种培育水中异养细菌数量Tab.1Heterotrophic bacterial counts in the water of scal-lop larva rearing tanks

图1 扇贝苗种培育水中可培养细菌的群落组成(纲水平)Fig.1Culturable bacterial community composition in the water of scallop larva rearing tanks(in class)
从图1可知,扇贝苗种培育水中酌-变形菌纲为优势纲,除W1外,其余4个时期水样还含有芽孢杆菌纲,只有W4和W5中含有琢-变形菌纲。
交替单胞菌目为W1~W5中第一优势目,占总数的比例依次为83.33%、73.33%、73.33%、80.00%和56.67%。其中,弧菌目为W1(13.33%)、W2(16.67%)、W3(20.00%)和W5(16.67%) 4个时期水样的第二优势目,芽孢杆菌目(10.00%)为W4的第二优势目,W5的优势菌目还有红杆菌目(13.33%)。
从图2可知:假交替单胞菌科是W1~W5中第一优势科;W1和W2的其余优势科依次为交替单胞菌科(23.33%和16.67%)和弧菌科(13.33%和16.67%),W3的其余优势科依次为弧菌科(20.00%)和交替单胞菌科(13.33%), W4的其余优势菌科依次为交替单胞菌科(13.33%)和芽孢杆菌科(10.00%),W5的其余优势菌科依次为弧菌科(16.67%)和红杆菌科(13.33%)。
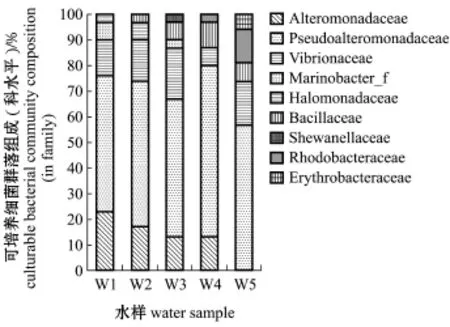
图2 扇贝苗种培育水中可培养细菌的群落组成(科水平)Fig.2Culturable bacterial community composition in the water of scallop larva rearing tanks(in fam ily)
从图3可知:假交替单胞菌属为W1~W5中第一优势属,所占比例依次为53.33%、56.67%、53.33%、66.67%和56.67%;W1的其余优势属依次为交替单胞菌属(23.33%)、弧菌属(13.33%)和海杆菌属(6.67%);W2的其余优势属依次为交替单胞菌属(16.67%)、弧菌属(16.67%)和Cobetia(6.67%);W3的其余优势属依次为弧菌属(20.00%)、交替单胞菌属(13.33%)和芽孢杆菌属(6.67%);W4的其余优势属依次为交替单胞菌属(13.33%)、芽孢杆菌属(10.00%)和弧菌属(6.67%);W5的其余优势属依次为弧菌属(16.67%)、Pseudophae-obacter(10.00%)、芽孢杆菌属(6.67%)和赤杆菌属(6.67%)。
综上所述,不同幼体发育时期苗种培育水中(W1~W5)异养细菌的群落组成存在差异,除各时期第一优势门(变形菌门)、纲(酌-变形菌纲)、目(交替单胞菌目)、科(假交替单胞菌科)和属(假交替单胞菌属)一致外,W4中第二优势目发生改变,W3和W5中第二优势科属与其他3个水样不同,W4和W5中第三优势科发生演替,W3、W4和W5中第三优势属发生改变且各不相同。
所有菌株与数据库中相关菌株的16S rRNA基因序列相似性为99%。按16S rRNA基因序列相似性大于97%的菌株归于同一种计[9],150株细菌可归为22个种。扇贝苗种培育水中种水平细菌群落组成见表2。
从Shannon指数和Pielou指数可以看出,幼体不同发育时期苗种培育水中细菌多样性稍有变化,细菌种类分布较为均匀(表3)。S覬rensen指数表明,幼体不同发育时期苗种培育水中细菌群落存在差异,其中W5与W1、W2、W3和W4,W4与W1细菌群落差异较为明显(表4)。

图3 扇贝苗种培育水中可培养细菌的群落组成(属水平)Fig.3Culturable bacterial community composition in the water of scallop larva rearing tanks(in ge-nus)
3 讨论
3.1 扇贝苗种培育水中异养细菌数量及群落变化
随着虾夷扇贝幼体的生长发育,其苗种培育水中异养细菌数量不断变化,受精卵期,水样中细菌数量为102CFU/mL,从担轮幼虫期到稚贝期水中细菌数量稳定在104CFU/mL。本试验中所得细菌数量与紫扇贝幼体养殖水中细菌数量[6]接近。大扇贝幼体发育早期阶段(4~11 d),静态养殖系统水中细菌群落略有波动,DGGE图谱中优势条带数目只有轻微变化[5]。tRFLP(限制性片段长度多态性)数据表明,太平洋牡蛎Crassostrea gigas苗种培育阶段,不同天清洁海水(经泡沫分离和过滤处理,加幼体或饵料之前)细菌群落显著不同[1]。本研究中,虾夷扇贝幼体不同发育时期苗种培育水中细菌群落组成存在差异,细菌群落演替的驱动力之一可能是进水细菌群落的变化[1,5],饵料(叉鞭金藻和新月菱形藻相关优势菌包括芽孢杆菌属、希瓦氏菌属、弧菌属和假交替单胞菌属等,未发表资料)和不同生长阶段幼体携带细菌的群落变化也可能影响水体细菌群落的演替[12]。

表2 扇贝苗种培育水中可培养细菌的群落组成(种水平)Tab.2Culturable bacterial community com position in the water of scallop larva rearing tanks(in species)

表3 扇贝苗种培育水中细菌群落的Shannon指数和Pielou指数Tab.3Shannon and Pielou indices of bacterial community in the water of scallop larva rearing tanks

表4 扇贝苗种培育水中细菌群落的S覬rensen指数Tab.4S覬rensen index of bacterial community in the wa-ter of scallop larva rearing tanks
3.2 扇贝苗种培育水中细菌群落组成
在纲水平,虾夷扇贝苗种培育水中优势纲为酌-变形菌纲,该结果与对大扇贝苗种培育系统水中相关细菌群落的研究结果一致[5],酌-变形菌纲也是紫扇贝幼体养殖水中的主要优势菌[6,13]。从虾夷扇贝苗种培育水中还可分离出属于芽孢杆菌纲和琢-变形菌纲的细菌,芽孢杆菌纲也存在于紫扇贝幼体养殖水中[6],琢-变形菌纲为太平洋牡蛎苗种培育用清洁海水中第一优势纲[1],也可在大扇贝苗种培育系统进水口水样中检测到[5]。在属水平,共分离到10个属。其中假交替单胞菌属、交替单胞菌属和弧菌属曾从包括虾夷扇贝在内的鱼和贝类养殖场周围海水中分离出[14]。大扇贝苗种培育系统水中可检测到假交替单胞菌属和弧菌属[5]。从紫扇贝幼体养殖水中可分离出交替单胞菌属、芽孢杆菌属和海杆菌属[6]。地中海牡蛎养殖海水中存在交替单胞菌属/假交替单胞菌属/希瓦氏菌属群[4]。在近岸海水中均可发现Cobetia、亚硫酸杆菌属、Pseudophaeobacter和Erythrobacter[15-18]。
已有研究表明,弧菌是扇贝幼体主要的致病菌[4,19-21]。在苗种培育过程中,虾夷扇贝幼体死亡现象发生在D形幼虫到壳顶幼虫过渡期和整个壳顶幼虫期。Liu等[4]通过人工感染试验证实,灿烂弧菌是幼体大量死亡的致病菌。本试验中,从D形幼虫期苗种培育水中分离出的灿烂弧菌可能是幼体病原菌的来源,因为同时进行的高通量测序数据分析表明,该菌的丰度在担轮幼虫和D型幼虫阶段明显高于受精卵、壳顶幼虫和稚贝3个阶段,而且幼体携带该菌的丰度是D型幼虫和壳顶幼虫阶段,高于其他3个阶段[22]。灿烂弧菌也是紫扇贝幼体大量死亡的病原菌[3]。Vibrio neocaledonicus在担轮幼虫、D型幼虫和壳顶幼虫期出现,可能存在潜在致病性,该菌曾从弧菌病暴发的菲律宾蛤仔Ruditapes philippinarum幼体中分离出[23]。除弧菌外,假交替单胞菌属的某些菌株也可能是潜在致病菌,如假交替单胞菌LT13菌株在攻毒试验中会导致10~16 d大扇贝幼体大量死亡[20]。另外,尤其要注意的是从水中分离出与人类病原菌炭疽杆菌Bacillus anthracis相似性为99%的菌株。因此,在扇贝苗种培育过程中应十分重视对包括细菌在内的水质监测。
本研究中,从扇贝苗种培育水中分离出的若干交替单胞菌、芽孢杆菌、弧菌、假交替单胞菌和Pseudophaeobacter arcticus,可能对扇贝幼体产生有益的影响。Douillet等[24]研究表明,交替单胞菌CA2菌株可提高太平洋牡蛎Crassostrea gigas幼体的存活率和生长率。麦氏交替单胞菌0444菌株可显著提高新西兰绿唇贻贝Perna canaliculus幼体的存活率,并增强其对病原菌弧菌DO1菌株的抵抗力[25],该菌株和Phaeobacter gallaeciensis可保护大扇贝幼体免受病原菌灿烂弧菌和Vibrio coralliilyticus的感染,保护欧洲牡蛎Ostrea edulis幼体免受V.coralliilyticus和Vibrio pectenicida的感染[26]。Phaeobacter sp.S4或短小芽孢杆菌Bacillus pumilus RI06-95可以保护美洲牡蛎Crassostrea virginica幼体免受病原菌Vibrio tubiashii RE22和Roseovarius crassostreae CV919-312T攻毒导致的死亡[27]。含弧菌C33菌株、假单胞菌11菌株和芽孢杆菌B2菌株的混合菌能够提高紫扇贝幼体存活率,使其在不使用抗生素的情况下顺利度过浮游幼体阶段[28]。弧菌OY15菌株可使美洲牡蛎幼体经病原菌弧菌B183菌株攻毒后48 h存活率提高[29],并且无论是否用B183菌株攻毒,该菌株也能显著提高美洲牡蛎在幼体变态过程中的存活率[30]。假交替单胞菌D41菌株可增强大扇贝幼体对灿烂弧菌感染的抵抗力,该菌株和P.gallaeciensis能保护太平洋牡蛎幼体免受V.coralliilyticus的感染[25]。本试验中分离的属于交替单胞菌属、芽孢杆菌属、弧菌属和假交替单胞菌属和P.arcticus的菌株是否可作为益生菌应用在虾夷扇贝苗种培育中有待进一步研究。
本研究表明,扇贝幼体不同发育时期苗种培育水中可培养细菌种类比较丰富,细菌群落结构存在差异。
[1]Powell SM,Chapman CC,BermudesM,etal.Dynamics of seawa-ter bacterial communities in a shellfish hatchery[J].Microbial E-cology,2013,66(2):245-256.
[2]Paillard C,Le Roux F,Borrego J J.Bacterial disease in marine bi-valves,a review of recent studies:trends and evolution[J].Aquatic Living Resources,2004,17(4):477-498.
[3]Rojas R,Miranda C D,Opazo R,et al.Characterization and patho- genicity of Vibrio splendidu s strains associated withmassivemortal-ities of commercial hatchery-reared larvae of scallop Argopecten purpuratus(Lamarck,1819)[J].Journal of Invertebrate Patholo-gy,2015,124:61-69.
[4]Liu Jichen,Sun Xueying,LiMing,et al.Vibrio infections associat-ed with Yesso scallop(Patinopecten yessoensis)larval culture[J]. Journal of Shellfish Research,2015,34(2):213-216.
[5]Sandaa R A,Magnesen T,Torkildsen L,et al.Characterisation of the bacterial community associated with early stages of great scallop (Pecten maximus),using denaturing gradient gel electrophoresis (DGGE)[J].Systematic and Applied Microbiology,2003,26 (2):302-311.
[6]Godoy F A,Espinoza M,Wittwer G,et al.Characterization of cul-turable bacteria in larval cultures of the Chilean scallop Argopecten purpuratus[J].Ciencias Marinas,2011,37(3):339-348.
[7]Lane D J.16S/23S rRNA sequencing[M]//Stackebrandt E, Goodfellow M D.Nucleic Acid Techniques in Bacterial Systematic. Chichester:John Wiley and Sons Press,1991:115-175.
[8]Stackebrandt E,Goebel B M.Taxonomic note:a place for DNADNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J].International Journal of Sys-tematic and Evolutionary Microbiology,1994,44(4):846-849.
[9]Shannon C E.A mathematical theory of communication[J].The Bell System Technical Journal,1948,27(3):379-423.
[10]Pielou EC.Themeasurementof diversity in different typesofbio-logical collections[J].Journal of Theoretical Biology,1966,13: 131-144.
[11]S覬rensen T.Amethod ofestablishing groups of equal amplitude in plant sociology based on similarity ofspeciesand itsapplication to analyses of the vegetation on Danish commons[J].Kongelige Danske Videnskabernes Selskab/Biologiske Skrifter,1948,5(4): 1-34.
[12]李明,刘继晨,孙雪莹,等.育苗阶段虾夷扇贝幼体中可培养细菌的多样性[J].大连海洋大学学报,2016,31(4):374-379.
[13]Jorquera M A,Lody M,Leyton Y,et al.Bacteria of subclass酌-Proteobacteria associated with commercial Argopecten purpuratus (Lamark,1819)hatcheries in Chile[J].Aquaculture,2004,236 (1-4):37-51.
[14]Schulze A D,Alabi A O,Tattersall-Sheldrake A R,etal.Bacteri-al diversity in amarine hatchery:balance between pathogenic and potentially probiotic bacterial strains[J].Aquaculture,2006,256 (1-4):50-73.
[15]Arahal D R,Castillo A M,Ludwig W,et al.Proposal of Cobetia marina gen.nov.,comb.nov.,within the family Halomonadace-ae,to include the species Halomonasmarina[J].Systematic and Applied Microbiology,2002,25(2):207-211.
[16]Yoon B J,Lee D H,Oh D C.Erythrobacter jejuensis sp.nov.,iso-lated from seawater[J].International Journal of Systematic and Evolutionary Microbiology,2013,63(4):1421-1426.
[17]Kwak M J,Lee J S,Lee K C,et al.Sulfitobacter geojensis sp. nov.,Sulfitobacter noctilucae sp.nov.,and Sulfitobacter noctiluci-cola sp.nov.,isolated from coastal seawater[J].InternationalJournal of Systematic and Evolutionary Microbiology,2014,64 (11):3760-3767.
[18]Bobrova O E,Kristoffersen JB,Ivanytsia V O.Metagenome 16S rRNA gene analysis of the Black seamicrobial diversity in the re-gion of the zmiiniy island[J].禺i剀剽剜匾i剜剌剜卦i伛i丌i剜劁刂冂剡剜剌剜卦i伛, 2015,(2):6-19.
[19]Nicolas JL,Corre S,Gauthier G,et al.Bacterial problems associ-ated with scallop Pecten maximus larval culture[J].Diseases of Aquatic Organisms,1996,27(1):67-76.
[20]Torkildsen L,LambertC,Nylund A,etal.Bacteria associated with early life stages of the great scallop,Pecten maximus:impact on larval survival[J].Aquaculture International,2005,13(6):575-592.
[21]Sandlund N,Torkildsen L,Magnesen T,et al.Immunohistochem-istry of great scallop Pecten maximus larvae experimentally chal-lenged with pathogenic bacteria[J].Diseases of Aquatic Organ-isms,2006,69(2-3):163-173.
[22]Sun Xueying,Liu Jichen,LiMing,etal.Characterization of bacte-rial communities associating with larval development of Yesso scallop(Patinopecten yessoensisis Jay,1857)by high-throughput sequencing[J].JOcean Univ China,2016,15(6):1067-1072.
[23]Dubert J,Osorio C R,Prado S,et al.Persistence of antibiotic re-sistant Vibrio spp.in shellfish hatchery environment[J].Microbial Ecology,2016,72(4):851-860.
[24]Douillet P,Langdon C J.Effects ofmarine bacteria on the culture of axenic oyster Crassostrea gigas(Thunberg)larvae[J].Biologi-cal Bulletin,1993,184(1):36-51.
[25]Kesarcodi-Watson A,Kaspar H,Lategan M J,et al.Alteromonas macleodii0444 and Neptunomonas sp.0536,two novel probiotics for hatchery-reared GreenshellTMmussel larvae,Perna canalicu-lus[J].Aquaculture,2012,309(1-4):49-55.
[26]Kesarcodi-Watson A,Miner P,Nicolas JL,et al.Protective effect of four potential probiotics against pathogen-challenge of the lar-vae of three bivalves:Pacific oyster(Crassostrea gigas),flat oys-ter(Ostrea edulis)and scallop(Pecten maximus)[J].Aquacul-ture,2012,344-349:29-34.
[27]Karim M,Zhao Wenjing,Rowley D,et al.Probiotic strains for shellfish aquaculture:protection of eastern oyster,Crassostrea vir-ginica,larvae and juveniles against bacterial challenge[J].Jour-nal of Shellfish Research,2013,32(2):401-408.
[28]Riquelme C E,Jorquera M A,Rojas A I,et al.Addition of inhibi-tor-producing bacteria to mass cultures of Argopecten purpuratus larvae(Lamarck,1819)[J].Aquaculture,2001,192(2-4): 111-119.
[29]Lim H J,Kapareiko D,Schott E J,et al.Isolation and evaluation of new,probiotic bacteria for use in shellfish hatcheries:I.isola-tion and screening for bioactivity[J].Journal of Shellfish Re-search,2011,30(3):609-615.
[30]Kapareiko D,Lim H J,Schott E J,et al.Isolation and evaluation of new probiotic bacteria for use in shellfish hatcheries:II.effects of a Vibrio sp.probiotic candidate upon survival of oyster larvae (Crassostrea virginica)in pilot-scale trials[J].Journal of Shell-fish Research,2011,30(3):617-625.
Characterization of culturable bacterial diversity in the water of Yesso scallop Patinopecten yessoensisis larva rearing tanks
SUN Xue-ying1,LIU Ji-chen1,LIMing2,ZHAO Xue-wei2,LIANG Jun2,SUN Pi-hai3,MA Yue-xin1
(1.Key Laboratory ofMariculture and Stock Enhancement in North China蒺s Sea,Ministry of Agriculture,Dalian Ocean University,Dalian 116023,Chi-na;2.Zhangzidao Island Group Company Limited,Dalian 116001,China;3.Seafood Seedling Breeding Base,Dalian Ocean University,Dalian 116023,China)
This study was aimed to examine the culturable bacterial community in the water of larva rearing tanks during Yesso scallop Patinopecten yessoensisis larval development stages(fertilized egg W1,trochophora W2,D-shaped larvaeW3,umbo larvaeW4,and juvenile scallop W5).Thirty strainswere isolated from each water sample by Zobell2216E,and 150 bacterial strainswere identified by polymerase chain reaction and partial16S rRNA gene sequencing.Results showed thatall isolateswere found to be in 2 phyla,3 classes,6 orders,9 families,10 genera and 22 species.At phylum level,Proteobacteria was themost dominant phylum in 5 water samples.At genus lev-el,the first dominantgenus in all sampleswas Pseudoalteromonas,followed by Alteromonas,Vibrio and Marinobact-er atW1 stage,Vibrio,Alteromonas and Cobetia atW2 stage,Vibrio,Alteromonas and Bacillus atW3 stage,Altero-monas,Bacillus and Vibrio atW4 stage,and Vibrio,Bacillus,Pseudophaeobacter and Erythrobacter at W5 stage. The findings show that there are abundant bacterial species and different community structure in the water of larva rearing tanks across different larval development stages.
Patinopecten yessoensisis;water in a larva rearing tank;16S rRNA gene;bacterial community
S917.1
A
10.16535/j.cnki.dlhyxb.2017.03.011
2095-1388(2017)03-0323-06
2016-06-02
国家科技支撑计划项目(2013BAD23B01);獐子岛集团资助项目(99801214)
孙雪莹(1989—),女,硕士研究生。E-mail:sunxueying1109@163.com
马悦欣(1963—),女,博士,教授。E-mail:mayuexin@dlou.edu.cn
