莆田黑猪不同组织DNA甲基化的MSAP分析
2017-06-15胡宇平张金耀林瑞意林威敏肖天放
胡宇平, 张金耀, 林瑞意, 林威敏, 陈 静, 汪 稳, 肖天放
(福建农林大学动物科学学院,福建 福州 350002)
莆田黑猪不同组织DNA甲基化的MSAP分析
胡宇平, 张金耀, 林瑞意, 林威敏, 陈 静, 汪 稳, 肖天放
(福建农林大学动物科学学院,福建 福州 350002)
为研究莆田黑猪不同组织间基因组DNA甲基化水平差异及各组织甲基化水平在不同生长发育阶段之间的变化规律,采用甲基化敏感扩增片段多态性(MSAP)方法检测了1、25和210日龄莆田黑猪的心脏、肝脏、肌肉、脂肪、耳和尾6个组织基因组DNA的甲基化水平.结果表明:心脏和肝脏组织的甲基化水平随日龄增长呈下降趋势,且日龄间的甲基化水平差异显著(P<0.05);25日龄肌肉组织的甲基化水平与1日龄的差异不显著(P>0.05),但显著高于210日龄(P<0.05);25日龄脂肪和耳组织的甲基化水平显著高于1和210日龄(P<0.05);3个日龄间尾组织的甲基化水平差异不显著(P>0.05);3个日龄的肝脏与脂肪组织之间、肝脏与耳组织之间的甲基化水平均存在显著差异(P<0.05),表明DNA甲基化在肝脏与脂肪组织之间、肝脏与耳组织之间存在组织特异性.
DNA甲基化; 组织; 甲基化敏感扩增多态性(MSAP); 莆田黑猪
DNA甲基化是目前研究较多的一种表观遗传修饰形式,是指在甲基转移酶的作用下,将一个甲基添加到胞嘧啶环的第5位碳原子上形成5-甲基胞嘧啶的过程[1].发生在CpG位点的甲基化修饰作为甲基化的一种主要模式,在胚胎发育、基因印记、X染色体失活、肿瘤和疾病的发生等过程中发挥着重要作用[2].DNA甲基化同样与生物体的组织发育和分化密切相关.目前,在哺乳动物[3]、植物[4]及低等海洋无脊椎动物[5]中都已发现组织间DNA甲基化差异片段.然而,DNA甲基化在发育过程中的调控作用在不同物种甚至不同组织中存在差异,如在人、鼠、牛上的研究显示,基因组DNA甲基化水平随年龄的增长呈下降趋势,并具有组织特异性[6];而班谦等[7]研究表明,在不同发育阶段的鸡胚中,DNA甲基化水平随鸡胚的发育呈上升趋势;将斑马鱼(Daniorerio)的DNA甲基转移酶基因(Dnmt1)敲除会导致肠等组织不能正常发育,但肝脏组织却不受影响[8];Warnecke et al[9]和Walsh et al[10]研究表明,在一些组织的特异性基因中,基因表达与甲基化水平之间并无关联.因此,分析不同组织之间及各组织不同发育阶段之间的甲基化水平对于理解DNA甲基化与组织生长发育之间的关系具有重要作用.检测DNA甲基化的方法主要有酶切法、亚硫酸盐测序法和甲基化特异性PCR等.相比较而言,甲基化敏感扩增片段多态性(methylation sensitive amplified polymorphism, MSAP)方法不但敏感性强,而且操作简单,适用于没有任何信息的全基因组水平的甲基化分析.MSAP方法使用一对同裂酶HpaⅡ和MspⅠ来检测基因组CCGG位点中DNA甲基化的多态性,它们均能识别CCGG位点,但对胞嘧啶的甲基化敏感性不同[11].HpaⅡ不能酶切双链都存在甲基化的位点, 而MspⅠ不能识别外侧甲基化胞嘧啶, 用它们处理基因组DNA就会产生甲基化敏感多态性片段.目前,关于猪不同组织间基因组DNA甲基化水平差异及各组织甲基化水平随日龄增长的变化规律鲜见报道.本试验以莆田黑猪为材料,采用MSAP方法检测其不同生长发育阶段、不同类型组织DNA的甲基化水平,探讨DNA甲基化在猪生长发育过程中的调控机制,旨在为猪基因组的甲基化研究积累基础性数据.
1 材料与方法
1.1 材料
选择莆田黑猪保种场同一批次遗传背景相同的莆田黑猪作为试验对象,采用相同营养水平的日粮和饲喂方式进行饲养.随机选取1、25和210日龄的莆田黑猪各6头进行屠宰,采集心脏、肝脏、肌肉、脂肪、耳和尾等组织样品,将样品置于冰箱(-20 ℃)中储存备用.
1.2 方法
1.2.1 基因组DNA的提取 用DNA提取试剂盒提取基因组DNA,采用1%琼脂糖凝胶电泳和NanoDrop 2000微量紫外可见分光光度计检验提取的DNA,将提取的DNA置于冰箱(-20 ℃)中储存备用.

表1 接头和引物Table 1 Adapters and primers
1.2.2 接头和引物的设计 接头和引物(表1)的设计参考Xu et al[12]所用的方法,略有改动.
1.2.3 MSAP方法的建立 (1)酶切反应:对每份基因组DNA分别用EcoRⅠ/HpaⅡ和EcoRⅠ/MspⅠ两种组合的酶切反应体系进行酶切.
(2)接头复性与连接反应:分别将HpaⅡ、MspⅠ接头与等量的EcoRⅠ接头混匀且短暂离心,置于PCR仪进行复性反应;然后把酶切产物与复性好的接头、反应物混合均匀,置于金属干浴锅(16 ℃)中过夜连接.
(3)预扩增反应:将连接产物的浓度稀释10倍,用PCR仪进行预扩增反应.预扩增反应体系含5 μL连接产物、1 μL E+1引物、7 μL 2×Taq MasterMix、1 μL HM+1引物,加ddH2O补齐至25 μL.PCR反应条件:94 ℃预变性5 min;94 ℃变性45 s,56 ℃退火1 min,72 ℃延伸45 s,32个循环;72 ℃延伸10 min.
(4)选择性扩增反应:将预扩增产物的浓度稀释15倍,用PCR仪进行选择性扩增反应.选择性扩增反应体系含2 μL连接产物、1 μL E+3引物、10 μL 2×Taq MasterMix、1 μL HM+3引物,加ddH2O补齐至20 μL.PCR反应条件:94 ℃预变性5 min;94 ℃变性45 s,65 ℃退火(每个循环递减0.7 ℃)45 s,72 ℃延伸1 min,13个递减循环;94 ℃变性45 s,56 ℃退火45 s,72 ℃延伸1 min,25个循环;72 ℃延伸7 min.
(5)选择性扩增产物的甲基化检测:对选择性扩增产物进行6%聚丙烯酰胺凝胶电泳及硝酸银染色,统计凝胶上各种模式的片段.
1.2.4 数据统计 采用MSAP方法得到的条带数据用SPSS(16.0)软件进行统计分析.
2 结果与分析
2.1 基因组DNA的甲基化模式
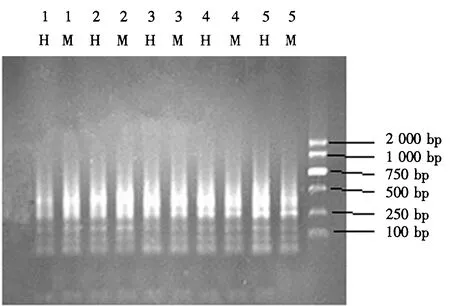
H:HpaⅡ/EcoRⅠ组合;M:MspⅠ/EcoRⅠ组合;1~5:5个样品.图1 基因组DNA选择性扩增产物的电泳检测Fig.1 Selective amplified product by electrophoresis
选用16对引物通过MSAP方法对莆田黑猪的基因组DNA甲基化状态进行检测,每个组织样本都获得了清晰的扩增谱带,每对引物组合扩增片段10条左右,100~500 bp处的扩增片段较多,大于1 000 bp的片段较少(图1).在MSAP电泳图谱上,每个组织基因组对应2个泳道,其中,H泳道的样品为使用HpaⅡ/EcoRⅠ处理的组织DNA样品,M泳道的样品为使用MspⅠ/EcoRⅠ处理的组织DNA样品.根据扩增产物在2个泳道出现的情况,在不同组织的MSAP电泳图谱上,每个组织基因组甲基化带型可分为3种模式:(1)半甲基化,即CCGG位点单链外侧的胞嘧啶甲基化(mCCGG),条带出现在H泳道而M泳道缺失,此类片段占扩增产物总数的20%左右;(2)全甲基化,即双链CCGG位点的内侧胞嘧啶甲基化(CmCGG),条带出现在M泳道而H泳道缺失,此类片段占扩增产物总数的30%左右;(3)非甲基化位点,即CCGG位点没有甲基化,条带同时出现在H和M泳道,这类片段最常见,占扩增产物总数的50%以上.
2.2 基因组DNA的甲基化检测结果
1、25和210日龄莆田黑猪心脏、肝脏、肌肉、脂肪、耳和尾6个组织甲基化状态的检测结果(表2)表明:1日龄共扩增12 324个条带,心脏组织的总甲基化水平最高,肝脏组织次之,耳组织最低;25日龄共扩增11 505个条带,脂肪组织的总甲基化水平最高,耳组织次之,肌肉组织最低;210日龄共扩增11 850个条带,尾组织的总甲基化水平最高,脂肪组织次之,肝脏组织最低.在3个日龄的甲基化水平中,各组织的全甲基化水平均要高于半甲基化水平,非甲基化水平均高于全甲基化水平.
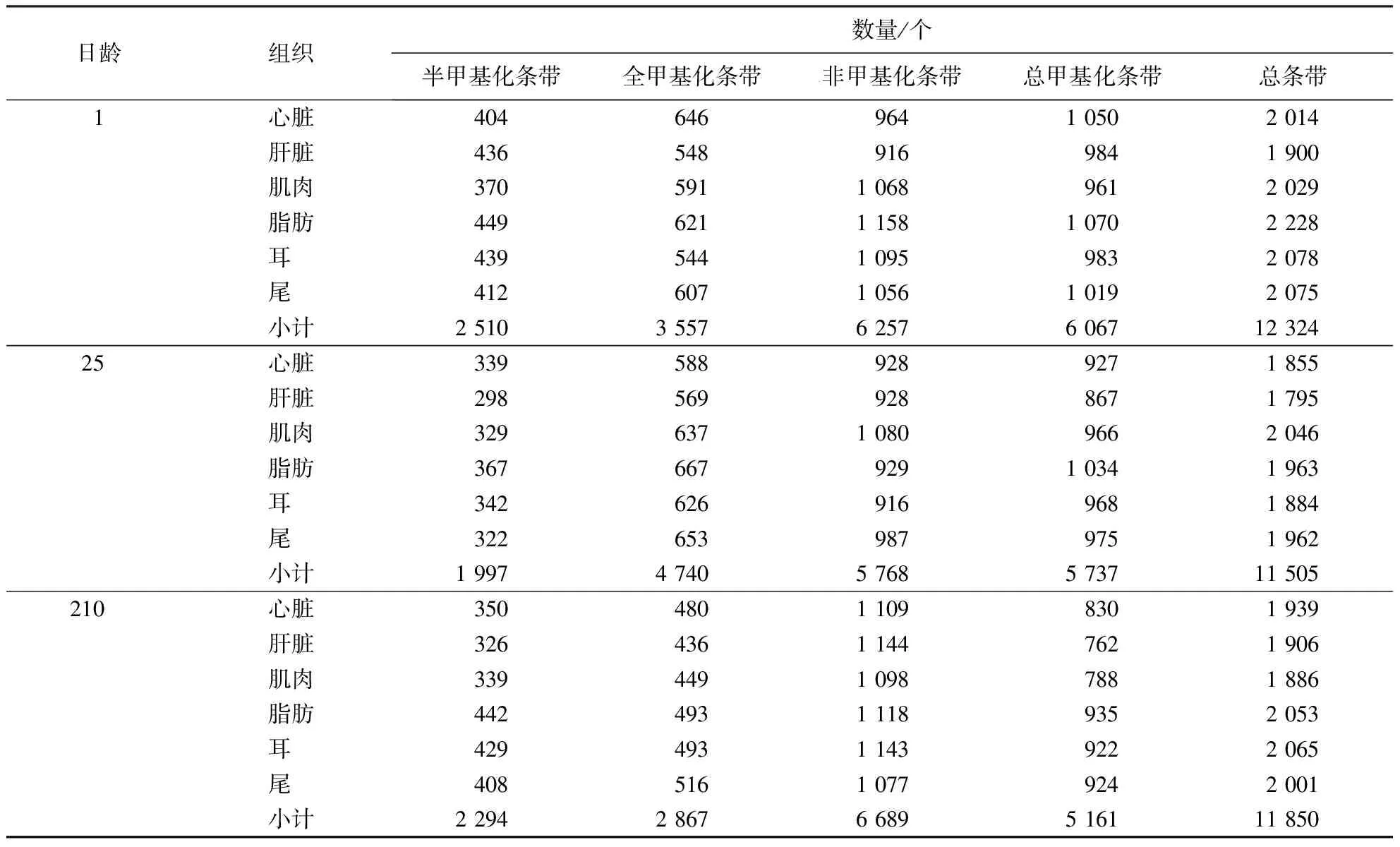
表2 莆田黑猪不同生长发育阶段、不同组织基因组DNA的甲基化检测结果Table 2 Methylation detection on various tissues of Putian black swine at different growth stages
2.3 不同生长发育阶段基因组DNA的甲基化水平
表3显示:莆田黑猪心脏和肝脏组织的总甲基化水平随日龄增长呈下降趋势,且不同日龄间的总甲基化水平差异显著(P<0.05);25日龄肌肉组织的总甲基化水平与1日龄的差异不显著(P>0.05),210日龄的总甲基化水平显著低于25日龄(P<0.05);25日龄脂肪和耳组织的总甲基化水平显著高于1和210日龄(P<0.05),210日龄脂肪组织的总甲基化水平显著低于1日龄(P<0.05),1日龄耳组织的总甲基化水平与210日龄间的差异不显著(P>0.05);尾组织的总甲基化水平总体呈先升后降的趋势,但各日龄间的差异不显著(P>0.05).在全甲基化水平上,各组织随日龄增长的变化规律与总甲基化水平的变化趋势基本一致.
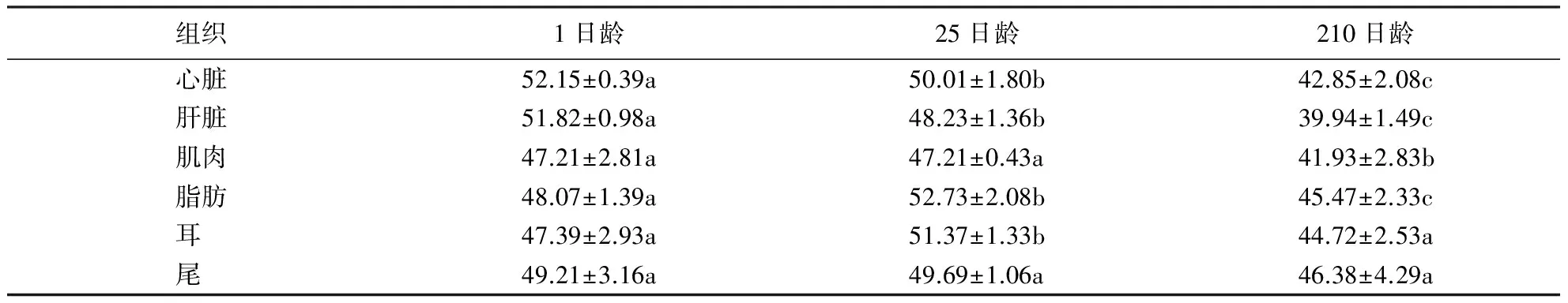
表3 莆田黑猪不同生长发育阶段基因组DNA的总甲基化水平1)Table 3 Methylation levels of various tissues of Putian black swine at different growth stages %
1)同行数值后附不同小写字母者表示差异显著(P<0.05),附相同字母者表示差异不显著(P>0.05).
2.4 不同组织基因组DNA的甲基化水平
表4显示:莆田黑猪肝脏组织的总甲基化水平在1日龄阶段均显著高于肌肉、脂肪和耳组织(P<0.05);在25日龄阶段显著低于脂肪、耳和尾组织(P<0.05);在210日龄阶段显著低于脂肪、耳和尾组织(P<0.05).脂肪组织的总甲基化水平在1日龄阶段显著低于心脏和肝脏组织(P<0.05);在25日龄阶段显著高于心脏、肝脏、肌肉和尾组织(P<0.05);在210日龄阶段显著高于肝脏和肌肉组织(P<0.05).耳组织的总甲基化水平在1日龄阶段显著低于心脏和肝脏组织(P<0.05);在25日龄阶段显著高于肝脏和肌肉组织(P<0.05);在210日龄阶段显著高于肝脏组织(P<0.05).可见,肝脏与脂肪组织之间、肝脏与耳组织之间的基因组DNA总甲基化水平在3个日龄均存在显著差异(P<0.05),表明DNA甲基化在肝脏与脂肪组织之间、肝脏与耳组织之间具有组织特异性.在全甲基化水平上,1日龄心脏组织的全甲基化水平显著高于肝脏、肌肉、脂肪、耳和尾组织(P<0.05),肝脏、肌肉、脂肪、耳和尾组织之间的的全甲基化水平差异不显著(P>0.05);25和210日龄所有组织之间的全甲基化水平差异均不显著(P>0.05).
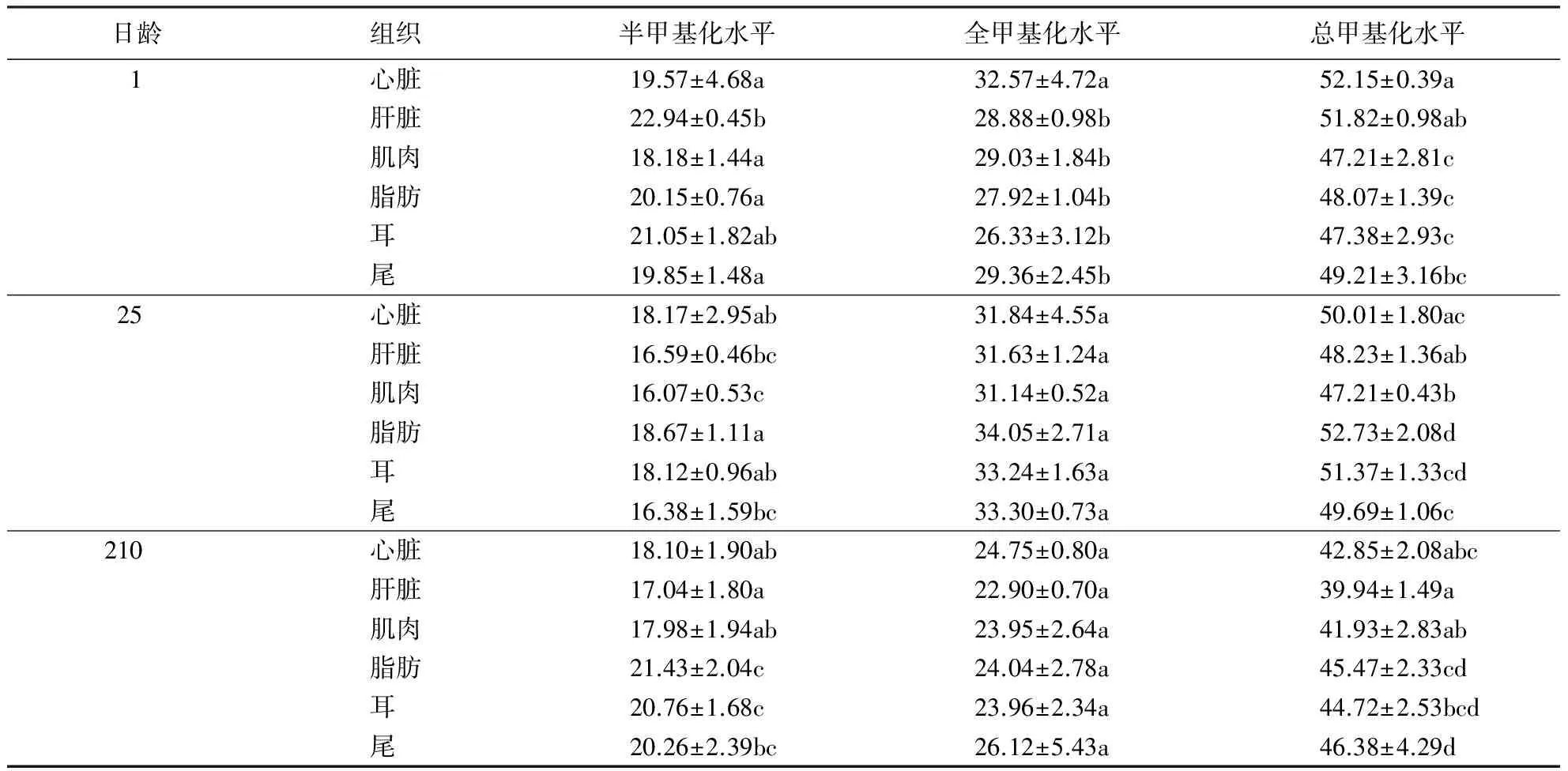
表4 莆田黑猪不同组织基因组DNA的甲基化水平1)Table 4 Methylation levels of tissue genomes of Putian black swine at different growth stages %
1)同列数值后附不同小写字母者表示差异显著(P<0.05),附相同字母者表示差异不显著(P>0.05).
3 讨论
莆田黑猪是福建省地方优质猪种,是国家级畜禽遗传资源保护品种之一,本试验探讨了其不同组织在3个生长发育阶段的甲基化水平及甲基化模式,为研究莆田黑猪表观遗传调控规律提供参考.本试验结果表明,莆田黑猪心脏和肝脏组织的总甲基化水平随日龄增长呈显著下降趋势(P<0.05),肌肉组织的总甲基化水平从25日龄开始显著下降(P<0.05),这与白小青等[6]的研究结果基本一致.白小青等[6]对荣昌猪不同生长发育阶段组织的基因组甲基化状态进行了研究,发现心脏、肝脏和半膜肌3个组织的基因组总甲基化水平随个体体重的增加呈下降趋势;而本试验结果显示,在莆田黑猪脂肪、耳和尾组织中,25日龄的甲基化水平要高于1日龄,表明不同组织的甲基化水平随体重增加的变化规律并不相同,不能一概而论.考虑到脂肪等组织的生长发育与猪的运动密切相关,推测这种差异可能与猪在这一时期的运动状态相关[13].
不同动物基因组中的不同甲基化类型的水平是不同的.杨春[14]对猪肌肉基因组DNA甲基化进行分析,发现大白猪、鲁莱猪和军牧一号猪的全甲基化水平要高于半甲基化水平.在一些植物也有类似的结论,如陈家慧等[15]发现,不同品种甘蔗的全甲基化水平也高于半甲基化水平.但蒋曹德[16]用MSAP方法检测了不同品种猪基因组的甲基化状态,发现梅山猪的全甲基化水平低于半甲基化水平.本试验结果表明,莆田黑猪各组织的全甲基化水平均要高于半甲基化水平.可见,品种不同、遗传背景不同和所选组织部位不同均可引起DNA甲基化的差异.
不同组织基因组中不同甲基化类型的水平也是不同的.徐青等[17]发现,在鸡的肌肉、肝脏、心脏和肾脏4个组织的基因组中,全甲基化水平差异显著,而半甲基化水平在所检测的各组织中差异均不显著.而本试验结果显示:1日龄莆田黑猪心脏的全甲基化水平显著高于肝脏、肌肉、脂肪、耳和尾组织(P<0.05),肝脏、肌肉、脂肪、耳和尾组织之间的全甲基化水平差异不显著(P>0.05);25和210日龄所有组织之间的全甲基化水平差异均不显著(P>0.05),而半甲基化水平在各组织间存在差异.这种不同的结果可能与选取样本的代表性或者采用的甲基化分析方法不同有关;另一方面也可能是不同的遗传背景,或不同的品种,或不同的组织和日龄,其甲基化模式和甲基化水平本身就存在差异[18].
DNA甲基化作为基因表达调控的一种方式,与组织特异基因表达模式密切相关.Ehrlich[19]研究了人体胸腺、脑和胎盘等7种组织和8种细胞的DNA甲基化水平,证实了DNA甲基化具有组织特异性和细胞类型特异性.本试验研究了1、25和210日龄莆田黑猪心脏、肝脏、肌肉、脂肪、耳和尾组织基因组DNA的甲基化水平,结果显示,在3个生长发育阶段,肝脏与脂肪组织之间、肝脏与耳组织之间的基因组DNA甲基化水平均存在显著差异(P<0.05),表明DNA甲基化在肝脏与脂肪组织之间、肝脏与耳组织之间具有组织特异性.
总之,莆田黑猪不同组织间的基因组DNA甲基化水平存在差异,且相同组织在不同生长发育阶段的甲基化水平也不尽相同,但不同组织基因组甲基化水平的差异是组织分化造成的结果,亦或是DNA甲基化对相关基因表达调控作用的结果目前还无法得知,有待于进一步研究.
[1] SINGAL R, GINDER G D. DNA methylation [J]. Blood, 1999,93(12):4 059-4 070.
[2] DAS P M, SINGAL R. DNA methylation and cancer [J]. J Clin Oncol, 2004,22(22):4 632-4 642.
[3] YANG C, ZHANG M, NIU W, et al. Analysis of DNA methylation in various swine tissues [J]. PloS One, 2011,6(1):e16229.
[4] PORTIS E, ACQUADRO A, COMINO C, et al. Analysis of DNA methylation during germination of pepper (CapsicumannuumL.) seeds using methylation-sensitive amplification polymorphism (MSAP) [J]. Plant Sci, 2004,166(1):169-178.
[5] 杜盈,何玉英,李健,等.野生和“黄海1号”中国明对虾不同组织基因组DNA的MSAP分析[J].中国水产科学,2013,20(3):536-543.
[6] 白小青,王金勇,陈英,等.荣昌猪基因组DNA甲基化水平分析[J].西北农林科技大学学报(自然科学版),2010,38(2):31-35,46.
[7] 班谦,赵宗胜,曹体婷.家鸡胚胎发育过程DNA甲基化的MSAP检测[J].安徽农业科学,2009,37(21):9 902-9 904.
[8] RAI K, NADAULD L D, CHIDESTER S, et al. Zebra fish Dnmt1 and Suv39h1 regulate organ-specific terminal differentiation during development [J]. Mol Cell Biol, 2006,26(19):7 077-7 085.
[9] WARNECKE P M, CLARK S J. DNA methylation profile of the mouse skeletal α-actin promoter during development and differentiation [J]. Mol Cell Biol, 1999,19(1):164-172.
[10] WALSH C P, BESTOR T H. Cytosine methylation and mammalian development [J]. Gene Dev, 1999,13(1):26-34.
[11] MCCLELLAND M, NELSON M, RASCHKE E. Effect of site-specific modification on restriction endonucleases and DNA modification methyltransferases [J]. Nucleic Acids Research, 1994,22:3 640-3 659.
[12] XU M L, LI X, KORBAN S S. AFLP-Based detection of DNA methylation [J]. Plant Molecular Biology Reporter, 2000,18(4):361-368.
[14] 杨春.猪不同组织和不同品种肌肉组织基因组DNA甲基化分析[D].长春:吉林大学,2011.
[15] 陈家慧,杨荣仲,周会.不同甘蔗品种DNA甲基化分析[J].农业研究与应用,2015(4):11-16.
[16] 蒋曹德.猪DNA甲基化与杂种表现的关系及遗传多样性分析[D].武汉:华中农业大学,2004.
[17] 徐青,张沅,孙东晓,等.应用MSAP方法检测鸡不同组织基因组的甲基化状态[J].遗传,2011,33(6):620-626.
[18] SHARMA R, MOHAN S R, MALIK G, et al. Rice cytosine DNA methyltransferases gene expression profiling during reproductive development and abiotic stress [J]. FEBS J, 2009,276(21):6 301-6 311.
[19] EHRLICH M. Expression of various genes is controlled by DNA methylation during mammalian development [J]. J Cell Bio-chem, 2003,88:899-910.
[20] SONG F, SMITH J F, KIMURA M T, et al. Association of tissue-specific differentially methylated regions (TDMs) with differential gene expression [J]. Proc Natl Acad Sci USA, 2005,102(9):3 336-3 341.
(责任编辑:施晓棠)
DNA methylation detection on different tissues of Putian black swine based on MSAP
HU Yuping, ZHANG Jinyao, LIN Ruiyi, LIN Weimin, CHEN Jing, WANG Wen, XIAO Tianfang
(College of Animal Science, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China)
Methylation sensitive amplification polymorphism (MSAP)method was used to detect methylation levels in various organs of Purain black swine at day 1, 25, and 100. The results showed that methylation levels of heart and liver tended to decrease with age, and varied significantly among different growth stages (P<0.05). Although methylation levels were not different between day 1 and 25 (P>0.05), methylation level at day 25 was significantly higher than that at day 210(P<0.05). Methylation levels of fat tissue and ear at day 25 were significantly higher than those at day 1 and 210 (P<0.05). Tail methylation level did not vary significantly among different growth stages (P>0.05). Furthermore, genomic DNA methylation levels varied significantly between liver and fat, and between liver and ear at 3 growth stages (P<0.05), indicating that tissue specificity of DNA methylation existed between liver and fat, and liver and ear tissues, respectively.
DNA methylation; tissue; methylation sensitive amplification polymorphism(MSAP); Putian black swine
2016-06-30
2016-10-31
科技部科技基础性工作专项(2014FY120800);福建省现代农业(生猪)产业技术体系建设项目(2014-357).
胡宇平(1990-),男,硕士研究生.研究方向:动物遗传育种.Email:2530727368@qq.com.通讯作者肖天放(1964-),男,教授.研究方向:动物遗传育种.Email:tfxiao@163.com.
S828.8+9
A
1671-5470(2017)03-0305-06
10.13323/j.cnki.j.fafu(nat.sci.).2017.03.012
