山西省植物病原镰孢菌种群分布及遗传变异分析
2017-06-15王琳李新凤徐玉梅畅引东王建明
王琳,李新凤,徐玉梅,畅引东,王建明
(山西农业大学农学院,山西太谷030801)
山西省植物病原镰孢菌种群分布及遗传变异分析
王琳,李新凤,徐玉梅,畅引东,王建明
(山西农业大学农学院,山西太谷030801)
【目的】通过对镰孢菌(Fusarium)ITS、EF-1α和β-tubulin 3个基因序列比较分析,筛选适合于镰孢菌种类鉴定的基因序列,并以此序列分析山西省植物病原镰孢菌种群分布及遗传变异情况。【方法】从2013—2015年在山西省11市28县(区)采集分离的625株镰孢菌菌株中选取形态学清晰的菌株进行ITS、EF-1α和β-tubulin基因序列联合分析,运用 Sequencher软件对序列进行拼接和校对,将测序结果与 NCBI及FUSARIUM-ID数据库中所有已公布的序列进行 BLAST分析,结合下载概念清晰或标准种序列,运用 ClustalX 和GelDoc软件进行序列对齐和编辑,运用MEGA、Excel、DNAstar和TaxonGap软件分析镰孢菌种内种间遗传变异情况,从ITS、EF-1α和β-tubulin中筛选适合镰孢菌种类鉴定的基因序列,并以此序列分析山西省镰孢菌的种群分布特点等。【结果】在3个候选基因序列中,EF-1α基因序列是最适用于镰孢菌种类鉴定的基因序列,其种间平均遗传距离分别是种内平均遗传距离的24倍,种内差异小于种间差异的种的数量最多,达到供试种的73%,物种鉴定准确率最强,达到87%。基于EF-1α基因片段的系统发育分析结果表明,在供试的 27种镰孢菌中,有 22种表现出单系性,相同种的不同菌株以较高支持率聚成独立支。在 27种镰孢菌中,F. oxysporum是山西省镰孢菌的优势种,分离频率最高(22.1%),且分布最广,在23个县(区)均有分布;其次是F. solani(13.8%),在14个县(区)有分布。从不同地区镰孢菌的种群结构及分布来看,运城、临汾、忻州、长治、吕梁、晋中和太原均以F. oxysporum为优势种,朔州以F. lateritium为优势种,大同以F. solani为优势种,晋城以F. verticillioides为优势种,阳泉以F. incarnatum为优势种;其中晋中和忻州的镰孢菌种类最丰富,临汾次之,朔州最少。从不同寄主镰孢菌的种群结构及分布来看,番茄上镰孢菌的种类最多(15种),其次是马铃薯(13种)和大豆(12种);番茄、黄瓜、西瓜、马铃薯、茄子、西葫芦和甘蓝,均以F. oxysporum为优势种,大豆和玉米以F. verticillioides为优势种,小麦以F. avenaceum和F. graminearum为优势种。【结论】镰孢菌种内种间存在丰富的遗传变异,其中 EF-1α基因序列的遗传变异最适用于镰孢菌种类的鉴定,但形态学种和系统发育学种不完全吻合。F. oxysporum是山西省镰孢菌的优势种,不同地区、不同寄主上镰孢菌种群存在明显的遗传分化;研究结果可为镰孢菌的分类鉴定、DNA条形码筛选、检验检疫及其综合防治提供理论依据。
镰孢菌;种群分布;种内种间差异;遗传变异;系统发育分析
0 引言
【研究意义】镰孢菌是一类重要的植物病原菌,它可侵染诱发多种植物的萎蔫、根腐以及腐烂病等,对人类生产生活影响极大。由于镰孢菌的多型性与易变异等特点,使得其被认为是所有真菌类群中在形态分类上最难鉴定的属之一[1]。而正确识别并鉴定其种类是镰孢菌各个研究领域的基础[2],因此,在形态学鉴定的基础上,选择准确的特异性分子鉴定方法,对镰孢菌的鉴定具有重要的理论和应用价值。【前人研究进展】近年来,传统形态学和分子鉴定相结合的方法已在镰孢菌分类鉴定中得到了广泛应用[3-4],但微乎其微的形态学差异以及单一序列信息的局限性,仍使镰孢菌属种的鉴定面临诸多困难。DIEPENINGEN 等[5]曾证实多基因位点联合分析能更准确对物种进行鉴定。所以采取多基因位点联合分析,是解决镰孢菌属种鉴定的有效途径,筛选适合的基因序列片段并明确其在鉴定中的特异性和适用性,是首先需要解决的一个重要的科学问题。目前,有关利用多基因位点联合分析对镰孢菌种进行鉴定的研究有些报道,曾莉莎等[6]评价了ITS、TEF-1α、IGS、histone H3、β-tubulin 等5个基因片段在香蕉枯萎病菌生理小种鉴定上的应用价值,发现TEF-1α、IGS和histone H3等3个基因片段可用于香蕉枯萎病菌生理小种鉴定;王容燕等[7]利用ITS、TEF-1α和β-tubulin 3个基因片段实现了对甘薯镰孢菌腐烂与溃疡病的病原菌鉴定。这些研究主要是用于少量镰孢菌种和生理小种的鉴定,利用多基因位点联合分析方法进行较大范围的镰孢菌种类鉴定和种群分布研究尚无系统详细的报道。【本研究切入点】山西省地形独特,气候特殊,作物种类繁杂,因此镰孢菌引起的病害数量和分布种类亦多。多年来,研究者对山西省多种植物病原镰孢菌做了大量的分类鉴定基础工作[8-12],但往往都只是采用形态学鉴定的方法,针对局部范围或局限在某一类寄主上,有关采用形态学结合分子鉴定方法分析山西省各地区的镰孢菌种群分布情况的研究尚未见报道。【拟解决的关键问题】选取ITS、EF-1α和β-tubulin 3个候选基因,从种内种间差异、遗传距离频率分布和构建系统发育树等方面,对镰孢菌的种内种间遗传变异及系统发育关系进行分析,评价3个候选基因在镰孢菌种类鉴定上的适用性,从中筛选适合镰孢菌种类鉴定的基因序列,对山西镰孢菌进行分子鉴定,并分析山西省镰孢菌种群分布特点和遗传分化情况,为今后开展镰孢菌的分类鉴定、筛选DNA条形码、检验检疫以及病害诊断提供科学依据。
1 材料与方法
1.1 供试菌株及序列信息
供试菌株共27种43株,由山西农业大学农学院植物病理重点实验室提供(所提供的菌株采集于2013 —2015年间,从山西省11个市共28个县(区)的罹病植物分离获得,分离部位包括果实、种子、茎部、根部、穗部、叶部等,并经过形态学鉴定),以Neonectria ramulariae为外群;供试序列共124条,其中自测序列39条,下载序列85条。供试菌株及序列详细信息见附表1。
1.2 模板DNA的制备
菌体获得:将活化后的镰孢菌单孢菌株接种于查彼培养液中[13],于25℃,150—200 r/min,恒温振荡培养5 d后,用真空抽滤泵抽滤菌丝,用无菌超纯水冲洗2遍,置于真空冷冻干燥机内冻干,回收备用。
镰孢菌基因组DNA制备:DNA提取采用CTAB 法[14],于凝胶成像仪(WD-9413A,ABI)内检测DNA质量,用紫外可见分光光度计(UV-2802S,ABI)检测DNA浓度,贮存于-20℃冰箱中备用。
1.3 PCR扩增及测序
PCR反应在BIO-RAD C1000 PCR仪上进行,所用引物信息见表 1,反应体系及条件参考赵鹏等[20],并稍加改动。PCR产物经 BigDye Terminator Ready Reaction Mix v3.1(Applied Biosystems,USA)纯化,使用ABI 3730型基因分析仪进行双向测序,由上海生工生物股份有限公司完成。
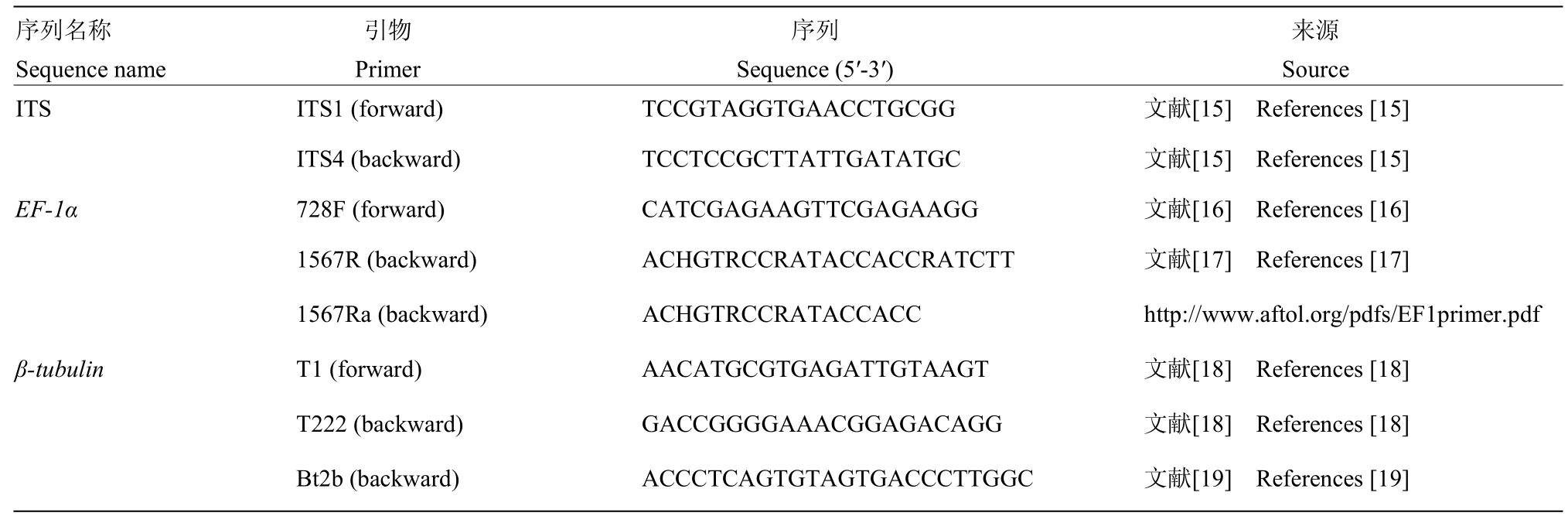
表1 PCR扩增和测序引物Table 1 Primers used for PCR amplification and sequencing
1.4 序列编辑及比对
用Sequencher 5.2.2(Gene Codes Corp.)对测序结果进行校对拼接,用ClustalX 1.81[21]进行序列比对,GelDoc[22]将比对结果进行编辑。登陆 NCBI数据库(http://blast.ncbi.nlm.nih.gov/blast.cgi)及 FUSARIUMID数据库(http://fusarium.cbio.psu.edu/),将 ITS、EF-1α和β-tubulin测序结果与数据库中所有已公布的序列进行BLAST分析。
1.5 序列数据分析
下载概念清晰或标准种的相关序列,结合本研究形态学清晰的5种13株菌株,基于ITS、EF-1α和β-tubulin 3个候选基因,共15种34株102条序列,以Neonectria ramulariae为外群,使用MEGA4.0.2[23]软件,分析序列特征,以 P-distancs模型计算种内种间遗传距离[24],结合Excel 2010分析种内种间遗传距离的频率分布情况[25],应用DNAstar 7.1.0[26](Lasergene,WI,USA)计算遗传相似性矩阵,将结果输入TaxonGap[27]分析种内种间序列差异情况,分析镰孢菌种内种间遗传变异情况;构建邻接树(NJ树,neighbor-joining tree),采用自展法进行1 000次检测分支支持率,选用K2P(Kimura’s two-parameter)模型,系统树支持率仅显示高于50%的数值,以检验种的单系性[28]。
1.6 种的鉴定方法
从形态学鉴定的种中选取一些代表性菌株,进行基于EF-1α基因序列的PCR扩增及测序,将测序结果分别在NCBI数据库和FUSARIUM-ID数据库中进行BLAST比对,并下载同源性较高并来源可靠的序列,构建邻接树。
1.7 山西省镰孢菌种群分布分析
根据菌株分离频率情况对山西省镰孢菌优势种进行分析;根据菌株类型和数量情况对不同地区和不同寄主镰孢菌种群结构及种群分布进行分析。
2 结果
2.1 ITS、EF-1α和β-tubulin 3个候选基因序列特征
序列经过拼接、编辑、对齐和整理后,ITS序列长度为405—541 bp,EF-1α序列长度为443—509 bp,β-tubulin序列长度为330—550 bp;碱基组成具有一定的 G+C含量偏向性;转换/颠换依次为β-tubulin>EF-1α>ITS;变异位点依次为β-tubulin >EF-1α>ITS;保守位点依次为 ITS>β-tubulin>EF-1α;简约信息位点依次为 EF-1α>β-tubulin>ITS(表2)。
2.2 基于遗传相似性的种内种间差异分析
基于EF-1α和β-tubulin基因序列分析的最小种间差异分别为3%和1%左右,均发生在F. graminearum 和F. culmorum之间,最大种间差异均发生在与外群之间;基于ITS序列分析的最小种间差异接近于0,发生在F. avenaceum和F. tricinctum之间,最大种间差异发生在F. lateritium和F. solani之间。ITS、EF-1α 和β-tubulin 3个候选基因,种内差异小于种间差异的种分别达到供试种的40%(6/15)、73%(11/15)和53%(8/15),而种内差异小于种间差异,有利于区分鉴定物种(图1)。故EF-1α基因序列是3个候选基因中最适合用作区分鉴定镰孢菌的基因序列。
2.3 基于遗传距离频率分布的种内种间差异
基于ITS、EF-1α和β-tubulin基因序列分析,遗传距离范围分别为0—0.17、0—0.76和0—0.52,种内平均遗传距离分别为0、0.01和0.01,种间平均遗传距离分别为0.09、0.24和0.18,种间平均遗传距离分别是种内平均遗传距离的9、24和18倍(图2)。故EF-1α基因序列是其中种间差异较种内差异最大的基因序列。
2.4 系统发育树的构建
由图3—图5可看出,分别基于EF-1α、β-tubulin 和ITS基因序列构建的NJ树均以较高支持率被划分为4个Clade(Clade I-IV)。基于EF-1α和β-tubulin序列分别构建的 NJ树的拓扑结构基本一致,与基于ITS序列构建的NJ树存在差异,且在个别种的区分上三者均存在一定差异。在图3中,有13个种表现出单系性,F. verticillioides和F. tricinctum虽未表现出单系性,但也与其他种明显区分开,物种鉴定率达到87% (13/15);在图4中,有11个种表现出单系性,F. tricinctum、F. subglutinans、F. incarnatum 和 F. lateritium 未表现出单系性,物种鉴定率为 73% (11/15);在图5中,有10个种表现出单系性,F. verticillioides、F. tricinctum、F. incarnatum、F. graminearum和F. avenaceum未表现出单系性,物种鉴定率为67%(10/15)。综上所述,EF-1α基因序列是3个候选基因中区分鉴定镰孢菌准确率最高的基因序列。
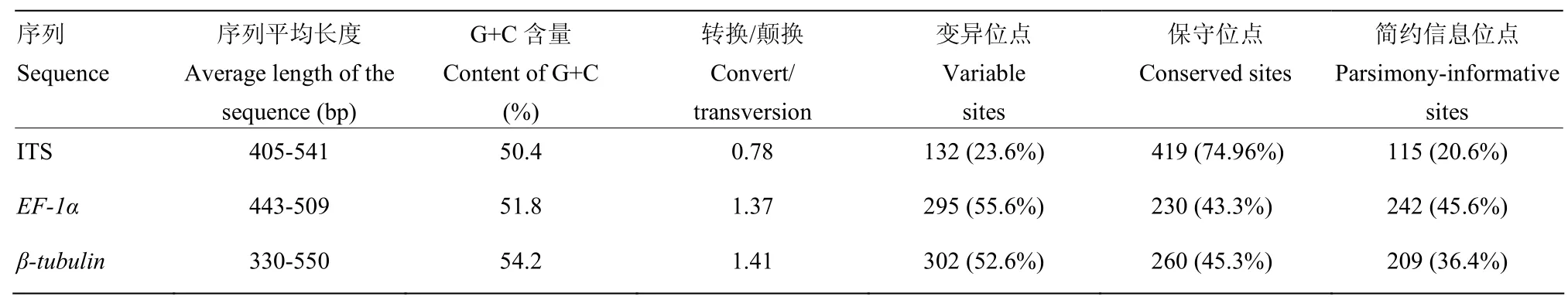
表2 序列碱基组成及变异率Table 2 Base composition and variation rate of sequences

图1 各基因片段的种内种间差异比较(使用TaxonGap和DNAstar软件)Fig. 1 Comparisons of intra- and inter-specific variations of the gene sequence generated by the softwares TaxonGap and DNAstar
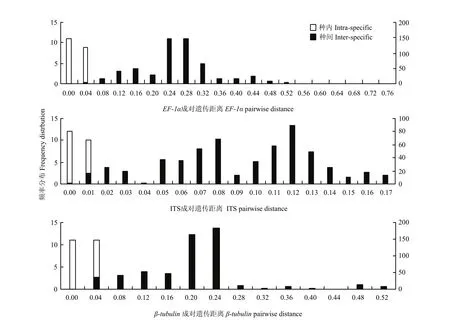
图2 各基因序列种内种间遗传距离的频率分布Fig.2 Frequency distribution of intra- and inter-specific pairwise distances of the gene sequence generated with MEGA and Excel
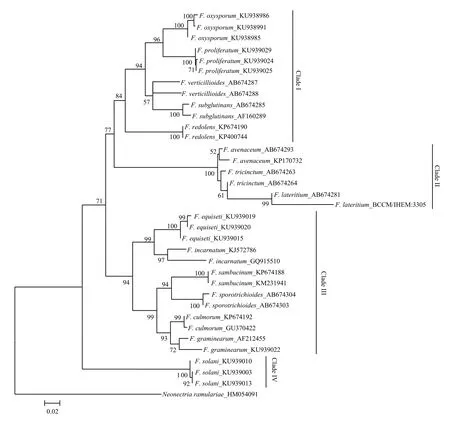
图3 构建基于EF-1α序列的邻接系统发育树Fig. 3 Neighbor-joining (NJ) phylogenetic tree inferred from EF-1α sequences
2.5 山西省镰孢菌种的鉴定
在GenBank和FUSARIUM-ID数据库中的比对结果如表3所示,40株菌株的测序结果与数据库中相关种的 EF-1α基因序列同源性均达 92%—98%,F. aquaeductuum、F. sambucinum var. coeruleum和 F. trichothecioides测序失败,未能进行比对。将获得的40条序列结合下载的24条同源性序列,以Neonectria ramulariae为外群,共65条序列进行比对分析,结果表明,EF-1α序列长度为423—666 bp,碱基组成具有一定的 G+C含量偏向性(52.1%);转换/颠换值为1.03;变异位点为 551(72.5%);保守位点为 193(25.4%);简约信息位点为460(60.5%);自裔位点为90(11.8%)。构建的系统发育树如图6所示,相同种的不同菌株均能聚在一起,除了 F. tricinctum 和F. acuminatum未聚成独立分支外,其余22种的不同菌株均以较高支持率分别聚成独立分支,表现出单系性。结果说明,系统发育学种的鉴定结果与形态学鉴定结果存在差异。
2.6 山西省镰孢菌种群分布分析
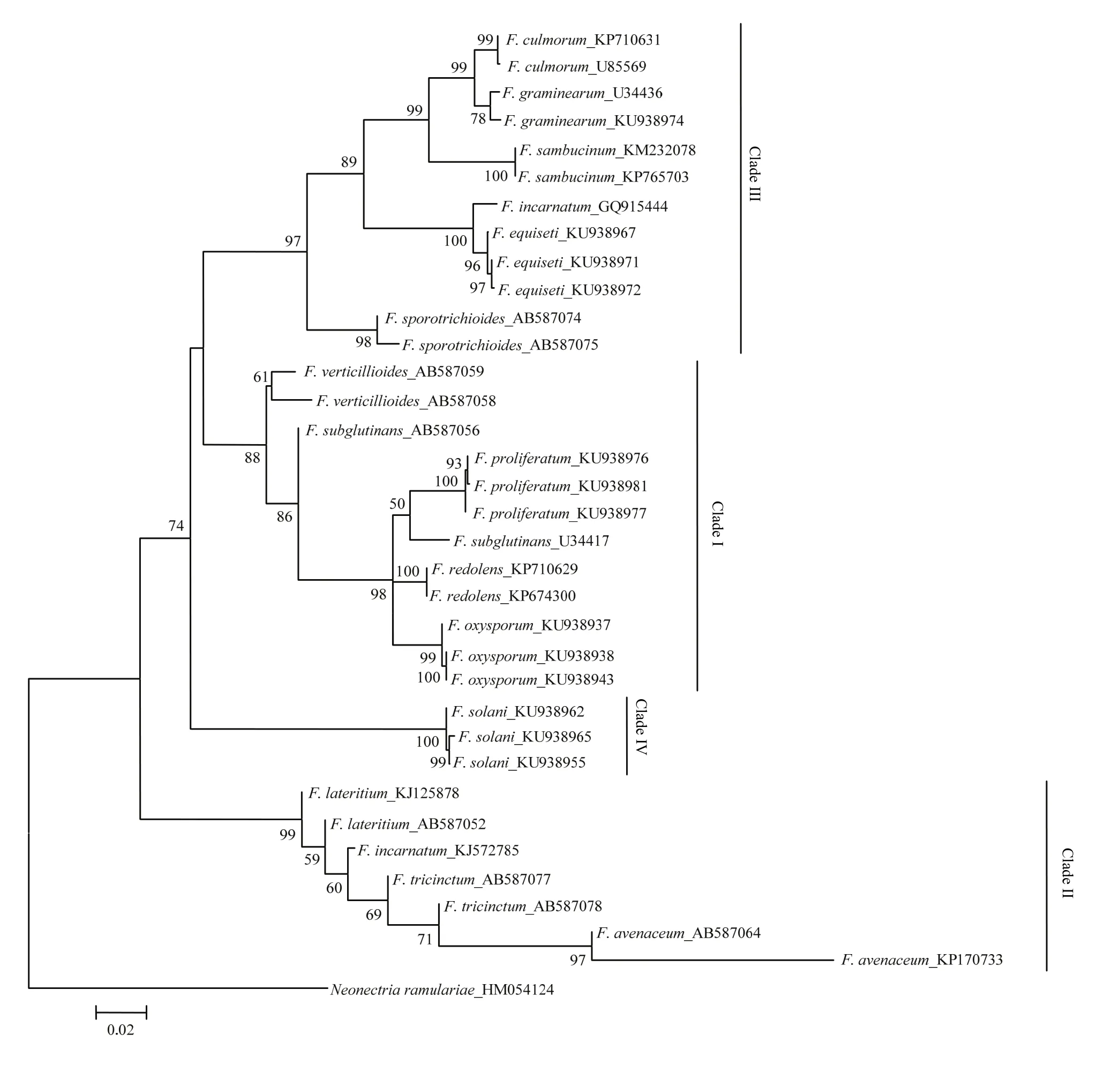
图4 构建基于β-tubulin序列的邻接系统发育树Fig. 4 Neighbor-joining (NJ) phylogenetic tree inferred from β-tubulin sequences
2.6.1 山西省镰孢菌分离频率及地理分布 分离频率>8%的有4种,其中最高的为F. oxysporum(22.1%);其他的依次为F. solani(13.8%)、F. verticillioides(10.1%)、F. incarnatum(8.6%);分离频率>2%的有8种,依次为F. equiseti(6.7%)、F. coeruleum (6.1%)、F. redolens(4.6%)、F. acuminatum(3.4%)、F. lateritium(3.1%)、F. proliferum(2.2%)、F. subglutinans(2.2%)和F. avenaceum(2.1%);分离频率<2%的有 15种,其中 F. heterosporum和 F. trichothecioides均为 1.8%,F. sporotrichioides、F. camptoceras、F. ventricosum和F. chlamydosporum均为1.3%,F. udum、F. tricinctum、F. culmorum和F. graminearum均为1%,F. illudens、F. sambucinum和F. aquaeductuum 均为 0.6%,F. sambucinum var. coeruleum 和 F. merismoides均为 0.3%。故 F. oxysporum为山西省镰孢菌的优势种(图7)。
以F. oxysporum分布范围最广泛,在23个县(区)均有分布;其次是F. solani和F. incarnatum,分别在14和 13个县(区)均有分布;F. sambucinum var. coeruleum和F. graminearum分布范围最小,仅在五寨分离到(图8)。

图5 构建基于ITS序列的邻接系统发育树Fig. 5 Neighbor-joining (NJ) phylogenetic tree inferred from ITS sequences
2.6.2 不同地区镰孢菌种群结构及分布 运城(33.3%)、临汾(16.1%)、忻州(18.4%)、长治(27.3%)、吕梁(15.8%)、晋中(23.1%)和太原(46.2%)均以F. oxysporum为主;朔州以F. lateritium为主(60%);大同以F. solani为主(33.3%);晋城以F. verticillioides为主(27.3%);阳泉以F. incarnatum为主(50%)(图 9)。晋中市和忻州市分离到镰孢菌种类最多(22种),其他依次为临汾(19种)、吕梁(12种)、长治(11种)、运城和晋城(8种)、大同(7种)、阳泉和太原(3种)和朔州(2种)(图10)。因此,晋中市和忻州市的镰孢菌种类最丰富,临汾市次之,朔州市最少。
2.6.3 不同寄主中镰孢菌种群结构及分布 不同寄主中镰孢菌种群结构及分布分析如表4所示。由表4可看出,在番茄(果实)、黄瓜(茎基、根部)和玉米(茎部、穗部、种子)等10个寄主上均能分离到不同种类的镰孢菌,分离种和菌株数量分别占到全部种和菌株数量的 92.6%和 71.5%。番茄上分离到的镰孢菌种类最多(15种);其次是马铃薯(13种)、大豆(12种)、黄瓜(6种)和玉米(8种),其余寄主也分离到3—4种。番茄、黄瓜、西瓜、马铃薯、茄子、西葫芦以及甘蓝,均以F. oxysporum为优势种;大豆和玉米以 F. verticillioides为优势种,小麦以 F. avenaceum 和 F. graminearum 为优势种;F. aquaeductuum和F. sambucinum var. coeruleum在上述几种寄主中未分离到。
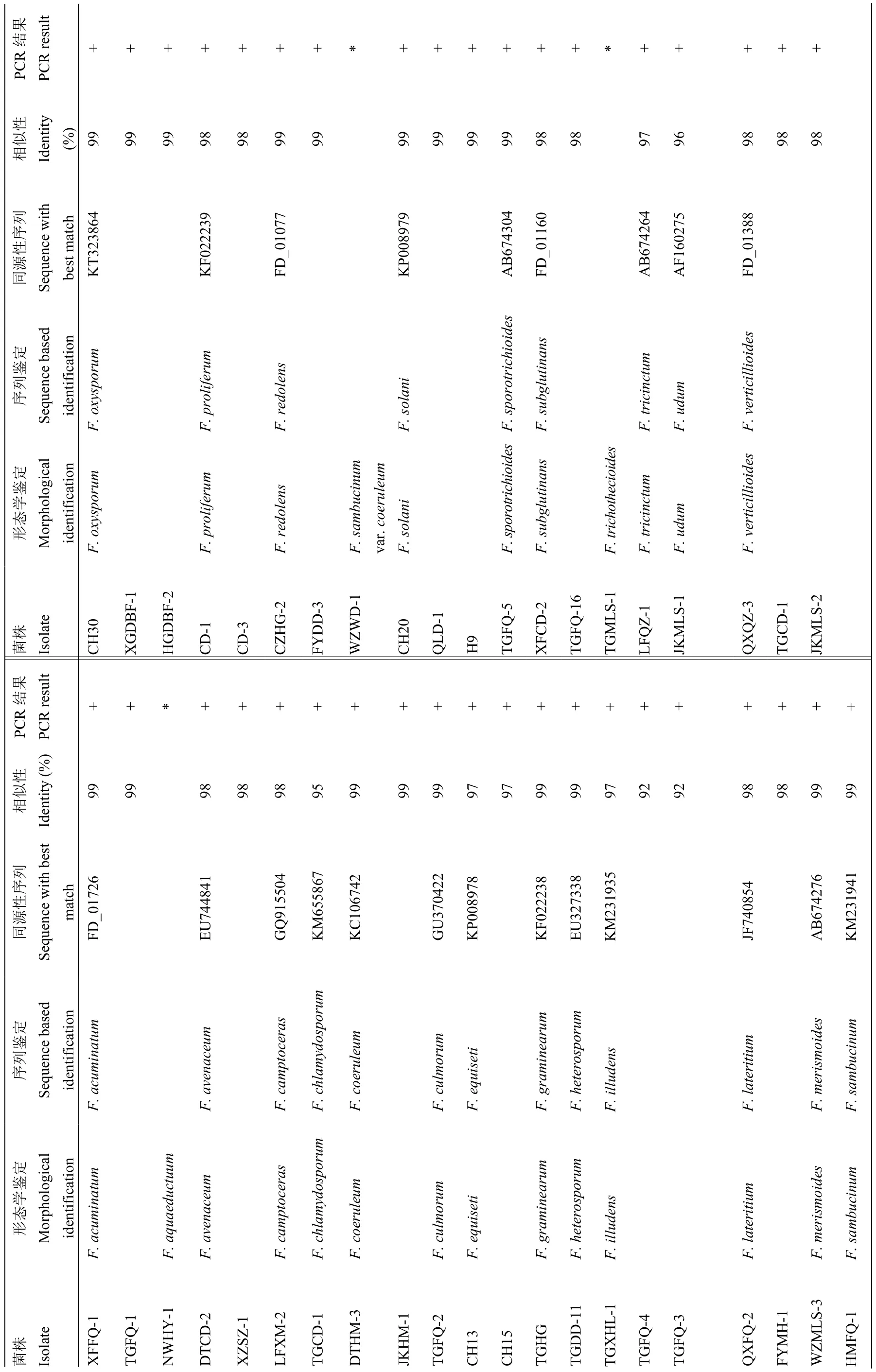
表3 基于EF-1α 基因序列的PCR 和分子鉴定结果Table 3 PCR result and molecular identification based on EF-1α sequences
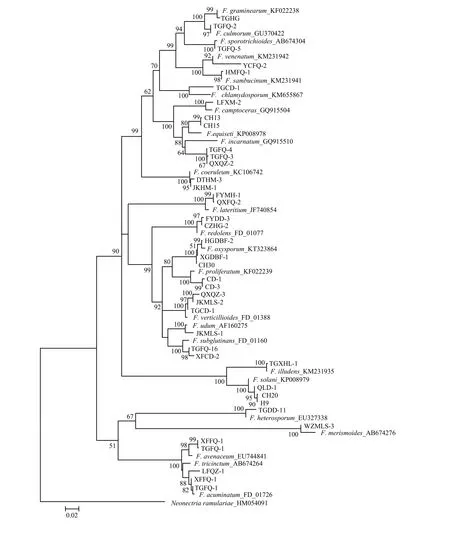
图6 基于EF-1α序列构建的山西省镰孢菌邻接系统发育树Fig. 6 Neighbor-joining (NJ) phylogenetic tree of Fusarium in Shanxi Province inferred from EF-1α sequences
3 讨论
镰孢菌种内种间遗传变异情况研究显示,ITS基因序列的鉴定能力最差,且反映出的亲缘关系与形态学分类存在差异,这可能与ITS基因序列进化相对保守有关。有学者证实EF-1α序列较ITS序列更能正确区分镰孢菌[29-30],WATANABE[31]也曾评价 β-tubulin基因序列是识别镰孢菌的有效标记,这些结果与本研究的结果一致。
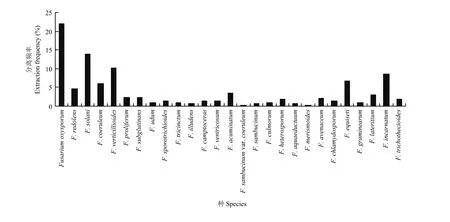
图7 山西省镰孢菌菌株分离频率Fig. 7 The Fusarium strains extraction frequency in Shanxi Province
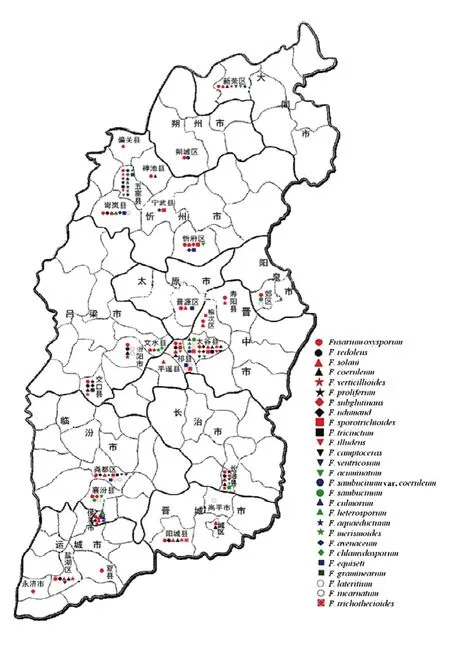
图8 山西省28个县(区)镰孢菌种群的地理分布Fig. 8 Geological distributions of Fusarium populations in 28 counties (areas) of Shanxi Province
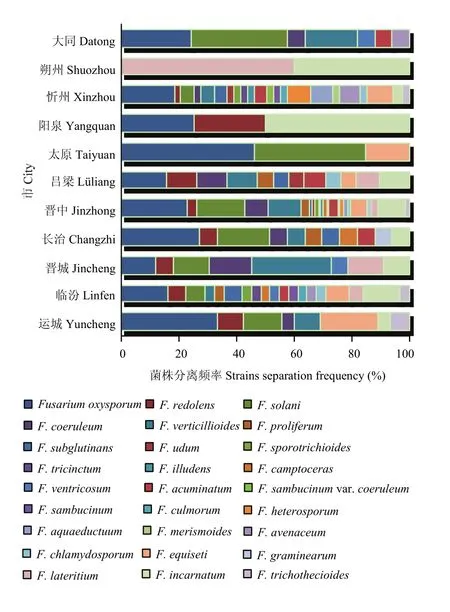
图9 山西省11个市镰孢菌菌株分离频率Fig. 9 The Fusarium strains extraction frequency in 11 cities of Shanxi Province
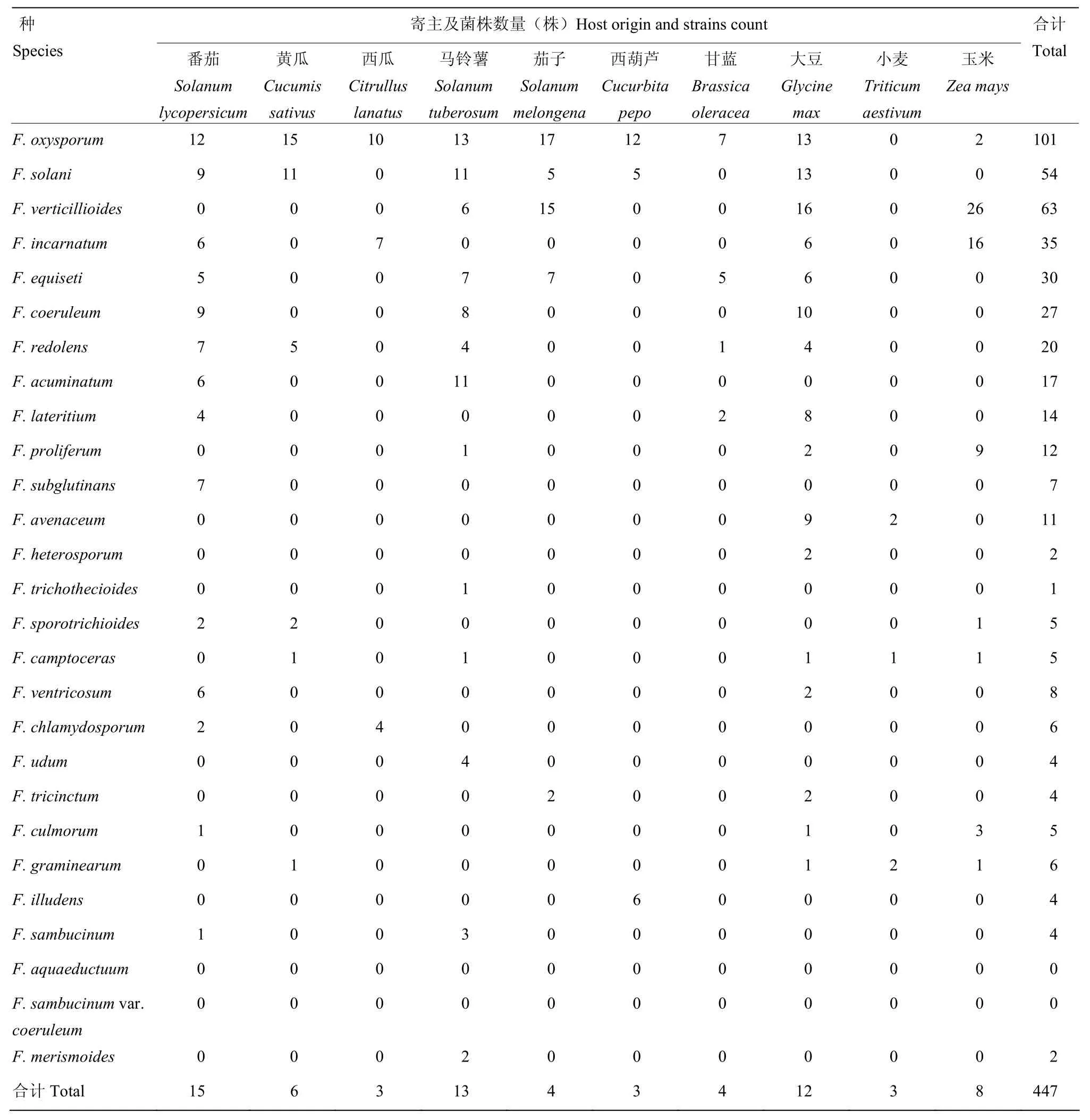
表4 山西省镰孢菌种在不同寄主中的分布Table 4 Distribution of Fusarium species in different hosts in Shanxi Province
基于EF-1α基因序列的系统发育分析表明,形态学上差异较大的 F. tricinctum和 F. acuminatum虽然未表现出单系性,但在系统树中表现较近的亲缘关系。说明形态学上的种和系统发育学种还是存在一定差异的,因此,在镰孢菌鉴定时,在形态学鉴定基础上,应结合分子鉴定才能作出更加准确的判断。
山西省不同地区、不同寄主的镰孢菌种群结构及优势种存在差异,说明山西省镰孢菌种群存在明显的遗传分化,可能与不同地区的耕作制度和气候环境有关。试验结果表明,山西马铃薯以F. oxysporum为优势种,这与黑龙江[32]和新疆[33]的马铃薯干腐病镰孢菌种群调查结果相似,但在优势种上存在差异,这可能与地域和气候差异有关;大豆上以F. verticillioides为优势种,其次为 F. oxysporum、F. solani和 F. coeruleum,魏巍等[34]也曾报道过上述4种镰孢菌为大豆根际土壤镰孢菌优势种群,与本研究结果一致;黄瓜上分获了 6种镰孢菌,以 F. oxysporum和 F. solani为优势种群,与东北三省[35-36]等地的调查结果一致。
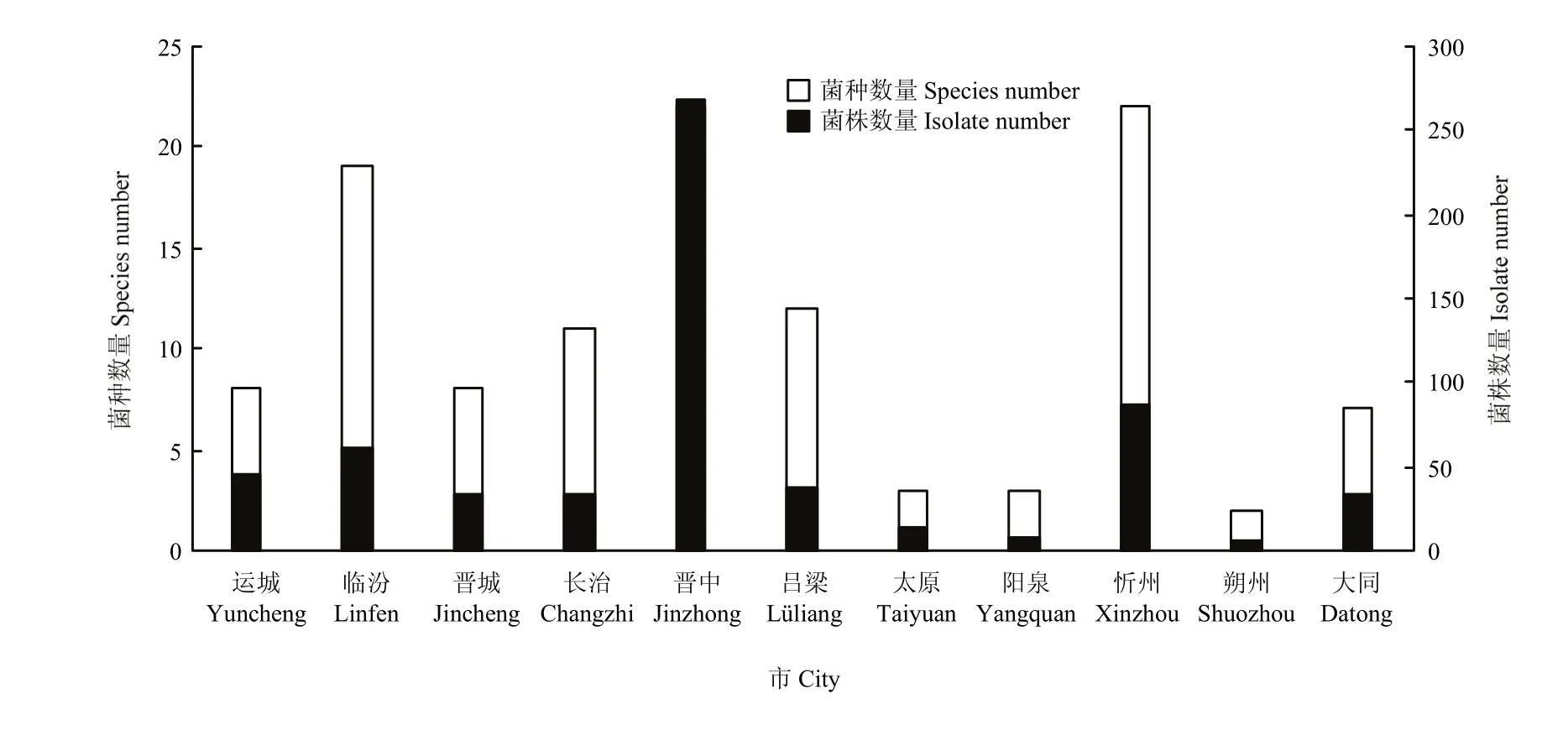
图10 山西省11个市镰孢菌种类及菌株数量Fig. 10 The species and amounts of Fusarium strains in 11 cities of Shanxi Province
本研究仅从不同地区和不同寄主上的镰孢菌种群结构及分布进行了研究,而且采集的地理范围和寄主范围有限,因此在今后的研究中,需要进一步扩大采样范围,以使其结果更加全面准确,同时应对镰孢菌年度间的种群结构变化加以分析,以便更加准确地了解其变化动态。
4 结论
从基于ITS、EF-1α和β-tubulin 3个基因的序列分析可看出,镰孢菌种内种间存在丰富的遗传变异,其中EF-1α基因序列的遗传变异最适用于镰孢菌种类的鉴定。基于EF-1α基因序列的系统发育分析显示,分子鉴定的结果与形态学鉴定的结果存在差异,所以在镰孢菌种的鉴定中,采用形态学结合EF-1α基因序列分子鉴定的方法,更能提高鉴定结果的准确性。F. oxysporum是山西省镰孢菌的优势种,不同地区、不同寄主上镰孢菌种群存在明显的遗传分化。
[1] 张向民. 镰刀菌属分类学研究历史与现状. 菌物研究, 2005, 3(2): 59-62. ZHANG X M. History and current research on taxonomy of the genus Fusarium. Journal of Fungal Research, 2005, 3(2): 59-62. (in Chinese)
[2] 俞大绂. 镰孢菌分类学的意义. 微生物学报, 1977, 17(2): 163-171. YU D F. The taxonomic significance of Fusarium. Acta Microbiologica Sinica, 1977, 17(2): 163-171. (in Chinese)
[3] 吴会杰, 范琳娟, 古勤生. 西瓜枯萎病菌的鉴定及申嗪霉素对其防效评价. 中国农学通报, 2016, 32(28): 131-135. WU H J, FAN L J, GU Q S. Identification of Fusarium wilt of watermelon and efficacy evaluation of shenqinmycin. Chinese Agricultural Science Bulletin, 2016, 32(28): 131-135. (in Chinese)
[4] 葛松, 蒋万, 何生虎, 余永涛, 张蕾蕾, 郭澍强, 王静. 宁夏肉牛皮肤病原真菌的分离与鉴定. 中国农业科学, 2015, 48(14): 2876-2883. GE S, JIANG W, HE S H, YU Y T, ZHANG L L, GUO S Q, WANG J. Isolation and identification of dermatophytes from beef cattle in Ningxia. Scientia Agricultura Sinica, 2015, 48(14): 2876-2883. (in Chinese)
[5] VAN DIEPENINGEN A D, BRANKOVICS B, ILTES J, VAN DER LEE T A, WAALWIJK C. Diagnosis of Fusarium infections: approaches to identification by the clinical mycology laboratory. Current Fungal Infection Reports, 2015, 9(3): 135-143.
[6] 曾莉莎, 吕顺, 刘文清, 赵志慧, 王芳, 周建坤, 李洪波, 陈石, 杜彩娴. 基于多基因序列分析对尖孢镰孢菌古巴专化型 (香蕉枯萎病菌)生理小种的鉴定. 菌物学报, 2014, 33(4): 867-882. ZENG L S, LÜ S, LIU W Q, ZHAO Z H, WANG F, ZHOU J K, LI HB, CHEN S, DU C X. The identification on race of Fusarium oxysporum f. sp. cubense based on multi-gene sequence analysis. Mycosystema, 2014, 33(4): 867-882. (in Chinese)
[7] 王容燕, 高波, 陈书龙, 马娟, 李秀花. 河北省甘薯镰孢菌腐烂与溃疡病的病原鉴定. 植物保护学报, 2016, 43(2): 241-247. WANG R Y, GAO B, CHEN S L, MA J, LI X H. Identification of the pathogens causing Fusarium root rot and stem canker on sweet potato in Hebei Province. Journal of Plant Protection, 2016, 43(2): 241-247. (in Chinese)
[8] 郭明霞, 贺运春, 洪坚平, 胡亚娟, 张宝俊. 山西省忻州地区土壤镰刀菌 (Fusarium)种类鉴定. 山西农业大学学报 (自然科学版), 2006, 26(2): 171-173. GUO M X, HE Y C, HONG J P, HU Y J, ZHANG B J. Identification of Fusarium in the soils collected from Xinzhou, Shanxi province. Journal of Shanxi Agricultural University (Natural Science), 2006, 26(2): 171-173. (in Chinese)
[9] 胡亚娟, 贺运春, 郭明霞. 山西晋中地区植物病原镰刀菌(Fusarium)种类鉴定. 山西农业大学学报 (自然科学版), 2007, 27(3): 276-280. HU Y J, HE Y C, GUO M X. Identification of phytopathogenic Fusarium in Jinzhong, Shanxi provine. Journal of Shanxi Agricultural University (Natural Science Edition), 2007, 27(3): 276-280. (in Chinese)
[10] 袁微微, 李新凤, 高俊明, 王建明, 郝晓娟, 贺运春. 山西省瓜类作物根茎部镰刀菌的分离与鉴定. 山西农业科学, 2013, 41(3): 246-250. YUAN W W, LI X F, GAO J M, WANG J M, HAO X J, HE Y C. Isolation and identification of Fusarium from roots of cucurbits crops in Shanxi province. Journal of Shanxi Agricultural Sciences, 2013, 41(3): 246-250. (in Chinese)
[11] 李新凤, 王建明, 高俊明, 贺运春. 山西省甘蓝黄叶病病原菌的分离与鉴定. 山西农业大学学报 (自然科学版), 2011, 31(6): 517-521. LI X F, WANG J M, GAO J M, HE Y C. Isolation and identification of the pathogens causing cabbage Fusarium wilt in Shanxi province. Journal of Shanxi Agricultural University (Natural Science Edition), 2011, 31(6): 517-521. (in Chinese)
[12] 李新凤, 王建明, 张作刚, 高俊明, 郝晓娟, 贺运春. 山西省玉米穗腐病病原镰孢菌的分离与鉴定. 山西农业大学学报 (自然科学版), 2012, 32(3): 218-223. LI X F, WANG J M, ZHANG Z G, GAO J M, HAO X J, HE Y C. Isolation and identification of the pathogen Fusarium causing maize ear rot in Shanxi province. Journal of Shanxi Agricultural University (Natural Science Edition), 2012, 32(3): 218-223. (in Chinese)
[13] 项勇, 崔京霞, 吕安国. 东北红豆杉内生真菌的分离和筛选. 东北林业大学学报, 2002, 30(2): 30-34. XIANG Y, CUI J X, LÜ A G. Isolation and screen of Taxus cuspidate Sieb. et Zucc. endophytic fungi. Journal of Northeast Forestry University, 2002, 30(2): 30-34. (in Chinese)
[14] WANG L, ZHUANG W Y. Designing primer sets for amplification of partial calmodulin genes from Penicillia. Mycosystema, 2004, 23(4): 466-473.
[15] WHITE T J, BRUNS T, LEE S, TAYLOR J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics// PCR Protocols: A Guide To Methods and Applications. New York: Academic Press, 1990: 315-322.
[16] CARBONE I, KOHN L M. A method for designing primer sets for speciation studies in filamentous ascomycetes. Mycologia, 1999, 91(3): 553-556.
[17] REHNER S A, BUCKLEY E. A Beauveria phylogeny inferred from nuclear ITS and EF1-alpha sequences: evidence for cryptic diversification and links to Cordyceps teleomorphs. Mycologia, 2005, 97(1): 84-98.
[18] O’DONNELL K, CIGELNIK E. Two divergent intragenomic rDNA ITS2 types within a monophyletic lineage of the fungus Fusarium are nonorthologous. Molecular Phylogenetics & Evolution, 1997, 7(1): 103-116.
[19] GLASS N L, DONALDSON G C. Development of primer sets designed for use with the PCR to amplify conserved genes from filamentous ascomycetes. Applied & Environmental Microbiology, 1995, 61(4): 1323-1330.
[20] 赵鹏, 罗晶, 庄文颖, 刘杏忠, 吴冰. 新丛赤壳属真菌DNA条形码研究. 中国科学(生命科学), 2011, 41(7): 565-576. ZHAO P, LUO J, ZHUANG W Y, LIU X Z, WU B. DNA barcoding of the genus Neonectria. Scientia Sinica Vitae, 2011, 41(7): 565-576. (in Chinese)
[21] THOMPSON J D, HIGGINS D G, GIBSON T J. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Research, 1994, 22(22): 4673-4680.
[22] HALL T A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series, 1999, 41: 95-98.
[23] TAMURA K, DUDLEY J, NEI M, KUMAR S. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0. Molecular Biology & Evolution, 2007, 24(8): 1596-1599.
[24] MEYER C P, PAULAY G. DNA barcoding: error rates based on comprehensive sampling. PLoS Biology, 2005, 3(12): e422.
[25] LAHAYE R, VAN DER BANK M, BOGARIN D, WARNER J, PUPULIN F, GIGOT G, MAURIN O, DUTHOIT S, BARRACLOUGH T G, SAVOLAIENE V. DNA barcoding the floras of biodiversity hotspots. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(8): 2923-2928.
[26] BURLAND T G. DNASTAR’s laser gene sequence analysis software. Methods in Molecular Biology, 2000, 132: 71-91.
[27] SLABBINCK B, DAWYNDT P, MARTENS M, DE VOS P, DE BAETS B. TaxonGap: a visualization tool for intra- and inter-species variation among individual biomarkers. Bioinformatics, 2008, 24(6): 866-867.
[28] 汤彦承, 路安民. 浅评当今植物系统学中争论的三个问题--并系类群、谱系法规和系统发育种概念. 植物分类学报, 2005, 43(5): 403-419. TANG Y C, LU A M. Paraphyletic group, PhyloCode and phylogenetic species-the current debate and a preliminary commentary. Acta Phytotaxonomica Sinica, 2005, 43(5): 403-419. (in Chinese)
[29] KRISTENSEN R, TORP M, KOSIAK B, JENSEN A H. Phylogeny and toxigenic potential is correlated in Fusarium species as revealed by partial translation elongation factor 1 alpha gene sequences. Mycological Research, 2005, 109(2): 173-186.
[30] KERENYI Z, ZELLER K, HORNOK L, LESLIE J F. Molecular standardization of mating type terminology in the Gibberella fujikuroi species complex. Applied & Environmental Microbiology, 1999, 65(9): 4071-4076.
[31] WATANABE M. Molecular phylogeny and identification of Fusarium species based on nucleotide sequences. Mycotoxins, 2013, 63(2): 133-142.
[32] 奚安. 马铃薯干腐病病原镰孢菌的分类研究[D]. 大连: 辽宁师范大学, 2015. XI A. Classification of Fusarium species from potato dry rot[D]. Dalian: Liaoning Normal University, 2015. (in Chinese)
[33] 王丽丽, 李洪涛, 日孜旺古丽·苏皮色来, 张磊, 王海云, 李克梅.乌昌地区窖藏马铃薯菌物病害调查及病原鉴定. 新疆农业大学学报, 2014, 37(6): 469-473. WANG L L, LI H T, RIZIWANGGULI S, ZHANG L, WANG H Y, LI K M. Investigation and identification of fungus disease and pathogens on storage period potato in Urumqi and Changji areas of Xinjiang. Journal of Xinjiang Agricultural University, 2014, 37(6): 469-473. (in Chinese)
[34] 魏巍, 许艳丽, 朱琳, 张思佳, 李淑娴. 长期连作对大豆根际土壤镰孢菌种群的影响. 应用生态学报, 2014, 25(2): 497-504. WEI W, XU Y L, ZHU L, ZHANG S J, LI S X. Impact of long-term continuous cropping on the Fusarium population in soybean rhizosphere. Chinese Journal of Applied Ecology, 2014, 25(2): 497-504. (in Chinese)
[35] 杨晓贺, 吕国忠, 赵志慧, 刘丽莉, 姚贤民. 东北地区蔬菜大棚内黄瓜枯萎病病株上镰孢菌的分离和鉴定. 沈阳农业大学学报, 2007, 38(3): 308-311. YANG X H, LÜ G Z, ZHAO Z H, LIU L L, YAO X M. Isolation and identification of Fusarium species from cucumber wilt diseased plants in vegetable greenhouse in Northeastern China. Journal of Shenyang Agricultural University, 2007, 38(3): 308-311. (in Chinese)
[36] 陈霞, 刘东, 张艳菊, 秦智伟, 周秀艳. 黄瓜枯萎病病株镰孢菌的分离与鉴定. 东北农业大学学报, 2010, 41(7): 37-44. CHEN X, LIU D, ZHANG Y J, QIN Z W, ZHOU X Y. Isolation and identification of Fusarium from cucumber wilt plants. Journal of Northeast Agricultural University, 2010, 41(7): 37-44. (in Chinese)
(责任编辑 岳梅)
Analysis of Population Distribution and Genetic Variation of Plant Pathogenic Fusarium in Shanxi Province
WANG Lin, LI XinFeng, XU YuMei, CHANG YinDong, WANG JianMing
(College of Agriculture, Shanxi Agricultural University, Taigu 030801, Shanxi)
【Objective】The objectives of this study are to obtain the sequences of ITS, EF-1α and β-tubulin gene regions from plant pathogen, Fusarium spp., compare the suitable gene sequences for species identification, and to analyze the populationdistribution and genetic variation of Fusarium spp. in Shanxi Province by using the suitable gene sequences. 【Method】 A total of 625 Fusarium strains were collected from 28 counties and 11 cities in Shanxi Province from 2013 to 2015. The ITS, EF-1α and β-tubulin gene fragments of the morphology of clear strains from 625 strains were sequenced and analyzed. The sequences were assembled and edited using Sequencher software, then blasted in GenBank in NCBI and FUSARIUM-ID databases. The sequences from the clearly documented and reference species were downloaded. All the sequences were aligned and edited manually using ClustalX and GelDoc. The inter- and intra-specific variations of Fusarium spp. were analyzed with MEGA, Excel, DNAstar and TaxonGap. All the three gene sequences consisting of ITS, EF-1α and β-tubulin were selected for the taxonomy of the Fusarium species and then the best optimal fragment genes were used in the analysis of the population distributions of Fusarium spp. in Shanxi Province. 【Result】EF-1α was the best gene fragment for identifying Fusarium species among the three candidate gene fragments. Intra-specific pairwise distances was 24 times higher than that of the inter-specific pairwise distances. The intra-specific variation was smaller than those of the inter-specific variation by 73% of the tested species. The accuracy rate for identification was the highest, reached 87%. The phylogenetic relationships derived from the EF-1α sequences showed that different strains in 22 of 27 Fusarium species were monophyly, clustering in a same clade with high supporting values. Among the 27 species of Fusarium, F. oxysporum was the dominant species with 22.1% frequency and covered the largest geographical distribution in the 23 counties or regions in Shanxi Province. Followed by F. solani with 13.8% in 14 counties or regions. The results of population distribution of Fusarium in different geographical regions in Shanxi Province showed that F. oxysporum was the dominant species in Yuncheng, Linfen, Xinzhou, Changzhi, Lüliang, Jinzhong and Taiyuan city; F. lateritium was the dominant species in Shuozhou, F. solani in Datong, F. verticillioides in Jincheng and F. incarnatum in Yangquan. Among the different regions in Shanxi Province, the most abundant Fusarium species was in Jinzhong and Xinzhou, followed by Linfen, and the least distribution was in Shuozhou. The results of the population distribution of Fusarium from different hosts showed that the 15 Fusarium species extracted from Lycopersicon esculentum, 13 species from Solanum tuberosum and 12 from Glycine max. In these hosts, F. oxysporum was the dominant species in Lycopersicon esculentum, Cucumis sativus, Citrullus lanatus, Solanum tuberosum, Solanum melongena, Cucurbita pepo and Brassica oleracea. F. verticillioides was the dominant species in Glycine max and Zea mays. F. avenaceum and F. graminearum were the dominant species in Triticum aestivum. 【Conclusion】There are abundant genetic variation between interand intra-specific of Fusarium. Of the three gene regions, EF-1α is the most suitable region for Fusarium species identification. Fusarium morphological taxonomy results are not fully agreed with the molecular phylogenetic results using EF-1α sequences. F. oxysporum is the dominant Fusarium species in Shanxi Province. There are obvious genetic variations of Fusarium populations in different districts and hosts. The research results will provide a theoretic and scientific basis for Fusarium taxonomy, DNA barcode screening, quarantine and integrated control.
Fusarium; population distribution; intra- and inter-specific variation; genetic variation; phylogenetic analysis
2017-01-25;接受日期:2017-03-27
山西省科技攻关项目(20120311019-3)、山西省科技基础条件平台建设项目(1105-0104)
联系方式:王琳,E-mail:wwll0621@163.com。通信作者王建明,E-mail:jm.w@163.com
