兰州熊蜂气味受体家族鉴定及分析
2017-06-15王烨韩蕾董捷黄家兴吴杰
王烨,韩蕾,董捷,黄家兴,吴杰
(中国农业科学院蜜蜂研究所农业部授粉昆虫生物学重点开放实验室,北京100093)
兰州熊蜂气味受体家族鉴定及分析
王烨,韩蕾,董捷,黄家兴,吴杰
(中国农业科学院蜜蜂研究所农业部授粉昆虫生物学重点开放实验室,北京100093)
【目的】了解兰州熊蜂(Bombus lantschouensis)基因组中气味受体基因(odorant receptors, Ors)情况,为进一步分析气味受体功能提供信息,从而为研究该基因家族在熊蜂觅食、交尾、通讯等行为中的重要功能打下基础。【方法】提取兰州熊蜂胸部基因组DNA,进行Illumina高通量测序,对测序所得原始序列进行质控并拼接获得基因组序列,然后使用本地 blast 2.2.28+对构建兰州熊蜂基因组本地数据库,使用已知的地熊蜂(Bombus terrestris)和意大利蜜蜂(Apis mellifera ligustica)的气味受体蛋白序列作为种子序列搜索数据库获得兰州熊蜂的气味受体序列;使用EMBOSS1.5对气味受体蛋白序列进行基本理化分析,利用GSDS2.0对家族序列的内含子与外显子位置进行分析,然后使用MEME 4.11.2对氨基酸序列进行保守基序分析,最后利用ClustaW 2.1、TrimAl 1.2、PhyML3.0对兰州熊蜂、地熊蜂和意大利蜜蜂气味受体家族序列进行系统进化分析。【结果】共获得165个兰州熊蜂气味受体基因,包括1个非典型气味受体(olfactory receptor co-receptor,Orco)、5个假基因和159个气味受体。基因结构分析发现,气味受体家族外显子数目从4个到9个不等。其中Or 47—57的序列中外显子数量最少,为4个;Or 128—161中外显子的数量最多,为9个。根据基因结构的不同,将气味受体分为10大类型,每个类型中的序列都有相似的外显子长度与数量。Or 1—38、Or 69—85、Or 128—164这3大类所包含成员数较多(分别为38、17、37),其他7类包含成员个数都约10个,且每个类型中序列在染色体上都呈串联排布,其中Or 1—38中都包含一个较长的第一外显子。保守基序分析发现,在检索的10个保守基序中,除基序5为未知基序外,其他9个基序均包含在其保守结构域(7tm_6 domain)中。 Or 1—38与Or 39—46多数成员包含全部10个保守基序,基序2、3、4、9广泛存在于该家族成员序列中,这4个基序可能为该家族关键的功能区域。系统发育分析将Ors分为5个亚家族(I—V),其中亚家族 II包含 2种基因结构类型序列(BlOr 97—100与BlOr 69—85),亚家族V包含4种(BlOr 1—46、47—57、86—95和101—107)。BlOr 150—155与AmOr 122—139分别聚为两个分支,BlOr 47—57与AmOr 63—65也发现类似的聚类,这表明在进化中,蜜蜂与熊蜂的Ors出现了特异性的扩张与缺失。在进化树中发现Or 115较早与其他成员分离,位于树的基部,推测该序列可能更接近气味受体家族的祖先序列。【结论】探明了兰州熊蜂的Or基因数量、基因结构及其进化关系;该基因家族在进化过程中部分成员保守基序丢失,这些结果为进一步了解 Or基因功能打下了基础。
兰州熊蜂;气味受体;基因结构;保守基序;进化分析
0 引言
【研究意义】熊蜂属于膜翅目蜜蜂科昆虫,是蜜蜂的近缘物种,在维持自然生态系统平衡和农业生产中发挥重要的作用[1-2]。兰州熊蜂(Bombus lantschouensis)是中国近年筛选出的优良蜂种,由于其易于人工饲养、蜂群群势大、温驯等优势,在设施作物授粉方面有较大的应用潜力[3]。嗅觉在昆虫感知外界环境、群内成员信息交流以及社会行为等方面都起到十分重要作用[4]。气味受体在嗅觉反应中起关键作用,其介导的气味分子与嗅觉神经元的专一性结合是昆虫嗅觉识别的重要基础[5-6]。因此,研究熊蜂中气味受体基因,对进一步了解气味受体在嗅觉反应中的作用具有重要意义。【前人研究进展】昆虫拥有复杂的嗅觉系统使其能够对外界环境进行识别,从而影响觅食、繁殖、群集及躲避敌害等行为[7]。在昆虫嗅觉识别的过程中有多种蛋白参与,主要包括气味结合蛋白(odorant binding proteins,OBPs)、气味受体(odorant receptors,Ors)、化学感受蛋白(chemosensory proteins,CSPs)以及感觉神经元膜蛋白(sensory neuronmembrane proteins,SNMPs)等[8],这些嗅觉蛋白中Ors是嗅觉识别过程中关键成分。当环境中气味分子通过昆虫嗅觉感受器表皮上的微孔进入感受器腔中,与OBP或是CSPs形成复合体,然后被运送到嗅觉神经元树突膜上与SNMPs和Ors相互作用,引起动作电位,从而传入中枢神经控制昆虫产生特定的生理与行为反应[9]。昆虫的气味受体为7次跨膜蛋白,其N端位于细胞膜内而C端位于膜外[6,10],嗅觉传导信号过程与传统G蛋白偶联受体不同,是一种独特的离子门控传递方式[11-12]。昆虫的嗅觉神经元一般表达两类气味受体:一类是传统气味受体,可以识别气味分子和信息素,在不同昆虫中同源性很低,具有高度的多样性;另一类受体在不同昆虫之间保守,不感受气味,与大多数传统气味受体共表达,称为非典型气味受体 Orco (olfactory receptor co-receptor)[4,13]。由于嗅觉在参与昆虫取食、繁殖以及个体之间信息交流中都发挥着重要作用,因此了解嗅觉在昆虫活动中发挥的作用以及调控机制尤为重要。自BUCK等[14]在褐家鼠(Rattus norvegicus)中发现嗅觉受体蛋白家族之后,在多种生物中都发现了气味受体,昆虫中气味受体家族的鉴定,直到1999年黑腹果蝇(Drosophila melanogaster)基因组测序后,在其基因组中鉴定出62个Ors基因[15]。随后在冈比亚按蚊(Anopheles gambiae)中鉴定到79 个Ors[16],家蚕(Bombyx mori)中有60个[17-19]。在膜翅目昆虫中,Or家族相对于果蝇与蚊类发生了扩展,目前在金小峰(Nasonia wasps)中发现300个Ors[20],在蚂蚁(Linepithema humile等)中甚至达到400个[21-24],在与熊蜂亲缘关系较近的意大利蜜蜂(Apis mellifera ligustica)中共发现170个Ors,其中7个为假基因,系统发育分析表明,这170个Ors分为5个亚家族,包含4个小亚家族和1个大亚家族(包含157个Ors)[25]。研究表明AmOr 151与AmOr 152在雌性蜂中具有较高的表达量,而且已被鉴定与花香气味分子靶定,AmOr 151主要靶标为大多数植物释放的芳香醇气味分子[26]。最近在小蜜蜂(Apis florea)的基因组中鉴定获得180个Ors,与意大利蜜蜂中的Ors序列十分相似,在小蜜蜂中还发现了一些与蜜蜂非同源的Ors和独立的假基因,说明在这两个物种的Ors进化中发生了分离[27]。目前对熊蜂中气味受体的研究很少,仅在B. terrestris全基因组测序后,鉴定获得165个Ors[28]。【本研究切入点】随着测序技术的日渐成熟,许多昆虫的基因组都已被测序,其气味受体也相继被发觉,而且对气味受体越来越深入的研究发现,气味受体对昆虫的社会活动起到十分重要的作用,但目前在熊蜂上气味受体相关的研究很少,对兰州熊蜂气味受体的研究尚未见报道。【拟解决的关键问题】通过对兰州熊蜂基因组测序挖掘气味受体家族,并利用生物信息学的方法对其特性、基因结构、保守结构域和进化关系进行分析,鉴定兰州熊蜂的气味受体家族,为克隆兰州熊蜂气味受体基因提供信息,并为研究气味受体家族在熊蜂中的功能打下基础。
1 材料与方法
试验于 2016年在中国农业科学院蜜蜂研究所昆虫授粉与生态实验室完成。
1.1 试验材料
兰州熊蜂为中国农业科学院蜜蜂研究所昆虫授粉与生态研究室繁育,饲养温度(28±0.5)℃,相对湿度为 50%±5%,无光照。取蜂群中雄性蜂,使用基因组 DNA提取试剂盒(Wizard Genomic DNA Purification Kit)提取其胸部基因组DNA,交由北京贝瑞和康生物技术有限公司进行 Illumina高通量测序,将测序所得原始序列进行质控,拼接获得兰州熊蜂的基因组。使用本地blast 2.2.28+对基因组进行数据库的构建及序列搜索。
1.2 序列鉴定与理化特性
使用地熊蜂与意大利蜜蜂的Ors序列作为参考序列进行tblastn搜索,搜索条件设定e值为 1×10-6。选取序列相似度最高部分进行拼接(相似度最低阈值50%)。然后,将拼接获得的编码区序列翻译获得对应氨基酸序列,最后,将氨基酸序列提交到 SMART (http://smart.embl–heidelberg .de/)数据库验证其保守结构区域7tm_6(PF02949)。
使用TMHMM对序列的跨膜结构域进行预测。使用EMBOSS1.5[29]中PEPSTATS程序对所获得的氨基酸序列进行分子量与等电点预测。
1.3 气味受体基因结构分析
将气味受体家族的每个成员的基因序列与编码区序列提交到在线基因结构分析工具 GSDS 2.0[30](http://gsds.cbi.pku.edu.cn/)中,获得基因序列的内含子与外显子位置信息。
1.4 基序分析
使用在线工具MEME 4.11.2[31]对Or序列的基序进行分析,设定motif长度在15—30,每个motif包含的位点最少7个,每个序列都进行10个motif的检索,其他参数均为默认参数。
1.5 多序列比对及进化分析
使用clustalW 2.1[32]进行多序列比对,利用TrimAl 1.2[33]去除冗余的片段(参数-resoverlap 0.8 -seqoverlap 80-gt 0.7),使用phyML 3.0[34]进行建树,替代模型为JTT,bootstrap为1 000次。
2 结果
2.1 兰州熊蜂气味受体序列鉴定
利用地熊蜂与意大利蜜蜂气味受体蛋白序列作为参考序列,通过tblastn搜索兰州熊蜂基因组获得气味受体,共获得165个气味受体蛋白序列,其中包括1个非典型气味受体Orco、4个假基因和159个气味受体。气味受体家族序列长度平均为392 bp,分子量为4.5 kD,等电点为7.97。共有41个序列预测到存在7个跨膜螺旋,81个序列预测到6个跨膜螺旋,且大部分序列都是缺少最后一个跨膜螺旋。从定位信息中发现Or 1—46、Or 58—68、Or 86—95、Or 128—139、Or 150—160分别串联排布在染色体2、13、9、11和4上,Or 47—57与Or 70—84都串联排布在15号染色体上(附表 1)。对序列分析发现多个序列在两种熊蜂中出现明显的长度差异(表 1)以及氨基酸位点的改变(图1)。
2.2 基因结构
对所有嗅觉受体基因序列进行结构分析,发现Or 101序列最长,超过13 kb,其第二内含子长度最长(图2-A)。在Or 102、127、148中均包含一个长内含子,导致这些基因序列的长度都超过了10 kb。气味受体家族基因序列外显子个数为4—9个,其中Or 47—57的序列中外显子数量最少,为4个;Or 128—161中外显子的数量最多,为9个。根据基因结构不同,可将所有序列分为10个类型,分别为Or 1—38、39—46、47—57、58—68、69—85、86—96、97—100、101—107、116—127、128—164,每个类型中的序列都有相似的外显子长度与数量。其中,Or 1—38、69—85、128—164这3个类型所包含成员数较多(分别为38、17、37),其他7类包含成员个数都约10个,Or 1 —38所有成员都具有较长的第一外显子。

图1 兰州熊蜂与地熊蜂气味受体序列氨基酸差异位点Fig. 1 Different amino acid site of Or sequences of B. lantschouensis and B. terrestris
2.3 保守基序分析
对所有蛋白序列进行保守基序分析(图 2-B),在检索的10个保守基序中(图3),除基序5为未知基序外,其他9个基序均包含在其保守结构域(7tm_6 domain)中。10种基因结构类型序列中Or 1—38与39—46、47—57与86—95、89—85与97—100具有相似的保守基序。在Or 1—46中大部分序列包含全部10个基序,其他序列中均缺失基序10;在Or 47—57与86—95中,除序列89序列长度缺失外,其他序列中仅缺少基序10;Or 58—68中主要缺少基序6与7;Or 69—85与Or 97—100中主要缺少基序6;Or 115 —127中除了缺失基序6之外,还缺少普遍存在基序1 与5;Or 128—164的序列中都缺少了基序7。在大部分序列中都包含了基序2、3、4、9,这4个基序可能为该家族关键的功能区域。
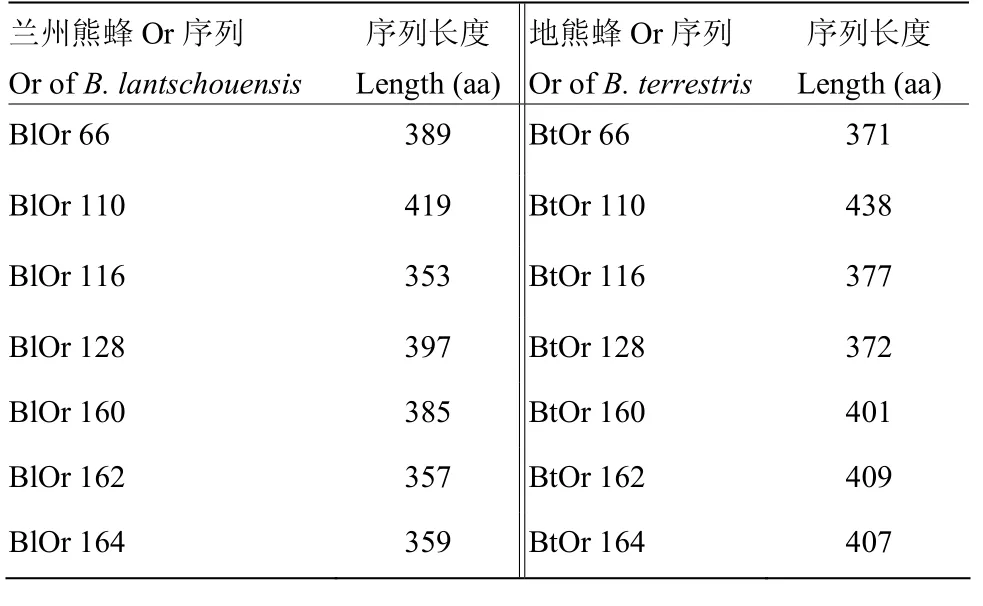
表1 兰州熊蜂与地熊蜂中Or序列长度差异Table 1 Difference of Or sequence length between B. lantschouensis and B. terrestris
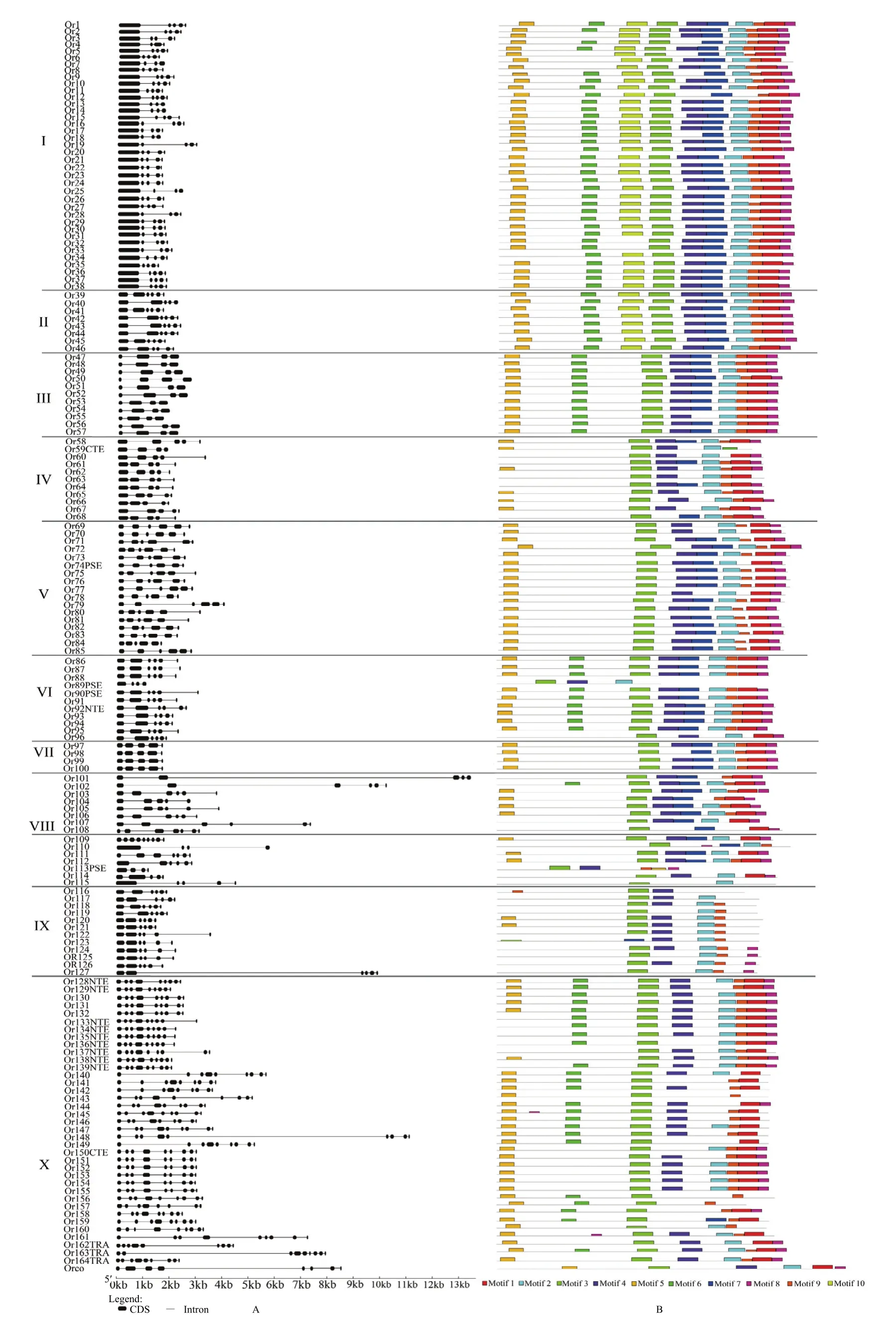
图2 兰州熊蜂气味受体基因结构与保守基序Fig. 2 Gene structure and conserved motif of Ors in B. lantschouensis

图3 气味受体家族蛋白序列中的保守基序及其序列LogoFig. 3 Sequences Logo of the MEME defined conserved motifs observed among odorant receptors proteins
2.4 气味受体家族进化树
以Orco作为外群,利用地熊蜂、兰州熊蜂、意大利蜜蜂的全部气味受体蛋白序列构建系统发育树(图4)。从系统发育树中可以看出,Or家族明显被分为5个亚家族(I—V)。所有BlOr 58—68成员都聚为亚家族I,BlOr 97—100与BlOr 69—85聚为亚家族II,BlOr 128—164聚为亚家族III,BlOr 86—95、101—107、47—57、1—46聚为亚家族IV,BlOr 110—127聚为亚家族 V。在亚家族 III中,BlOr 150—155与AmOr 122—139分别聚为两个分支,在亚家族IV的中,BlOr 47—57与AmOr 63—65也发现类似的聚类,这表明在进化中,蜜蜂与熊蜂的Ors出现了特异性的扩张与缺失。在进化树中发现BlOr 115较早与其他成员分离,位于树的基部,而且在结构域分析中 Or115中包含的保守基序较少,说明该序列更为接近气味受体家族的祖先序列。
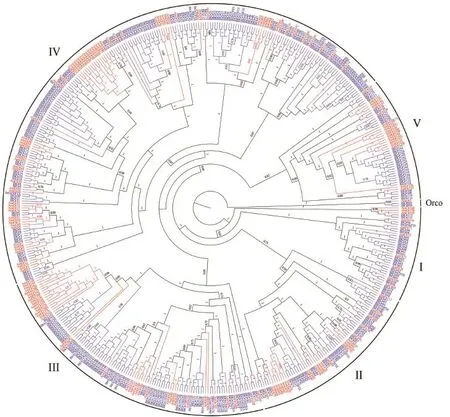
图4 意大利蜜蜂、地熊蜂、兰州熊蜂Ors 系统发育树Fig. 4 Phylogenetic tree of odorant receptors from A. m. ligustica, B. terrestris and B. lantschouensis
3 讨论
气味受体家族是昆虫化学感受器家族中一类重要的蛋白家族,在昆虫的生存及繁衍生息中都发挥十分重要的作用[12]。目前在许多昆虫中已经发现气味受体家族的存在,例如在果蝇中发现存在62个气味受体[15],家蚕中存在60个[19],金小峰中发现300个[20],蚂蚁中多达400个[21]。在不同的昆虫中,Or家族的成员数量差异很大,这可能是由于在长期进化中,不同物种对嗅觉的依赖程度不同而造成。熊蜂与蜜蜂中假基因的个数比果蝇与蚊类中要多,但是远少于哺乳动物中的假基因(比例高达25%—100%)[35]。在鉴定的所有气味受体中,包括一个非典型气味受体Orco,这个蛋白与果蝇中的 DmOr83b同源,序列相似度为61.41%,与蜜蜂中的AmOr2相似度为76.88%,与地熊蜂中的Orco相似度为99.17%[36],这表明该蛋白在不同物种之间高度保守,可以作为判断昆虫进化参数。在兰州熊蜂中,Or家族的成员共有165个基因,其中包括5个假基因,与地熊蜂中气味受体家族成员数目相同,在聚类分析中也出现一一对应的现象,这可能是由于它们具有较近的亲缘关系,导致气味受体序列之间相似度很高,同时这说明这两种熊蜂中对各种化学气味识别机制有可能相近。但是通过对序列的分析发现,两种熊蜂的某些气味受体在序列长度与氨基酸的变异程度上有十分明显的差异(表1、图1),这说明在兰州熊蜂中气味受体序列可能开始出现特异性改变。
BlOr家族成员存在串联分布于某条染色体上的现象,这些串联序列具有相似的基因结构,表明 Or家族的起源可能是通过祖先序列的多重复制产生。进化分析发现Or家族包含5个亚家族,10种类型的基因结构,在亚家族IV的BlOr 1—46分支中,所有成员的序列中包含一个特有的保守基序10,该基序可能使它们具有特有的功能,而且该分支与蜜蜂中AmOr 1 —61聚在一起,AmOr 1—61是已知的昆虫化学感受器中最大的串联重复,其中包含一个已经明确功能的成员AmOr 11,该成员只对蜂王信息素中9-氧代-癸二烯酸(9-ODA)产生特异反应[37]。在另一个成员较多的亚家族IV中,成员序列都含有9个外显子,与蜜蜂中一个同样包含 9个外显子的亚家族 AmOr 97— 113、122—140、159、162聚在一起,该亚家族在蚂蚁中也存在,而且被鉴定为一类参与认巢及同伴识别的表皮烃类受体[31-32,34]。
在序列拼接中发现,BlOr 162—164的第一外显子与其他外显子的编码方向相反,其位置处于其他外显子的下游,这种反式剪切的现象在蜜蜂的气味受体AmOr 140中首次被发现[36],本文的结果也证实了这种现象的存在。
在蜜蜂中这些气味受体与蜜蜂触角叶神经小球的数目相似,而且具有一个近似一对一的关系,这说明一个嗅觉神经元可能仅表达一个气味受体蛋白,而且触角叶神经的轴突突触为单个小球[36],果蝇中也发现类似现象[4,8,38]。在蜜蜂中发现Ors数量远超于果蝇,这可能是由于蜜蜂主要食物为花蜜和花粉,长期进化使得其需要更多Ors识别不同的花香及其复杂社会行为[36]。熊蜂进化程度低于蜜蜂,虽在社会性及采集能力上不及蜜蜂,但是对于一些特定作物如茄科植物的授粉能力却高于蜜蜂,这种现象可能是因为熊蜂与蜜蜂中Ors差异所致。关于熊蜂与蜜蜂之间Ors差异研究较少,故对其进行生物信息学分析有助于揭示其重要功能进化,进一步帮助揭示蜜蜂中复杂的社会行为。
4 结论
通过对兰州基因组的鉴定获得165个气味受体家族成员,包含1个非典型气味受体Orco,5个假基因,159个气味受体。该家族中包含有多个亚家族,家族内成员之间具有相似的基因结构与保守结构域。在进化中由于蜜蜂与熊蜂物种的分化,导致气味受体在进化中发生了物种特异性的扩展与缺失。
[1] GOULSON D, LYE G C, DARVILL B. Decline and conservation of bumble bees. Annual Review of Entomology, 2008, 53(3): 191-208.
[2] VERGARA C H, FONSECABUENDIA P. Pollination of greenhouse tomatoes by the mexican bumblebee Bombus ephippiatus (Hymenoptera: Apidae). Journal of Pollination Ecology, 2012, 7(4): 27-30.
[3] 周志勇, 张红, 梁铖, 邹宇, 董捷, 袁晓龙, 黄家兴, 安建东. 西方蜜蜂和兰州熊蜂在设施桃园的访花偏好性比较. 昆虫学报, 2015, 58(12): 1315-1321. ZHOU Z Y, ZHANG H, LIANG C, ZOU Y, DONG J, YUAN X L, HUANG J X, AN J D. Foraging preference of the honeybee Apis mellifera and the bumblebee Bombus lantschouensis (Hymenoptera:Apidae) in peach greenhouse. Acta Entomologica Sinica, 2015, 58(12): 1315-1321. (in Chinese)
[4] 乔奇, 原国辉, 李海超, 郭线茹, 罗梅浩. 昆虫气味受体研究进展.昆虫学报, 2008, 51(1): 75-80. QIAO Q, YUAN G H, LI H C, GUO X R, LUO M H. Research advances in odorant receptors in insects. Acta Entomologica Sinica, 2008, 51(1): 75-80. (in Chinese)
[5] VOSSHALL L B, WONG A M, AXEL R. An olfactory sensory map in the fly brain. Cell, 2000, 102(2): 147-159.
[6] BENTON R, SACHSE S, MICHNICKM S W, VOSSHALL L B. Atypical membrane topology and heteromeric function of Drosophila odorant receptors in vivo. PLoS Biology, 2006, 4(2): e20.
[7] FIELD L, PICKETT J, WADHAMS L. Molecular studies in insect olfaction. Insect Molecular Biology, 2008, 9(6): 545-551.
[8] PELOSI P, ZHOU J J, BAN L P, CALVELLO M. Soluble proteins in insect chemical communication. Cellular & Molecular Life Sciences, 2006, 63(14): 1658-1676.
[9] RÜTZLER M, ZWIEBEL L J. Molecular biology of insect olfaction: recent progress and conceptual models. Journal of Comparative Physiology A Neuroethology Sensory Neural & Behavioral Physiology, 2005, 191(9): 777-790.
[10] LUNDIN C, KÄLL L, KREHER S A, KAPP K, SONNHAMMER E L, CARLSON J R, GUNNAR V H, NILSSON I. Membrane topology of the Drosophila OR83b odorant receptor. FEBS Letters, 2007, 581(29): 5601-5604.
[11] SATO K, PELLEGRINO M, NAKAGAWA T, NAKAGAWA T, VOSSHALL L B, TOUHARA K. Insect olfactory receptors are heteromeric ligand-gated ion channels. Nature, 2008, 452(7190): 1002-1006.
[12] WICHER D, SCHAFER R, BAUERNFEIND R, STENSMYR M C, HELLER R, HEINEMANN S H, HANSSON B S. Drosophila odorant receptors are both ligand-gated and cyclic-nucleotideactivated cation channels. Nature, 2008, 452(7190): 1007-1011.
[13] VOSSHALL L B, AMREIN H, MOROZOV P S, RZHETSKY A, AXEL R. A spatial map of olfactory receptor expression in the Drosophila antenna. Cell, 1999, 96(5): 725-736.
[14] BUCK L, AXEL R. A novel multigene family may encode odorant receptors: a molecular basis for odor recognition. Cell, 1991, 65(1): 175-187.
[15] ROBERTSON H M, WARR C G, CARLSON J R. Molecular evolution of the insect chemoreceptor gene superfamily in Drosophila melanogaster. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(Suppl. 2): 14537-14542.
[16] FOX A N, PITTS R J, ROBERTSON H M, CARLSON J R, ZWIEBEL L J. Candidate odorant receptors from the malaria vector mosquito Anopheles gambiae and evidence of down-regulation in response to blood feeding. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(25): 14693-14697. [17] SAKURAI T, NAKAGAWA T, MITSUNO H, MORI H, ENDO Y, TANOUE S, YASUKOCHI Y, TOUHARA K, NISHIOKA T. Identification and functional characterization of a sex pheromone receptor in the silkmoth Bombyx mori. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(47): 16653-16658.
[18] NAKAGAWA T, SAKURAI T, NISHIOKA T, TOUHARA K. Insect sex-pheromone signals mediated by specific combinations of olfactory receptors. Science, 2005, 307(5715): 1638-1642.
[19] WANNER K W, ANDERSON A R, TROWELL S C, THEILMANN D A, ROBERTSON H M, NEWCOMB R D. Female-biased expression of odourant receptor genes in the adult antennae of the silkworm, Bombyx mori. Insect Molecular Biology, 2007, 16(1): 107-119.
[20] ROBERTSON H M, GADAU J, WANNER K W. The insect chemoreceptor superfamily of the parasitoid jewel wasp Nasonia vitripennis. Insect Molecular Biology, 2010, 19(Suppl. 1): 121-136.
[21] SMITH C D, ZIMIN A, HOLT C, ABOUHEIF E, BENTON R, CASH E, CROSET V, CURRIE C R, ELHAIK E, ELSIK C G, et al. Draft genome of the globally widespread and invasive Argentine ant (Linepithema humile). Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(14): 5673-5678.
[22] SMITH C R, SMITH C D, ROBERTSON H M, HELMKAMPF M, ZIMIN A, YANDELL M, HOLT C, HU H, ABOUHEIF E, BENTON R, et al. Draft genome of the red harvester ant Pogonomyrmex barbatus. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(14): 5667-5672.
[23] WURM Y, WANG J, RIBA-GROGNUZ O, CORONA M, NYGAARD S, HUNT B G, INGRAM K K, FALQUET L, NIPITWATTANAPHON M, GOTZEK D, et al. The genome of the fire ant Solenopsis invicta. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(14): 5679-5684.
[24] ZHOU X, SLONE J D, ROKAS A, BERGER S L, LIEBIG J R, RAY A, REINBERG D, ZWIEBEL L J. Phylogenetic and transcriptomic analysis of chemosensory receptors in a pair of divergent ant species reveals sex-specific signatures of odor coding. PLoS Genetics, 2012, 8(8): e1002930.
[25] WEINSTOCK G M, ROBINSON G E, GIBBS R A, WEINSTOCK GM, WORLEY K C. Insights into social insects from the genome of the honeybee Apis mellifera. Nature, 2006, 443(7114): 931-949.
[26] CLAUDIANOS C, LIM J, YOUNG M, YAN S Z, CRISTINO A S, NEWCOMB R D, GUNASEKARAN N, REINHARD J. Odor memories regulate olfactory receptor expression in the sensory periphery. European Journal of Neuroscience, 2014, 39(10): 1642-1654.
[27] KARPE S D, JAIN R, BROCKMANN A, SOWDHAMINI R. Identification of complete repertoire of Apis florea odorant receptors reveals complex orthologous relationships with Apis mellifera. Genome Biology & Evolution, 2016, 8(9): 2879-2895.
[28] SADD B M, BARRIBEAU S M, BLOCH G, DE GRAAF D C, DEARDEN P, ELSIK C G, GADAU J, GRIMMELIKHUIJZEN C, HASSELMANN M, LOZIER J D, et al. The genomes of two key bumblebee species with primitive eusocial organization. Genome Biology, 2015, 16: 76.
[29] RICE P, LONGDEN I, BLEASBY A. EMBOSS: the European molecular biology open software suite. Trends in Genetics, 2000, 16(6): 276-277.
[30] HU B, JIN J, GUO A Y, ZHANG H, LUO J, GAO G. GSDS 2.0: an upgraded gene feature visualization server. Bioinformatics, 2015, 31(8): 1296-1297.
[31] BAILEY T L, BODÉN M, BUSKE F A, FRITH M, GRANT C E, CLEMENTI L, REN J Y, LI W W, NOBLE W S. MEME SUITE: tools for motif discovery and searching. Nucleic Acids Research, 2009, 37(Suppl. 2): W202-W208.
[32] CAPELLA-GUTIÉRREZ S, SILLA-MARTÍNEZ J M, GABALDÓN T. trimAl: a tool for automated alignment trimming in large-scale phylogenetic analyses. Bioinformatics, 2009, 25(15): 1972-1973.
[33] LARKIN M A, BLACKSHIELDS G, BROWN N P, CHENNA R, MCGETTIGAN P A, MCWILLIAM H, VALENTIN F, WALLACE I M, WILM A, LOPEZ R, et al. Clustal W and Clustal X version 2.0. Bioinformatics, 2007, 23(21): 2947-2948.
[34] GUINDON S, DUFAYARD J F, LEFORT V, ANISIMOVA M, HORDIJK W, GASCUEL O. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0. Systematic Biology, 2010, 59(3): 307-321.
[35] YOUNG J M, TRASK B J. The sense of smell: genomics of vertebrate odorant receptors. Human Molecular Genetics, 2002, 11(10): 1153-1160.
[36] ROBERTSON H M, WANNER K W. The chemoreceptor superfamily in the honey bee, Apis mellifera: expansion of the odorant, but not gustatory, receptor family. Genome Research, 2006, 16(11): 1395-1403. [37] WANNE K W, NICHOLS A S, WALDEN K K O, BROCKMANN A, LUETJE C W, ROBERTSON H M. A honey bee odorant receptor for the queen substance 9-oxo-2-decenoic acid. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(36): 14383-14388.
[38] HALLEM E A, DAHANUKAR A, CARLSON J R. Insect odor and taste receptors. Annual Review of Entomology, 2006, 51(10): 113-135.
(责任编辑 岳梅)
Identification and Characteristics of Odorant Receptors in Bumblebee, Bombus lantschouensis
WANG Ye, HAN Lei, DONG Jie, HUANG JiaXing, WU Jie
(Key Laboratory for Insect-Pollinator Biology of the Ministry of Agriculture, Institute of Apicultural Research, Chinese Academy of Agricultural Sciences, Beijing 100093)
Bombus lantschouensis; odorant receptors; gene structure; conservative motif; phylogenetic analysis
2016-11-01;接受日期:2016-12-16
国家蜂产业技术体系专项资金(CARS-45)、中国农业科学院科技创新中心工程项目(CAASASTIP-2016-IAR)
联系方式:王烨,E-mail:wangye9276@foxmail.com。通信作者吴杰,Tel:010-62591543;E-mail:apis@vip.sina.com。通信作者黄家兴,E-mail:huangjiaxing@caas.cn
Abstract:【Objective】Bumblebee Bombus lantschouensis is one of the most important pollinators for wild plants and crops in North China. Odorant receptors (Ors) gene family from the genome of B. lantschouensis was identified and characterized. Result of this study will help to explore the function of this gene family in foraging, mating and other social behaviors in bumblebee.【Method】Genomic DNA of thorax of B. lantschouensis was extracted and sequenced by the high-output of next generation sequencing. The original sequence of the sequencing was quality controlled and assembled to the genomic sequence. The contigs and scaffolds were used to build local sequence database. Gene sequences of Bombus terresters and Apis mellifera ligustica were used to query the local database. The characteristics and gene structure of Ors were analyzed by EMBOSS1.5 and GSDS2.0 software, respectively. Conservative motif analysis of amino acid sequences was performed using MEME 4.11.2. The phylogenetic analysis of Ors of B. lantschouensis, B. terresters and A. mellifera was studied by ClustaW 2.1, TrimAl 1.2 and PhyML3.0.【Result】One hundred and sixty-five Ors were identified from the genome of B. lantschouensis, including an Orco, 5 pseudogenes and 159 Ors. Gene structure analysis showed that the number of exons of these Ors are varies from 4 to 9. The least number of exons found in Or 47-57 was 4 and the largest number of exons found in Or 128-161 was 9. According to gene structure, Or was classified into 10 groups. Similarity in exon lengths and numbers was identified in the sequence of each group. The numbers of subgroup members were about 10, except in Or 1-38, Or 69-85 and Or 128-164 (which has 38, 17 and 37 members, respectively). The members of each subgroup are arranged in tandem on chromosomes and Or 1-38 have a longer first exon. Conserved motif analysis revealed that 9 motifs are present in Ors conserved domains (7tm_6 domain) except for motif 5 in all 10 motifs. While Or 1-38 and Or 39-46 have all predicted motifs and motifs 2, 3, 4, and 9 are widely present in sequences, which may be the key functional regions of the family. Phylogenetic analysis showed the 5 subfamilies of Ors family. Whereas the subfamily II contains 2 groups (BlOr 97-100 and BlOr 69-85) and subfamily V contains 4 groups (BlOr 1-46, 47-57, 86-95 and 101-107). BlOr 150-155 and AmOr 122-139 were clustered into two branches, and similar phenomena were also found in BlOr 47-57 and AmOr 63-65. It indicates that Ors has species-specific loss or expansion in evolution of Apis and Bombus. Or 115 is located at the base of the tree, suggesting that the sequence may be closer to the ancestor sequence of the odor receptor family.【Conclusion】Numbers, gene structure and phylogenetic relationships of Ors from the genome of B. lantschouensis were clarified in current study. Conserved motif analysis indicated that motif has been missed during the evolution of Ors family among bees. These results would provide an important information for exploring the gene evolution and function of Ors in future.
