静电相互作用对驱动蛋白马达运动的影响
2017-06-05黄艳宾郭海军李洁刘秀红纪青河北工程大学数理学院河北邯郸05608邯郸学院机电学院河北邯郸056005河北工业大学生物物理研究所天津0040
黄艳宾,郭海军,李洁,刘秀红,纪青(.河北工程大学 数理学院,河北 邯郸 05608;.邯郸学院 机电学院,河北 邯郸 056005;.河北工业大学 生物物理研究所,天津 0040)
静电相互作用对驱动蛋白马达运动的影响
黄艳宾1,郭海军1,李洁2,刘秀红1,纪青3
(1.河北工程大学 数理学院,河北 邯郸 056038;2.邯郸学院 机电学院,河北 邯郸 056005;3.河北工业大学 生物物理研究所,天津 300401)
利用一种常见驱动蛋白(1BG2)和微管蛋白(1TUB)的结晶结构,通过静电计算得到了马达头部和微管蛋白表面的电势分布,并对比晶体结构分析了微管势场对马达头部正电势区域的作用.此外,利用解泊松方程的方法计算了驱动蛋白与微管间的静电相互作用,并发现马达沿微管轴向运动时所处的微管电势场具有周期性,其周期与马达步进周期有很好的一致性.最后,结合计算结果提出了驱动蛋白在静电作用下的运动机制.
驱动蛋白马达;微管;静电相互作用;泊松方程
驱动蛋白(kinesin)是由一系列沿微管运动的马达蛋白超家族(KIF family)构成的,目前已有15个驱动蛋白家族被发现,所有的驱动蛋白马达都有一个共同的特征,即能够高效地利用水解ATP释放的能量来完成沿微管的定向物质输运[1].驱动蛋白马达在细胞内的物质运输、细胞骨架重组、有丝分裂、细胞功能等方面起着至关重要的作用.驱动蛋白的变异或功能缺失,将直接导致记忆功能衰退、神经系统病变、癌症、多囊肾和耳聋等多种疾病,可以说驱动蛋白是生命赖以生存的基础之一[2-4].
双头驱动蛋白马达主要以一种非对称的步进机制(hand-over-hand)向微管正向端前进,而能够很好描述单头驱动蛋白马达运动的是噪声驱动的棘轮运动机制(ratchet mechanism),虽然这2种运动机制已经能够成功解释驱动蛋白个体和协作运动的许多问题[5-6],但是马达化学能转化成机械能的力学过程和影响其运动的详细力学机制仍没有解决.研究表明在一些非马达的蛋白系统里,静电相互作用可以有效操控相关蛋白结合成一个合适的结构[7-8],为了研究影响驱动蛋白运动的力学机制,尤其是微管长程静电相互作用对驱动蛋白运动的操控和影响,本文依据蛋白质数据库中驱动蛋白和微管蛋白的结构信息,通过静电计算软件模拟和解泊松方程计算的方法,得到了驱动蛋白马达头部和微管蛋白的电势分布以及它们间的静电相互作用,并且结合晶体结构分析了静电相互作用对马达关键区域的影响,最后得到了驱动蛋白伴随核苷酸轮换过程和静电相互作用影响的运动机制.
1 材料和计算方法
1.1 蛋白质三维结构数据文件
建模和模拟计算时所有结构信息均来自专业的蛋白质数据库——PDB库[9],具体用到了4种蛋白质晶体结构:1)一种人体内普遍存在的处于ADP结合态的驱动蛋白马达头部结构(PDB:1BG2)[10];2)由α和β微管蛋白(tubulin)二聚体构成的微管蛋白结构(PDB:1TUB)[11];3)2种驱动蛋白马达头部和微管蛋白的复合体结构,其中一种马达头部处于ADP结合态(PDB:2HXH)[12],另外一种马达头部处于ATP结合态(PDB:1IA0)[13].
1.2 计算方法
1.2.1 驱动蛋白和微管蛋白的静电势分布
利用静电计算软件APBS(1.3版)[14],分别计算了驱动蛋白(PDB:1BG2)和微管蛋白(PDB:1TUB)的静电势分布,计算时选用了AMBER力场提供的原子电荷分布和原子半径信息文件,并利用PDB2PQR公共服务平台(http://www.poissonboltzmann.org/docs/calculating/)生成了所需的驱动蛋白和微管蛋白的PQR格式文件.
1.2.2 驱动蛋白与微管间静电相互作用的计算
根据马达微管复合结构(PDB:1IA0和2HXH)给出的信息,利用马达和微管的晶体结构(PDB:1BG2和1TUB)构建了驱动蛋白马达头部1BG2和微管蛋白的复合结构,构建的微管模型中微丝有15条.由于微管蛋白是由α微管蛋白和β微管蛋白2个单体组成的二聚体,所以模建时微管蛋白的长度为7.944 250 9 nm,相邻微丝上的2个紧邻的微管蛋白位错差为1.059 233 4 nm.模拟计算静电相互作用时所参照的是1IA0晶体结构所处的坐标系,马达头部和微管的结构信息都统一到了该坐标系下,计算模型和坐标系如图1所示.
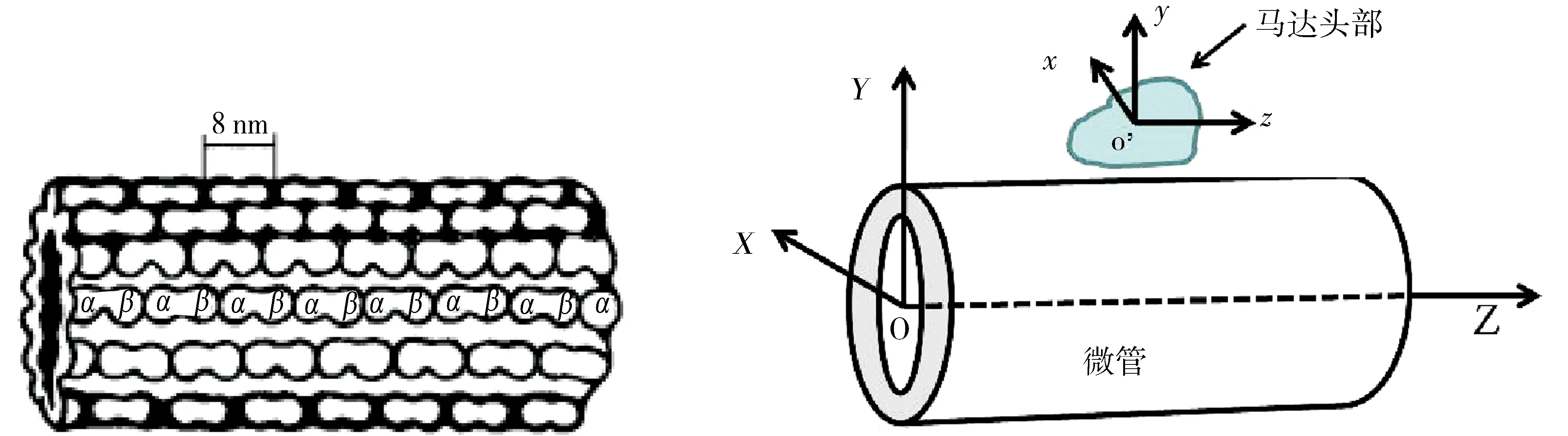
图1 微管结构模型和坐标系Fig.1 Structure model of microtubule and the coordinate system
根据解泊松方程的结果[15],微管周围的电势可由下式给出:
(1)
根据方程(1)可得,在微管上运行的驱动蛋白马达,受到微管电场的静电相互作用可表示为
(2)
2 结果与讨论
2.1 驱动蛋白和微管蛋白的电势分布
通过计算发现驱动蛋白(1BG2)头部的电势呈明显的不对称分布如图2所示,马达头部上半部分呈现明显的负电势分布(浅色区域),下半部分则呈现正电性(深色区域)(图2a),对于微管蛋白在上表面呈现明显的负电势分布,根据物理学同性相斥异性相吸的基本原理,微管表面的电势分布将直接影响马达头部在其势场中运行的构象,尤其是马达头部在扩散搜寻结合位点过程中的构象取向,即在静电相互作用下,电势分布不对称的马达头部朝向微管的一侧将尽量呈现的正电势,而在背向微管的一侧尽量呈现负电势(图2b),这种构象取向与驱动蛋白马达和微管蛋白的复合晶体结构里马达头部取向非常一致.同时,在长程静电相互作用引导下,马达头部通过头部构象的不断变化而逐渐靠近微管上的结合位点(图2c),计算结果表明静电相互作用在引导驱动蛋白马达头部和微管的结合时,可能发挥了重要的调控作用,它使马达头部不断调整到最适宜结合的构象,这与生物系统里蛋白质间结合时的静电操控现象是一致的[16-17].
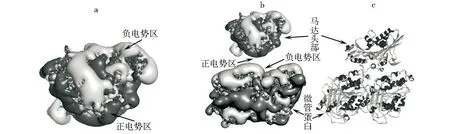
a.驱动蛋白马达头部电势分布(浅色区域为负电势分布区域,深色区域为正电势分布区域);b.马达头部和微管蛋白复合结构表面电势分布;c.马达头部和微管蛋白复合结构[12].图2 马达头部和微管蛋白的复合结构及其电势分布Fig.2 Kinesin head-tubulin complex structure and its electrostatic potential maps
2.2 驱动蛋白马达头部(1BG2)的正电势区域
由于微管表面主要是负电势分布,所以本文主要分析了驱动蛋白运动中起关键作用的正电势分布区域,对比马达头部的电势分布和结晶结构(PDB:1BG2)发现,马达头部正电势主要分布在4个区域如图3所示,具体为:1)Helix-3(α4)、Helix-4(α4)的一部分、Loop-11(L11)(部分)和Loop-12(L12)4个结构区域(图3a),这些区域组成的结构通常被称为开关区域Ⅱ(SwitchⅡ)[12],它们是马达头部与微管表面结合的主要区域,这些区域通过自身构象的变化来调整电荷分布,从而改变与负电势分布的微管间的静电相互作用,导致马达头部和微管的结合或解离,可见开关区域与微管间静电相互作用的变化,对头部在微管结合位点上的状态有直接的影响;2)Loop-7(L7)和Loop-8(L8)2个区域(图3a),L7通过氢键与核苷酸结合位点和2个开关区域(SwitchI、SwitchII)相连,并且L7也是马达能够识别微管结合位点的关键结构,它的构象变化将导致核苷酸结合位点的打开和开关区域的构象变化[18-20],所以微管上关键位点对正电势区域Loop-7的静电相互作用,可能是引起L7构象的变化从而导致马达识别结合位点和头部开启核苷酸轮转的重要保证;3)Helix-6(α6)区域(图3a),与α6直接相连的是马达的颈链(Neck-linker),颈链区域的构象变化对马达头部核苷酸结合位点的开关和马达的动力冲程有重要的作用[21],静电相互作用引起α6区域构象变化可能是开启颈链区域构象变化的关键;4)核苷酸结合位点区域(图3b),正电势分布的核苷酸结合位点更有利于显负电性的ATP的结合,从而有效促进马达头部核苷酸的轮转,保持驱动蛋白沿微管的定向连续步进.
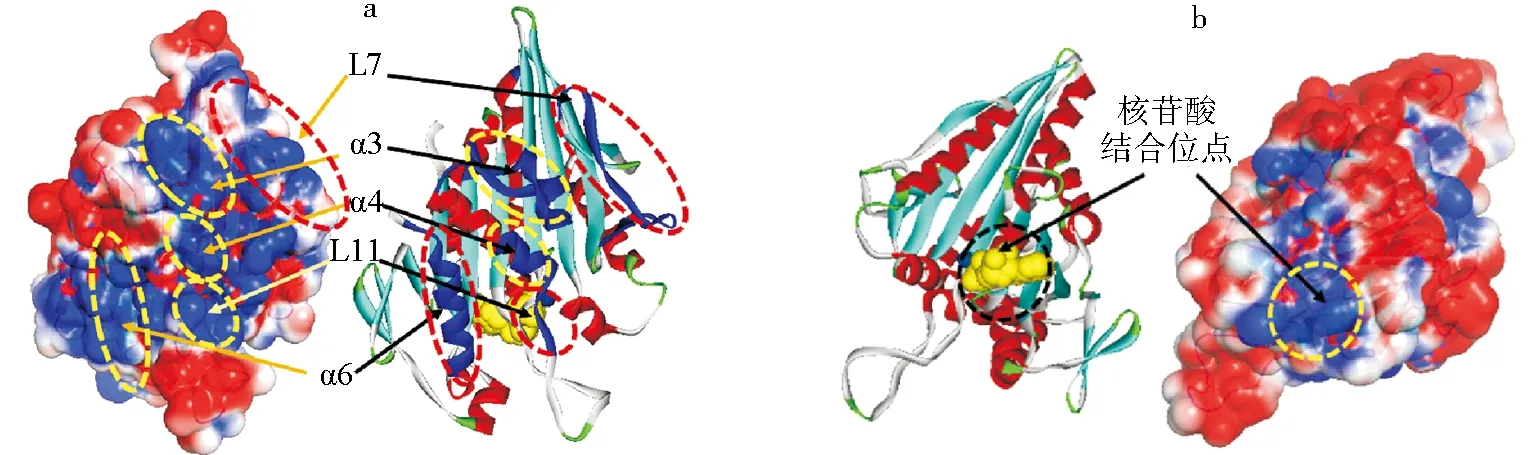
a.马达与微管结合面的相关区域;b.核苷酸结合位点区域.图3 马达头部正电势分布区域和晶体结构对比Fig.3 Comparison of the positive electric potential region and their crystal structure
2.3 驱动蛋白沿微管轴向的静电势能
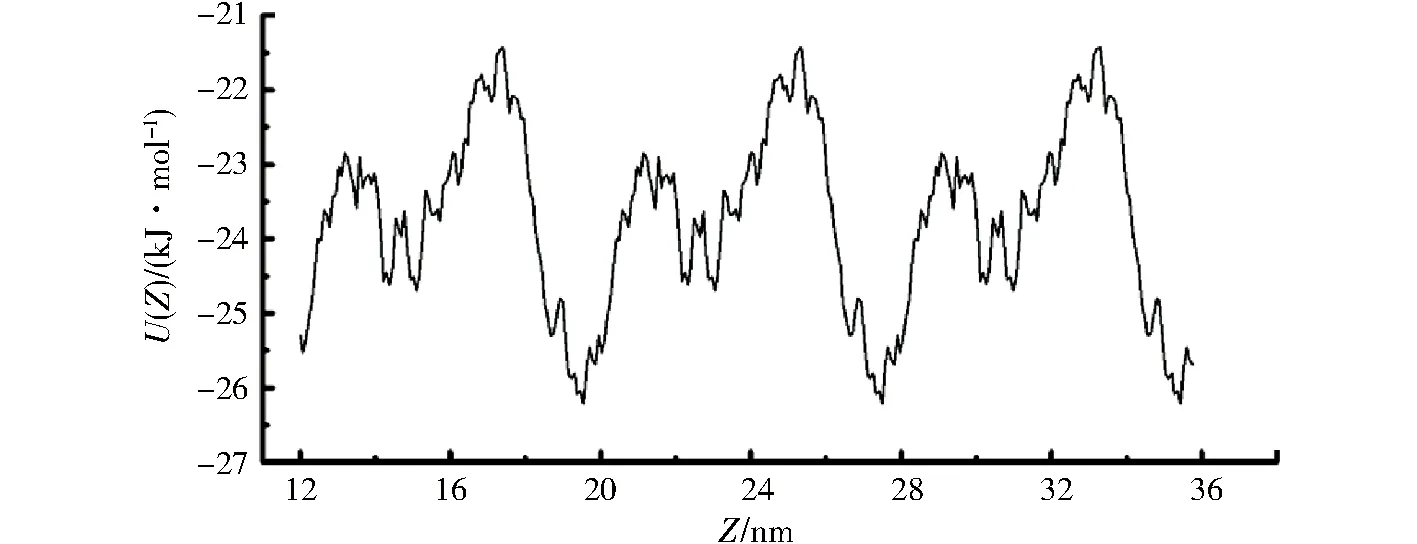
图4 驱动蛋白马达沿微管轴心方向的静电势曲线Fig.4 Electrostatic potential curve of kinesin along the axis direction of microtubule
为了研究长程静电相互作用对马达运动的影响,根据方程(2)模拟计算了马达头部在距离微管表面1.5 nm时沿其轴向的静电相互作用(图4),通过计算发现:首先,驱动蛋白马达与微管间的静电势能为负,即马达头部在扩散运动中始终受到了微管势场的吸引作用,这也意味着在静电相互作用的驱使下,马达在与微管结合位点结合前将不断调整构象,从而使主要的正电区域尽量朝向微管表面,静电相互作用的这种影响已在芽孢杆菌RNA酶和其抑制剂的研究中得到了证实[22],同时相互吸引的静电相互作用也确保了马达头部不能轻易偏离轨道,这也是驱动蛋白沿微管定向输运物质的保证.其次,马达头部沿微管轴向的静电势能是周期性变化的,而且其周期与驱动蛋白马达沿微管定向运动的步长相等(约8 nm),这意味着微管的周期性势场轨道可能是驱动蛋白马达周期性步进一个重要原因.最后,驱动蛋白沿微管轴向的静电势能曲线上有一些势能最低点,并且它们周期性的出现在接近β微管蛋白上的马达结合位点位置,这种能量最低点的出现可能是由马达和微管蛋白带电氨基酸的不均匀分布引起的,而其分布的周期性是由于α微管蛋白和β微管蛋白的结构不同造成的,马达沿微管运动时必将优先趋于稳定的能量最低点,这暗示了马达在扩散运动时尤其是搜索结合位点阶段,结构不同的微管蛋白所产生的周期性静电势场,对马达的运动方向和结合位点的选择起到了重要的指引作用.
2.4 静电作用下的驱动蛋白运动机制
通过计算发现,静电相互作用对驱动蛋白马达沿微管的定向运动有重要的影响,并且该作用与ATP水解释放的化学能向机械能转化的力产生过程协调配合,最终使驱动蛋白实现连续的定向步进,因此可以推断一个配合化学能转化机械能的静电运动机制,如图5,具体为:1)设驱动蛋白马达的头部A和B分别处于ATP和ADP结合态为初始状态(图5a),此时头部A与微管处于紧密结合态,其核酸结合位点的ATP开始水解,而水解释放的化学能将转化成头部A构象变化的机械能,从而引起头部电荷分布的改变和静电相互作用的减小,导致头部A与微管间的结合力减弱,随着磷酸基团的释放,微管与ADP结合态的头部A处于最弱结合态;对于头部B,在微管电势场的作用下,其头部的SwitchII和Loop-7区域将发生构象变化,Loop-7的构象变化将引起头部B核苷酸结合位点打开并释放ADP,同时随着SwitchII区域的构象变化,头部B与微管逐渐趋于紧密结合的空态.2)由于微管电势场与头部B上Helix-6区域的作用,导致该区域构象变化并促使与其相连的颈链区域开始与头部B对接,颈链区域的对接将使马达两头间的自由颈链长度逐渐缩短,从而不断增加两头部间的张力,最终导致处于弱结合态的头部A在颈链张力的作用下逐渐从结合位点上解离(图5b),同时颈链的对接将保持头部B上的核酸结合位点处于开启状态,使头部B迅速结合新的ATP并关闭核苷酸结合位点.3)在颈链张力的作用下,头部A穿越头部B并处于其前面的位置(图5c),由于此时颈链间张力已经不能驱使头部A的运动,根据前面计算结果可以推测静电相互作用此后将扮演重要角色,正是在微管周期性电势场作用下,头部A不断调整自身的构象,使其负电势分布区域背向微管而正电势分布区域面对微管,同时随着微管与马达头部主要结合区域的构象变化,这些区域的电荷分布也将不断变化,最终使头部A识别并与微管结合位点紧密结合(图5d),此时头部A和B的前后位置实现轮换,并恢复与开始阶段相同的核苷酸结合态,马达完成一个约8 nm的步进,此后,马达将重复前面的过程并开始新的步进.
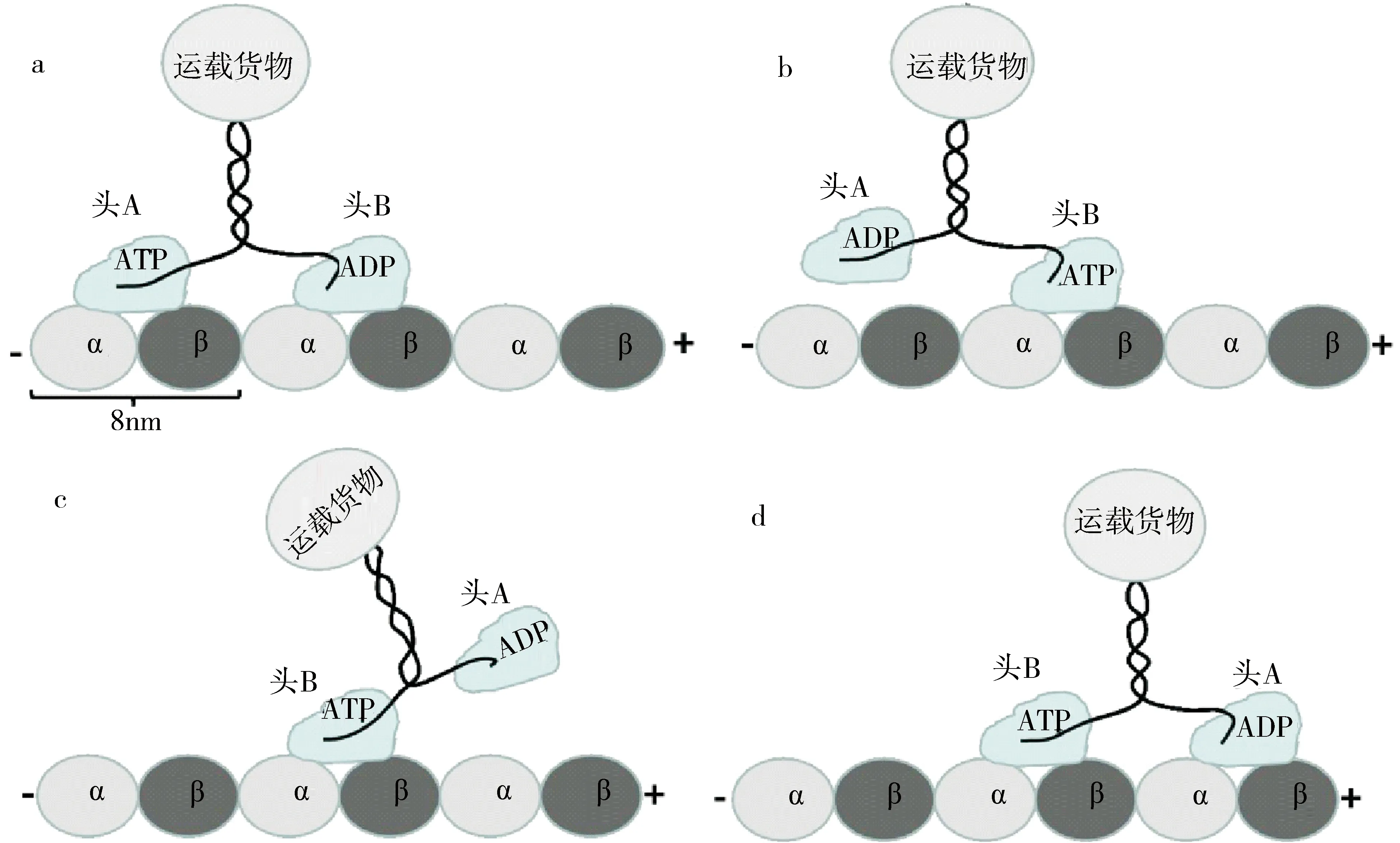
图5 静电作用下驱动蛋白马达的运动机制Fig.5 The electrostatic mechanism of kinesin
3 结论
利用蛋白数据库内分子马达(1BG2)和微管蛋白(1TUB)的结构信息,首先通过静电计算软件计算了二者的电势分布,结果表明马达头部电势呈正负电势非对称性分布,而微管表面主要是负电势分布,它们间的静电相互作用对于调控马达头部与微管结合为点的结合,以及马达在扩散运动时的构象变化发挥着重要的作用.同时对比晶体结构分析了驱动蛋白马达头部的正电势分布区域,发现这些区域都是在驱动蛋白运动和其头部核苷酸水解轮转中起关键作用的区域,这些区域的构象改变将引起自身电荷分布的改变,从而导致与微管间的静电相互作用发生变化,最终影响结合位点的识别、头部与微管的结合或解离、核苷酸的轮换等过程的发生.此外,利用解泊松方程的方法计算了驱动蛋白马达运动过程中与微管间的静电相互作用,结果表明它们间的静电相互作用呈周期性变化,并且其周期与马达步进长度相等,这种周期性的相互作用有效保证了驱动蛋白连续的周期性步进,并且对马达的运动方向和结合位点选择也有关键的指引作用.
[1] HIROKAWA N,NODA Y,TANAKA Y,et al.Kinesin superfamily motor proteins and intracellular transport[J].Nature Reviews Molecular Cell Biology,2009,10(10):682-696.DOI:10.1038/nrm2774.
[2] MORFINI G,SCHMIDT N,WEISSMAN C,et al.Conventional kinesin:Biochemical heterogeneity and functional implications in health and disease[J].Brain Research Bulletin,2016,126:347-353.DOI:10.1016/j.brainresbull.2016.06.009.
[3] HIROKAWA N,TANAKA Y.Kinesin superfamily proteins (KIFs):Various functions and their relevance for important phenomena in life and diseases[J].Experimental Cell Research,2015,334(1):16-25.DOI:10.1016/j.yexcr.2015.02.016.
[4] MYERS S M,COLLINS I.Recent findings and future directions for interpolar mitotic kinesin inhibitors in cancer therapy[J].Future Medicinal Chemistry,2016,8(4):463-489.DOI:10.4155/fmc.16.5.
[5] ISOJIMA H,IINO R,NIITANI Y,et al.Direct observation of intermediate states during the stepping motion of kinesin-1[J].Nature Chemical Biology,2016,12(4):290-297.DOI:10.1038/nchembio.2028.
[6] KANADA R,KUWATA T,KENZAKI H,et al.Structure-based molecular simulations reveal the enhancement of biased brownian motions in single-headed kinesin[J].Plos Computational Biology,2013,9(2):e1002907.DOI:10.1371/journal.pcbi.1002907.
[7] MALEKI M,VASUDEV G,RUEDA L.The role of electrostatic energy in prediction of obligate protein-protein interactions[J].Proteome Science,2013,11(1):1-12.DOI:10.1186/1477-5956-11-S1-S11.
[8] DE JESUS M C,INGLE B L,BARAKAT K A,et al.The role of strong electrostatic interactions at the dimer interface of human glutathione synthetase.[J].The Protein Journal,2014,33(5):403-409.DOI:10.1007/s10930-014-9573-y.
[9] BENMAN H M,WESTBROOK J,FENG Z,et al.The protein data bank[J].Nucleic Acids Research,2000,28(1):235-242.DOI:10.1093/nar/28.1.235.
[10] KULL F J,SABLIN E P,LAU R,et al.Crystal structure of the kinesin motor domain reveals a structural similarity to myosin[J].Nature,1996,380(6574):550-555.DOI:10.1038/380550a0.
[11] NOGALES E,WOLF S G,DOWNING K H.Structure of the alpha beta tubulin dimer by electron crystallography[J].Nature,1998,391(6663):199-203.DOI:10.1038/34465.
[12] KIKKAWA M,HIROKAWA N.High-resolution cryo-EM maps show the nucleotide binding pocket of KIF1A in open and closed conformations[J].Embo Journal,2006,25(18):4187-4194.DOI:10.1038/sj.emboj.7601299.
[13] KIKKAWA M,SABLIN E P,OKADA Y,et al.Switch-based mechanism of kinesin motors[J].Nature,2001,411(6836):439-445.DOI:10.1038/35078000.
[14] BAKER N A,SEPT D,JOSEPH S,et al.Electrostatics of nanosystems:application to microtubules and the ribosome.[J].PNAS,2001,98(18):10037-10041.DOI:10.1073/pnas.181342398.
[15] YAN L.Electrostatic correlations:from plasma to biology[J].Reports on Progress in Physics,2002,65(11):1577-1632.DOI:http://dx.doi.org/10.1088/0034-4885/65/11/201.
[16] ZHANG L Q,BORTHAKUR S,BUCK M.Dissociation of a dynamic protein complex studied by all-atom molecular simulations.[J].Biophysical Journal,2016,110(4):877-886.DOI:10.1016/j.bpj.2015.12.036.
[17] MCCAMMON J A.Darwinian biophysics:Electrostatics and evolution in the kinetics of molecular binding[J].PNAS,2009,106(19):7683-7684.DOI:10.1073/pnas.0902767106.
[18] HIROKAWA N,NITTA R,OKADA Y.The mechanisms of kinesin motor motility:lessons from the monomeric motor KIF1A[J].Nature Reviews Molecular Cell Biology,2009,10(12):877-884.DOI:10.1038/nrm2807.
[19] ATHERTON J,FARABELLA I,YU I M,et al.Conserved mechanisms of microtubule-stimulated ADP release,ATP binding,and force generation in transport kinesins[J].Elife Sciences,2014,3(5):1209-1224.DOI:10.7554/eLife.03680.
[20] ZHENG W J,CHAKRABORTY S.Decrypting the structural,dynamic and energetic basis of kinesin interacting with tubulin dimer in three atpase states by all-atom molecular dynamics simulation[J].Biochemistry,2015,54(3):859-869.DOI:10.1021/bi501056h.
[21] ZHAO Y C,KULL F J,COCHRAN J C.Modulation of the kinesin ATPase cycle by neck linker docking and microtubule binding[J].Journal of Biological Chemistry,2010,285(33):25213-25220.DOI:10.1074/jbc.M110.123067.
[22] WANG L,SIU S W,GU W,et al.Downhill binding energy surface of the barnase-barstar complex [J].Biopolymers,2010,93(11):977-985.DOI:10.1002/bip.21507.
(责任编辑:孟素兰)
Effects of electrostatic interactions on the movement of kinesin
HUANG Yanbin1,GUO Haijun1,LI Jie2,LIU Xiuhong1,JI Qing3
(1.School of Mathematics and Physics,Hebei University of Engineering,Handan 056038,China;2.School of Mechanical and Electric Engineering,Handan College,Handan 056005,China;3.Institute of Biophysics,Hebei University of Technology,Tianjin 300401,China)
Based on the crystal structure of a kinesin motor(1BG2) and tubulin(1TUB),the electric potential distribution on the surface of the motor head and the tubulin is obtained by electrostatic calculation,and the electrostatic interactions between the microtubule and the positive potential regions of kinesin are analyzed by comparing the crystal structures of them.Furthermore,through simulation by solving Poisson's equation,the electrostatic interactions of kinesin along the microtubule have been calculated,the results show that microtubule provides a periodic potential field for motor movement,and the period is consistent with that of the kinesin steps.Then combined with the calculated results, we present the electrostatic mechanism of kinesin movement.
kinesin;microtubule;the electrostatic interaction;Poisson's equation.
2016-12-10
国家自然科学基金资助项目(11505045);河北省自然科学基金资助项目(A2015402035);河北省教育厅资助项目(QN2014134);邯郸市科学技术研究与发展计划项目(1221107079-6;1121103183);邯郸学院课题(15219)
黄艳宾(1981—),男,河北邯郸人,河北工程大学讲师,主要从事分子马达和大分子动力学方面的研究. E-mail:496444091@qq.com
10.3969/j.issn.1000-1565.2017.03.003
Q615
A
1000-1565(2017)03-0237-06
