利用EMA-qPCR建立快速检测猕猴桃溃疡病菌活菌的方法
2017-06-05周大祥殷幼平王中康
周大祥, 殷幼平, 王中康, 熊 书
(1. 重庆三峡学院生命科学与工程学院, 万州 404100; 2. 重庆大学生命科学学院, 重庆市基因功能与调控重点实验室, 重庆 400030; 3. 重庆三峡医药高等专科学校基础医学部, 万州 404120)
实验方法与技术
ExperimentalMethod&Technology
利用EMA-qPCR建立快速检测猕猴桃溃疡病菌活菌的方法
周大祥1,2, 殷幼平2, 王中康2, 熊 书3*
(1. 重庆三峡学院生命科学与工程学院, 万州 404100; 2. 重庆大学生命科学学院, 重庆市基因功能与调控重点实验室, 重庆 400030; 3. 重庆三峡医药高等专科学校基础医学部, 万州 404120)
利用叠氮溴乙锭(ethidium monoazide bromide,EMA)与实时荧光定量PCR技术相结合(EMA-qPCR),建立了一种有效快速检测猕猴桃溃疡病菌活菌的方法。以猕猴桃溃疡病菌ITS序列为检测靶标,菌体经EMA渗透处理,再进行qPCR特异性扩增。结果显示,qPCR检测灵敏度为2 cfu;当EMA的浓度为2.0 μg/mL时,能有效抑制1.0×107cfu/mL经高温灭活的死菌的扩增,对活菌的扩增没有影响。当活菌数在1.0×101~1.0×105cfu范围内,每个qPCR反应体系中活菌数与Ct值呈线性相关(R2=0.988)。不同温度处理活菌菌悬液后用EMA-qPCR检测猕猴桃溃疡病菌的存活情况并与平板计数法进行比较,结果表明待检样品可在4℃和20℃短期保存。对疑似带病猕猴桃材料进行EMA-qPCR检测,结果表明能减少猕猴桃溃疡病菌PCR的假阳性结果。本研究建立的EMA-qPCR方法是一种有效检测猕猴桃溃疡病菌活菌的方法,能有效避免PCR检测实际样品可能造成的假阳性结果。
猕猴桃溃疡病菌; 叠氮溴乙锭; 实时荧光定量PCR; 活菌检测
猕猴桃溃疡病由丁香假单胞杆菌猕猴桃致病变种Pseudomonassyringaepv.actinidiae(Psa)引起,是猕猴桃生产中最严重的病害之一[1]。目前,猕猴桃溃疡病菌的检测主要采用PCR技术,常规的PCR技术不能区分样品中病原菌的生活状态,在对活菌DNA进行扩增时,也会扩增自由状态的DNA或死菌的DNA[2],导致检测出现假阳性,因此急需建立一种快速有效检测待检样品中猕猴桃溃疡病菌的方法。
叠氮溴乙锭(ethidium monoazide bromide, EMA)是一种插入型荧光核酸结合染料,其光解后生成的氮宾化合物能够穿过死菌的细胞膜,与菌体DNA发生不可逆共价结合,因而可抑制死菌中DNA的扩增,但不会抑制细胞膜完整的活菌DNA的扩增[3-5]。EMA-PCR方法仅以活菌DNA分子为检测靶标,克服了传统PCR检测假阳性较高的缺点,使结果更加有效、可靠,已经广泛应用于各种病原微生物的检测[6-7]。本研究拟通过EMA-qPCR对猕猴桃溃疡病菌进行快速检测,分析EMA对PCR检测猕猴桃溃疡病菌死活细菌的影响;同时,初步优化了枝条中猕猴桃溃疡病菌活菌的EMA-qPCR检测体系,为猕猴桃溃疡病的检验检疫提供技术支持。
1 材料与方法
1.1 材料
菌株:猕猴桃溃疡病菌Pseudomonassyringaepv.actinidiae菌株C33从重庆黔江染病猕猴桃枝条上分离,由西南大学植物保护学院提供,本实验室保存。
试剂:叠氮溴乙锭(ethidium monoazide bromide,EMA,Invitrogen)、SYBR®PremixExTaqTMⅡ Mix (TaKaRa)、牛肉膏蛋白胨培养基(BPA)、LB液体培养基。
1.2 方法
1.2.1 猕猴桃溃疡病菌活菌和死菌的制备
从BPA培养基上挑取猕猴桃溃疡病菌,接种到20 mL LB液体培养基中,25℃下250 r/min培养过夜,调整菌悬液浓度为1.0×107cfu/mL。取一半活菌悬液100℃水浴加热10 min,得到死菌悬液。
1.2.2 DNA模板制备
将猕猴桃溃疡病菌接种至LB液体培养基,25℃下250 r/min培养过夜,A600值在0.4~0.8时,根据《新编分子生物学实验指南》提供的方法提取DNA[8]。
1.2.3 猕猴桃溃疡病菌特异性检测引物以及荧光定量PCR反应条件
猕猴桃溃疡病菌的ITS序列特异引物[9]PsaF1(5′-TTTTGCTTTGCACACCCGATTTT-3′)和PsaR2(5′- CACGCACCCTTCAATCAGGATG-3′)由北京华大基因公司合成。扩增目的片段长度为280 bp。PCR反应体系为25 μL: 12.5 μL SYBR PremixExTaqMix,10 mmol/L PsaF1/PsaR2各0.75 μL,模板DNA 2 μL,加超纯水补至25 μL。qPCR反应在CFX96 Real-Time PCR System(Bio-Rad,USA)中进行,PCR反应程序为:94℃预变性2 min;94℃ 15 s,58℃ 30 s,72℃30 s, 30个循环,qPCR扩增完成后随即分析熔解曲线,扩增特异性验证。
1.2.4 荧光定量PCR灵敏度检测
将新鲜培养的猕猴桃溃疡病菌进行稀释,取稀释后的菌液2 μL直接进行荧光定量PCR扩增,同时取对应稀释菌液1 mL涂布BPA平板,25℃培养48~72 h计数,重复3次。
1.2.5 EMA-qPCR条件优化
于黑暗中分别取1 mL猕猴桃溃疡病菌的活菌和死菌悬液(1.0×107cfu/mL)避光加入不同浓度的EMA,避光室温静置5 min,将离心管置于冰上,打开管盖,距离650 W卤钨灯15 cm,持续曝光10 min[10-11],激活EMA,取2 μL EMA处理的菌液进行荧光定量PCR扩增。
1.2.6 死活菌混合体系中EMA-qPCR的选择性扩增
将1.0×107cfu/mL的死菌悬液0.5 mL分别与1.0×1010、5×108、1.0×108、5×107、1.0×107、5×106、1.0×106、5×105、1.0×105、5×104、1.0×104cfu/mL的活菌悬液0.5 mL混合均匀,形成不同比例的死、活菌混悬液,取1 mL混合的菌液按1.2.5的方法,用优化浓度的EMA处理菌悬液,取2 μL菌液进行荧光定量PCR扩增。
1.2.7 EMA-qPCR法和平板计数法比较不同温度处理后猕猴桃溃疡病菌的存活情况
取1 mL浓度为1.0×107cfu/mL的新鲜猕猴桃溃疡病菌活菌悬液,10 000 r/min离心5 min,然后加1 mL的超纯水中混匀,分别进行-20℃冷冻、4℃冷藏和20℃常温各处理24 h、48 h和72 h后,按1.2.5的方法,用优化浓度的EMA处理菌悬液,取2 μL菌液进行荧光定量PCR扩增。同时取相应的菌液涂板,25℃培养48~72 h计数。
1.2.8 EMA-qPCR检测方法的假阳性验证
按1.2.1制备浓度为107、106、105cfu/mL的猕猴桃溃疡病菌死菌溶液,比较常规qPCR法、EMA-qPCR法和平板计数法对灭活猕猴桃溃疡病菌的检测差异,验证EMA-qPCR方法检测是否存在假阳性。
1.2.9 猕猴桃枝干样品中猕猴桃溃疡病菌的EMA-qPCR扩增
将四川、重庆等地采集的10份疑似猕猴桃溃疡病症状的枝干洗净后切成4 mm×4 mm的小块,无菌水冲洗3次,置于无菌试管中用玻璃棒碾碎,随后加入800 μL无菌水浸泡2 h,将上层浸泡液分成3部分,一部分进行6 h增殖培养备用;一部分直接加入终浓度为3.0 μg/mL的EMA,按1.2.5的方法进行静置曝光处理;第三部分浸泡液经热致死处理后再添加3.0 μg/mL的EMA(对照),并按1.2.5的方法进行静置曝光处理,提取DNA,按1.2.3的方法进行PCR扩增。
1.2.10 数据分析
采用SPSS 19.0对试验数据进行t检验,P<0.05表示具有显著性差异。
2 结果与分析
2.1 荧光定量PCR灵敏度
随着qPCR反应体系中猕猴桃溃疡病菌活菌数减少,Ct值逐渐增大,当qPCR反应体系中的活菌数为1 cfu时,荧光定量PCR检测结果为阴性,因此,荧光定量PCR检测灵敏度为2 cfu(图1)。
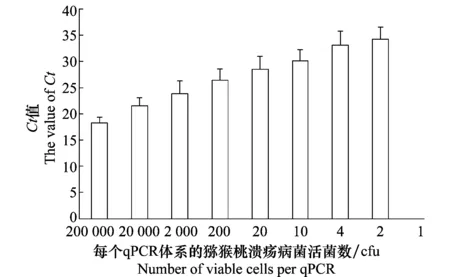
图1 荧光定量PCR检测猕猴桃溃疡病菌的灵敏度Fig.1 The detection limit of qPCR for Psa
2.2 抑制纯培养死菌DNA扩增的最适EMA浓度
对死菌的qPCR检测结果显示,添加EMA的各组Ct值与未添加EMA的对照组相比均显著增加(P<0.05),当EMA终浓度≥2.0 μg/mL时,死菌的PCR扩增被彻底抑制(图2)。低浓度的EMA(≤10 μg/mL)对活菌PCR扩增抑制不显著 (P>0.05)。但是,当EMA≥20 μg/mL时,可显著抑制活菌的PCR扩增(P<0.05)(图2)。可见,抑制死菌DNA扩增的最低EMA浓度(2.0 μg/mL)远远小于影响活菌DNA扩增的最小EMA浓度(20 μg/mL)。因此,我们使用终浓度为2.0 μg/mL的EMA作为纯菌培养后续试验的最佳浓度。
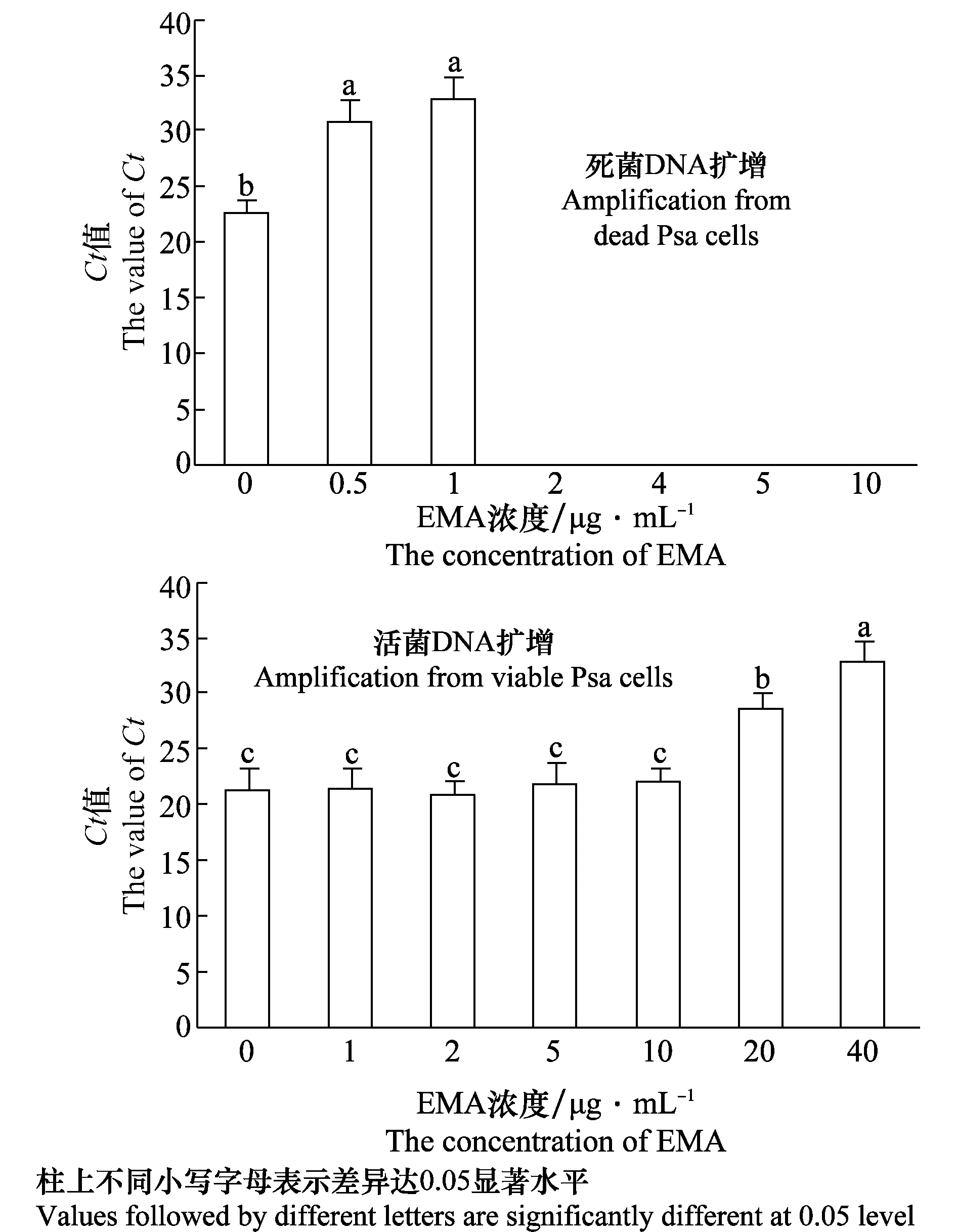
图2 猕猴桃溃疡病菌EMA-qPCR检测体系中EMA浓度优化Fig.2 Optimization of the EMA concentration in EMA-qPCR
2.3 死活菌混合体系中EMA-qPCR选择性扩增活菌
用5.0×106cfu的死菌与不同数量的活菌混合后加入EMA(终浓度为2.0 mg/L)。结果发现,死菌DNA的扩增被彻底抑制,随着活菌数目的降低,Ct值逐渐增加,检测最低限度达到1.0×101cfu (Ct为35.12)(图3a)。在活菌数为1.0×101~1.0×105cfu范围内,其cfu数与Ct值呈线性相关,y=-1.915 8x+37.546,相关系数为R2=0.988(图3b)。此时,通过Ct值可对qPCR中活菌cfu数进行快速定量测定。
2.4 EMA-qPCR检测不同温度处理后猕猴桃溃疡病菌的存活情况
当检测实际样品时,可能需要暂时保存待检样品,为了明确保存温度对待检样品检测结果的影响,设计了-20℃、4℃和20℃ 3个常规保存温度分别保存24、48和72 h,EMA-qPCR检测猕猴桃溃疡病菌的存活情况并与平板培养计数法进行比较。结果表明,-20℃冷冻处理时间越长,EMA-qPCR和平板计数结果越低,处理72 h后,EMA-qPCR检测活菌数为3.7×105cfu,平板计数检测活菌数为4.1×103cfu,两种结果显著差异(P<0.05),显示猕猴桃溃疡病菌低温处理会部分死亡。4℃冷藏和20℃常温放置24、48和72 h后,两种方法的活菌测定结果无显著性差异(P>0.05),且接近对照活菌数,结果表明待检样品可在4℃和20℃短期保存(表1)。

图3 qPCR反应体系中活菌数与Ct值的关系Fig.3 Relationship between the Ct values from EMA-qPCR and the varying number of genomic targets per PCR

处理时间/hTreatmenttime-20℃EMA-qPCR/cfu平板计数/cfu·板-1Platecounting4℃EMA-qPCR/cfu平板计数/cfu·板-1Platecounting20℃EMA-qPCR/cfu平板计数/cfu·板-1Platecounting01.0×107aA1.0×107aA1.0×107aA1.0×107aA1.0×107aA1.0×107aA243.8×106bA6.9×105bB9.9×106aA1.0×107aA9.9×106aA9.9×106aA487.2×105cA6.3×104cB9.9×106aA9.8×106aA1.0×107aA9.8×106aA723.7×105dA4.1×103dB1.0×107aA9.9×106aA9.9×106aA1.0×107aA
1) 同列不同小写字母表示不同时间处理之间差异显著(P<0.05);同行不同大写字母表示相同温度处理下两种方法之间差异显著(P<0.05)。 Different small letters in the same column indicated significant differences between different time treatments (P<0.05); Different capital letters in the same line indicate significant differences between two methods under the same temperature (P<0.05).
2.5 EMA-qPCR检测方法的假阳性验证
以常规qPCR法、EMA-qPCR法和平板计数法检测3个不同浓度的灭活猕猴桃溃疡病菌,验证EMA-qPCR 方法对该菌的测定是否会出现假阳性。结果表明,除了常规qPCR法测定到猕猴桃溃疡病菌外,平板计数法和EMA-qPCR法的测定结果皆为阴性(表2)。表明猕猴桃溃疡病死菌DNA在短时间内也可作为PCR扩增的模板,常规qPCR法不能有效区分猕猴桃溃疡病菌死活菌。而EMA-qPCR法仅以活菌DNA为检测靶标,不会扩增死菌DNA,没有出现假阳性结果,平板计数法进一步证明了EMA-qPCR法的可靠性。

表2 常规qPCR方法、EMA-qPCR方法和平板计数法测定灭活猕猴桃溃疡病菌数的比较1)
1) NA: 阴性。 NA: Negative.
2.6 EMA-qPCR检测疑似猕猴桃溃疡病枝干样品
对四川、重庆等地采集的10份疑似猕猴桃溃疡病枝干样品(浸泡液未经增殖培养)进行测定,结果发现对照EMA-qPCR测定结果均为阴性,说明猕猴桃枝干样品中猕猴桃溃疡病死菌DNA的扩增被完全抑制。来自重庆万州、四川邛崃和四川崇州的样品EMA-qPCR检测结果为阴性,与平板培养结果一致,这3份样品经过培养后,EMA-qPCR检测结果还是阴性(数据未列出),而qPCR检测结果均为阳性,说明这3份样品中的猕猴桃溃疡病菌虽然已经死亡,但DNA序列还没有完全降解。结果表明EMA-qPCR法可以代替平板培养法快速检验猕猴桃溃疡病。

表3 疑似感染猕猴桃溃疡病的枝干样品的检测
3 讨论
普通PCR技术可以快速鉴定细菌,但不能将死菌和活菌区分开。由于EMA具有选择性[12-13],它只能进入细胞壁(膜)不完整的死细胞内,不能进入细胞壁(膜)完整的活细胞。因此,将EMA与qPCR方法相结合,能快速、准确地区分实际样品中的死菌和活菌,有效降低传统PCR检测的假阳性。EMA-qPCR目前已经广泛应用于食源致病微生物[14-16]和医学病原微生物[17]的检测中,但在果树病原菌的检测上报道极少[18]。
本研究首先选用纯化的猕猴桃溃疡病菌进行EMA-qPCR方法研究,结果表明,当EMA终浓度≥2.0 μg/mL时,107cfu/mL浓度死菌的DNA扩增可被彻底抑制。死菌和活菌混合试验表明,检测最低限度为1.0× 101个活菌,在活菌数为1.0×101~1.0×105cfu内,其cfu与Ct值呈线性相关,此时,以Ct值可对qPCR中活菌cfu进行快速定量测定。
在实际样品测定时,可能需要暂时保存待检样品。我们测试了不同温度短时间保存猕猴桃溃疡病菌后其存活情况。结果表明,活菌经过-20℃保存72 h后,EMA-qPCR法检出的活菌数高于平板计数法,原因可能是低温短期处理后的死亡菌体仍能保持细胞壁(膜)完整,使EMA染料无法渗透进入[19-20]。4℃和20℃保存72 h,两种方法的活菌检测结果一致,且接近对照活菌数,但EMA-qPCR法耗时更短,大大缩短了测定时间。综合试验结果表明,猕猴桃溃疡病待检样品可在4℃和20℃短期保存,不会影响测定结果,但不能在-20℃冷冻短期保存。
目前,EMA结合PCR技术检测实际样品中的病原菌报道极少[18]。EMA-qPCR检测疑似猕猴桃溃疡病枝干样品时,因为猕猴桃枝干中未经增殖培养的浸泡液细菌总浓度不会超过1.0×107cfu/mL,同时猕猴桃枝干中的非靶标死菌可能会消耗掉一部分EMA,因此我们选择的EMA浓度为3.0 μg/mL(比纯菌条件下稍高)。结果表明,10份对照样品EMA-qPCR检测结果都是阴性,说明EMA在猕猴桃枝干样品检测中抑制猕猴桃溃疡病菌死菌DNA被扩增的效果比较理想。其中3份样品的EMA-qPCR测定结果与qPCR测定结果不同,与平板培养结果相同,表明EMA-qPCR方法比qPCR法更能克服假阳性的出现,准确反映实际样品的带菌情况,并且比平板培养法检测时间更短。
有时,实际样品中存在的活菌数可能非常少,低于qPCR的最低检测下限,这样EMA-qPCR检测可能会出现假阴性[21]。因此当未经增殖培养的浸泡液检测结果为阴性时,必须经过增殖培养后再次进行EMA-qPCR检测,才能确定实际样品中是否有活菌存在。
[1] Vanneste J, Poliakoff F, Audusseau C, et al. First report ofPseudomonassyringaepv.actinidiae, the causal agent of bacterial canker of kiwifruit onActinidiadeliciosain France [J]. Plant Disease, 2011, 95(10): 1311.
[2] Wang Shishan, Levin R E. Discrimination of viableVibriovulnificuscells from dead cells in real-time PCR [J]. Journal of Microbiological Methods, 2006, 64(1): 1-8.
[3] Gu Weimin, Levin R E. Quantification of viablePlesiomonasshigelloidesin a mixture of viable and dead cells using ethidium bromide monoazide and conventional PCR [J]. Food Biotechnology, 2007, 21(2): 145-149.
[4] 赵素君, 谢晶, 曹冶, 等. 肠出血性大肠杆菌O157∶H7 EMA-PCR检测技术的建立[J]. 中国草食动物科学, 2012, 32(4): 50-53.
[5] Wu Guoping, Chen Suhua, Levin R E. Application of ethidium bromide monoazide for quantification of viable and dead cells ofSalmonellaentericaby real-time loop-mediated isothermal amplification [J]. Journal of Microbiological Methods,2015, 117:41-48.
[6] Luo Gang, Angelidaki I. Analysis of bacterial communities and bacterial pathogens in a biogas plant by the combination of ethidium monoazide, PCR and Ion Torrent sequencing [J]. Water Research, 2014, 60:156-163.
[7] Shi Hui, Xu Wentao, Trinh Q, et al. Establishment of a viable cell detection system for microorganisms in wine based on ethidium monoazide and quantitative PCR [J]. Food Control, 2012, 27(1): 81-86.
[8] 奥斯伯 F,布伦特 R,金斯顿 R E,等.新编分子生物学实验指南[M].颜子颖,王海林 译.北京:科学出版社,1998:78-79.
[9] Rees-George J, Vannestej L, Cornish D A, et al. Detection ofPseudomonassyringaepv.actinidiaeusing polymerase chain reaction(PCR) primers based on the 16S-23S rDNA intertranscribed spacer region and comparison with PCR primers based on other gene regions [J]. Plant Pathology, 2010, 59(3): 453-464.
[10]赵瑜, 李金磊, 董鹏, 等. EMA与PCR结合检测沙门氏菌方法的研究 [J]. 饲料与畜牧科学, 2013, 34(11): 113-115.
[11]Lee J L, Levin R E. Discrimination of viable and deadVibriovulnificusafter refrigerated and frozen storage using EMA, sodium deoxycholate and real-time PCR [J]. Journal of Microbiological Methods, 2009, 79(2): 184-188.
[12]Howard Z R, Abryan C A, Phylip G C, et al.Salmonellaenteritidisin shell eggs: Current issues and prospects for control [J]. Food Research International, 2012, 45(2): 755-764.
[13]Liang Ningjian, Dong Jin, Luo Laixin, et al. Detection of viableSalmonellain lettuce by propidium monoazide real-time PCR [J]. Journal of Food Science, 2011, 76(4): 234-237.
[14]Rudi K, Moen B, Dromtorp S M, et al. Use of ethidium monoazide and PCR in combination for quantification of viable and dead cells in complex samples [J]. Applied and Environmental Microbiology, 2005, 71(2): 1018-1024.
[15]Lee J L, Levin R E. A comparative study of the ability of EMA and PMA to distinguish viable from heat killed mixed bacterial flora from fish fillets [J]. Journal of Microbiological Methods, 2009, 76(1): 93-96.
[16]Inoue H, Takama T, Yoshizaki M, et al. Detection ofLegionellaspecies in environmental water by the quantitative PCR method in combination with ethidium monoazide treatment [J]. Biocontrol Science, 2015, 20(1):71-74.
[17]Seinige D, Krischek C, Klein C, et al. Comparative analysis and limitations of ethidium monoazide and propidium monoazide treatments for the differentiation of viable and nonviable campylobacter cells [J]. Applied & Environmental Microbiology, 2014, 80(7):2186-92.
[18]熊书, 王中康, 卢小林, 等. 柑橘溃疡病菌EMA-PCR快速活体检测技术的建立[J]. 植物保护, 2013, 39(4): 78-84.
[19]Kell D B, Keprelyants A S, Weichart D H, et al. Viability and activity in readily culturable bacteria: a review and discussion of the practical issues [J]. Antonie van Leeuwenhoek, 1998, 73(2): 169-187.
[20]Novak J S, Juneja V K. Detection of heat injury inListeriamonocytogenesScott A [J]. Journal of Food Protect, 2001, 64(11): 1739-1743.
[21]石慧, 程国灵, 翟百强, 等. 泡菜中乳酸菌活菌的EMA结合定量PCR检测方法的建立[J]. 农业生物技术学报, 2013, 21(3): 373-378.
(责任编辑:杨明丽)
Establishment of a method to rapidly detect only viable cells ofPseudomonassyringaepv.actinidiaeby EMA-qPCR
Zhou Daxiang1,2, Yin Youping2, Wang Zhongkang2, Xiong Shu3
(1. College of Life Science & Engineering, Chongqing Three Gorges University, Wanzhou 404100, China; 2. College ofLife Science, Chongqing University, Chongqing Key Lab of Genetic Function and Regulation, Chongqing 400030, China;3. Department of Basic Medicine, Chongqing Three Gorges Medical College, Wanzhou 404120, China)
A method to rapidly detect only viable cells ofPseudomonassyringaepv.actinidiae(Psa) was established by EMA-qPCR. The ITS sequence was used as the target gene for qPCR detection of Psa. Samples were treated with EMA prior to DNA extraction. DNA was then amplified by qPCR to detect only viable Psa cells. The sensitivity of qPCR detection was 2 cfu. 2 μg/mL EMA could completely inhibit the PCR amplification of DNA derived from dead cells with the concentration of 1.0×107cfu/mL, but no inhibition to viable cells. A standard curve was generated relating the number of viable cells to theCtvalues of the EMA-qPCR. A linear range of DNA amplification was observed from 1.0×101-1.0×105cfu genomic targets per PCR. EMA-qPCR method was used to evaluate the survival rate of Psa treated with different temperatures for a short time, and compared with the method of plate counting. The results indicated that samples can be stored for a short time under 4℃ and 20℃. The data of EMA-qPCR detection on kiwifruit field samples indicated that 3 μg/mL EMA could successfully inhibit PCR amplification of DNA from dead bacteria in filed samples. The EMA-qPCR method established in this study can effectively avoid false positive results of Psa detection.
Pseudomonassyringaepv.actinidiae; ethidium monoazide bromide; real-time PCR; detection of viable bacteria
2016-05-21
2016-07-20
重庆市自然科学基金(cstc2016jcyjA2026);重庆市教委科学技术研究项目(KJ1502601);重庆三峡学院校级重点项目
S 436.634.12
A
10.3969/j.issn.0529-1542.2017.03.024
* 通信作者 E-mail: dqzhou79@163.com
