甘薯TD—SSR—PCR反应体系的优化
2017-05-30南文卓郑丹炜朱国鹏张小贝
南文卓 郑丹炜 朱国鹏 张小贝

摘 要 为探索甘薯最佳TD-SSR-PCR体系,利用L16(43)正交设计研究2×PCR Mix、引物、模板DNA等主要影响因素的适宜浓度,并在此基础上对扩增程序和聚丙烯酰胺凝胶电泳上样量进行优化。结果表明,优化后的TD-SSR-PCR反应体系包括:10 μL 2×PCR Mix、100 ng模板DNA、0.4 μmol/L引物,1 μL甘油,总体积20 μL;优化后的扩增程序为:94 ℃预变性4 min,94 ℃变性45 s、Tm+5 ℃~Tm-5 ℃(每循环退火温度下降0.5 ℃,Tm选用一对引物中的较小Tm值)退火30 s(退火时间因扩增片段大小而异)、72 ℃延伸1 min共20个循环,94 ℃变性45 s、Tm-5 ℃退火30 s、72 ℃延伸1 min共15个循环,最后72 ℃延伸7 min,4 ℃保存;聚丙烯酰胺电泳上样量以1.5~2 μL为宜。在此条件下,利用引物Z37对10份甘薯材料进行PCR扩增,得到的条带清晰、多态性高,表明此条件适用于甘薯的TD-SSR-PCR反应体系。
关键词 甘薯;TD-SSR-PCR;正交设计;2×PCR Mix;最佳上样量
中图分类号 Q945 文献标识码 A
Optimization of TD-SSR-PCR System on Sweet Potato
NAN Wenzhuo,ZHEGN Danwei,ZHU Guopeng*,ZHANG Xiaobei
College of Horticulture and Landscape Architecture,Hainan University,Haikou, Hainan 570228, China
Abstract To explore the best TD-SSR-PCR system on sweet potato, an L16(43)orthogonal design was used to study the suitable concentration of the main influence factors such as 2×PCR Mix, primers and template DNA. And on this basis, the amplification process and the best sample amount of polyacrylamide gel electrophoresis were optimized. The results showed that the optimized 20 μL TD-SSR-PCR reaction system included: 10 μL 2×PCR Mix, 100 ng template DNA, 0.4 μmol/L primers, 1 μL glycerol, total volume is 20 μL; The optimized PCR amplification system started at initial denaturation for 4 min at 94 ℃, followed by 20 cycles of denaturation for 45 s at 94 ℃, anneal for 30 s(Annealing time varied depending on the DNA fragment size)at Tm+5~Tm-5 ℃(annealing temperature decreased 0.5 ℃ per cycle, chose the smaller Tm of a pair of primers as the using one), extension for 1 min at 72℃, then 15 cycles of denaturation for 45 s at 94 ℃, anneal for 30 s at Tm-5 ℃, extension for 1 min at 72 ℃, the amplification was completed after extension for 7 min at 72 ℃, finally stored at 4 ℃. The best sample amount of polyacrylamide gel electrophoresis was 1.5-2 μL. Under this condition, clear banding pattern with high polymorphic was obtained by using primer Z37 to amplify 10 sweet potato materials. It indicated that this condition was suitable for TD-SSR-PCR of sweet potato.
Key words sweet potato; TD-SSR-PCR; orthogonal design; 2×PCR Mix; the best sample amount
doi 10.3969/j.issn.1000-2561.2017.01.016
甘薯[Ipomoea batatas(L)Lam.]作為主要粮食作物,不论在发达国家还是发展中国家都具有重要意义[1]。甘薯为同源六倍体作物,染色体数量多,遗传背景复杂[2],且繁殖方式为无性繁殖,严重制约甘薯育种和生产的发展。
SSR(Simple sequence repeat)即简单序列重复,通常又称为微卫星,是一类主要由1~6个碱基组成的基序串联重复而成的DNA序列。每个SSR位点两侧一般是相对保守的单拷贝序列,根据这个特点可设计一对特异引物来扩增不同重复次数的SSR序列。SSR标记为共显性标记,多态性高,并可区分种质资源的纯合和杂合状况。另外SSR在实验中对DNA模板的用量和质量要求相对较低,结果重复性好[3],相比于AFLP、RAPD、ISSR等分子标记技术具有更高的可靠性[4]。
对甘薯材料进行PCR反应体系优化方面的研究已有报道。宋吉轩[5]和张世安[6]建立了甘薯ISSR-PCR反应体系,雷剑[7]、蒲志刚[8]分别优化了甘薯RAPD-PCR和AFLP-PCR反应体系,而关于甘薯SSR-PCR反应体系的优化则未见报道。多数已报道的其他物种[9-11]的SSR-PCR反应体系优化都是根据单个影响因素进行不同梯度间的组合从而选择最适合的反应体系,关于现今广为应用的商品化PCR Mix体系优化研究则相对较少[12-13]。不同研究对象从SSR扩增到最后的电泳,每个步骤都会影响到最终效果。本研究拟利用正交试验设计对甘薯SSR-PCR反应体系(2×PCR Mix、引物和模板3个因素4个水平)进行优化,并在此基础上对SSR-PCR反应程序、聚丙烯酰胺凝胶电泳上样量继续优化,以期建立一套适用于甘薯TD-SSR-PCR的反应体系。
1 材料与方法
1.1 材料
10份甘薯材料取自海南大学海甸校区试验基地。
2×PCR Mix为东盛生物产品,其组分包括100 mmol/L KCL、20 mmol/L Tris-HCL、3 mmol/L MgCL2、400 umol Dntps、0.1 U/μL TaqDNA聚合酶、溴酚蓝、其他增强剂与稳定剂。
试验引物ZY5序列Fwd:TCGTCACTTTCTCTC
TCCTG;Rev:GCTCTCCTCCATCTCTTCTG,重复序列为(AG)10。
结论验证引物Z37序列Fwd:GGCGACTGTAA
TGTGGTGAA;Rev:CGGGAGGTATCTTGGATTGA,重复序列为(CTGCTC)3。引物均由北京三博远志生物技术有限责任公司合成。
Marker为东盛生物公司的DS 2 000。
1.2 方法
1.2.1 DNA的提取与检测 采用CTAB法提取基因组DNA,用NanoDrop检测DNA的浓度和质量,并用琼脂糖跑胶。将样品稀释到100 ng/μL后,置于4 ℃冰箱中保存备用。
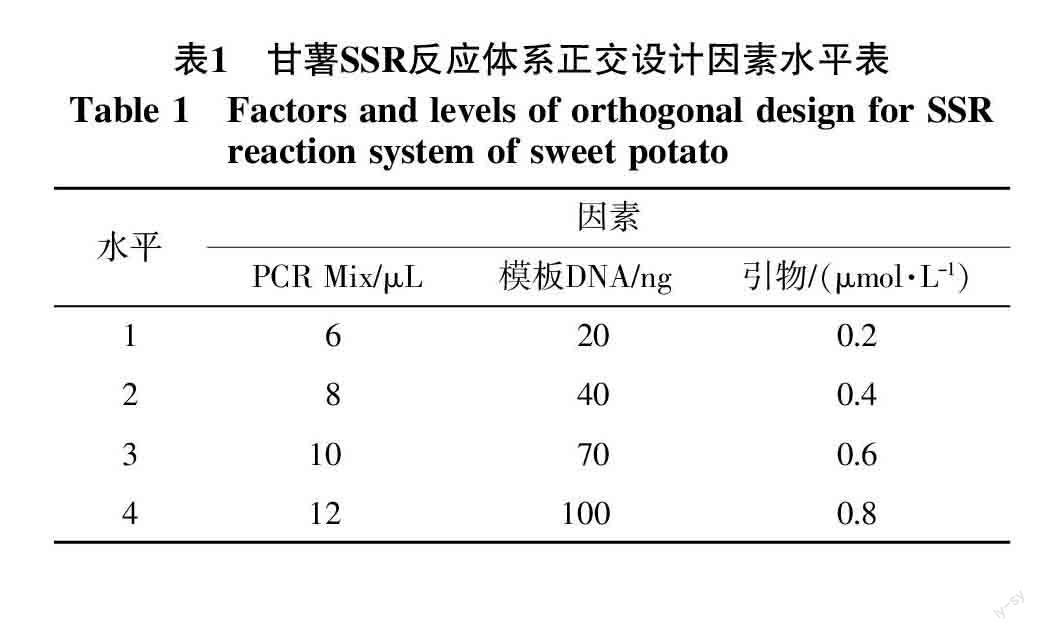
1.2.2 SSR-PCR扩增体系优化 对编号16的甘薯材料设计3因素、4个水平的L16(43)正交表(表1、2),选择最佳SSR扩增体系。SSR扩增在Heal Force T960 PCR仪上进行。
1.2.3 甘薯SSR-PCR方法的选择 对优化后的体系分别进行梯度PCR(G-PCR)和降落PCR(TD-PCR)。G-PCR反应程序:94 ℃预变性4 min;94 ℃变性45 s,55 ℃退火30 s(55 ℃为预试验中确定的最佳退火温度),72 ℃延伸1 min,共32个循环;然后,72 ℃延伸7 min,4 ℃保存。TD-PCR反应程序:94 ℃预变性4 min,94 ℃变性45 s、Tm+5 ℃~Tm-5 ℃退火30 s(每循环退火温度降低0.5 ℃,Tm选用一对引物中的较小Tm值)、72 ℃延伸1 min共20个循环,94 ℃变性45 s、Tm-5 ℃退火30 s、72 ℃延伸1 min共12个循环,最后72 ℃延伸7 min,4 ℃保存。
扩增产物在6%非变性聚丙烯酰胺凝胶上检测,每孔点样4 μL。所用电泳仪为北京六一电泳仪厂的DYCZ-30C型电泳仪,1.0 mm 40齿梳子,100 V恒压电泳2 h,银染显色,照相保存。
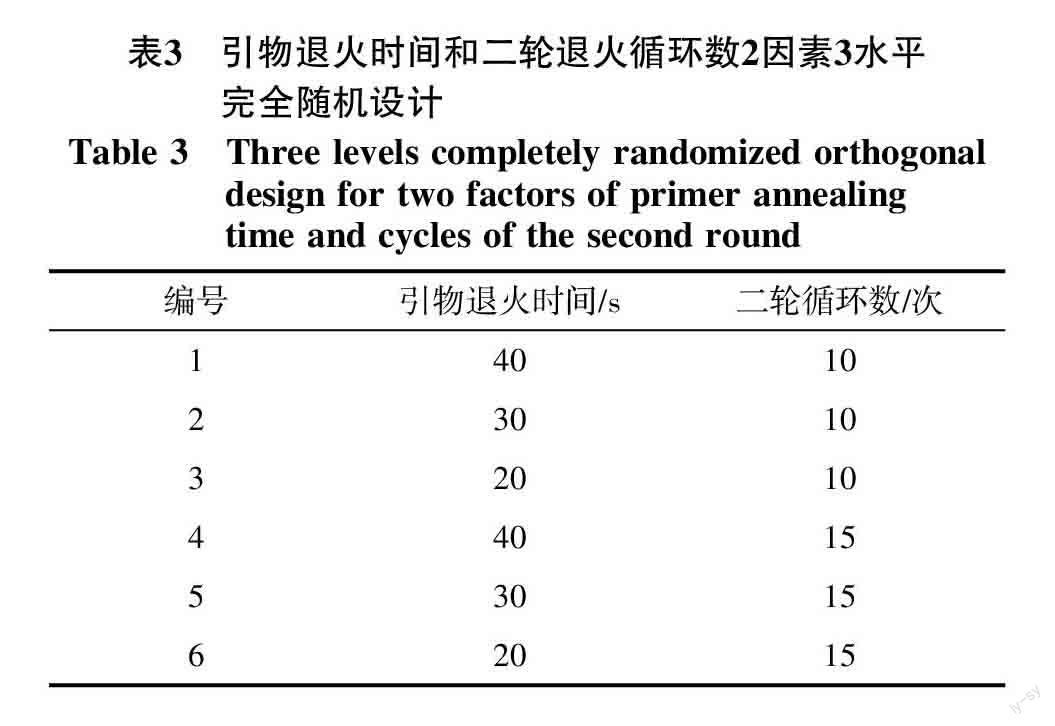
1.2.4 TD-SSR-PCR反应程序的优化 以一对上下游引物的较低Tm±5 ℃值为范围对10份甘薯材料进行TD-PCR。对退火时间和二轮循环次数(一轮循环退火温度渐降,二轮循环退火温度不变)2个因素设置3个水平完全随机设计,选择最优TD-SSR-PCR組合。
1.2.5 聚丙烯酰胺凝胶电泳上样量的优化 利用优化后的TD-SSR-PCR最佳反应体系对上样量进行优化,共设置5个梯度,分别为1、1.5、2、2.5、3 μL,选择最佳上样量。
2 结果与分析
2.1 正交设计SSR-PCR扩增结果分析
正交设计SSR-PCR反应体系扩增见图1。从图1可看出,16个组合都可以扩增出条带,其中第1、2、5、6、9、13组合扩增条带较为清晰但敏感性差,且每组相同PCR Mix梯度的条带随着DNA含量的增加逐渐变强,说明模板DNA为PCR扩增体系主要影响因素。其余组合敏感性较好,尤其第12组合扩增条带最为清晰,为本研究最优组合,即20 μL PCR体系包含:10 μL PCR Mix、100 ng模板DNA、0.4 μmol/L引物,5%甘油,其余加ddH2O补齐。
2.2 G-PCR与TD-PCR结果比较
G-PCR与TD-PCR的扩增结果见图2。由图2可看出,TD-PCR较G-PCR特异性更高,但部分条带颜色较浅。考虑到试验结果可靠性,TD-PCR更适合SSR的扩增,条带深浅则进一步优化。
2.3 不同TD-SSR-PCR程序扩增结果分析
不同TD-SSR-PCR程序对SSR扩增的影响见图3。由图3可看出,在一定循环数和退火时间范围内,循环数与PCR扩增条带强度正相关,循环数越多,条带越强;特异性扩增则与退火时间成负相关,退火时间越短,特异性越强。图3中的组合1,由于其退火时间较组合2、3长,非特异性扩增较多,组合2和3虽然特异性较强,但产物量较少,条带较浅。相较而言,组合4~6扩增产物量较多,特异性与非特异性扩增也可以区分,其中组合5、6由于退火时间的缩短,有效抑制了非特异性条带的扩增,使条带更加清晰易读,为较适宜TD-SSR-PCR组合。
2.4 聚丙烯酰胺凝胶不同上样量的电泳结果分析
不同上样量对聚丙烯酰胺凝胶电泳的影响见图4。由图4可知,上样量的多少几乎不影响特异性和非特异性条带的区分,但是在上样量为1.5~2 μL时,泳道背景浅且原本堆积成片的较小片段也可以被分离成条,使条带更加清晰易读。因此,聚丙烯酰胺电泳建议上样量为1.5~2 μL。
2.5 TD-SSR-PCR优化体系的验证
引物Z37的TD-SSR扩增结果见图5。由图5可看出,根据优化结果利用引物Z37进行TD-SSR-PCR扩增时,退火30 s和20 s都可扩增出条带,但退火30 s时较大片段更加清晰。与引物ZY5更适合的20 s之所以造成差异可能是扩增片段大小不一,扩增片段大则所需退火时间长,反之则短。
3 讨论
在优化PCR反应条件时需考虑到各因素对反应的影响。梁玉琴等[12]在优化柿树反应体系中发现影响PCR反应最显著的因素是Mix,而本研究中模板DNA含量为最主要影响因素,可能与柿树为乔木而甘薯为草本植物有关。李玉恒等[14]对大鼠RNA的扩增结果表明,TD-PCR省略了G-PCR摸索最适退火温度的过程,且其灵敏度和特异性均高于G-PCR;张瑞强等[15]发现相同的反应体系,TD-PCR更有益于提高PCR特异性,这些观点在本研究中也得到验证。
本研究证明TD-PCR是甘薯PCR扩增较好的选择,但其起始退火温度的升高在提高PCR扩增特异性的同时,也提高了引物结合难度,降低了扩增效率。一般普通PCR的扩增30个循环即可,但本研究同样循环数的TD-PCR产物却较少,电泳条带较浅。故使用TD-PCR时应根据试验需要适当增加循环数,得到足够产物量。
本研究预试验发现,PCR的优化有时很难区分设计的各组合的优劣,对此建议:①优化PCR反应体系,可先利用稍低的退火温度,温度太高可能某些特异性的条带也消失,条带结果无二,难以区分;温度稍低虽然有非特异性条带产生,但容易区分组合优劣。②优化PCR退火时间,要根据体系中的酶延伸速度和扩增片段大小适当选择,然后根据结果决定是否继续优化。③优化聚丙烯酰胺凝胶电泳最佳上样量,参考本研究设计梯度,择优选用。本研究中,3 μL上样量也可读带,但泳道背景深,若增加样本数量,则整个电泳图会显得杂乱。反之,1.5~2 μL时泳道背景清亮,一目了然,且长度接近的较小片段也可很好地区分。究其原因,进行SSR-PCR的扩增最重要的是引物,特异性高的引物不用繁琐的优化也可得到漂亮且可靠的条带,特异性低的引物则需要一步步的优化以达到理想的效果。
4 结论
利用不同分子标记对不同物种进行扩增时使用的反应体系各有差异,对所研究物种建立稳定的PCR反应体系是深入分子研究的基础。本研究建立了适用于甘薯材料的20 μL TD-SSR-PCR反应体系:10 μL 2×PCR Mix、100 ng模板DNA、0.4 μmol/L引物,1 μL甘油,其余加ddH2O补齐;优化后的反应程序为:94 ℃预变性4 min,94 ℃变性45 s、Tm+5 ℃~Tm-5 ℃(每循环退火温度下降0.5 ℃,Tm选用一对引物中的较小Tm值)退火30 s(使用DNA聚合酶聚合速度约1 kbp/min,退火时间因DNA片段大小而异)、72 ℃延伸1 min共20个循环,94 ℃变性45 s、Tm-5 ℃退火30 s、72 ℃延伸1 min共15个循环,最后72 ℃延伸7 min,4 ℃保存;聚丙烯酰胺电泳上样量以1.5~2 μL为宜。
参考文献
[1] 马剑凤, 程金花, 汪 洁, 等. 国内外甘薯产业发展概况[J]. 江苏农业科学, 2012(12): 1-5.
[2]唐 茜, 何凤发, 王季春, 等. 甘薯SRAP遗传图谱构建及淀粉含量QTL初步定位[J]. 西南大学学报(自然科学版), 2010, 32(6): 40-45.
[3] 方宣钧, 无为人, 唐纪良. 作物DNA标记辅助育种[M]. 北京: 科学出版社, 2000.
[4] Schlotterer C. Opinion: The evolution of molecular markers-just a matter of fashion[J]. Nature Reviews Genetics, 2004, 5(1): 63-69.
[5] 宋吉轩, 雷尊国, 黄 团, 等. 甘薯ISSR-PCR反应体系的优化[J]. 贵州农业科学, 2009(11): 25-26, 29.
[6] 张安世, 张利民, 刘 莹. 均匀设计优化甘薯ISSR-PCR反应体系[J]. 焦作师范高等专科学校学报, 2015(1): 66-68.
[7] 雷 剑, 杨新笋. 甘薯RAPD-PCR反应条件的优化[J]. 湖北农业科学, 2009(10): 2 343-2 346.
[8] 蒲志刚, 唐 静, 钟昌松, 等. 甘薯AFLP分子标记体系建立[J]. 分子植物育种, 2006(S1): 40-44.
[9] 謝文刚, 张新全, 彭 燕, 等. 鸭茅SSR-PCR反应体系优化及引物筛选[J]. 分子植物育种, 2008(2): 381-386.
[10] 李亚利, 陈书霞, 孟焕文, 等. 利用正交设计优化黄瓜的SSR-PCR反应体系[J]. 西北农业学报, 2008(3): 280-284.
[11] 范小宁, 林 萍, 张盛周. 油茶SSR-PCR反应体系的优化研究[J]. 安徽农业科学, 2011(23): 14 098-14 102.
[12]梁玉琴, 李芳东, 傅建敏, 等. 正交设计优化柿属植物SSR-PCR反应体系[J]. 经济林研究, 2011(4): 17-22.
[13] 李 季, 黄天带, 华玉伟, 等. 多重PCR技术在橡胶树转基因分子鉴定中的应用[J]. 热带作物学报, 2014, 35(5): 882-889.
[14] 李玉恒, 赵明慧, 赵紫阳, 等. 利用梯度PCR和降落PCR扩增CIRP基因的比较[J]. 黑龙江八一农垦大学学报, 2011(5): 46-49, 75.
[15] 张瑞强, 繁 萍, 张红见, 等. 利用降落PCR扩增KMT-1基因[J]. 青海大学学报(自然科学版), 2010(2): 1-3.
