山蚂蝗属植物遗传多样性的AFLP分析
2017-05-30王春梅任健兰平秀马向丽李阳春白昌军徐立
王春梅 任健 兰平秀 马向丽 李阳春 白昌军 徐立


摘 要 为探讨山蚂蝗属植物遗传多样性和亲缘关系,利用扩增片段长度多态性(AFLP)标记技术对46份山蚂蝗属植物种质进行遗传多样性分析,从64对引物中筛选出条带清晰、多态性高的2对引物E-AAC/M-CAA、E-AAG/M-CAA,共扩增出172条谱带,其中154条为多态性带,多态性检出率为89.53%,表明在分子水平上山蚂蝗属植物遗传多样性丰富。采用离差平方和法将46个供试材料分为6类,聚类结果与《中国植物志》的分类体系相吻合;其中,卵叶山蚂蝗和糙伏山蚂蝗可能也属于三点金亚属,卵叶山蚂蝗亲缘关系离大叶山蚂蝗较近,糙伏山蚂蝗离假地豆较近,这个结论可以为中国植物志的修订提供一个依据;引种材料CIAT350(29)与其他卵叶山蚂蝗的亲缘關系较远。总体来看,聚类结果与地理来源没有必然联系。此外,从本实验来看,AFLP技术扩增效率高,信息量大、结果稳定、重复性好,可用于山蚂蝗属植物的遗传多样性分析和鉴定。
关键词 山蚂蝗属;AFLP;遗传多样性;聚类分析
中图分类号 S567;S54 文献标识码 A
Abstract In order to classify the genetic diversity and genetic relationship of Desmodium species, 46 Desmodium Desv. lines were studied using amplified fragment length polymorphism(AFLP)markers. Two primer combinations (E-AAC/M-CAA, E-AAG/M-CAA)with higher polymorphism were selected from 64 combinations. A total of 172 AFLP markers were generated from the selected two primer combinations and of which, 154 were polymorphic (89.53%), indicating that the genetic diversity of Desmodium was abundant. According to AFLP marker data, the experimented 46 materials were clustered into 6 groups by using NTSYS software, and the cluster analysis results were consistent with the classification system published in-the Chinese flora. D. ovalifolium and D. strgillosum may also be classified to the Subgen. Sagotia(Duchass. et Walpers)Baker. D. ovalifolium and D. strgillosum was closely related, and similar results were observed between D. strgillosum and D. heterocarpon. In contrast, relationships between the introduced species-CIAT350(29)and other D. ovalifolium materials were far. Overall, there was not much relationship between the clustering results and the geographical origin. Regarded to AFLP technique, it is noted in efficiency and replicate, stable results as well as more information, which contributes to the genetic diversity analysis and identification of Desmodium species.
Key words Desmodium Desv; AFLP; genetic diversity; clustering analysis
doi 10.3969/j.issn.1000-2561.2017.03.022
山蚂蝗属(Desmodium Desv.)为豆科碟形花亚科一年生或多年生草本或灌木植物。全世界约350~450种,多分布于亚热带和热带地区,我国有27种5变种,大部分布于西南经中南部至东南部,仅1种产陕、甘西南部[1]。目前,对山蚂蝗属植物的药用价值[2-8],细胞学[9-10],饲草与饲料[11-14]等方面研究较多,但在山蚂蝗属植物遗传多样性方面的研究较少,特别是山蚂蝗属植物从广义山蚂蝗属中独立出来后,在分子评定方面更是鲜有报道。
扩增片段长度多态性(Amplified Fragment Length Polymorphism,AFLP)是荷兰科学家Zabeau[15]和Vos[16]于1992年建立起来的一种检测DNA多态性的新方法,其原理是基于PCR技术选择性扩增基因组DNA限制性酶切片段。AFLP综合了RFLP和RAPD的优点,既具有RFLP的稳定性和PCR反应快速、灵敏的特点,又具有RAPD的方便性,同时还克服了RFLP操作繁琐,周期长和RAPD重复性较差的缺点[17-21],自问世以来就被广泛地用于分类和系统发育方面的研究,在鉴定与评估种质资源方面也有广泛的应用前景[22-31]。
因此,本研究采用AFLP分子标记技术,对山蚂蝗属植物的5个国内种和2个引进种共计46份材料进行了遗传多样性和亲缘关系的分析,旨在为山蚂蝗属植物的分类提供分子水平的证据。
1 材料与方法
1.1 材料
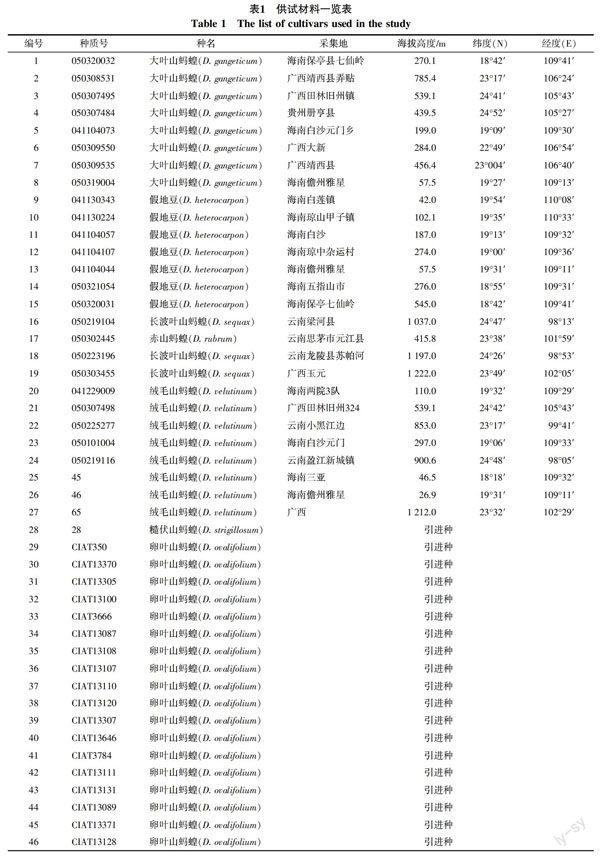
本试验选用 27份国内的野生山蚂蝗属植物种质和19份国外引进的山蚂蝗种质,共46份,采集信息见表1。种质保存在中国热带农业科学院热带作物品种资源研究所牧草中心,并在中国热带农业科学院热带作物品种资源研究所资源圃内少量种植。每份种质选10粒经过发芽处理的种子,分别播种于大棚的花盆里。3片真叶后开始采样提取DNA。
1.2 方法
1.2.1 DNA提取 选择生长良好无病虫害感染的植物嫩叶提取DNA,DNA提取采用改进的SDS法[32]。DNA浓度和纯度用紫外分光光度计检测,OD260/OD280比值在1.6~1.8之间,OD260/OD230的比值都大于2.0,表明所提的DNA纯度高,可用于AFLP的操作。将DNA浓度稀释至50 ng/μL,-20 ℃保存备用。
1.2.2 引物筛选 先用差异性较大的2个种(卵叶山蚂蝗和大叶山蚂蝗)对64对引物进行了筛选。选出了8对主带在5条或5条以上而且比较清晰的引物。然后对这8对引物进行二次筛选,最终选择了2对种间主带较多、种内差异较大而且较清晰的引物E-AAC/M-CAA、E-AAG/M-CAA,对所有供试材料进行选择性扩增。
1.2.3 AFLP分析
(1)限制性酶切与连接。酶切体系为25 μL,包含DNA模板4 μL,EcoRⅠ/MseⅠ 2 μL,10×Reaction buffer 2.5 μL,ddH2O 16.5 μL,混匀后37 ℃水浴8 h。70 ℃水浴15 min。再加入Adapter 1 μL,10 mmol/L ATP 2.5 μL,T4 Ligase 1 μL,18 ℃水浴连接过夜。
(2)预扩增。反应总体系为25 μL,包含模板-DNA 2 μL,Pre-ampmix 1 μL,dNTPs 0.5 μL,10×PCR buffer 2.5 μL,Taq DNA polymease 0.5 μL,ddH2O 16.5 μL,混匀后37 ℃水浴8 h。70 ℃水浴15 min。再加入Adapter 1 μL,10 mmol/L ATP 2.5 μL,离心混匀后用以下程序进行PCR扩增:94 ℃ 2 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 80 s,25个循环;72 ℃ 5 min;4 ℃保存。预扩增产物稀释50倍后于-20 ℃保存备用。
(3)选择性扩增反应。反应总体系为25 μL,包含稀释后的预扩增产物2 μL,EcoRⅠ和MseⅠ引物各1 μL,dNTPs 0.5 μL,10×PCR buffer 2.5 μL,Taq DNApolymease 0.5 μL,ddH2O 17.5 μL,离心混勻后用以下程序进行扩增:94 ℃ 2 min;94 ℃ 30 s,65 ℃ 30 s,72 ℃ 80 s,12个循环,每循环降低0.7 ℃;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 80 s,30个循环;72 ℃ 5 min;4 ℃保存。
(4)扩增产物检测。选择扩增产物中加入等体积的loading buffer,95 ℃变性5 min后立即置于冰浴中冷却。在80 W下用6%的聚丙烯酰胺凝胶电泳2 h,最后银染检测扩增产物。
1.3 数据处理
对扩增产物的电泳结果采用“0-1”系统记录谱带位置,统计多态性带谱带百分比。采用NTSYS分析软件进行数据处理与聚类分析。
2 结果与分析
2.1 多态性分析
采用2对引物,对46份供试材料进行检测,部分扩增结果见图1、2。2对引物共扩增出172条谱带,其中具有多态性的条带为154条,多态性检出率为89.53%。表明在分子水平上,山蚂蝗属植物遗传多样性丰富。同时说明,用AFLP标记揭示山蚂蝗属植物不同基因型的遗传多样性效率较高。2对引物中,引物组合E-AAC/M-CAA扩增的条带较多,共113条,有7条共带,106条多态性条带,其多态性检出率较高,为93.81%;引物组合E-AAG/M-CAA扩增的条带相对较少,共59条,其中有11条共带,48条多态性条带,多态性检出率为81.36%(表2)。
2.2 聚类分析
利用NTSYS进行聚类分析,结果见图3。聚类结果表明,46份山蚂蝗属植物野生种质资源种间遗传距离明显,种内遗传多样性也较丰富。在阈值为0.77时,将46份山蚂蝗属植物分为6大类。第一类中,大叶山蚂蝗与赤山蚂蝗聚在了一起,各为1个亚类。8份大叶山蚂蝗又分为4个组,其中3份海南(1、5、8)的种质首先聚在一起;贵州册亨(4)和广西靖西(2)的聚为第2个组;另一份广西靖西(7)和广西大新(6)聚为第3组,较其他种质来说,这一组的两份种质的相似系数相对较高,亲缘关系较近;广西田林(3)为第4组。第二类中,绒毛山蚂蝗的8份种质聚为一类,且地域性差异不明显,说明绒毛山蚂蝗的起源和亲缘关系较为复杂。第三类中,早年从国外引种的一份糙伏山蚂蝗种质单独分出来,由于其耐盐性和饲用价值,在海南得以保存与推广。第四类中,假地豆的7份种质聚在一起。选用的假地豆种质大多采集自海南,地域差异不明显,但种间差异明显,表明假地豆遗传多样性丰富。第五类中,卵叶山蚂蝗的18份种质聚在一起。这18份种质都是引进的野生种质,从聚类图上可以看出遗传多样性比较复杂:当阈值为0.95时,又可分为5个亚类:第1个亚类有一份种质(29);第2个亚类有一份种质(34);第3个亚类有两份种质(32和33);第4个亚类有两份种质(46和43),它们之间的相似度较低;第5个亚类中包含了其余的12份种质,其中第1个亚组的44和45号材料之间的相似系数在整个聚类图中最大(0.99),说明这两份种质的亲缘关系最接近。第六类中,长波叶山蚂蝗的3份种质聚在一起,3个不同地域的长波叶山蚂蝗的相似度比较高,表明它们的遗传多样性较为单一,亲缘关系较近。
整体来看,第二类(绒毛山蚂蝗)和第三类(糙伏山蚂蝗)首先聚在一起,说明这两类的亲缘关系较近,再和第一类(大叶山蚂蝗+赤山蚂蝗)聚在一起;第四类(假地豆)和第五类(卵叶山蚂蝗)首先聚到一起,说明这两类亲缘关系相对较近。聚类结果与地理来源有一定的一致性,但没有必然联系。
3 讨论
3.1 应用银染AFLP技术进行山蚂蝗属植物野生种质指纹分析的优越性
目前,在种质资源遗传多样性研究中使用较多的分子标记有RAPD、RFLP、SSR、ISSR、AFLP等。RAPD简单易行,DNA用量少(15~25 ng),但是对反应条件敏感,重复性较差;RFLP对DNA的需要量较大(5~10 μg),操作繁琐,周期长,成本较高;SSR虽然具有共显性标记和特种特异性以及发现的标记引物可以共用等优点,但是非常费时,耗资。ISSR虽然具有很好的稳定性和多态性,技术要求低,但其与RAPD类似,不能鉴别检测位点的纯合与杂合状态;而AFLP技术结合了RFLP和RAPD各自的优点,既有RFLP的可靠性,又有RAPD的简便性,较之其他分子标记技术更能覆盖整个基因组,多态性丰富,灵敏度高,被认为是迄今为止最有效的分子标记。王斌等[33]对水稻的研究表明,多态性检出效率为AFLP>RAPD>RFLP。Russell[34]对大麦基因组的多样性进行RFLP、RAPD、SSR、AFLP分析,结果发现揭示多样性最多的是AFLP,最低的是RFLP。Nakajima[35]以胡萝卜为材料进行了RAPD和AFLP分析,发现AFLP能产生4倍于RAPD的条带数,具有更高的分辨能力。
本研究利用银染AFLP技术对46份山蚂蝗属植物进行遗传多样性分析,所有供试材料全部得到了区分,而且所获得的系统分析结果与《中国植物志》上山蚂蝗属分类大致相同,为山蚂蝗属植物的分类提供了分子水平的依据。研究结果證明,采用银染AFLP技术对山蚂蝗属植物进行遗传多样性分析具有很多优越性。首先,AFLP扩增效率高,信息量大。本试验采用2对引物组合进行扩增,获得多态性条带154条,多态性比率达89.53%,多态性丰富,比较深入地反映了山蚂蝗属植物的遗传变异信息。其次,AFLP分析结果稳定、重复性好。对46个材料的同一扩增产物做多次重复电泳试验,扩增的带型完全一致,表现出良好的重复性。第三,理想的三碱基引物还可以防止错配产生的带达到检测水平。综上所述,利用银染AFLP技术进行山蚂蝗的指纹分析,具有其他分子标记所不能比拟的优越性,可以用于山蚂蝗属植物的遗传多样性分析和鉴定。
3.2 山蚂蝗属植物的亲缘关系和分类系统
依据本研究所获得的山蚂蝗属植物亲缘关系聚类图可以看出各种质之间的亲缘关系远近,及其在分类上的系统位置。本实验选用的5个国内种中,大叶山蚂蝗、赤山蚂蝗、绒毛山蚂蝗、假地豆在中国植物志分类系统中都属于三点金亚属,长波叶山蚂蝗属于饿蚂蝗亚属;大叶山蚂蝗、赤山蚂蝗、绒毛山蚂蝗的形态学距离较近,假地豆相对其他3种较远。本实验聚类分析结果与《中国植物志》的分类体系[1]以及刘苗苗等[36]利用形态特征对山蚂蝗植物进行聚类分析的结果相吻合。
从聚类图上看,长波叶山蚂蝗作为一个独立的分支而从其他供试山蚂蝗中分离出来,其亲缘关系与其他种较远,这与形态学分类系统一致。大叶山蚂蝗、赤山蚂蝗、绒毛山蚂蝗和糙伏山蚂蝗首先聚在一起,假地豆和卵叶山蚂蝗首先聚在一起,然后这6个种聚在一起,从分子标记的结果来看卵叶山蚂蝗和糙伏山蚂蝗可能也属于三点金亚属,卵叶山蚂蝗可能离大叶山蚂蝗更近一些,糙伏山蚂蝗可能离假地豆更近一些。这个结论可以为中国植物志的修订提供一个依据。
在假地豆种间,形态差异较大。基于AFLP的聚类图上也可以看出,虽然材料均采自海南,但是种间相似系数较小,遗传距离较大,从分子角度支持了假地豆种间遗传多样性丰富这一结论。
卵叶山蚂蝗CIAT350(29)是1981年从澳大利亚引入,经过多年的驯化,已于2005年已经申报为品种——热研16号卵叶山蚂蝗。从聚类图上可以看出CIAT350(29)和其他卵叶山蚂蝗的亲缘关系较远,说明人为的干预对种质的遗传多样性的作用很明显。
总体来看,聚类结果与地理来源没有必然的联系,这可能是山蚂蝗属植物的起源和亲缘关系较为复杂所致,也可能反映了地区之间山蚂蝗属植物的广泛交流。
参考文献
[1] 中国科学院中国植物志编辑委员会. 中国植物志[D]. 北京: 科学出版社, 1997: 15-47.
[2] Bentham G, Hooker J D. Genera plantarum[M]. London, 1965.
[3] 张渝文, 李 鑫. 山蚂蝗植物的免疫双扩散研究[J]. 重庆工商大学学报, 2004(8): 339-342.
[4] 国家药典委员会. 中华人民共和国药典[M]. 北京: 化学工业出版社, 2000: 32.
[5] 许实波, 针如芸, 冼顺英. 广金钱草总黄酮对心脑血管的效应[J]. 中山大学学报, 1980, 11(6): 265.
[6] 简洁莹, 宋丽芬, 陈再智. 广金钱草提取物对狗脑血流量及脑血管阻力等效应的研究[J]. 中草药, 1980, 11(8): 362.
[7] 王植柔, 白先忠, 覃光熙. 广金钱草主要成分防治尿石症的实验研究[J]. 中华泌尿外科杂志, 1991, 12(1): 13.
[8] 杨衔晋, 黄普华. 豆科-新属-长柄山蚂蝗属[J]. 东北林学院植物研究室汇刊, 1979(40): 1-15.
[9] 高成艺, 邹琦丽. 广西广义山蚂蝗属部分种类的核型及染色体数目报道[J]. 广西植物, 1995, 15(2): 166-171.
[10] 張渝文, 熊济华. 山蚂蝗属植物过氧化物酶同工酶研究[J]. 西南农业大学学报, 1990, 12(4): 387-388.
[11] 张渝文, 李 鑫. 重庆地区山蚂蝗属植物的花粉形态[J]. 渝州大学学报, 1992(4): 30-35.
[12] 陈艳琴, 周汉林, 刘国道. 山蚂蝗饲料资源研究进展[J]. 草业科学, 2010, 10(10): 173-178.
[13] 龙会英, 张 德, 朱红业, 等. 元谋干热河谷豆科牧草的引种试验[J]. 草业科学, 2011, 28(8): 1 485-1 490.
[14] 刘苗苗, 尹小畅, 严琳琳, 等. 山蚂蝗属(Desmodium Desv.)营养期营养成分分析[J]. 热带作物学报, 2015, 36(1): 125-131.
[15] Zabeau M, Vos P. Selective restriction fragment amplification: a general method for DNA fingerprinting[M]. European Application 92402629.7(Publication No.0534858AI). Paris: European Patent Office, 1993.
[16] Vos P, Hogers R, Zabeau M. AFLP: a new technique for DNA fingerprinting[J]. Nucleic Acids Research, 1995, 23(21): 4 407-4 417.
[17] Cho Y G, Blair M W, Panaud O. Cloning and mapping of variety-specific rice genomic DNA sequences: AFLP from silver-stained polyacrylamide gels[J]. Genome, 1996, 39(2): 373-378.
[18] Chalhoub B A, Thibault S, Lauco V. Silver staining and recovery of AFLP amplification products on large denaturing polyacrylamide gels[J]. Biotechniques, 1997, 22(2): 216-218.
[19] Akopyanz N, Bukanov N, Westblom T U. PCR-based RFLP analysis of DNA sequence diversity in the gastric pathogen Helicobacter pylori[J]. Nucleic Acids Research, 1992, 20: 6 221-6 225.
[20] Koniecyzn A, Ausubel F M. A procedure from mapping Arabidopsis mutations using codominant ecotype specific PCR-based markers[J]. Plant Journal, 1993, 4: 403-410.
[21] Jarvis P, Lister C, Szabo V. Integration of cAPs markers into the AFLP map generated using recombinant inbred lines of Arabidopsis thaliana[J]. Plant Molecular Biology, 1994, 24: 685-687.
[22] Paul S, Wachira F N, Powell W. Diversity and genetic differentiation among populations of Indian and Kenyan tea (Camelia sinensis(L.)O. kuntze) revealed by AFLP[J]. Theoretical and Applied Genetics, 1997, 94(2): 255-263.
[23] He q Prakash C S. Identification of polymorphic DNA markers in cultivated peanut(Arachis hyogaea L.)[J]. Euphytica, 1997, 97(2): 143-149.
[24] Rahman M S M, Shimada T, Yamamoto T. Genetic diversity of cherimoya cultivars revealed by amplified fragment length polymorphism(AFLP) analysis[J]. Breeding Science, 1998, 48(1): 5-10.
[25] Hongtrkul, Huestis G M, Knapp S. Amplified fragment length polyrnorphisms as a tool for DNA fingerprinting sunflower germplasm: Genetic diversity among oilseed inbred lines[J]. Theoretical and Applied Genetics, 1994, 89: 9-13.
[26] Hill M, Witseboer H, Zabeau. PCR-based fingerprinting using AFLPs as a tool for studying genetic relationships in Lactuca spp[J]. Theoretical and Applied Genetics, 1996, 93(8): 1 202-1 210.
[27] Koch q, Jung C. Phylogenetic relationship of industrial chicory varieties revealed by RAPDs and AFLPs[J]. Agronoma Paris, 1997, 17(6-7): 323-333.
[28] 李 鸣, 梁朝旭, 方位宽, 等. AFLP分子标记技术在甘蔗遗传育种上的应用[J]. 中国糖料, 2006(1): 43-46
[29] Sharma S K, Knox M R, Ellis T H N. AFLP analysis of the diversity and phylogeny of Lens and its comparison with RAPD analysis[J]. Theoretical and Applied Genetics, 1996, 93(5-6): 751-758.
[30] Cervera M T, Gusmao J, Steenackers M. Identification of AFLP molecular markers for resistance against Melampsora larice-populina in popular[J]. Theoretical and Applied Genetics, 1996, 93(5-6): 733-737.
[31] 刘 欢, 赵桂琴, 耿小丽, 等. 燕麦AFLP 反应体系的建立与优化[J]. 草业科学, 2008, 25(2): 84-89.
[32] 王春梅, 徐 娜, 李阳春, 等. 山蚂蝗属植物DNA提取方法研究[J]. 草业与畜牧, 2011, 188(5): 8-11.
[33] 王 斌, 李传友, 郑洪刚, 等. 水稻的AFLP研究初报——反应条件的优化及对温敏核不育等位突变系的分析[J]. 植物学报, 1999, 41(5): 502-507.
[34] Russell. Direct comparision of levels of genetics variation among barley accession detected by RFLPs, AFLPs, SSRs and RAPDs[J]. Theoretical and Applied Genetics, 1997, 95(4): 714-722.
[35] Nakajima Y, Deda K, Yamanoto T. Characterization of genetic diversity of nuclear and mitochondrial genomes in Daucus varieties by RAPD and AFLP[J]. Plant Cell Repress, 1998, 17: 848-853.
[36] 劉苗苗, 严琳琳, 张 瑜, 等. 山蚂蝗属(Desmodium Desv.)种质资源形态多样性研究[J]. 热带作物学报, 2014, 35(10): 1 897-1 904.
