利用CRISPR/Cas9系统定向改良水稻粒长和穗粒数性状
2017-05-19沈兰李健付亚萍王俊杰华宇峰焦晓真严长杰王克剑
沈兰李健付亚萍王俊杰华宇峰焦晓真严长杰王克剑,*
(1扬州大学农学院/江苏省作物遗传生理国家重点实验室培育点/江苏省现代粮食作物生产协同创新中心/教育部植物功能基因组学重点实验室,江苏扬州225009;2中国水稻研究所水稻生物学国家重点实验室,杭州310006;3连云港市农业科学院,江苏连云港222000;#共同第一作者;*通讯联系人,E-mail:cjyan@yzu.edu.cn;wangkejian@caas.cn)
利用CRISPR/Cas9系统定向改良水稻粒长和穗粒数性状
沈兰1,2,#李健3,#付亚萍2王俊杰2华宇峰2焦晓真2严长杰1,*王克剑2,*
(1扬州大学农学院/江苏省作物遗传生理国家重点实验室培育点/江苏省现代粮食作物生产协同创新中心/教育部植物功能基因组学重点实验室,江苏扬州225009;2中国水稻研究所水稻生物学国家重点实验室,杭州310006;3连云港市农业科学院,江苏连云港222000;#共同第一作者;*通讯联系人,E-mail:cjyan@yzu.edu.cn;wangkejian@caas.cn)
【目的】基因组定点编辑技术已成为分子育种的重要手段。本研究拟对GS3和Gn1a功能缺失突变对目标性状的改良效应进行分析,以期为培育高产水稻提供理论基础。【方法】利用CRISPR/Cas9系统,以控制粒型基因GS3和控制每穗粒数基因Gn1a为编辑对象,构建了共敲除载体pC1300-2×35S::Cas9-gGS3-gGn1a,用农杆菌介导法转化4个优质水稻品种,分析了基因突变的特征和相应农艺性状。【结果】构建的敲除载体成功地实现了对GS3和Gn1a基因的定点编辑。在4个转化受体的T0代均分别获得了gs3和gs3gn1a的移码突变体。对T1代中无选择标记突变体的农艺性状分析表明,突变体gs3和gs3gn1a与野生型相比粒长变长,千粒重增加;突变体gs3gn1a与突变体gs3相比,每穗粒数显著增加。【结论】利用CRISPR/Cas9系统进行水稻基因编辑可以快速改良品种的目标性状,在水稻品种的定向改良方面具有巨大的潜力。
CRISPR/Cas9;基因编辑;水稻;GS3;Gn1a
在传统水稻品种改良过程中,常见的方法是通过杂交、回交的方式将目标基因导入受体品种,实现优良基因的聚合。然而,传统的育种方法周期长,费时耗力;加上不可避免存在连锁累赘,在将目标基因导入时往往会带入其他不利基因,因而育种的效率较低。因此,利用分子生物学技术,实现对基因组的定点修饰,克服传统育种中的连锁累赘,将会大大提高育种的效率。近年来,基因组编辑技术的兴起,为水稻实现基因的编辑带来了新的契机。基因组定点编辑技术是通过编码核酸酶切割基因组特定位点,进而诱导基因组定点突变。近几年由微生物适应性免疫系统发展而来的一种基因编辑工具CRISPR/Cas9(clustered regularly interspaced short palindromic repeats/CRISPR-associated protein 9)系统[1-3]已经在微生物[1,4,5]、动物[6-10]和植物[11-14]中得到广泛应用。CRISPR/Cas9系统主要包括Cas9蛋白和sgRNA两个重要组分,sgRNA主要识别靶向的DNA序列并引导Cas9蛋白对识别的位点进行切割,引起DNA双链断裂,并通过非同源重组方式和同源重组方式对DNA双链断裂进行修复形成碱基插入、碱基缺失、碱基替换等突变类型。Shan等[3]利用CRISPR/Cas9技术首次成功地编辑水稻OsPDS-P1和OsBADH2基因;Wang等[15]结合同尾酶连接的原理建立水稻中CRISPR/Cas9多基因敲除系统;Zhang等[16]、Ma等[17,18]结合Golden Gate克隆和Gibson组装技术建立水稻、拟南芥CRISPR/ Cas9多基因敲除系统。这些CRISPR/Cas9基因编辑技术的发展使该技术在水稻基因编辑中的应用越来越广泛[19-21]。
前人研究表明,GS3是控制水稻粒重和粒长的主效QTL,同时也是控制水稻粒宽和籽粒充实度的主效QTL[22,23]。位于GS3基因N端的调节器官大小的结构域(OSR)的功能缺失导致水稻籽粒变长以及产量增加[22,23]。Gn1a是影响水稻每穗实粒数的主效QTL,Gn1a基因的表达量降低引起花序分生组织中细胞分裂素的积累,促进颖花的分化,最终导致产量增加[24]。已有研究表明,部分籼稻品种同时带有这两个主效QTL座位上的有利等位基因gs3和gn1a,而在粳稻品种中却很少[25]。
本研究选取4个东北优质粳型常规的栽培水稻品种吉粳102、垦鉴稻6号、空育131和长白25为研究受体,以GS3和Gn1a为靶基因,运用CRISPR/ Cas9系统进行基因的定点编辑,分析了GS3和Gn1a功能缺失突变对目标性状改良的效应,旨在为培育高产水稻品种提供参考。
1 材料与方法
1.1 实验材料及种植
转基因受体材料为4个优质东北栽培品种,分别为中熟粳稻品种吉粳102和中早熟粳稻品种长白25,中早熟粳稻品种垦鉴稻6号,以及早熟粳稻品种空育131。所有受体材料和转基因受体均种植于浙江杭州中国水稻所实验基地。常规水肥管理。
1.2 靶位点设计
GS3(Os03g0407400)和Gn1a(Os01g0197700)靶序列参照已有研究设计的靶位点[25],在GS3第1外显子上设计19 bp靶位点序列AGTGACATGGC AATGGCGG,PAM序列为CGG(图1)。同样在Gn1a第1外显子上设计19 bp靶位点序列AATGA AGCAAGAGCAGGTC,PAM序列为AGG(图1)。其中GS3,Gn1a靶位点中都包含了ATG启动密码子以便更容易获得基因功能缺失的突变体。GS3和Gn1a基因靶序列利用NCBI网站上的BLAST程序进行筛选,未找到脱靶序列。
1.3 CRISPR/Cas9表达载体的构建

图1 GS3和Gn1a靶点位置Fig.1.Schematic diagram of the targeted sites in GS3 and Gn1a.

表1 本研究所用的引物Table 1.Primers used in this research.

图2 CRISPR/Cas9载体构建流程Fig.2.Flow diagram of CRISPR/Cas9 system for construction.
利用合成引物(表1),GS3-g++/GS3-g--和 Gn1a-g++/Gn1a-g--分别制成靶位点接头GS3和Gn1a。用AarⅠ酶切SK-gRNA载体,再用T4连接酶将GS3和Gn1a接头连接AarⅠ酶切回收过的SK-gRNA载体。连接产物转化大肠杆菌DH5α,用SK-gRNA上的引物T3分别与引物GS3-g--和Gn1a-g--进行菌落PCR,挑选阳性克隆送公司测序,筛选正确的中间载体SK-gRNA-GS3和SK-gRNA-Gn1a。分别用KpnⅠ/SalⅠ和XhoⅠ/BglⅡ酶切两个中间载体,用KpnⅠ/BamHⅠ酶切CRISPR/Cas9载体,回收酶切产物,再通过T4连接酶连接,构建pC1300-2×35S::Cas9-gGS3-gGn1a表达载体,通过酶切和测序的方法确认植物表达载体构建成功后转化农杆菌EHA105。载体构建的具体流程见图2。研究所用的中间载体SK-gRNA和转基因表达载体CRISPR/Cas9由实验室保存[15]。
阳性的农杆菌菌株用于转化受体品种,获得转基因T0代苗。用载体上特异引物(Hyg-F1/Hyg-R1和Cas9-F2/pC1300-R2)鉴定组培苗中是否成功整合T-DNA区,不能扩增出目的条带的为假阳性苗。
1.4 转基因阳性株的获得
用阳性农杆菌株转化吉粳102、垦鉴稻6号、空育131和长白25愈伤组织。提取T0代植株的基因组DNA,用载体pC1300-2×35S::Cas9特异引物Hyg-F1/Hyg-R1和Cas9-F2/pC1300-R2进行PCR扩增,扩增产物大小分别为615 bp和600 bp(表1),能扩增出目的片段的植株为转基因阳性植株。
1.5 测序检测靶位点
为了检测靶位点的突变情况,在GS3和Gn1a的靶位点分别设计检测引物GS3-JC-F/GS3-JC-R和 Gn1a-JC-F/Gn1a-JC-R(表1),扩增产物大小分别为326 bp和334 bp。用引物GS3-JC-F/GS3-JC-R和Gn1a-JC-F/Gn1a-JC-R扩增T0代阳性株的目的条带,将目的条带送杭州擎科测序公司测序。测序结果通过刘耀光课题组研发的序列解码方法[18]进行分析(http://dsdecode.scgene.com/)。以吉粳102、垦鉴稻6号、空育131和长白25水稻品种基因组为模板,扩增目的条带的比对模板序列。
1.6 T1代无选择标记基因突变株的获得
对挑选出靶基因突变株用引物Hyg-F1/Hyg-R1和Cas9-F2/pC1300-R2进行PCR扩增,两对引物都不能扩增出目的条带的植株为无选择标记基因的突变株,Actin基因为对照。
1.7 主要农艺性状考查与分析
于成熟期选择每个水稻背景中野生型和突变株各5株,取所选单株的主穗5个,考查植株每穗粒数。烘干种子,每个单株随机选30粒饱满的种子测量粒长、粒宽;随机称取200粒饱满种子的质量并换算成千粒重。利用SAS软件进行分析。
2 结果与分析
2.1 转基因阳性苗的获得
用带有表达载体pC1300-2×35S::Cas9-gGS3-gGn1a的农杆菌分别转化吉粳102、垦鉴稻6号、空育131和长白25,获得的转基因T0苗[26]用载体上特异引物Hyg-F1/Hyg-R1和Cas9-F2/pC1300-R2(表1)鉴定转化体中是否成功整合T-DNA区。结果表明在吉粳102、垦鉴稻6号、空育131和长白25中分别获得42株、29株、52株和30株组培苗,其中吉粳102有38株转基因阳性苗,垦鉴稻6号有20株转基因阳性苗,空育131有42株转基因阳性苗,长白25有28株转基因阳性苗。
2.2 GS3和Gn1a基因靶位点突变类型分析
为了解4个水稻品种中GS3和Gn1a基因的突变情况,将转基因阳性苗进行测序分析(图3)。测序分析结果表明,4个水稻品种中均筛选到gs3和gn1a突变,所有T0代突变株GS3基因的突变率都是100%,且均为纯合突变,而Gn1a的突变类型包括杂合突变和纯合突变。因此,综合两个基因位点,在转基因后代中获得了gs3和gs3gn1a两种类型的突变体。从T0阳性植株中选择在GS3和Gn1a基因座位上发生移码突变的植株用于农艺性状分析。GS3的突变类型包括单碱基(A,T,G,C)插入,1个碱基、2个碱基、4个碱基和11个碱基缺失。Gn1a基因的突变类型包括单碱基(A,T,G)插入和单碱基缺失。
2.3 T1代无选择标记基因突变株的获得
将T1代所有的gs3和gs3gn1a突变体用两对引物筛选T-DNA片段,一对引物Hyg-F1/Hyg-R1扩增潮霉素上615 bp片段,另一对引物Cas9-F2/ pC1300-R2扩增的600 bp片段包含Cas9基因和pC1300载体上的部分序列(图4)。两对引物同时扩增不出条带的为无选择标记基因的突变株,其中OsActin基因为对照。利用该PCR方法分别筛选吉粳102、长白25、垦鉴稻6号和空育131背景的转基因后代113、114、104和102株,选出gs3和gs3gn1a无选择标记基因的突变株植株数量分别为吉粳102背景18株,长白25背景23株,垦鉴稻6号背景10株和空育131背景20株。筛选出来的无选择标记基因的突变株进一步进行小区试验,分析植株的粒长和每穗粒数等性状。

图3 4个水稻品种T0代GS3和Gn1a突变类型分析Fig.3.Mutation types at the GS3 and Gn1a loci of the four varieties in T0generation.

图4 PCR鉴定无选择标记基因的突变株Fig.4.PCR identification of the marker-free transgenic plants.

图5 4个品种T1代株型Fig.5.Grass morphology of the plants of the four varieties in T1generation.
2.4 T1代植株性状分析
利用测序的方法筛选T1所有的移码突变gs3和gs3gn1a突变体,GS3和Gn1a是控制粒形和每穗粒数的主效QTL,前人的研究结果表明这两个主效基因不影响植株的正常生长,4个水稻品种中无论是单敲GS3还是共敲除GS3和Gn1a,植株的株型正常(图5)。选择T1代后代中生长状况良好的植株进行粒型、每穗粒数的分析。
我们统计并分析了4个粳稻品种中gs3和gs3gn1a突变体的粒形和每穗粒数等性状。总的来说,4个粳稻品种中gs3和gs3gn1a突变体的粒长较野生型植株增加8%~18%,并且都达到了极显著水平(图6)。值得注意的是水稻品种长白25和空育131中gs3gn1a突变体的粒长比gs3突变体长1%~4%;吉粳102和垦鉴稻6号中gs3gn1a突变体的粒长比gs3突变体短4%(图6)。而在相同品种中,不同基因型植株的粒宽变化都未达到显著水平,吉粳102、长白25和空育131的突变体的粒宽相比野生型的短0%~4%;而垦鉴稻6号中gs3gn1a突变体的粒宽比野生型增加2%(图6-C,F,I,L)。千粒重的分析结果表明,4个品种中突变体的千粒重较对应野生型植株均增加,gs3突变体千粒重增加2%~21%,gs3gn1a突变体千粒重增加7%~16%(图7)。
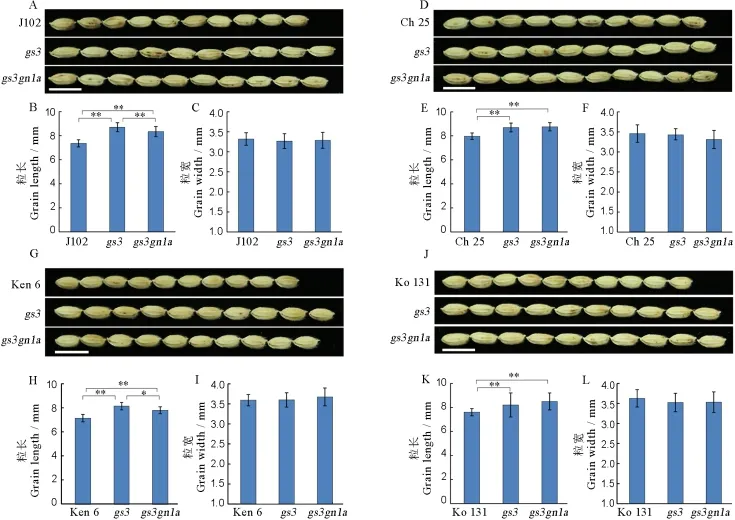
图6 T1代4个水稻品种及其突变体粒型Fig.6.Grain size of the four varieties and their mutants in T1generation.

图7 T1代4个水稻品种及其突变体的千粒重Fig.7.1000-grain weight among the four varietiesand their mutantsin T1generation.
比较gs3gn1a突变体和gs3突变体的每穗粒数发现(图8),4个水稻品种中gs3gn1a突变体的每穗粒数比gs3突变体增加11%~51%,而结实率出现相反的结果。4个水稻品种中gs3gn1a突变体的结实率比gs3突变体下降1%~8%。4个品种中突变体的结实率都比野生型的低,而且单突变体结实率降幅比双突变体小。

图8 T1代4个水稻品种及其突变体的每穗粒数和结实率Fig.8.Grain number per panicle and the seed-setting rate among the four varieties in T1generation and their mutants.
3 讨论
CRISPR/Cas9基因编辑技术具有成本低,操作简单,技术门槛低等优点。利用该基因编辑技术来获得植物基因功能缺失型突变体的方法已经成功地应用到许多作物中,如水稻[25,27]、玉米[28,29]、小麦[30,31]、大麦[32]、大豆[33,34]等。Shen等[25]敲除5个江浙水稻栽培品种的GS3和Gn1a基因,发现这两个基因在不同遗传背景下对产量的影响是多样的。本研究中,我们为了研究GS3和Gn1a基因功能缺失在东北品种中的效应,利用CRISPR/Cas9技术在吉粳102、垦鉴稻6号、空育131和长白25水稻品种中同时敲除GS3和Gn1a基因,成功获得功能缺失的突变体gs3和gs3gn1a,并在T1代考查粒形相关和穗型相关性状,发现敲除突变体的粒长和每穗粒数都与预期结果一致,且突变体的种子千粒重都比各自品种的野生型增加。
本研究中将设计的GS3和Gn1a的两个靶位点序列并联连入CRISPR/Cas9表达载体中,T0代植株中Gn1a的突变类型有纯合突变(包括纯合突变和双等位突变)和杂合突变,而GS3的突变类型均为纯合突变,且突变效率在4个水稻品种中都高达100%,以至于后代未能获得单敲除的gn1a突变体。有研究表明,靶序列的GC含量影响突变效率[17],GS3靶序列的GC含量(61%)比Gn1a(50%)高22%,因此,GS3靶序列的高GC含量可能是GS3基因突变效率高的原因之一。
我们发现敲除GS3和Gn1a基因产生的效应在不同地区的水稻背景中表现不同。Shen等[25]的研究结果表明,在5个江浙水稻品种中,突变体gs3和gs3gn1a植株的粒长与野生型相比增加1%~4%;同时gs3gn1a突变体的每穗粒数相较于gs3突变体增加3%~15%。而本研究在4个东北水稻品种中gs3和gs3gn1a突变株的粒长与野生型相比差异都达到极显著水平,增幅为8%~18%(表2),远大于5个江浙水稻品种;每穗粒数的增幅也比江浙水稻品种大,gs3gn1a突变体的每穗粒数相较于gs3突变体增加11%~51%(表2)。此外,东北品种gs3和gs3gn1a突变体的千粒重与野生型相比增幅为2%~21%,远大于5个江浙品种(0%~8%)[25]。以上结果表明,通过基因编辑技术对GS3和Gn1a进行编辑,在理论上可以选育出更加高产的品种。
产量是作物最为重要和最复杂的性状之一,产量的决定因素主要有三个方面:千粒重、每穗实粒数和有效穗数[35]。GS3主要控制粒长,进而影响籽粒的千粒重[22,23],Gn1a增加穗粒数[24]。理论上结合这两个主效QTL可以获得千粒重和每穗粒数都增加的水稻株系,然而我们发现突变株系的结实率下降,长白25号转基因背景中gs3gn1a突变体的每穗总粒数比gs3突变体增加11%(表2),而gs3gn1a突变体的每穗实粒数比gs3突变体仅增加2%(表2),其他的水稻背景下也是同样的变化趋势。结合水稻栽培中的源库流理论,我们推测可能是因为增加了水稻籽粒的库容量,而光合作用产生的源不够充足,使得每穗上的瘪粒数增加,从而降低了结实率。当然,环境条件也可能是导致转化体结实率降低的因素之一。
传统的杂交育种技术通过杂交、回交进行遗传改良,周期长,耗费大量的人力和物力资源,且难以突破基因连锁带来的连锁累赘。CRISPR/Cas9技术不但可以较容易地获得单个或多个基因定向改良的植株,而且在自交后代中获得无选择标记的株系,极大地加快了育种的进程。本研究利用基因编辑技术定向敲除4个东北品种中GS3和Gn1a基因,在T0代获得gs3和gs3gn1a突变体,通过对突变体T1代株系考查发现突变体除了目标性状得到改良外,其他性状未受到明显影响,说明通过基因编辑技术定向改良水稻品种是完全可行的。当然,由于本研究中选择的转基因受体均是来自于东北的粳稻品种,在浙江杭州试验基地种植会缩短生育期,因此本研究没有详细分析转基因植株的产量水平,需要将gs3和gs3gn1a突变体种植于吉林和黑龙江等东北地区进一步研究其产量性状。
[1]Wiedenheft B,Sternberg S H,Doudna J A.RNA-guided genetic silencing systems in bacteria and archaea.Nature, 2012,482:331-338.
[2]Cong L,Ran F A,Cox D,Lin S,Barretto R,Habib N, Hsu P D,Wu X,Jiang W,Marraffini L A,Zhang F. MultiplexgenomeengineeringusingCRISPR/Cas systems.Science,2013,339:819-823.
[3]Shan Q,Wang Y,Li J,Zhang Y,Chen K,Liang Z,Zhang K,Liu J,Xi J J,Qiu J L,Gao C.Targeted genome modification of crop plants using a CRISPR/Cas system. Nat Biotechnol,2013,31:686-688.
[4]DiCarlo J E,Norville J E,Mali P,Rios X,Aach J,Church G M.Genome engineering in Saccharomyces cerevisiae using CRISPR/Cas systems.Nucleic Acids Res,2013,41: 4336-4343.
[5]Jakociunas T,Bonde I,Herrgard M,Harrison SJ, Kristensen M,Pedersen L E,Jensen M K,Keasling J D. Multiplexmetabolicpathwayengineeringusing CRISPR/Cas9 in Saccharomyces cerevisiae.Metab Eng, 2015,28:213-222.
[6]Zhou X,Xin J,Fan N,Zou Q,Huang J,Ouyang Z,Zhao Y,Zhao B,Liu Z,Lai S,Yi X,Guo L,Esteban M A, Zeng Y,Yang H,Lai L.Generation of CRISPR/Cas 9-mediated gene-targeted pigs via somatic cell nuclear transfer.Cell Mol Life Sci,2015,72:1175-1184.
[7]Zou Q,Wang X,Liu Y,Ouyang Z,Long H,Wei S,Xin J, Zhao B,Lai S,Shen J,Ni Q,Yang H,Zhong H,Li L,Hu M,Zhang Q,Zhou Z,He J,Yan Q,Fan N,Zhao Y,Liu Z, Guo L,Huang J,Zhang G,Ying J,Lai L,Gao X. Generation of gene-target dogs using CRISPR/Cas 9 system.J Mol Cell Biol,2015,7:580-583.
[8]Wang H,Yang H,Shivalila C S,Dawlaty M M,Cheng A W,Zhang F,Jaenisch R.One-step generation of mice carrying mutations in multiple genes by CRISPR/Casmediated genome engineering.Cell,2013,153:910-918.
[9]Hoshijima K,Jurynec M J,Grunwald D J.Precise editing of the zebrafish genome made simple and efficient.Dev Cell,2016,36:654-667.
[10]Gratz S J,Harrison M M,Wildonger J,O'Connor-Giles K M.Precisegenomeeditingofdrosophilawith CRISPR/Cas RNA-guided Cas9.Methods Mol Biol,2015, 1311:335-348.
[11]Mao Y,Zhang H,Xu N,Zhang B,Gou F,Zhu J K. Application of the CRISPR-Cas system for efficient genome engineering in plants.Mol Plant,2013,6: 2008-2011.
[12]Puchta H.Applying crispr/cas for genome engineering in plants:The best is yet to come.Curr Opin Plant Biol, 2016,36:1-8.
[13]Jiang W,Zhou H,Bi H,Fromm M,Yang B,Weeks D P. DemonstrationofCRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis,tobacco, sorghum and rice.Nucleic Acids Res,2013,41:e188.
[14]Feng Z,Zhang B,Ding W,Liu X,Yang D L,Wei P,Cao F,Zhu S,Zhang F,Mao Y,Zhu J K.Efficient genome editing in plants using a CRISPR/Cas system.Cell Res, 2013,23:1229-1232.
[15]Wang C,Shen L,Fu Y,Yan C,Wang K.A simple crispr/cas9 system for multiplex genome editing in rice.J Genet Genom,2015,42:703-706.
[16]Zhang Z J,Mao Y F,Ha S,Liu W S,Botella J R,Zhu J K. A multiplex CRISPR/Cas9 platform for fast and efficient editing of multiple genes in Arabidopsis.Plant Cell Rep, 2016,35:1519-1533.
[17]Ma X L,Zhang Q Y,Zhu Q L,Liu W,Chen Y,Qiu R, Wang B,Yang Z F,Li H Y,Lin Y R,Xie Y Y,Shen R X, Chen S F,Wang Z,Chen Y L,Guo J X,Chen L T,Zhao X C,Dong Z C,Liu Y G.A robust CRISPR/Cas9 system for convenient,high-efficiency multiplex genome editing in monocot and dicot plants.Mol Plant,2015,8: 1274-1284.
[18]Ma X,Chen L,Zhu Q,Chen Y,Liu Y G.Rapid decoding of sequence-specific nuclease-induced heterozygous and biallelic mutations by direct sequencing of PCR products. Mol Plant,2015,8:1285-1287.
[19]Xu R F,Li H,Qin R Y,Li J,Qiu C H,Yang Y C,Ma H, Li L,Wei P C,Yang J B.Generation of inheritable and "transgene clean"targeted genome-modified rice in later generations using the CRISPR/Cas9 system.Sci Rep, 2015,5:11491
[20]Li J,Sun Y,Du J,Zhao Y,Xia L.Generation of targeted point mutations in rice by a modified CRISPR/CAS9 system.Mol Plant,2017,10(3):526-529.
[21]Lu Y,Zhu J K.Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system.Mol Plant,2017,10(3):523-525.
[22]Fan C,Xing Y,Mao H,Lu T,Han B,Xu C,Li X,Zhang Q.Gs3,a major QTL for grain length and weight and minor QTL for grain width and thickness in rice,encodes a putative transmembrane protein.Theor Appl Genet, 2006,112:1164-1171.
[23]Mao H,Sun S,Yao J,Wang C,Yu S,Xu C,Li X,Zhang Q.Linking differential domain functions of theGS3 protein to natural variation of grain size in rice.Proc Natl Acad Sci U S A,2010,107:19579-19584.
[24]Ashikari M,Sakakibara H,Lin S,Yamamoto T,Takashi T,Nishimura A,Angeles E R,Qian Q,Kitano H, Matsuoka M.Cytokinin oxidase regulates rice grain production.Science,2005,309:741-745.
[25]Shen L,Wang C,Fu Y,Wang J,Liu Q,Zhang X,Yan C, Qian Q,Wang K.QTL editing confers opposing yield performance in different rice varieties.J Integr Plant Biol, 2016,DOI:10.1111/jipb.12501
[26]Hiei Y,Ohta S,Komari T,Kumashiro T.Efficient transformation of rice(Oryza sativa L.)mediated by agrobacterium and sequence analysis of the boundaries of the t-DNA.Plant J,1994,6:271-282.
[27]Li M,Li X,Zhou Z,Wu P,Fang M,Pan X,Lin Q,Luo W,Wu G,Li H.Reassessment of the four yield-related genes Gn1a,DEP1,GS3,and IPA1 in rice using a CRISPR/Cas9 system.Front Plant Sci,2016,7:
[28]Feng C,Yuan J,Wang R,Liu Y,Birchler J A,Han F. Efficient targeted genome modification in maize using CRISPR/Cas 9 system.J Genet Genom,2016,43:37-43.
[29]Svitashev S,Young J K,Schwartz C,Gao H,Falco S C, Cigan A M.Targeted mutagenesis,precise gene editing, and site-specific gene insertion in maize using Cas9 and guide RNA.Plant Physiol,2015,169:931-945
[30]Shan Q,Wang Y,Li J,Gao C.Genome editing in rice and wheat using the CRISPR/Cas system.Nat Protoc, 2014,9:2395-2410.
[31]Wang Y,Cheng X,Shan Q,Zhang Y,Liu J,Gao C,Qiu J L.Simultaneouseditingofthreehomoeoallelesin hexaploid bread wheat confers heritable resistance to powdery mildew.Nat Biotechnol,2014,32:947-951.
[32]Lawrenson T,Shorinola O,Stacey N,Li C,Ostergaard L, Patron N,Uauy C,Harwood W.Induction of targeted, heritable mutations in barley and Brassica oleracea using RNA-guidedCas9 nuclease.Genome Biol,2015,16:258.
[33]Cai Y,Chen L,Liu X,Sun S,Wu C,Jiang B,Han T,Hou W.CRISPR/Cas 9-mediated genome editing in soybean hairy roots.PLoS One,2015,10:e0136064.
[34]Jacobs T B,LaFayette P R,Schmitz R J,Parrott W A. Targetedgenomemodificationsinsoybeanwith CRISPR/Cas 9.BMC Biotechnol,2015,15:16.
[35]Xing Y,Zhang Q.Genetic and molecular bases of rice yield.Annu Rev Plant Biol,2010,61:421-442.
Orientation Improvement of Grain Length and Grain Number in Rice by Using CRISPR/Cas9 System
SHEN Lan1,2,#,LI Jian3,#,FU Yaping2,WANG Junjie2,HUA Yufeng2,JIAO Xiaozhen2,YAN Changjie1,*, WANG Kejian2,*
(1Jiangsu Key Laboratory of Crop Genetics and Physiology/Co-Innovation Center for Modern Production Technology of Grain Crops/Key Laboratory of Plant Functional Genomics of the Ministry of Education/College of Agronomy,Yangzhou University,Yangzhou 225009,China;2State Key Laboratory of Rice Biology,China National Rice Research Institute,Hangzhou 310006,China;3Lianyungang Academy of Agricultural Sciences;Lianyungang 222000,China;#These authors contributed equally to this work;*Corresponding author,E-mail:cjyan@yzu.edu.cn,wangkejian@caas.cn)
【Objective】Gene orientation editing has become an important way for molecular breeding.We evaluated the improvement effects on the target traits following the construction of GS3 and Gn1a loss-of-function mutants so as to lay a solid foundation for high-yielding rice breeding.【Method】GS3 and Gn1a were selected as targets for gene editing, which control the grain size and grain number in rice,respectively.The pC1300-2×35S::Cas9-gGS3-gGn1aexpression vector was constructed for knocking out both GS3 and Gn1a by using CRISPR/Cas9 system,and transformed into four good quality rice varieties by the Agrobacterium-mediated method.And the properties of mutations and the target traits were analyzed in the transgenic lines.【Result】The sequencing results showed the GS3 and Gn1a in four rice varieties were successfully edited.In T0generation,we obtained mutants with frame shift mutations in GS3 and Gn1a in four rice varieties.In T1generation,the marker-free plants were screened for analyzing the agronomic traits in four genetic backgrounds.For the agronomic characters,the gs3 and gs3gn1a mutants had the longer grain length and the increased 1000-grain weight compared to the wild type,and the gs3gn1a mutants had more grains per panicle compared to the gs3 mutants.【Conclusion】CRISPR/Cas9-mediated gene editing can improve rice target traits,which has great potential in orientation improvement of rice varieties.
CRISPR/Cas9;gene editing;rice;GS3;Gn1a
Q755;S511.032
:A
:1001-7216(2017)03-0223-09
2017-03-09;修改稿收到日期:2017-03-20。
国家自然科学基金资助项目(31371233);高校自然科学基金重大项目(14KJA210002);中国农业科学院科技创新工程资助项目;江苏省农业科技自主创新资金资助项目[CX(13)5075]。
