开口箭种质资源及其混伪品的SSR指纹图谱研究
2017-05-16周天华
陈 征, 周天华
(1.陕西理工大学 生物科学与工程学院, 陕西 汉中 723000;2.陕西汉中朱鹮国家级自然保护区, 陕西 洋县 723301)
开口箭种质资源及其混伪品的SSR指纹图谱研究
陈 征1,2, 周天华1
(1.陕西理工大学 生物科学与工程学院, 陕西 汉中 723000;2.陕西汉中朱鹮国家级自然保护区, 陕西 洋县 723301)
利用SSR分子标记技术研究开口箭种质资源的DNA鉴定方法,构建其指纹图谱,为开口箭及其混伪品鉴定提供参考依据。应用7对多态性丰富的SSR引物对10份开口箭种质资源和5份混伪品进行了PCR扩增和聚丙烯酰胺凝胶电泳检测。结果表明,开口箭种质资源及其混伪品的遗传多样性丰富,7对SSR引物共检测出21个多态性位点,平均每对引物的多态性位点数为3.0;基于SSR分子标记的DNA指纹图谱可将开口箭种质及其混伪品完全区分开。该研究建立的SSR指纹图谱可用于开口箭种质资源标准化分析及市场化检测。
开口箭; SSR标记; 指纹图谱; 种质资源; 混伪品鉴定
开口箭(TupistrachinensisBaker),又名牛尾七、岩七、竹根七,为百合科开口箭属多年生草本植物,主要分布于陕西(秦岭以南)、四川、湖北等地,生于海拔1200~2200 m的林下、溪边、路旁或灌木丛中潮湿处[1]。其根状茎可入药,味苦、辛,性寒,有毒,具有清热解毒、散瘀止痛、祛风除湿等功效[2]。开口箭含有甾体皂苷、甾体皂甙元等有效化学成分,是秦巴山区常见的中药材资源[3]。
不同产地的开口箭生长环境不同,其药材质量存在显著差异。此外,开口箭的近缘种弯蕊开口箭(T.wattiiHook)、剑叶开口箭(T.ensifoliaWang & Tang)、筒花开口箭(T.delavayiFranchet)在形态特征上与其极为相似,常作为伪品与开口箭混淆。如开口箭的近缘种弯蕊开口箭(T.wattii),仅在花序下部苞片的长短上与开口箭有差异[4]。因此该药材在种植和采收上较为混乱,造成药材品质良莠不齐。鉴于不同物种及不同品种药材的有效成分普遍存在差异,有必要开发出适合开口箭属植物种质鉴定的分子生物学方法,为开口箭属植物进行药材鉴定提供强有力的保障。
近年发展起来的DNA指纹图谱技术在近缘种鉴定方面显示出了明显的优势[5]。简单重复序列(Simple Sequence Rrepeat,SSR)又称微卫星DNA,是近几年在PCR基础上发展起来的DNA分子标记技术[6]。SSR标记具有结果可靠,重复性好,多态性丰富,操作简单,呈共显性遗传等特点。与以往的分子标记相比,由于微卫星DNA广泛存在于真核生物基因组内,且有以孟德尔方式分离、选择中性、呈共显性等优点,因此SSR技术已成为研究种质资源遗传多样性、基因组作图、农作物品种指纹图谱的研究等工作的首选标记[7-8]。利用SSR指纹图谱技术鉴定是在传统形态分类学和物种准确鉴定研究基础上的一大改革,该方法还不受环境因素的影响、不受样本形态和材料部位的限制,可为中药原植物和中药材的品种鉴定提供更加准确可靠的手段,是中药分子鉴定方法学上的创新[9]。因此,本研究将采用SSR指纹图谱对开口箭及其混伪品进行DNA分子鉴定,建立开口箭的DNA指纹图谱鉴定体系,为开口箭种质资源鉴定和混伪品鉴定提供技术支持。
1 材 料
1.1 实验材料
4个不同种的开口箭属植物开口箭(T.chinensisBaker)、弯蕊开口箭(T.wattiiHook)、剑叶开口箭(T.ensifoliaWang & Tang)、筒花开口箭(T.delavayiFranchet)及3个不同种的百合科铃兰族其它属植物万年青(RohdeajaponicaRoth)、吉祥草(ReineckiacarneaKunth)、蜘蛛抱蛋(AspidistraelatiorBlume)的新鲜叶片采于陕西、湖北及云南等地(见表1)。野外采集新鲜的幼嫩叶片,放置于干净自封袋中,硅胶干燥,常温保存,供实验使用。
1.2 实验试剂与仪器
PowerPacTMHV型电泳仪(BIO-RAD公司),UP H-II-40型超纯水机(优普),KG-SX-500型灭菌锅(Chamber公司),Gel DocTMXR+型凝胶成像系统(BIO-RAD公司),Eppendorf型移液器等。DNA Marker(pUC18 DNA/Mspl,北京天根生化科技有限公司),PCR All-in-One PreMix(西安热默尔生物科技有限公司)。
2 方法与结果
2.1 基因组DNA提取
开口箭基因组DNA的提取采用改良CTAB法[10]。抽提出来的DNA经过浓度测定,A260nm/A280nm值在1.8~2.0为可用基因组DNA。
2.2 PCR扩增和电泳检测
PCR反应体系的总体积为10 μL[11],其中Mix 5 μL,上下游引物各1 μL,DNA模板1 μL,ddH2O 2 μL。PCR反应在PCR扩增仪(Bio-rad S1000TM)上的运行程序为:94 ℃预变性3 min;94 ℃变性30 s,退火温度(每组引物的退火温度见表2)下持续45 s,72 ℃延伸45 s,共35个循环;最后72 ℃延伸7 min,4 ℃保存。
在完成PCR循环48 h内,取2 μL PCR扩增产物以165 V的恒定电压在浓度为10%的非变性聚丙烯酰胺凝胶进行电泳15 h,然后对电泳后的聚丙烯酰胺凝胶进行银染和显影。显影完成之后,用凝胶成像系统(BIO-RAD Gel DOCTM XR+)采集图像。
2.3 数据统计分析与指纹图谱构建
对电泳后的凝胶图像进行条带统计,数据统计与分析PCR扩增产物的电泳图谱采用人工读带的方式,只记录主要带型,忽略弱杂带。根据每对SSR引物生成带型间的差别直接对带型进行分类编号,可得到不同个体的带型编号,一个个体的DNA经过不同引物扩增后,将获得一系列带型编号,按照固定的引物顺序,串联各带型编号,形成一组数据(见表1),便形成了该个体的分子指纹图谱[12]。
将7对SSR引物在15份开口箭种质及其混伪品材料扩增的电泳条带记录进行整理,再利用NTSYSpc 2.1统计分析软件按Nei的方法计算开口箭种质及其混伪品间的遗传相似系数[13]。用算术平均非加权配组法(Unweighted Pair-Groupmethod with Arithmetic Mean,UPGMA)进行遗传相似聚类分析并绘制各材料间的亲缘关系聚类树状图。
本研究SSR位点及其引物序列是基于开口箭设计开发的(由陕西理工大学植物分子生物学实验室开发,将另文发表),在基于2轮SSR多态性分析的基础上,筛选能够扩增出稳定的带型且不同材料之间差异明显的SSR引物,从中选取了7个多态性高、重复性好、分辨率高的SSR引物对(见表2)作为扩增7个不同种的SSR谱带,通过聚丙烯酰胺凝胶电泳,结果表明,扩增条带清晰,可以有效区分。统计分析了7对引物对全部15份开口箭种质及其混伪品的扩增带型后,构建其指纹图谱。

2.4 PCR扩增和电泳检测
本研究所用7对SSR引物均能对15份样本进行扩增和电泳(图1)。7对SSR引物扩增共产生了21个复等位基因位点,平均每对引物的复等位基因数为3.0;其中引物KKJSSR15的等位位点数最少,为2个;引物KKJSSR16的等位位点最多,为4个(见表2)。这表明本研究所用的SSR分子标记具有较高的多态性,而开口箭种质资源及其混伪品表现出了较高的遗传多样性。研究同时发现,多态性位点比较丰富的单个SSR引物,如KKJSSR16,由于其复等位基因数及特征谱带有限,单一引物(位点)仅能将部分开口箭种质进行区分,但采用多对引物(位点)的多个基因型的组合能有效地将15份开口箭种质资源及其混伪品进行区分和准确鉴定。
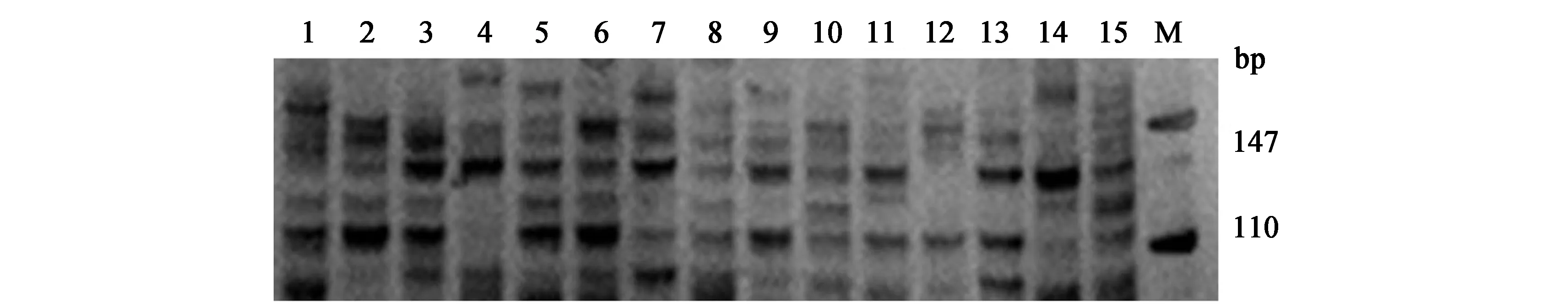
注:1-KFP; 2-KLBGHZ; 3-KLBDDS; 4-KHFS; 5-KQS; 6-WHLS; 7-WHY1; 8-WHY2; 9-WZLM;10-WZKS; 11-JYKKJ; 12-THKKJ; 13-WNQ; 14-JXC; 15-ZZBD; M-DNA Marker
利用软件NTSYS-pc2.10通过非加权平均法(UPMGA)计算开口箭及其混伪品种质间的遗传相似系数,构建了15份开口箭及其混伪品种质的UPGMA聚类分析图,如图2所示。
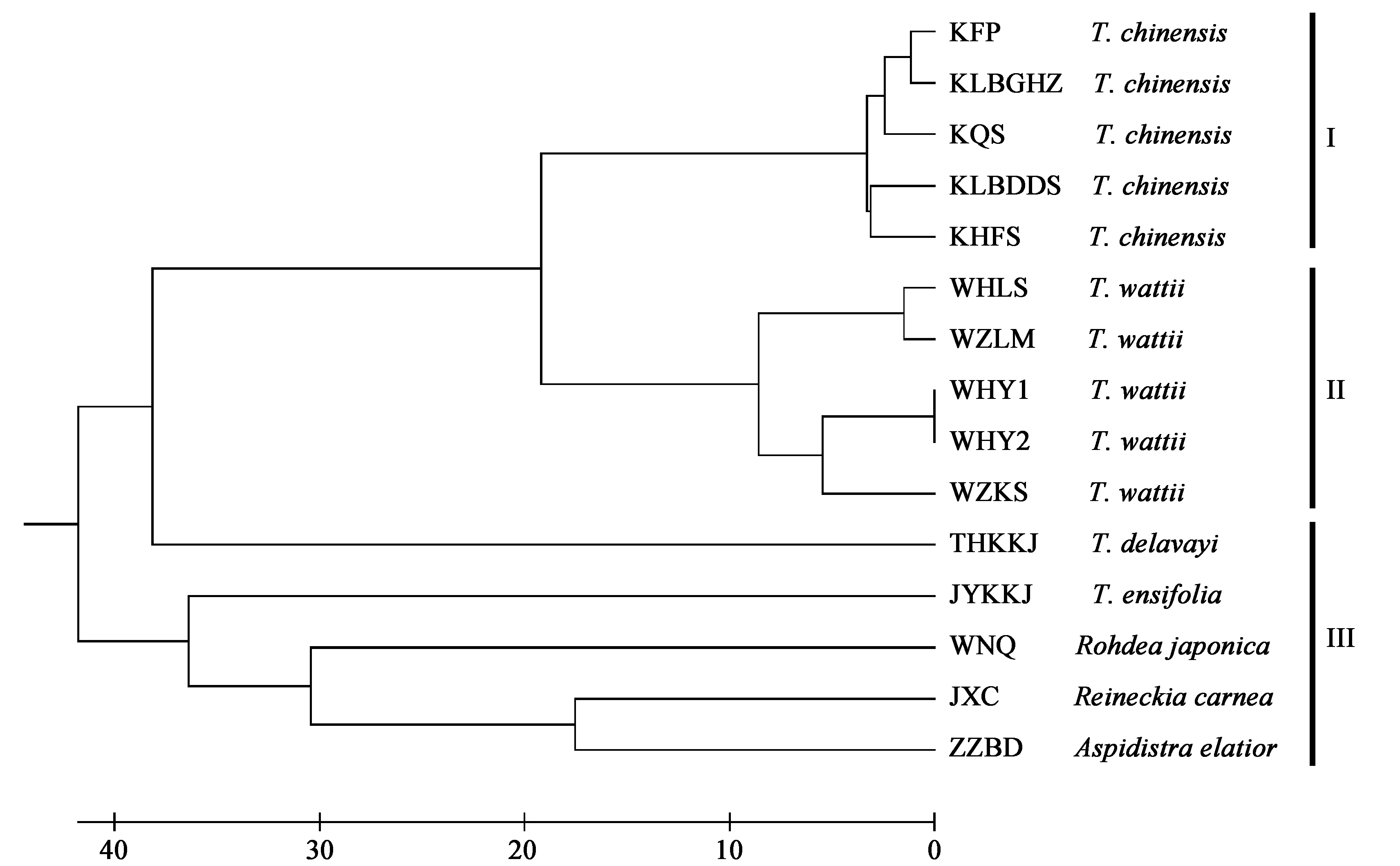
图2 15份开口箭种质资源与混伪品的UPGMA聚类图
利用软件GenAlEx 6.3 for Excel 2007对统计的条带进行分析,基于SSR标记检测获得的Nei’s遗传相似系数,构建主相关性分析(Principal Coordinated Analysis,PCoA)图,结果如图3所示。
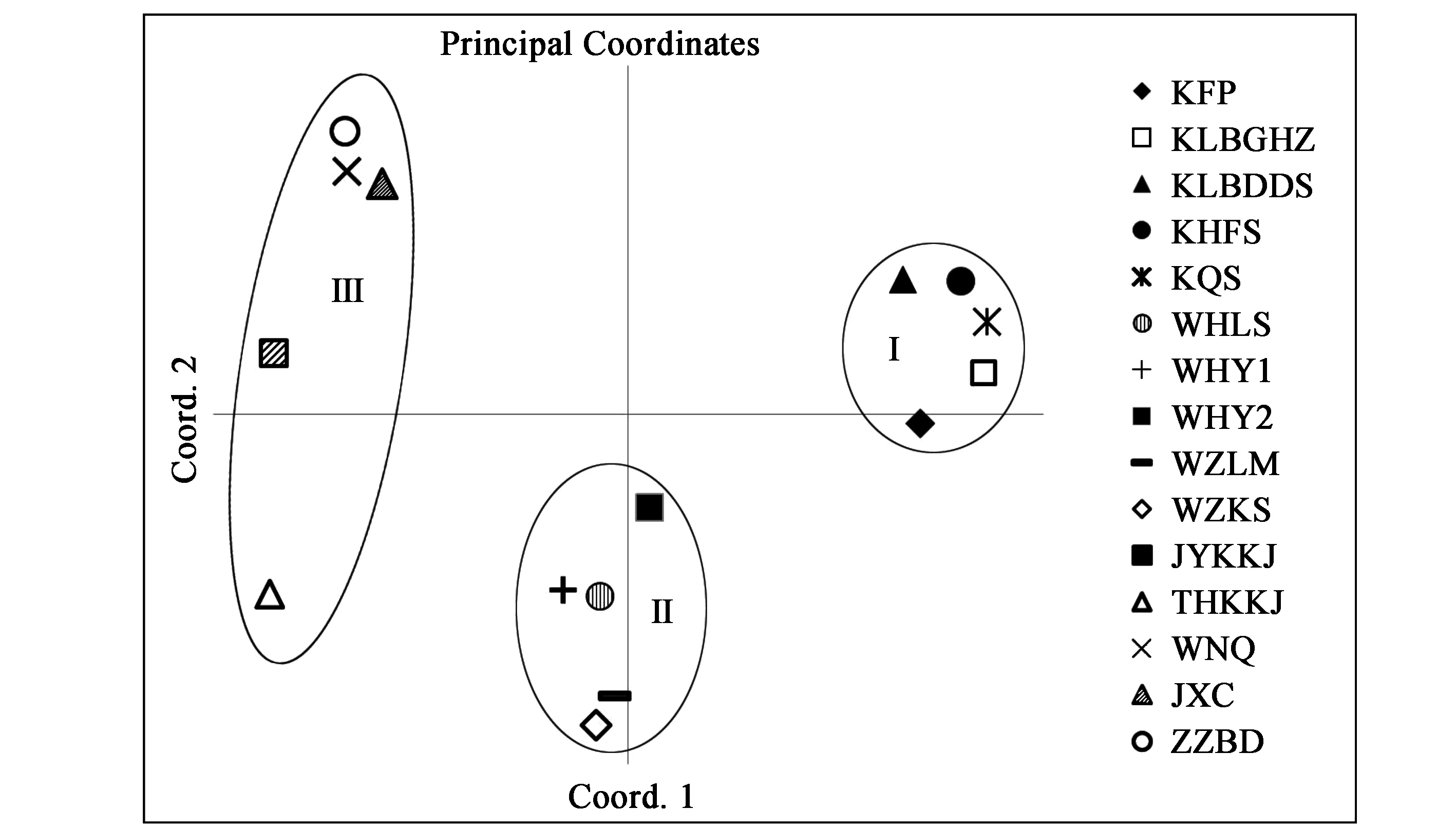
图3 15份开口箭种质资源与混伪品的主相关性分析(PCoA)图
从图2和图3中可见,根据遗传关系的远近,15份开口箭及其混伪品种质大致可划分为3大组,即组Ⅰ、组Ⅱ和组Ⅲ。5个开口箭种质(KFP、KLBGHZ、KQS、KLBDDS和KHFS)首先聚在一起组成组Ⅰ,5个不同产地的弯蕊开口箭个体(WHLS、WZLM、WHY1、WHY2和WZKS)聚在一起组成组Ⅱ,而与开口箭形态学相似的5个近缘种(THKKJ、JYKKJ、WNQ、JXC和ZZBD)因其亲缘关系相对较远,分散在组Ⅰ和组Ⅱ的外缘一起组成了组Ⅲ。
在组Ⅰ中,其亲缘关系均比较近,遗传相似度高,可分为两个亚组,KFP、KLBGHZ、KQS为一个亚组,KLBDDS和KHFS为一个亚组;在第Ⅱ类群中,也可分为两个亚组,WHLS和WZLM聚为一个亚组,剩下的聚为一个亚组,其中WHY1、WHY2因都采自陕西洋县华阳,因此亲缘关系近,遗传相似度较高;第Ⅲ类群因其均为与开口箭形态学相似的近缘种,在植物分类学上同属于百合科铃兰族,但毕竟不同种甚至不同属,因此亲缘关系较远,遗传相似度低。综上所述,本研究所用的SSR分子标记能清楚地将开口箭种质资源及其混伪品区分开,且区分度大,效果良好。
3 讨 论
SSR标记数量丰富,覆盖整个基因组,揭示的多态性高;具有复等位基因的特性,提供的信息量大;以孟德尔方式遗传,呈共显性[14];设计的引物序列决定每一个位点的位置,便于开发引物时不同实验室相互间进行交流合作,获得的资料能够在不同实验室重复并且共享[15],因此SSR标记将会在开口箭种质鉴定工作中发挥更加重要的作用。
本研究利用开口箭的7对SSR引物构建了开口箭种质资源和混伪品的DNA指纹图谱,7对SSR引物所产生的21个有效基因位点可对15份开口箭种质及其混伪品进行准确有效地区分。此外,在开口箭DNA指纹图谱构建的过程中,选用SSR标记的数量也是非常关键的[16],这不仅因为其影响着DNA指纹图谱的有效性和准确性,也决定着它所能区分开口箭种质的数量,而SSR标记数量的确定主要取决于单个SSR标记扩增多态性位点的多少,对某一特定数量开口箭种质而言,单个SSR标记扩增位点越多,DNA指纹图谱构建中选用的SSR标记数量则越少[14]。
传统的品种鉴定一般采用形态标记进行,但是鉴定周期往往比较长,存在着较大的误差,一些性状差异小的品种间鉴定困难[17]。DNA分子标记反映的是遗传本质上的差异,用于品种鉴定时具有周期短,不受环境条件的影响,真实可靠等优点[18]。开口箭及其混伪品之间性状差异小,利用形态标记很难进行区分,发展DNA分子标记指纹图谱对这些品种的鉴定具有重要意义。利用SSR标记构建开口箭种质资源的指纹图谱数据库,为这些中药材的鉴定奠定了基础。
[1] 中国科学院植物志编辑委员会.中国植物志:第十五卷[M].北京:科学出版社,1978:6-16.
[2] 贾敏如,李星炜.中国民族药志要[M].北京:中国医药科技出版社,2005:622.
[3] 李玥.秦巴山区三种开口箭植物的遗传多样性分析[D].汉中:陕西理工大学,2014:4-5.
[4] 濮社班,钱士辉,丁志遵,等.开口箭属五种植物叶的组织学研究[J].中国野生植物资源,1999,19(4):44-46.
[5] 王忠华. DNA指纹图谱技术及其在作物品种资源中的应用[J].分子植物育种,2006,4(3):425-430.
[6] 张立荣,徐大庆,刘大群.SSR和ISSR分子标记及其在植物遗传育种研究中的应用[J].河北农业大学学报,2002,25(1):90-94.
[7] 张晓峰,刘福艳.DNA指纹图谱技术在中药研究中的应用进展[J].齐鲁药事,2012,31(12):722-725.
[8] 王小童,陈征,周天华.开口箭种质资源的SSR遗传多样性分析[J].陕西理工学院学报:自然科学版,2016,32(1):70-75.
[9] 魏臻武,符昕,耿小丽,等.苜蓿遗传多样性和亲缘关系的SSR和ISSR分析[J].草地学报,2007,15(2):118-123.
[10] 周天华,陈征,李康,等.开口箭DNA提取方法比较[J].中国实验方剂学杂志,2015,21(19):10-13.
[11] 李玥,周天华,赵桦.开口箭ISSR-PCR实验反应体系条件的优化[J].中国实验方剂学杂志,2013,19(13):149-153.
[12] 王立新,赵昌平,邱军,等.记录小麦SSR带型的快捷方法[J].麦类作物学报,2006,26(4):164-168.
[13] 童和林,冯素萍,贺军虎,等.菠萝微卫星指纹图谱的构建[J].果树学报,2011,28(2):240-245.
[14] 王健胜,贺军虎,陈华蕊,等.基于SSR标记的菠萝种质DNA指纹图谱构建[J].热带作物学报,2015,36(7):1274-1279.
[15] 王丽,赵桂仿.植物不同种属间公用微卫星引物的研究[J].西北植物学报,2005,25(8):1540-1546.
[16] 李忠虎,刘晓东.党参微卫星引物筛选及群体遗传多样性研究[J].中草药,2013,1(2):210-214.
[17] 余守武,刘宜柏.微卫星标记在水稻遗传育种中的应用[J].江西农业大学学报,2003,25(2):157-163.
[18] 张立荣,徐大庆,刘大群.SSR和ISSR分子标记及其在植物遗传育种研究中的应用[J].河北农业大学学报,2002,25(1):90-94.
[责任编辑:魏 强]
SSR fingerprinting study onTupistrachinensisgermplasms and their adulterants
CHEN Zheng1,2, ZHOU Tian-hua1
(1.College of Biological Science and Engineering, Shaanxi University of Technology,Hanzhong 723000, China;2.Shaanxi Hanzhong Crested Ibis National Nature Reserve,Yangxian 723301, China)
With the aim to set up a molecular identification way forTupistrachinensisgermplasms and their adulterants, SSR DNA markers were employed to construct their DNA fingerprinting. PCR and polyacrylamide gel electrophoresis detection was carried out on 10T.chinensisgermplasms and 5 adulterants with 7 pair of polymorphism primers. As a result, theT.chinensisand its adulterants performed a high genetic diversity, 21 polymophic alleles were detected in total, with the average of 3.0 per loci; the DNA fingerprinting based on SSR molecular markers could distinguish the 10T.chinensisgermplasms and 5 adulterants from one another completely. The study provides a way of standardized analysis and market identification for theT.chinensisgermplasms and their adulterants.
TupistrachinensisBaker; SSR marker; fingerprinting; germplasm resources; adulterants identification
2016-11-19
2017-02-24
陕西省自然科学基础研究计划项目(2015JM3115);陕西省教育厅重点实验室科研计划项目(14JS017)
陈征(1992—),男,陕西省洋县人,陕西汉中朱鹮国家级自然保护区助理工程师,主要研究方向为野生动植物保护;[通信作者]周天华(1976—),男,陕西省汉中市人,陕西理工大学讲师,硕士生导师,博士,主要研究方向为植物分子生物学。
1673-2944(2017)02-0074-06
Q943.2
A
