仿刺参“化皮病”体壁组织DNA 甲基化的MSAP 分析
2017-05-16杨爱馥蒋经伟关晓燕姜孙红娟周遵春
高 杉 杨爱馥 董 颖 王 摆 陈 仲 蒋经伟 关晓燕姜 北 孙红娟 周遵春
(辽宁省海洋水产科学研究院, 辽宁省海洋水产分子生物学重点实验室, 大连 116023)
仿刺参“化皮病”体壁组织DNA 甲基化的MSAP 分析
高 杉 杨爱馥 董 颖 王 摆 陈 仲 蒋经伟 关晓燕姜 北 孙红娟 周遵春
(辽宁省海洋水产科学研究院, 辽宁省海洋水产分子生物学重点实验室, 大连 116023)
利用甲基化敏感扩增多态性(Methylation-sensitive amplified polymorphism, MSAP)技术分析了健康仿刺参(Apostichopus japonicus)体壁和“化皮病”仿刺参病变体壁、正常体壁DNA序列中CCGG位点的甲基化情况。结果显示, 健康仿刺参体壁和“化皮病”仿刺参病变体壁、正常体壁总甲基化水平分别为(18.60±5.61)%、(26.70±6.82)%和(19.53±3.34)%, 其中全甲基化水平分别为(13.97±4.86)%、(20.08±5.26)%和(15.42±2.61)%, 半甲基化水平分别为(4.63±3.59)%、(6.62±3.80)%和(4.11±2.08)%。“化皮病”仿刺参病变体壁总甲基化水平和全甲基化水平显著高于健康仿刺参体壁和“化皮病”仿刺参正常体壁(P<0.05), 健康仿刺参体壁与“化皮病”仿刺参正常体壁总甲基化水平和全甲基化水平差异不显著(P>0.05); 三者的半甲基化水平无显著差异(P>0.05)。因此, 推测仿刺参体壁“化皮”与DNA甲基化有关。
仿刺参; 体壁; 甲基化; MSAP; 化皮病
DNA甲基化(DNA methylation)是表观遗传学重要的修饰方式之一, 主要是指在DNA甲基转移酶的作用下, 将由S-腺苷甲硫氨酸提供的甲基转移到胞嘧啶C5位上, 形成5-甲基胞嘧啶的过程[1], 也有少量转移到腺嘌呤的N-6位和鸟嘌呤的N-7位形成N6-甲基腺嘌呤和N7-甲基鸟嘌呤[2,3]。DNA甲基化可以在不改变DNA序列的情况下通过基因沉默等方式调控基因的表达[4,5], 在X染色体失活[6]、进化[7]、肿瘤的产生[8]、基因印记[9]等生物过程中发挥重要的作用。DNA甲基化在细菌、植物、水产动物及人类(Homo sapiens)中有较广泛研究: 细菌可以通过DNA甲基化的方式调控母细胞基因的表达, 使子细胞更适应环境[10]; DNA甲基化在调控高粱(Sorghum bicolor)的基因表达方面可能有重要的作用[11]; Cd2+和Pb2+的离子种类、浓度和作用时间与泥鳅(Misgurnus anguillicaudatus)DNA的甲基化水平有相关性[12]; 人类DNA甲基化与DNA损伤应答之间联系紧密[13]等。DNA甲基化在海洋无脊椎动物中的研究主要集中在DNA甲基化在生物不同组织之间的差异[14,15], 以及对环境的适应机制方面[16]。
甲基化敏感扩增多态性(Methylation-sensitive amplified polymorphism, MSAP)技术是在扩增片段长度多态性(Amplified fragment length polymorphism, AFLP)技术的基础上建立起来的一种简便可靠的DNA甲基化检测技术。对胞嘧啶甲基化敏感性不同的同裂酶HpaⅡ和MspⅠ都能识别并切割CCGG序列, 分别与对甲基化不敏感的EcoRⅠ组成两对内切酶, 经过将DNA双酶切、连接接头、两步PCR、凝胶电泳等实验步骤后可以得到不同的特异性扩增图谱, 可以通过图谱判断甲基化的存在及状态。尽管对非CCGG位点上的胞嘧啶甲基化有着无法检测的局限性, 但相比于其他DNA甲基化检测技术, MSAP具有通用性强、操作简单、效率高, 且无论有无基因组背景信息, 均可通过检测获得丰富的甲基化位点等诸多优点, 因此受到广泛应用。
仿刺参(Apostichopus japonicus)又称刺参, 是我国北方重要的增养殖品种之一, 具有较高的经济价值。近年来随着养殖规模的扩大、养殖池塘的老化、集约化程度的提升, 仿刺参在养殖过程中时有病害发生, 仿刺参“化皮病”(腐皮综合征)就是其中之一, 该病具较强的传染性、较广的波动性和较高的死亡率, 大量病原学研究表明, 灿烂弧菌(Vibrio splendidus)是其主要病原之一[17], 并常伴以霉菌和寄生虫产生继发性感染[18], 感染初期的仿刺参表现为厌食、“摇头”、并对外界反应迟钝, 感染中期仿刺参表现为体壁“化皮”(白点状溃疡)、排脏, 感染后期仿刺参“化皮”的体壁逐渐恶化为大面积腐烂,直至死亡。本文以仿刺参为试验材料, 应用MSAP技术检测和分析健康仿刺参体壁和“化皮病”仿刺参病变体壁、正常体壁的甲基化程度的差异, 以期为研究仿刺参“化皮”过程中的分子调控机制积累资料。
1 材料与方法
1.1 试验材料
试验幼参(10—12) g取自辽宁省海洋水产科学研究院引育种中心, 于育苗池暂养一周, 暂养期间持续充气, 水温12℃左右, pH 8.1—8.3, 盐度32, 每日换水量25%。
试验用本实验室前期筛选的灿烂弧菌感染仿刺参[19,20], 灿烂弧菌于2216E培养基中在28℃下培养24h, 10000 r/min离心分离后收集。
1.2 菌液刺激及样品收集
将80只健康幼参等量分为两组, 分别放入两个长方形水族箱中(长0.80 m, 宽0.45 m, 高0.45 m), 其中一组为健康组, 每日换水量为25%; 另一组为“化皮病”组, 在每只幼参背部体壁用解剖刀轻划0.5 cm× 0.5 cm小口(头部下方), 并投入收集的菌液, 使养殖水体菌液终浓度为5×109CFU/mL, 每日换水量为25%, 并补充适量菌液维持菌液浓度。每日观察“化皮病”组仿刺参体壁(非切口处)是否发生病变(化皮)。当仿刺参体壁出现两处化皮部位(非切口处, 化皮部位直径大于0.2 cm)时开始取样, 健康仿刺参的体壁及“化皮病”仿刺参病变体壁和同一个体的正常体壁取样方法参照Yang等[21]。累计收集15只健康仿刺参的体壁, 以及15只“化皮病”仿刺参的病变体壁和正常体壁。每次将收集的样品投入液氮中迅速冷冻后, 放入–80℃冰箱中保存。
1.3 DNA提取
采用海洋动物基因组DNA提取试剂盒(天根生化科技有限公司)提取DNA, Implen NanoPhotometer核酸蛋白分析仪(德国)检测DNA的质量和浓度, 4℃保存备用。
1.4 引物和接头
所有引物和接头均由深圳华大基因科技有限公司合成。引物和接头序列见表 1。
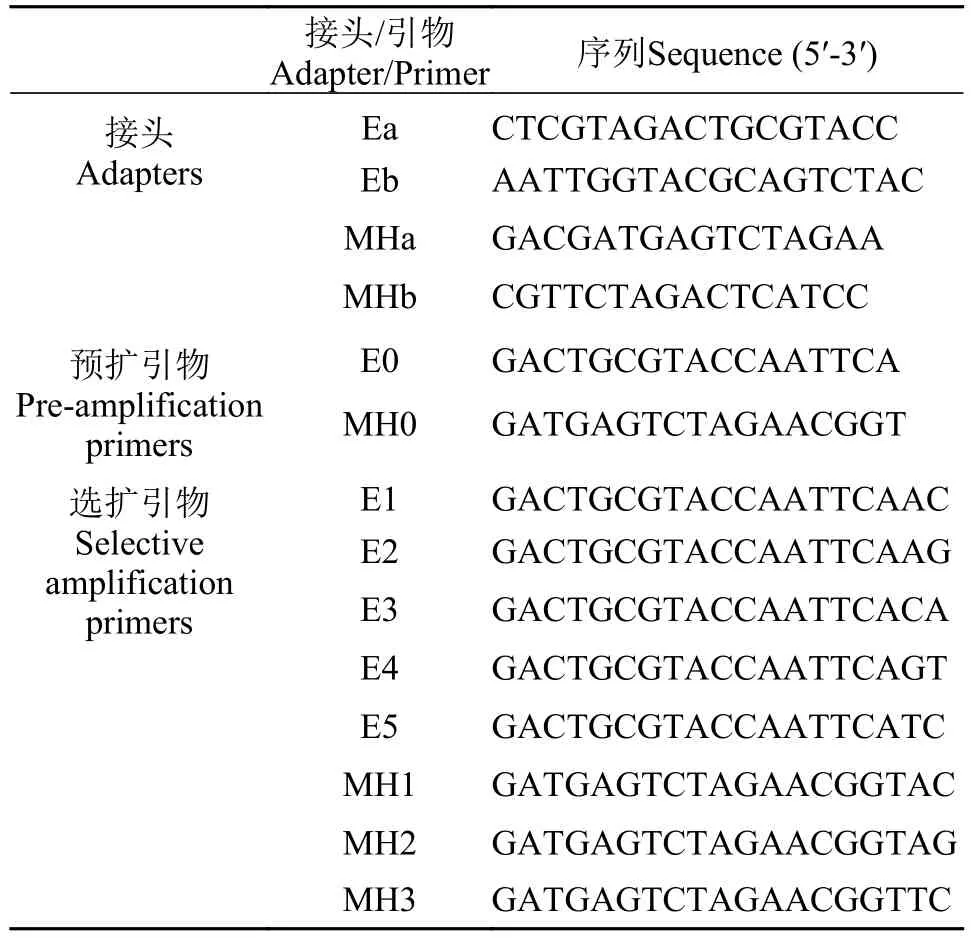
表 1 MSAP中所用的接头与引物序列Tab. 1 Adapters and primers used for MSAP analysis
1.5 MSAP分析
在Xiong等[22]的方法基础上进行适当修改。每个样品设置EcoRⅠ+MspⅠ和EcoRⅠ+HpaⅡ两组双酶切组合。酶切反应体系为: 100 ng DNA, 5 U EcoRⅠ(NEB HF), 5 U HpaⅡ/MspⅠ(NEB), 2 μL 10×NEB Buffer (NEB), ddH20补足至20 μL。该反应体系经过37℃金属浴4h, 80℃ 20min灭活内切酶。然后进行连接反应, 反应体系为: 酶切产物15 μL, 10 μmol/L Ea、Eb各0.2 μL, 100 μmol/L MHa、MHb各0.2 μL, T4 连接酶 200 U(NEB), T4 10×Buffer 2 μL (NEB), ddH2O补足至20 μL, 16℃连接过夜。预扩增反应体系: 连接产物2 μL, E0 0.4 μL, MH0 0.4 μL, Premix TaqTM(TaKaRa) 10 μL, ddH2O补足至20 μL。反应条件: 94℃ 3min; 94℃ 30s, 56℃ 30s, 72℃ 1min, 30个循环; 72℃ 10min; 4℃保存。将预扩增产物稀释40倍作为选择性扩增模板。选择性扩增反应体系: 稀释后的预扩增产物2 μL, Ex (E1-E5) 0.4 μL, MHx (MH1-MH3) 0.4 μL, Premix TaqTM(TaKaRa) 10 μL, ddH2O补足至20 μL。反应条件: 94℃ 3min; 94℃ 30s, 65℃ 30s (每个循环递减0.7℃), 72℃ 1min, 13个循环; 94℃ 30s, 56℃ 30s, 72℃ 1min, 23个循环; 72℃ 10min; 4℃保存。将扩增产物经12%非变性聚丙烯酰胺凝胶电泳分离检测, 经GeneFinder染色后统计凝胶上各种甲基化模式的片段条带。
1.6 数据分析
统计各甲基化模式条带后通过SPSS 16.0软件进行ANOVA分析和Duncan多重比较, 获得不同甲基化模式之间的差异, P<0.05表明差异显著。
2 结果
2.1 仿刺参的甲基化模式
每个DNA样品利用MSAP方法, 在EcoRⅠ和MspⅠ、EcoRⅠ和HpaⅡ两组双酶切处理后, 经过预扩增和15对选扩引物选择性扩增后, 电泳凝胶上呈现清晰扩增条带(图 1)。每个凝胶为同一DNA样品, 每对引物对应M、H两条泳道。其中, M泳道上有而H泳道上无的条带, 为全甲基化位点(A型); M泳道无而H泳道上有的条带为半甲基化位点(B型); M、H泳道均有的条带为非甲基化位点(C型)。扩增片段主要在100—800 bp。

图 1 不同引物组合在仿刺参基因组DNA中的MSAP图谱Fig. 1 DNA methylation profiles in A. japonicus amplified using different primer pairs第一条泳道为Marker(m); M表示EcoR Ⅰ/Msp Ⅰ酶切; H表示EcoR Ⅰ/Hpa Ⅱ酶切; 1—15表示Ex(E1-E5)和MHx(MH1-MH3)不同组合形成的15对引物。A. 全甲基化带型; B. 半甲基化带型; C. 非甲基化带型The first lane is marker(m); M means digested by EcoR Ⅰ/MspⅠ, H means digested by EcoR Ⅰ/Hpa Ⅱ; 1—15 means different combination forms of 15 primer pairs by Ex (E1-E5) and MHx (MH1-MH3). A. Full methylated bands; B. Hemi-methylated bands; C. Non-methylated bands
2.2 健康和“化皮病”仿刺参体壁组织的甲基化条带统计
利用15对引物分别对健康仿刺参体壁和“化皮病”仿刺参病变体壁、正常体壁进行扩增, 发现每个DNA样品均存在A型、B型和C型三种甲基化模式条带, 统计各模式下100—800 bp的扩增条带, 获得的甲基化多态性片段结果见表 2。健康仿刺参体壁和“化皮病”仿刺参病变体壁、正常体壁的总扩增条带分别为1916条、2124条和2204条, 以非甲基化条带为主, 分别为1567条、1560条和1772条; 总甲基化条带分别为349条、564条和432条, 以全甲基化条带为主, 分别为263条、427条和342条。
2.3 健康和“化皮病”仿刺参体壁组织的甲基化水平分析
仿刺参体壁总甲基化的水平为(A+B)/(A+B+C),全甲基化的水平为A/(A+B+C), 半甲基化的水平为B/(A+B+C), 非甲基化水平为C/(A+B+C)。结果显示(表 3), 健康仿刺参体壁的总甲基化水平为(18.60± 5.61)%, 其中全甲基化(13.97±4.86)%, 半甲基化(4.63±3.59)%; “化皮病”仿刺参病变体壁的总甲基化水平为(26.70±6.82)%, 其中全甲基化(20.08± 5.26)%, 半甲基化(6.62±3.80)%; “化皮病”仿刺参正常体壁的总甲基化水平为(19.53±3.34)%, 其中全甲基化(15.42±2.61)%, 半甲基化(4.11±2.08)%。“化皮病”仿刺参病变体壁总甲基水平和全甲基化水平均显著高于健康仿刺参体壁和“化皮病”仿刺参正常体壁(P<0.05), 而健康仿刺参体壁与“化皮病”仿刺参正常体壁的总甲基化水平和全甲基化水平差异不显著(P>0.05)。三者间的半甲基化水平无显著差异(P>0.05)。
3 讨论
3.1 MSAP在水产动物DNA甲基化研究中的应用
在水产动物甲基化研究方面, Jiang等[23]应用MSAP方法对太平洋牡蛎(Crassostrea gigas)闭壳肌的甲基化情况进行了研究, 发现总甲基化水平为26.4%, 其中全甲基化水平24.8%, 半甲基化水平1.6%; 吕佳等[24]利用MSAP方法检测到海湾扇贝(Argopecten irradias)闭壳肌总甲基化水平25.99%,其中全甲基化水平18.90%, 半甲基化水平7.09%; 虾夷扇贝(Patinopecten yessoensis)闭壳肌的总甲基化水平32.88%, 其中全甲基化水平25.85%, 半甲基化水平7.03%; 吴彪等[25]通过MSAP方法得到虾夷扇贝闭壳肌的总甲基化水平23.65%, 其中全甲基化水平16.37%, 半甲基化水平7.28%。郭婷婷等[14]采用MSAP方法分析了仿刺参不同组织基因组甲基化情况, 发现体壁中总甲基化水平33.51%, 其中全甲基化水平为18.39%, 半甲基化水平为15.12%, 高于本试验用同一方法得出的结果。同一物种相同组织表现出不同的甲基化水平在虾夷扇贝中也有类似的报道: 吴彪等[25]研究表明, 虾夷扇贝闭壳肌的总甲基化和全甲基化水平低于吕佳等[24]的研究结果,而在半甲基化水平间差别较小。Roberts和Gavery[26]报道了环境胁迫可能改变甲基化的模式; 曹家雪等[27]认为环境可通过影响DNA的甲基化水平从而调控基因的表达。因此本次试验仿刺参体壁总甲基化水平低于前人的研究结果可能与同一物种分属于不同的群落或者同一物种处于不同的生长环境有关。在本试验中, 健康仿刺参体壁和“化皮病”仿刺参病变体壁、正常体壁中半甲基化水平均低于全甲基化水平, 说明体壁的全甲基化水平占有优势,栉孔扇贝(Chlamys farreri)、虾夷扇贝、太平洋牡蛎等都有相似的结果, 而耐寒品系的尼罗罗非鱼(Oreochromis niloticus)半甲基化和全甲基化水平相当[28], 鸡(Gallus domestiaus)的半甲基化水平高于全甲基化水平[29], 体现不同物种间的甲基化水平呈现多元化。
3.2 DNA甲基化在水产动物病害研究中的应用
迄今为止, DNA甲基化作为表观遗传学研究重要的内容之一, 较多的应用于人类医学方面的研究,肿瘤、免疫系统疾病、心脑血管疾病等都与其有关, 例如抑癌基因和DNA修复基因呈现高甲基化水平、整个基因组呈现低甲基化水平与多种肿瘤的发生联系紧密[30]; T淋巴细胞基因组的低甲基化与系统性红斑狼疮密切相关[31]; 冠心病患者外周淋巴细胞的基因组甲基化水平显著高于健康人[32]等。水产动物中甲基化研究更多是在育种方面, 虾夷扇贝选育品种“玉贝”的总甲基化水平和全甲基化水平均低于对照组, 半甲基化水平高于对照组[25]; 尼罗罗非鱼耐寒品系的总甲基化水平、全甲基化水平和半甲基化水平均低于对照组[28]; 中国明对虾(Fenneropenaeus chinensis)选育品种“黄海1号”鳃、血液、肌肉三个组中的甲基化水平均低于对照组三个相同组织的甲基化水平[33]。
关于甲基化对水产动物病害免疫方面的研究还处于起步阶段, 较多都与基因的调控有关: Li等[34]研究表明半乳凝素基因Galectin可能参与珍珠贝(Pinctada fucata)外套膜损伤愈合的免疫应答, DNA的甲基化作用可能是Galectin基因表达的调控因子之一; Shang等[35]通过对呼肠病毒(GCRV)感染草鱼(Ctenopharyngodon idella)的试验推断出DNA甲基化在草鱼LGP2基因抗病毒转录中起到重要的调控作用。在本研究中, “化皮病”仿刺参病变体壁的总甲基化水平和全甲基化水平均显著高于健康仿刺参体壁和“化皮病”仿刺参正常体壁(P<0.05), 而“化皮病”仿刺参的正常体壁与健康仿刺参体壁的总甲基化水平、全甲基化水平和半甲基化水平均无显著差异(P>0.05), 推测仿刺参体壁的“化皮”可能与DNA甲基化相关。在通常情况下, DNA低甲基化会激活基因的转录, 而高甲基化则抑制基因的转录[36], 仿刺参“化皮”体壁组织中DNA甲基化位点和甲基化水平与基因表达存在怎样的相关性, 较高的DNA甲基化水平是否与“化皮”过程中基因的转录抑制有关, 我们将会做进一步的研究, 为揭示仿刺参“化皮”过程中的分子调控机制提供科学依据。
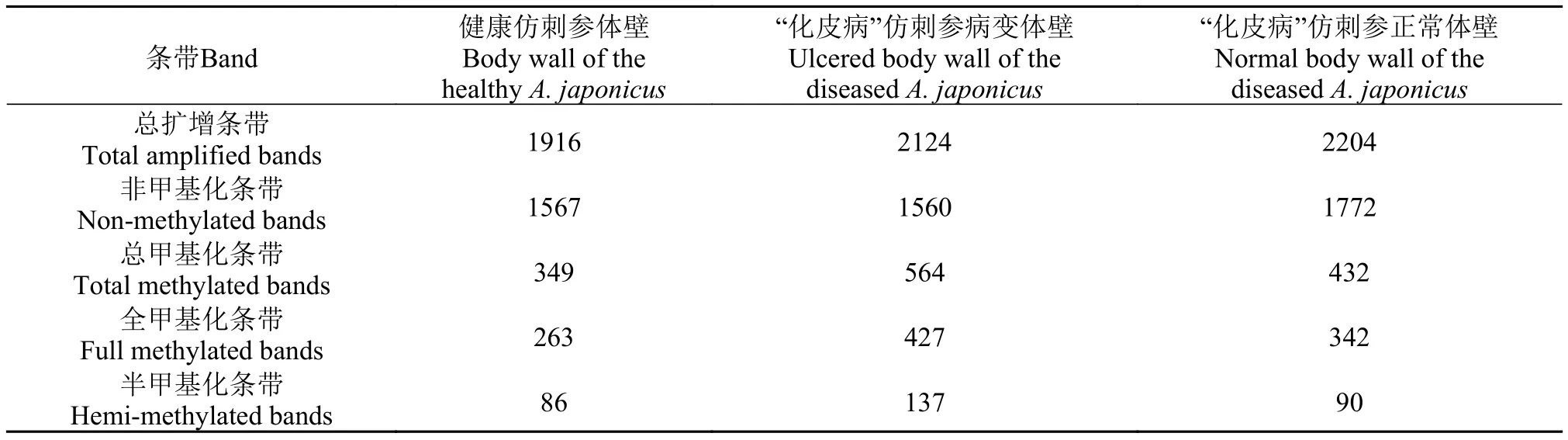
表 2 健康仿刺参体壁和“化皮病”仿刺参病变体壁、正常体壁DNA甲基化扩增条带Tab. 2 The amplified bands of DNA methylation of body wall from healthy A. japonicus, and diseased A. japonicus

表 3 健康仿刺参体壁和“化皮病”仿刺参病变体壁、正常体壁的甲基化程度Tab. 3 The methylation levels of body wall from the healthy and diseased A. japonicus
[1]Singal R, Ginder G D. DNA methylation [J]. Blood, 1999, 93(12): 4059—4070
[2]Von K T, Huber A R. DNA methylation analysis [J]. Swiss Medical Weekly, 2013, 143(5): 249—260
[3]Dodge J E, Ramsahoye B H, Wo Z G, et al. De novo methylation of MMLV provirus in embryonic stem cells: CpG versus non-CpG methylation [J]. Gene, 2002, 289(1—2): 41—48
[4]Morris J R. Genes, genetics, and epigenetics: a correspondence [J]. Science, 2001, 293(5532): 1103—1105
[5]Bird A. DNA methylation patterns and epigenetic memory [J]. Genes & Development, 2002, 16(1): 6—21
[6]Allen R C, Zoghbi H Y, Moseley A B, et al. Methylation of Hpa Ⅱ and Hha Ⅰ sites near the polymorphic CAG repeat in the human androgen-receptor gene correlates with X chromosome inactivation [J]. American Journal of Human Genetics, 1992, 51(6): 1229—1239
[7]Choe J. DNA methylation in development [J]. Journal of Genetic Medicine, 2008, 5(2): 100—104
[8]Das P M, Singal R. DNA methylation and cancer [J]. Journal of Clinical Oncology, 2004, 22(22): 4632—4642
[9]Autran D, Huanca-Mamani W, Vielle-Calzada J P. Genomic imprinting in plants: the epigenetic version of an Oedipus complex [J]. Current Opinion in Plant Biology, 2005, 8(1): 19—25
[10]Lecturer A H S. Epigenetic gene regulation in the bacterial world [J]. Microbiology & Molecular Biology Reviews Mmbr, 2006, 70(3): 830—856
[11]Meishan Z, Chunming X, Diter V W, et al. Tissue-specific differences in cytosine methylation and their association with differential gene expression in sorghum [J]. Plant Physiology, 2011, 156(4): 1955—1966
[12]Wang B L, Zhang Y M, Tan Y F, et al. Influence of cadmium and lead on the DNA methylation level of loach Misgurnus anguillicaudatus [J]. Journal of Toxicology, 2006, 20(2): 78—80 [王丙莲, 张迎梅, 谭玉凤, 等. 镉铅对泥鳅DNA甲基化水平的影响. 毒理学杂志, 2006, 20(2): 78—80]
[13]Jin B, Robertson K D. DNA methyltransferases, DNA damage repair, and cancer [J]. Advances in Experimental Medicine & Biology, 2013, 754: 3—29
[14]Guo T T, Sun G H, Yang J M, et al. MSAP analysis of genome DNA methylation in different tissues of Apostichopus japonicus [J]. Oceanologia et Limnologia Sinica, 2013, 44(1): 77—82 [郭婷婷, 孙国华, 杨建敏,等. 刺参(Apostichopus japonicus)不同组织基因组甲基化状态MSAP分析. 海洋与湖沼, 2013, 44(1): 77—82]
[15]Sun Y, Hou R, Fu X T, et al. Genome-Wide analysis of DNA methylation in five tissues of Zhikong Scallop, Chlamys farreri [J]. PloS One, 2014, 9(1): e86232
[16]Gavery M R, Roberts S B. Predominant intragenic methylation is associated with gene expression characteristics in a bivalve mollusk [J]. Peerj, 2013, 1(1): e215
[17]Gao Q, Liao M, Wang Y, et al. Transcriptome analysis and discovery of genes involved in immune pathways from coelomocytes of Sea Cucumber (Apostichopus japonicus) after Vibrio splendidus challenge [J]. International Journal of Molecular Sciences, 2015, 16(7): 16347—16377
[18]Wang Y G, Rong X J, Zhang C Y, et al. Main diseases of cultured Apostichopus japonicus: prevention and treatment [J]. Marine Sciences, 2005, 29(3): 1—7 [王印庚, 荣小军, 张春云, 等. 养殖海参主要疾病及防治技术. 海洋科学, 2005, 29(3): 1—7]
[19]Deng H, He C, Zhou Z, et al. Isolation and pathogenicity of pathogens from skin ulceration disease and viscera ejection syndrome of the sea cucumber Apostichopus japonicas [J]. Aquaculture, 2009, 287(s1—s2): 18—27
[20]Sun H, Zhou Z, Dong Y, et al. Expression analysis of microRNAs related to the skin ulceration syndrome of sea cucumber Apostichopus japonicas [J]. Fish & Shellfish Immunology, 2015, 49: 205—212
[21]Yang A, Zhou Z, Pan Y, et al. RNA sequencing analysis to capture the transcriptome landscape during skin ulceration syndrome progression in sea cucumber Apostichopus japonicus [J]. BMC Genomics, 2016, 17(1):1—16
[22]Xiong L Z, Xu C G, Maroof M A S, et al. Patterns of cytosine methylation in an elite rice hybrid and its parental lines, detected by a methylation-sensitive amplification polymorphism technique [J]. Molecular & General Genetics, 1999, 261(3):439—446
[23]Jiang Q, Li Q, Yu H, et al. Genetic and epigenetic variation in mass selection populations of Pacific oyster Crassostrea gigas [J]. Genes & Genomics, 2013, 35(5): 641—647
[24]Lü J, Hou R, Li N, et al. Application of MSAP technique for investigation of genome-wide DNA methylation level in scallops [J]. Periodical of Ocean University of China, 2013, 43(10): 48—53 [吕佳, 侯睿, 李宁, 等. 应用MSAP技术研究扇贝全基因组DNA甲基化水平. 中国海洋大学学报:自然科学版, 2013, 43(10): 48—53]
[25]Wu B, Yang A G, Dong Y H, et al. MSAP analysis on Genome-Wide DNA methylation in selected and wild Japanese scallop Patinopecten yessoensis [J]. Oceanologia et Limnologia Sinica, 2015, 46(4): 976—982 [吴彪, 杨爱国, 董迎辉, 等. 虾夷扇贝选育群体与野生群体基因组DNA甲基化的MSAP分析. 海洋与湖沼, 2015, 46(4): 976—982]
[26]Roberts S B, Gavery M R. Is there a relationship between DNA methylation and phenotypic plasticity in invertebrates [J]? Frontiers in Physiology, 2012, 2: 116
[27]Cao J X, Zhang H P, Du L X. Influence of environmental factors on DNA methylation [J]. Hereditas, 2013, 35(7): 839—846 [曹家雪, 张红平, 杜立新. 环境因素对DNA甲基化的影响. 遗传, 2013, 35(7): 839—846]
[28]Zhu H P, Lu M X, Huang Z H, et al. Effect of low temperature on genomic DNA methylation in Nile tilapia (Oreochromis niloticus) [J]. Journal of Fisheries of China, 2013, 37(10): 1460—1467 [朱华平, 卢迈新, 黄樟翰, 等. 低温对罗非鱼基因组DNA甲基化的影响. 水产学报, 2013, 37(10): 1460—1467]
[29]Xu Q, Zhang Y, Sun D X, et al. Analysis of DNA methylation in different chicken tissues with MSAP [J]. Hereditas, 2011, 33(6): 620—626 [徐青, 张沅, 孙东晓,等. 应用MSAP方法检测鸡不同组织基因组的甲基化状态. 遗传, 2011, 33(6): 620—626]
[30]Zhang L L, Wu J X. DNA methylation: An epigenetic mechanism for tumorigenesis [J]. Hereditas, 2006, 28(7): 880—885 [张丽丽, 吴建新. DNA甲基化——肿瘤产生的一种表观遗传学机制. 遗传, 2006, 28(7): 880—885]
[31]Shi W D, Xu Y. DNA methylation and common autoimmune diseases [J]. Chinese Journal of Allergy & Clinical Immunology, 2012, 6(2): 136—141 [石文丹, 许昱. DNA甲基化与常见免疫性疾病. 中华临床免疫和变态反应杂志, 2012, 6(2): 136—141]
[32]Priyanka S, Jitender K, Gaurav G, et al. Detection of altered global DNA methylation in coronary artery disease patients [J]. DNA & Cell Biology, 2008, 27(27): 357—365
[33]Du Ying, He Y Y, Li J, et al. MSAP analysis of genomic DNA in the tissues of wild and “Huanghai No.1” Fenneropenaeus chinensis [J]. Journal of Fishery Sciences of China, 2013, 20(3): 536—543 [杜盈, 何玉英, 李健, 等.野生和“黄海1号”中国明对虾不同组织基因组DNA的MSAP分析. 中国水产科学, 2013, 20(3): 536—543]
[34]Li Y, Huang X, Guan Y, et al. DNA methylation is associated with expression level changes of galectin gene in mantle wound healing process of pearl oyster, Pinctada fucata [J]. Fish & Shellfish Immunology, 2015, 45(2): 912—918
[35]Xueying S, Jianguo S, Quanyuan W, et al. CpG methylation in the 5′-flanking region of LGP2 gene lacks association with resistance/susceptibility to GCRV but contributes to the differential expression between muscle and spleen tissues in grass carp, Ctenopharyngodon idella [J]. Fish & Shellfish Immunology, 2014, 40(1): 154—163
[36]Adrian B. DNA methylation patterns and epigenetic memory [J]. Genes & Development, 2002, 16(1): 6—21
ANALYSIS OF DNA METHYLATION IN THE BODY WALL OF SEA CUCUMBER APOSTICHOPUS JAPONICUS WITH SKIN ULCERATION BY THE METHYLATION-SENSITIVE AMPLIFIED POLYMORPHISM (MSAP)
GAO Shan, YANG Ai-Fu, DONG Ying, WANG Bai, CHEN Zhong, JIANG Jing-Wei, GUAN Xiao-Yan, JIANG Bei, SUN Hong-Juan and ZHOU Zun-Chun
(Liaoning Key Lab of Marine Fishery Molecular Biology, Liaoning Ocean and Fisheries Science Research Institute, Dalian 116023, China)
The technology of methylation-sensitive amplification polymorphism (MSAP) was used to investigate the DNA methylation patterns at CCGG sites in the body wall of the healthy and diseased Apostichopus japonicus. The results showed that the methylation levels of body wall of the healthy A. japonicus, ulcered and normal body wall of the diseased ones were (18.60±5.61)%, (26.70±6.82)% and (19.53±3.34)%, among which the full methylations were (13.97±4.86)%, (20.08±5.26)% and (15.42±2.61)%, and the hemi-methylations were (4.63±3.59)%, (6.62±3.80)% and (4.11±2.08)%, respectively. The total methylation and full methylation levels in ulcer body wall of diseased A. japonicus were obviously higher than those of normal body wall in both healthy and diseased A. japonicas that have no difference in the total methylation level and full methylation level in the body wall. There were no significant differences in the hemi-methylations among three groups. These results suggest that the ulceration of body wall may be associated with DNA methylation.
Apostichopus japonicus; Body wall; Methylation; Methylation-sensitive amplified polymorphism (MSAP); Skin ulceration
S917
A
1000-3207(2017)03-0637-06
10.7541/2017.81
2016-06-06;
2016-11-18
国家自然科学基金(31672688); 辽宁省自然科学基金(2015020786); 辽宁省海洋与渔业厅科研项目(201503)资助 [Supported by the National Natural Science Foundation of China (31672688); the Natural Science Foundation of Liaoning Province, China (2015020786); the Ocean & Fisheries Project of Liaoning Province (201503)]
高杉(1983—), 男, 辽宁沈阳人; 硕士, 助理研究员; 主要从事分子生物学的研究。E-mail: gs_7920@163.com
周遵春(1967—), 男, 博士, 研究员; 主要从事海洋生物技术的研究。E-mail: zunchunz@hotmail.com
