途径工程及Tn5转座子介导突变提高大肠杆菌丙酮酸生产
2017-04-16史晓荣刘俊彭彦峰李林王文科王钦宏
史晓荣,刘俊,彭彦峰,李林,王文科,王钦宏
1 山西师范大学 生命科学学院,山西 临汾 041004
2 中国科学院天津工业生物技术研究所 中国科学院系统微生物工程重点实验室,天津 300308
丙酮酸是生物代谢过程中承上启下的关键中间产物,同时它为糖酵解途径和三羧酸循环提供原料,并且为生物的各项生命活动提供必需的能量,此外,它还广泛应用于食品、农药、生化等工业中[1-3]。随着我们对丙酮酸需求量的不断增加,如何找到一种环保、可持续的大量获得丙酮酸的方法显得尤为重要。
生产丙酮酸的方法主要有化学合成法、酶转化法和微生物发酵法 3种[4-5],由于发酵法具有产品纯度高、成本低、转化率高、对环境污染小等优点,已成为国内外最受青睐的生产方法。对生物发酵法来讲,寻找合适的发酵工程菌是最关键的,自然界中有很多的微生物可以合成丙酮酸,如球拟酵母、假丝酵母、酿酒酵母、肠杆菌属等。通过代谢工程手段有效地改造上述微生物的代谢途径,可以获得优良高产菌株。沈冬钱等[2]敲除大肠杆菌lpdA基因后,突变菌可以在以不同糖为碳源下积累丙酮酸,得率都达到了0.8 g丙酮酸/g糖以上。Zhu等[6]在大肠杆菌中敲除多个丙酮酸副产物基因,经发酵优化后,发酵罐分批补料44 h产丙酮酸90 g/L,得率0.68 g丙酮酸/g葡萄糖。Causey等[7]构建了1株大肠杆菌TC44,敲除了丙酮酸代谢途径中生成副产物的基因,它在以葡糖糖作为唯一碳源的矿物盐培养基中发酵43 h,丙酮酸的产量52 g/L,葡萄糖转换成丙酮酸的转化率为 0.75 g丙酮酸/g葡萄糖。于莹等[8]在谷氨酸棒杆菌中敲除了丙酮酸支流代谢途径基因,利用4.5%的葡萄糖复合培养基,工程菌经摇瓶发酵48 h,丙酮酸的浓度达到14.6 g/L,比野生菌提高了 32.5倍。Kawata等[9]发现在培养盐单胞菌Halomonassp. KM-1时,当培养基中硝酸钠的浓度增加时,导致丙酮酸的分泌增强,培养48 h,野生型Halomonassp. KM-1可以产生63.3 g/L丙酮酸。Kamzolova等[10]在食腺嘌呤节孢酵母Blastobotrys adeninivorans发酵中限制硫胺素来干扰硫胺素依赖的丙酮酸脱氢酶功能,可以产生43.2 g/L的丙酮酸。目前积累丙酮酸代谢产物主要集中于副产物代谢相关基因的改造和优化发酵条件[11-12]。由于进一步改造的靶点基因不明确,通过诱变是一种方法。
基于传统诱变的方法,难以对发生有效突变的菌株进行快速基因位点定位,无法精确地发现控制菌株高产的关键基因。而利用Tn5转座子来构建载体进行随机突变则不然,它通常被用于发现特定功能的未知基因[13],能够保证单基因插入突变来进行全基因改造,而且没有大量的无效突变背景干扰,能够进行基因组水平的随机插入。Wang等[14]利用Tn5转座子插入诱变用于构建运动发酵单胞菌中的较高耐盐性菌株,诱变产生 200个运动发酵单胞菌的突变体,筛选到耐盐性比野生型高达2%的突变菌株ZMT2,并且找到himA基因,该基因的破坏在响应耐盐中起着重要的作用。因此我们把这种方法应用到菌株的遗传改造过程中,为工程菌提供有效的改造靶点[15]。
本研究中我们以大肠杆菌 MG1655为出发菌株,通过基因操作得到积累丙酮酸的菌株KLPP,进一步利用Tn5转座子[16–17]对KLPP进行了基因组水平的随机插入,构建了1个突变体文库,与高通量筛选方法相结合,通过对文库的两轮筛选得到了丙酮酸产量稳定提高的突变体,再对转座子插入位点进行定位,找出了可能影响丙酮酸积累的基因靶点,为构建更高效的产丙酮酸大肠杆菌提供指导。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
E. coliK12 substr. MG 1655是构建产丙酮酸大肠杆菌的出发菌株。本研究所用菌株和质粒如表1所示。
1.1.2 主要试剂
氨苄青霉素、氯霉素、硫酸卡那霉素购自生工生物工程(上海)股份有限公司;质粒小量快速提取试剂盒购自美国Axygen公司;DNA回收试剂盒购自康为世纪公司;TransStart Fast Pfu DNA聚合酶、DNA marker、pEASY-Blunt Zero Cloning Kit试剂盒购自北京全式金生物技术有限公司;限制性内切酶、T4 DNA连接酶、T4多聚核苷酸激酶购自NEB公司;二硝基苯肼购自国药集团化学试剂有限公司;其他试剂均为分析纯试剂。
1.1.3 培养基
每升LB培养基包括10 g胰蛋白胨、5 g酵母提取物和10 g氯化钠 (固体LB加入1.5%的琼脂粉);发酵培养基为M9培养基,其1 L组成为:85.5 g Na2HPO4·12H2O,15 g KH2PO4,2.5 g NaCl,5 g NH4Cl,2 mmol MgSO4,0.1 mol CaCl2。氨苄青霉素、硫酸卡那霉素、氯霉素终浓度分别为 100、50、34 μg/mL。

表1 本研究的菌株和质粒Table 1 Strains and plasmids used in this study
1.2 方法
1.2.1 目标基因敲除
采用基于重复引物和同源重组的方法进行大肠杆菌相关基因的敲除[18]。使用这种方法,只需要一步转化,基因敲除后没有抗性基因或者其他序列存在于染色体上。基因敲除过程以ldhA的敲除为例说明。引物设计用于 PCR扩增cat-sacB序列,正向引物Sens-ldhA-CS包含ldhA的50 bp上游序列和50 bp的下游序列,之后是cat-sacB的扩增序列 (5ʹ-TCCTGGTGTCCCTGTT GATA-3ʹ);反向引物Anti-ldhA-CS包含ldhA的50 bp下游序列,之后是cat-sacB的扩增序列(5ʹ-ATAGATACATCAGAGCTTTTACGAG-3ʹ) 。扩增后DNA片段经过胶回收后电击转化到含有pKD46的感受态细胞。使用CmR筛选克隆,并进一步使用引物Sens-ldhA-yz和CS-yz进行PCR验证。正确的克隆接入含有10%葡萄糖的LB培养基中通过胞内重组以消除cat-sacB基因。使用氯霉素抗性和引物 Sens-ldhA-yz、Anti-ldhA-yz检测cat-sacB的消除。本研究使用引物见表2。
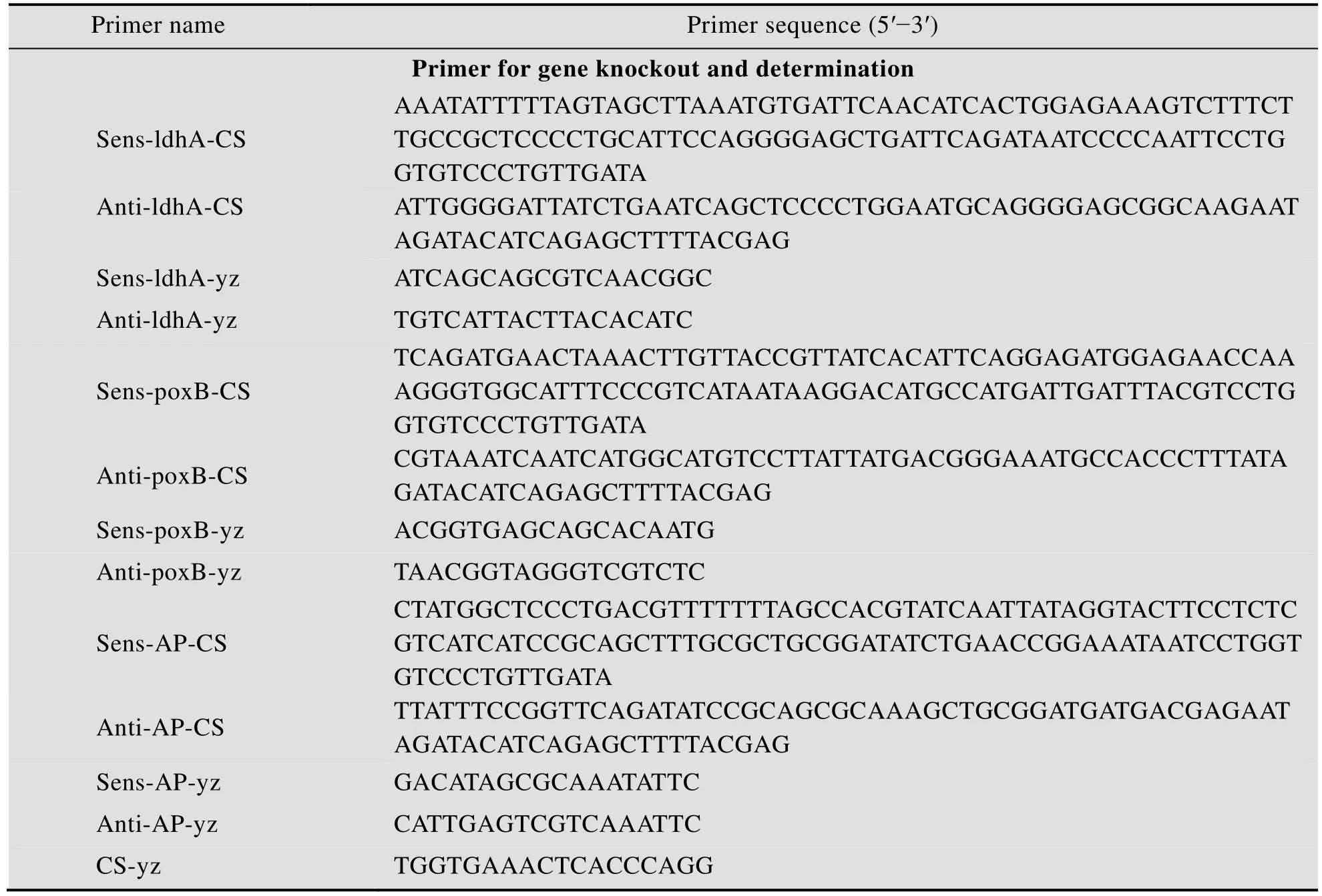
表2 本研究所用引物Table 2 Primers used in this study
1.2.2 突变文库的建立
将pUT Mini-Tn5 Cm质粒转入到大肠杆菌S17-1λpir中,涂布于含有氯霉素的平板,然后于37 ℃的培养箱中培养,挑选正确克隆。以含有 pUT Mini-Tn5 Cm 大肠杆菌 S17-1λpir为供体,受体为 KLPP菌株 (带有卡那霉素抗性)。这两种细菌分别在含氯霉素和卡那霉素的LB培养基中,37 ℃、220 r/min条件下培养5 h。将两株菌株在 5000×g条件下离心 2 min以后,用10 mmol/L MgSO4溶液重新悬浮并离心,重复洗涤3次,并最终用MgSO4悬浮并调整到合适的浓度后将二者混合。在含有0.1 mol/L柠檬酸钠的LB平板中心放置一张0.45 μm的无菌滤膜,将上述混合菌液添加至无菌滤膜上,放置于超净台中,吹干后于37 ℃的培养箱中培养10 h。
将滤膜上的菌苔刮下,并用少量无菌水进行清洗,涂布于含有卡那霉素和氯霉素的平板上进行选择培养。
1.2.3 高通量筛选及检测方法
丙酮酸在酸性条件下与 2,4-二硝基苯肼发生缩合反应形成丙酮酸二硝基苯腙。二硝基苯腙在碱性条件下呈现红色,可在520 nm处比色后灵敏地反映出丙酮酸的含量[19]。
配置6.25 g/L 的丙酮酸母液,再稀释成0、0.625、1.250、1.875、2.5、3.125、3.75、5 g/L的溶液,取40 μL不同稀释度丙酮酸溶液、40 μL 0.033%的二硝基苯肼、40 μL 2.81 mol/L NaOH混合,静置反应10 min,利用Molecular Device Spectra Max M2 酶标仪在520 nm的波长下测定吸光度。每个梯度设置3个平行。酶标仪可以直接测定0−140 mg/L浓度范围内的丙酮酸,并呈现良好的线性关系,线性回归方程为:y=0.005x–0.007,R2=0.993,用该法测出的发酵丙酮酸浓度与HPLC测定浓度一致,说明菌体及发酵液其他成分不对丙酮酸测定形成干扰。
为了测定细胞产丙酮酸的能力,我们设计了以下筛选流程:1) 突变库单克隆接种至96孔深孔板培养,摇床培养20 h;2) 菌体吹吸均匀后,先后加入40 μL菌液、40 μL 0.033%的二硝基苯肼和40 μL 2.81 mol/L NaOH进行显色反应,测定反应液的OD520值;3) 根据丙酮酸标准曲线计算出发酵液中丙酮酸的浓度,最后根据突变菌株与对照菌株丙酮酸的浓度比筛选出丙酮酸产量提高的菌株。
1.2.4 菌株的发酵评价
将最终筛选到的菌株进行摇瓶发酵验证。首先将保存于−80 ℃的突变菌在加有氯霉素和卡那抗生素的平板划线,培养过夜,挑取单菌落到含有3 mL LB的试管中,37 ℃、220 r/min培养12 h,测OD600值,保证起始OD一致,按相应比例接种到15 mL摇瓶发酵培养基中 (100 mL摇瓶),37 ℃、220 r/min培养24 h。取1 mL发酵液12000×g下离心10 min,将上清液稀释10倍后利用 HPLC (高效液相色谱仪) 检测丙酮酸的含量,确定丙酮酸产量明显提高的菌株,对高产丙酮酸的菌株进行3轮发酵重复实验,每轮3个平行。
发酵罐使用上海保兴BioTech-5BG全自动5 L发酵系统,装2 L发酵液。挑单克隆到装有3 mL LB试管 37 ℃培养OD600至 3.0,转接到装量200 mL LB的1 L摇瓶,37 ℃培养过夜后,以10%接种量转接入发酵罐,发酵罐 pH控制在6.5,通气量1 vvm,控制溶氧不低于30%。
1.2.5 利用HPLC对丙酮酸产量进行测定
丙酮酸产量利用高效液相色谱仪 (HPLC system 1260 Infinity Series, Agilent Technologies)测定。所用色谱柱为Aminex HPX-87H色谱柱,流动相为5 mmol/L硫酸溶液,流速为0.6 mL/min,柱温是63 ℃;检测器为紫外检测器,检测波长210 nm,检测时间为30 min。
1.2.6 全基因组重测序确定Tn5插入位置
将筛选到的突变菌株经过培养离心后,去掉上清液,将菌体送至广州基迪奥生物科技有限公司进行全基因组重测序,基因组重测序后获得原始数据,经过过滤去污染、比对参考基因组,来确定转座子插入位点。
2 结果与分析
2.1 积累丙酮酸菌株的构建
在有氧代谢途径中,丙酮酸会经过丙酮酸脱氢酶PDH作用进一步脱羧反应生成CO2和乙酰辅酶A,大部分乙酰辅酶A会进入TCA循环,但仍有大量乙酰辅酶 A会直接代谢为乙酸。有研究通过敲除PDH编码基因或者硫辛酸编码基因降低PDH活性来积累大量的丙酮酸[4-5],然而,需要添加乙酸或者硫辛酸维持细胞生长。本研究中,PDH活性保留,而由丙酮酸代谢到乙酸和乳酸的途径被阻断。
本研究中乳酸产生途径基因ldhA被敲除,得到菌株KL01 (ΔldhA)。进一步敲除乙酸途径,在大肠杆菌中,乙酸生成途径主要有两个:一是经由乙酰辅酶 A由磷酸转乙酰酶 (pta) 和乙酸激酶 (ackA) 作用生成乙酸,二是被丙酮酸氧化酶 (poxB) 直接氧化生成乙酸。在大肠杆菌基因组上ackA和pta是相邻的基因,因此可以同时敲除,得到菌株KLP01 (ΔldhA,ΔackA-pta)。进一步敲除poxB基因得到KLPP (ΔldhA,ΔackA-pta,ΔpoxB) 菌株,即本研究进行高通量筛选的出发菌株。野生型MG1655发酵未检测到丙酮酸积累,KLPP菌株摇瓶发酵丙酮酸可以达到3 g/L,5 L发酵罐发酵产量达到45 g/L,转化率达到0.55 g丙酮酸/g葡萄糖。
2.2 突变体筛选结果
将进行结合转移后的菌液稀释至合适浓度涂布于氯霉素和卡那霉素双抗性筛选平板生长,得到每个平板有 100个左右的单菌落的专座插入突变库用于后续筛选。我们总共筛选了7197个克隆。
第一轮筛选:以KLPP为出发菌株,基于转座子突变的方法筛选到了 7197个突变体 (图1A),以突变菌株和出发菌株中丙酮酸浓度的比值评价菌株。在7197个样本中,筛选到的突变菌株与KLPP的丙酮酸比值大于8%的共有661个,占文库的9%。
第二轮筛选:经过第一轮的筛选后,得到了661个突变菌株 (图1B),我们选择比值大于8%的菌株作为潜力菌株,通过96深孔板进一步筛选到了50个菌株,经过再次评价,筛选出了丙酮酸产量得到稳定提高的6个菌株进行后续评价。
2.3 突变菌株发酵能力分析
把上述筛选到的菌株接种于含 M9培养基的试管中进行发酵评价,用HPLC对发酵产物进行分析 (图2),发现这6株菌的丙酮酸产量确实得到了稳定的提高。其中K5、K12、K13、K22、K30和K36丙酮酸产量分别提高了38%、31%、19%、28%、44%和14%。
2.4 突变体菌株转座子插入位置的确定
通过对 K5、K12、K13、K22、K30、K36六株菌进行基因组重测序,经过对数据的分析和与出发菌株基因组进行对比,我们找到了五株菌的插入位点分别为intR基因、nanC基因 (K12和K13插入位点的基因是同一个)、yagU基因、ydfJ基因 (表3),K22的插入位点未找到。
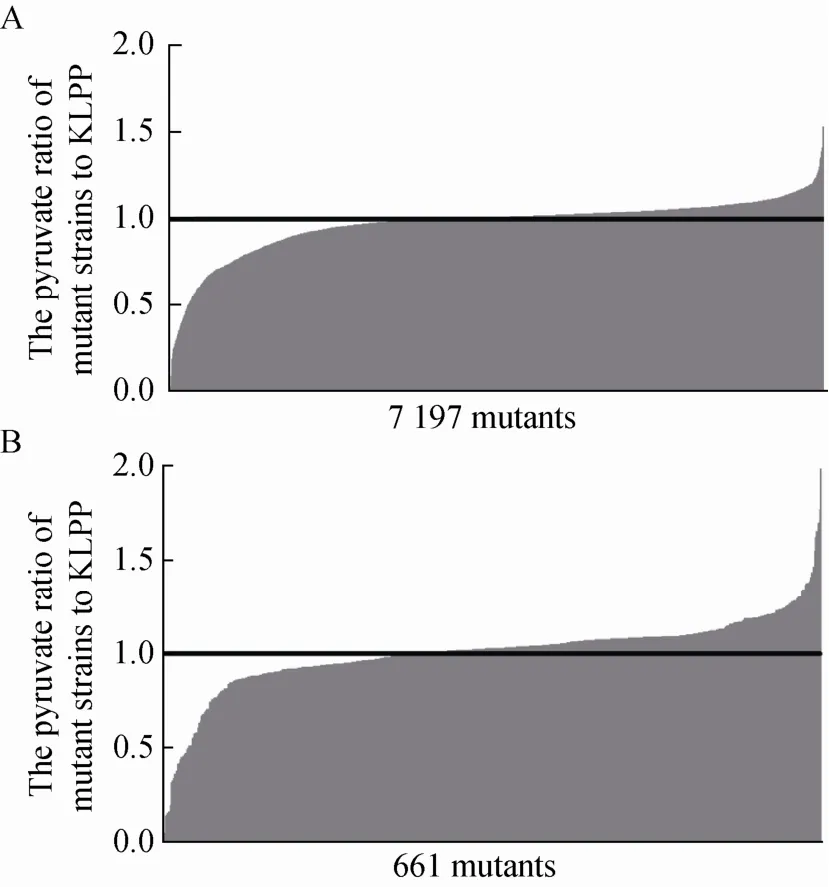
图1 突变菌株的丙酮酸浓度/出发菌株KLPP比值Fig. 1 The pyruvate concentration of the mutant strain/starting strain KLPP. (A) The ratio of pyruvate of 7197 mutant strains (Solid black line: OD520 is 1). (B)The ratio of pyruvate of 661 mutant strains (Solid black line: OD520 is 1).
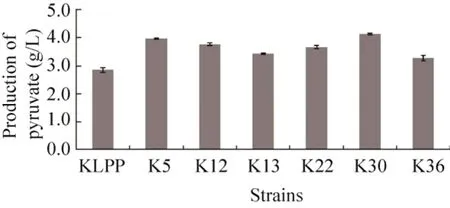
图2 不同突变菌株的丙酮酸产量Fig. 2 Production of pyruvate by different mutant strains.
2.5 突变株K30的发酵罐培养
为进一步检测筛选菌株的丙酮酸生产效果,我们选取摇瓶验证丙酮酸产量提高最多的菌株K30进行了5 L发酵罐的分批补料发酵。结果如图 3所示,发酵 47 h,丙酮酸产量可以积累到63 g/L,转化率在50%,在放大到5 L发酵罐时发酵丙酮酸产量较出发菌株KLPP提升约40%。
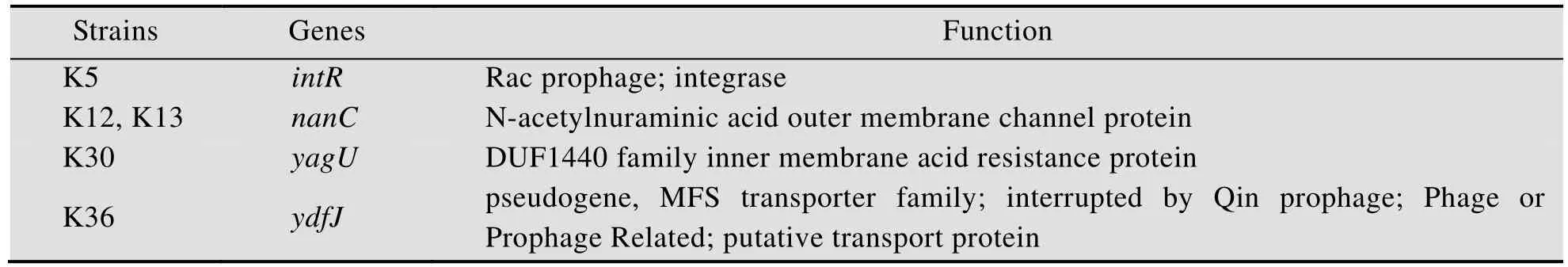
表3 转座子插入位点的鉴定Table 3 Identification of the transposon insertion sites

图3 K30分批补料发酵Fig. 3 Results of fed batch process using K30.
3 讨论
我们首先以大肠杆菌MG1655出发,经过敲除乙酸和乳酸积累的途径,获得了积累丙酮酸的菌株KLPP,此菌株在摇瓶发酵有3 g/L的水平,放大到5 L发酵罐时,丙酮酸产量可以达到45 g/L,摇瓶丙酮酸积累较低,主要原因是丙酮酸为酸性物质,即使有缓冲体系存在,后期pH也下降至5左右,细胞停止生长,因此我们后续高通量筛选的检测时间控制在pH过低之前,以区别不同突变株的发酵能力。
为了提高丙酮酸的产量,并发现可能影响丙酮酸积累的基因,我们以KLPP为出发菌株,使用 Tn5转座子突变的方法构建了 1个突变体文库,然后对突变库进行基于酶标仪的快速筛选,成功地筛选到了丙酮酸产量稳定提高的突变菌株。Tn5转座插入为单插入突变,可以克服利用传统诱变的方法难以对发生有效突变的菌株进行快速基因位点定位的缺点。通过筛选得到丙酮酸产量提高的突变菌株,进一步定位突变菌株的Tn5插入位点发现了4个可能影响丙酮酸产量的基因nanC、yagU、ydfJ和intR。
其中nanC基因编码N-乙酰神经氨酸外膜通道蛋白[20]。nanC(yjhA) 是yjhATS操纵子中的第一个基因。有研究报道nanC基因编码外膜特异性通道的KdgM家族的蛋白质,nanC的表达是由N-乙酰神经氨酸诱导,同时受N-乙酰葡萄糖胺调节[21]。大肠杆菌能够使用N-乙酰神经氨酸作为唯一的碳源生长,当缺失孔蛋白 OmpF和OmpC时,NanC是N-乙酰神经氨酸进入胞内的唯一通道。但是nanC与丙酮酸产量提高的关系还不清楚,需要进一步研究。
yagU编码一种DUF1440家族内膜酸抗性蛋白,在高度酸诱导时,大肠杆菌K-12 W3110中的yagU的表达量快速提高,当yagU缺失时,细胞在高酸度下 (pH 2) 存活能力显著降低,抗酸性降低3倍左右[22-23]。丙酮酸生产菌在快速积累丙酮酸时发酵液pH处于快速降低过程,在发酵末期时发酵pH低于5,细胞已基本不生长。但仍不清楚酸抗性蛋白的缺失与促进丙酮酸的积累是否有关。
在大肠杆菌的基因组注释中ydfJ基因有多个注释:MFS超家族 (Major facilitator superfamily)转运蛋白[24],一种可能的转运蛋白,噬菌体或原噬菌体相关基因;Tang等[25]研究发现ydfJ与哺乳动物钾通道基因亚基Kir6.1具有8.6%的同源性,与Kir6.2基因具有8.3%的同源性,将ydfJ在哺乳动物细胞中表达可以产生一个新的 K+通道。但是ydfJ基因的缺失与丙酮酸的积累是否相关还不清楚。
intR基因组注释为Rac原噬菌体或整合酶,根据UniProt上的功能注释,intR有助于噬菌体定点整合到宿主基因组上,在和切除酶(Excisionase) 共同作用时有助于原噬菌体从宿主基因组剪切下来。到目前为止还没有文献报道该基因的详细功能。
我们用 Tn5转座子突变库筛选到的可能影响丙酮酸合成基因,根据文献分析都没有找到其与丙酮酸积累的关系,但是分析这几个基因发现,4个基因中有3个明确与细胞膜相关,intR没有研究确定是否与膜相关。这其中2个是与转运相关,2个与原噬菌体相关。几个基因功能比较集中,但是与丙酮酸积累的关系仍需进一步研究。
K12和K13的Tn5转座子插入位置相同,但产量有较明显差异,通过进一步分析两株菌的基因组差异,发现K13相对K12在cpdB基因位置有点突变,cpdB基因是一个有647个氨基酸的环核苷酸磷酸二酯酶,其481位色氨酸密码子TGG突变为终止密码子TAG而提前终止,这可能是造成K12和K13菌株丙酮酸产量差异的原因,但并没有报道发现cpdB和丙酮酸积累有关,还需要进一步研究。
Tn5转座子插入突变是一种早就认识的遗传学机制,通常被用于发现特定功能的未知基因,主要应用于微生物遗传学研究。我们把这种方法应用到菌株的遗传改造过程中,为高产菌株构建寻找有效的靶点,相比于传统的诱变,它没有大量的无效突变背景干扰,将是代谢工程的一种有效策略。
[1] Guo Y, Fu T. A tentative study of the pyruvic acid industrialization in zymotechnics. J Shijiazhuang Vocat Technol Inst, 2010, 22(2): 26–28 (in Chinese).郭英, 付涛. 发酵法生产丙酮酸的工艺条件优化研究. 石家庄职业技术学院学报, 2010, 22(2):26–28.
[2] Shen DQ, Feng XY, Lin DQ, et al. Effect of different carbon sources on pyruvic acid production by usinglpdAgene knockoutEscherichiacoli. Chin J Biotech, 2009, 25(9): 1345–1351 (in Chinese).沈冬钱, 冯晓雨, 林东强, 等. 不同碳源对大肠杆菌lpdA突变菌累积丙酮酸的影响. 生物工程学报, 2009, 25(9): 1345–1351.
[3] Ma CQ. The study on pyruvate production by biocatalysis [D]. Ji’nan: Shandong University, 2003(in Chinese).马翠卿. 生物催化法生产丙酮酸(盐)的研究[D].济南: 山东大学, 2003.
[4] Liu LM, Li Y, Du GC, et al. Progress in biotechnological production of pyruvic acid. Chin J Biotech, 2002, 18(6): 651–655 (in Chinese).刘立明, 李寅, 堵国成, 等. 生物技术法生产丙酮酸的研究进展. 生物工程学报, 2002, 18(6):651–655.
[5] Han GD, Zhao SW, Li SW, et al. Handbook of Organic Preparation Chemistry (II). Beijing: China Petroleum Chemical Industry Press, 1977: 235–236(in Chinese).韩广甸, 赵树纬, 李述文, 等. 有机制备化学手册(中册). 北京: 石油化学工业出版社, 1977:235–236.
[6] Zhu YH, Eiteman MA, Altman R, et al. High glycolytic flux improves pyruvate production by a metabolically engineeredEscherichiacolistrain.Appl Environ Microbiol, 2008, 74(21): 6649–6655.
[7] Causey TB, Shanmugam KT, Yomano LP, et al.EngineeringEscherichiacolifor efficient conversion of glucose to pyruvate. Proc Natl Acad Sci USA, 2004, 101(8): 2235–2240.
[8] Yu Y, Xu MX, Liu JL, et al. Metabolic engineering for modifyingCorynebacteriumglutamicumto produce more pyruvate. Biotechnol Bull, 2016,32(8): 226–232 (in Chinese).于莹, 许湄雪, 刘金雷, 等. 代谢工程改造谷氨酸棒杆菌生成丙酮酸. 生物技术通报, 2016, 32(8):226–232.
[9] Kawata Y, Nishimura T, Matsushita I, et al. Efficient production and secretion of pyruvate fromHalomonassp. KM-1 under aerobic conditions.AMB Express, 2016, 6: 22, doi:10.1186/s13568-016-0195-y.
[10] Kamzolova SV, Morgunov IG. Biosynthesis of pyruvic acid from glucose byBlastobotrys adeninivorans. Appl Microbiol Biotechnol, 2016,100(17): 7689–7697.
[11] Zelić B, Gostović S, Vuorilehto K, et al. Process strategies to enhance pyruvate production with recombinantEscherichiacoli: from repetitive fed-batch toin situproduct recovery with fully integrated electrodialysis. Biotechnol Bioeng, 2004,85(6): 638–646.
[12] Tomar A, Eiteman MA, Altman E. The effect of acetate pathway mutations on the production of pyruvate inEscherichiacoli. Appl Microbiol Biotechnol, 2003, 62(1): 76–82.
[13] Reznikoff WS, Goryshin IY, Jendrisak JJ. Tn5 as a molecular genetics tool:in vitrotransposition and the coupling ofin vitrotechnologies within vivotransposition//Miller WJ, Capy P, Eds. Mobile Genetic Elements: Protocols and Genomic Applications. New Jersey: Humana Press, 2004: 83–96.
[14] Wang JL, Wu B, Qin H, et al. EngineeredZymomonasmobilisfor salt tolerance using EZ-Tn5-based transposon insertion mutagenesis system. Microb Cell Fact, 2016, 15: 101.
[15] Lin Z, Dong HJ, Li Y. Improvement of butanol production byEscherichiacolivia Tn5 transposon mediated mutagenesis. Chin J Biotech, 2015,31(12): 1711−1719 (in Chinese).林兆, 董红军, 李寅. 利用 Tn5转座子介导突变提高大肠杆菌丁醇生产水平. 生物工程学报,2015, 31(12): 1711−1719.
[16] Reznikoff WS. Transposon Tn5. Annu Rev Genet,2008, 42(1): 269–286.
[17] Goryshin IY, Jendrisak J, Hoffman LM, et al.Insertional transposon mutagenesis by electroporation of released Tn5 transposition complexes. Nat Biotechnol, 2000, 18(1): 97–100.
[18] Liu YL, Yang MH, Chen JJ, et al. PCR-based seamless genome editing with high efficiency and fidelity inEscherichiacoli. PLoS ONE, 2016, 11(3):e0149762, doi: 10.1371/journal.pone. 0149762.
[19] Eze LC, Echetebu CO. Some properties of aspartate and alanine aminotransferases fromTrichoderma virie. J Gen Microbiol, 1980, 120(2): 523–527.
[20] Zhai YF, Saier MH Jr. The β-barrel finder (BBF)program, allowing identification of outer membrane β-barrel proteins encoded within prokaryotic genomes. Protein Sci, 2002, 11(9): 2196–2207.
[21] Condemine G, Berrier C, Plumbridge J, et al.Function and expression of anN-acetylneuraminic acid-inducible outer membrane channel inEscherichiacoli. J Bacteriol, 2005, 187(6):1959–1965.
[22] Hayes ET, Wilks JC, Sanfilippo P, et al. Oxygen limitation modulates pH regulation of catabolism and hydrogenases, multidrug transporters, and envelope composition inEscherichiacoliK-12.BMC Microbiol, 2006, 6: 89.
[23] Kannan G, Wilks JC, Fitzgerald DM, et al. Rapid acid treatment ofEscherichiacoli: transcriptomic response and recovery. BMC Microbiol, 2008, 8: 37.
[24] Pao SS, Paulsen IT, Saier MH Jr. Major facilitator superfamily. Microbiol Mol Biol Rev, 1998, 62(1):1–34.
[25] Tang GH, Jiang B, Huang Y, et al. Identification of a novel bacterial K+channel. J Membr Biol, 2011,242(3): 153–164.
