工业微生物的噬菌体感染与防治策略
2017-04-16沈俊涛修志龙
沈俊涛,修志龙
大连理工大学 生命科学与技术学院,辽宁 大连 116024
在人类社会从石油经济向生物质经济转型的过程中,以细菌为基础的生物转化过程必将扮演越来越重要的角色。噬菌体感染是生物转化过程中一直存在的一个问题,至今仍无良策,其实质是噬菌体与细菌之间复杂的共进化关系。随着对噬菌体与宿主相互作用关系认识的深入,尤其是近年来CRISPR-Cas (Clustered regularly interspaced short palindromic repeats (CRISPR)-associated proteins) 和群体感应信号分子对细菌的噬菌体防御作用的发现,为这一问题的解决带来了更多希望。因此,本文从噬菌体与宿主的相互作用关系入手,对工业微生物领域急需解决的噬菌体感染问题进行了综述。
1 噬菌体及其感染的危害
噬菌体是一类感染细菌并在细菌内完成复制的病毒。病理学家 Twort和微生物学家D’Herelle在1915年和1917年分别独立发现了噬菌体[1-2]。一个世纪以来,围绕噬菌体的相关研究吸引了各领域大批研究者的关注,这些研究对生物学尤其是分子生物学的发展起到了举足轻重的作用[3]。这其中包括近来引发基因编辑领域革命的 CRISPR-Cas系统的发现[4]。现在的研究表明噬菌体是地球上最丰富的生命体,在地球的物质循环、细菌的进化和致病性等方面都扮演着重要的角色[5]。
然而,噬菌体感染一直是微生物发酵过程中最为常见的感染问题,会给发酵企业带来灾难性损失,并且至今没有良策[6-8]。噬菌体的几个特征决定了噬菌体感染的普遍性和顽固性:1) 噬菌体在自然界中有着极高的丰富性和多样性,数量在细菌的10倍之上[5]。2) 除了以游离态存在于环境的外源噬菌体外,内源性噬菌体 (来源于细菌自身染色体上噬菌体序列的诱导) 也是常见的感染形式[9]。3) 噬菌体体积小,可以顺利通过一般的膜过滤系统 (如0.22 μm),易于扩散[7]。4) 噬菌体基因组具有高度的可塑性 (噬菌体基因组的典型特征是模块化的组织结构,不同模块往往具有完全不同的来源[10]) 和较快的增殖速率 (噬菌体一次感染可以繁殖几十个甚至几百个子代噬菌体[11]),能够通过突变和重组快速适应多种不利因素。5) 在漫长的进化过程中,噬菌体已经形成了多种针对细菌抗性系统的逃逸机制[12]。
事实上,噬菌体可以感染任何利用细菌或以细菌为产品的微生物发酵过程[8]。在乳制品行业,对这一问题的认识和研究是最为详细的,如 1935年研究者就发现了噬菌体感染现象,然而,80多年过去了,时至今日这一问题并没有得到有效解决,噬菌体感染仍然是乳制品行业发酵失败的主要原因[13]。在丙酮丁醇的工业化发酵过程中,噬菌体感染也在全球多个地方 (包括美国、日本、南非等) 有过不同程度的暴发,结果往往导致一个工厂长时间停产甚至是彻底关闭[14]。据国内相关研究团队报告,噬菌体感染在苏云金芽孢杆菌发酵过程中的发生率曾高达15%–30%,即使后来采取了相应的防范措施 (包括合理的工厂设计和抗性菌株筛选),噬菌体感染的发生率仍在 1%左右[15-17]。本课题组曾在和某公司合作进行的克雷伯杆菌千吨级规模的中试发酵产1,3-丙二醇的试验中发现了噬菌体感染现象[18],结果往往导致发酵过程中菌体大量死亡,发酵长时间停滞甚至是彻底失败,大大降低了克雷伯杆菌发酵产1,3-丙二醇过程的经济性[19]。在这些噬菌体感染的案例中,来源于环境的噬菌体和来源于宿主自身染色体上的噬菌体诱导共同导致了噬菌体感染的普遍性和顽固性[8-9]。关于噬菌体感染的报道较少的主要原因是噬菌体感染是一个阴性结果,一般的学术期刊并不支持这样的结果发表,并且企业也不愿意将失败的结果公布于众。但在实际的发酵生产过程中,噬菌体感染经常发生,难以控制。虽然合理的工厂设计可以减少噬菌体感染,但是并不能彻底清除。更糟糕的是,一旦噬菌体感染在工厂中暴发就会迅速扩散,从而给发酵工厂带来巨大的损失,因为不仅要对所有设备进行彻底的消毒,还要对菌种进行重新选育,这往往会导致工厂长时间停产甚至破产。而且微生物发酵尤其是大宗化学品的发酵每一批次的规模都达成百上千立方米,因此每一次的感染倒罐都会带来巨大的经济损失[20-21]。
2 噬菌体感染及其与宿主的共进化机制
2.1 噬菌体的感染、繁殖机制
噬菌体感染一般包括6个阶段:噬菌体吸附、DNA注入、DNA复制、转录和翻译、噬菌体组装和释放[22]。所有噬菌体都需要依赖宿主完成自己的生活史。根据生活史的不同,可以将这些噬菌体分为几类,其中大部分属于烈性噬菌体或温和噬菌体[23]。烈性噬菌体感染一般都会经历上述六个阶段;而温和噬菌体感染后还可以将其基因组整合在宿主染色体上,并随着宿主的繁殖而复制、传代,在某些因素的刺激下也可以从染色体上解离并导致宿主裂解。另外,也有少部分噬菌体处于假溶原状态或噬菌体携带状态,这是一种噬菌体和宿主共存的状态[24],在这种状态下噬菌体既不会整合到宿主的染色体上也不会导致宿主裂解。区别在于,假溶原状态只会在宿主处于饥饿的环境时出现;而噬菌体携带状态则可以出现在营养丰富的条件下[25]。
2.2 噬菌体和宿主的共进化关系
在自然界中,噬菌体和宿主总是共同存在和进化的[24]。噬菌体抗性菌株可以帮助宿主逃过噬菌体的捕食,而新的噬菌体突变体又会继续威胁着宿主的安全[23]。在这一过程中,宿主的噬菌体抗性机制和噬菌体的适应机制扮演着重要的角色 (图1)[26]。
2.2.1 宿主的噬菌体抗性机制
从免疫学的角度看,细菌的噬菌体抗性机制可以分为两大类:固有免疫和获得性免疫[22]。前者出现在噬菌体生活史的各个阶段,包括阻止噬菌体吸附,阻止DNA注入、剪切噬菌体核酸和流产感染系统[27]。后者主要针对噬菌体的核酸,目前,在细菌中发现了 CRISPR-Cas和 pAgo(Prokaryotic Argonaute family proteins) 两种获得性免疫系统[26,28]。
噬菌体吸附是其感染的第一个阶段,细菌可以通过几种方式来阻止噬菌体吸附[22],包括吸附受体的丢失或表达下调、受体的突变和受体的空间阻遏。阻止噬菌体DNA注入一般是通过超感染排斥蛋白来完成的[29],这类蛋白常常由前噬菌体编码。而当噬菌体的核酸注入细胞后,限制性修饰系统会对其进行剪切,从而破坏噬菌体感染[30]。另外,也有些细菌在感染噬菌体后会启动程序性死亡[31],从而避免噬菌体的繁殖和扩散,俗称流产感染系统。流产感染系统主要是通过宿主中的一对毒性和抗毒性蛋白来实现的,正常情况下这两种蛋白结合在一起不表现毒性,当噬菌体入侵时会导致不稳定的抗毒性蛋白失活,在毒性蛋白的作用下引发宿主死亡,从而终止噬菌体的繁殖和复制[32]。CRISPRCas和pAgo两种获得性免疫系统都是针对入侵的核酸的,都会在第一次入侵时获得一小段核酸片段,并利用Cas或Ago蛋白在DNA或RNA的引导下对二次入侵的核酸进行定向切割[33-34],从而导致噬菌体或质粒的感染失败。区别在于前者仅以RNA为向导[35],后者既可以利用DNA也可以利用RNA作为向导[36]。
另外,最近的研究也显示,细菌的噬菌体抗性机制并不只是单个细胞的孤立行为,而是会受到群体信号分子的调控[37]。在自然界中,群体信号分子和噬菌体都对菌群密度的调节起着关键的作用[38]。研究表明Ⅰ类群体信号分子AHL (高丝氨酰酯类群体信号分子) 可以减少大肠杆菌表面噬菌体受体的表达,从而减小 λ和 χ的吸附[39]。在鳗弧菌的研究中,两种噬菌体抗性策略的选择 (噬菌体受体表达降低和生物膜形成增加) 也是由Ⅰ类群体信号分子 AHL所决定的[40]。Patterson和 Høyland-Kroghsbo等最新的研究则显示群体信号分子还可以通过调控CRISPR-Cas系统的活性增强细菌在高密度时的噬菌体抗性[41-42]。
2.2.2 噬菌体的适应机制
尽管细菌有多种噬菌体抗性策略,在自然界中,噬菌体仍然可以以10倍于细菌的数量存在[5]。这说明噬菌体可以成功地避开细菌的多种抗病毒系统[12]。而这主要是由于噬菌体基因组的高度可塑性和较快的增殖速率。
噬菌体的吸附过程主要是由噬菌体的受体结合蛋白和宿主表面的受体分子 (主要是多糖和脂多糖) 的特异性结合实现的[43]。噬菌体可以分泌一些水解酶去破坏覆盖在细胞表面的荚膜或多聚糖类物质等,而挖掘出受空间阻遏的受体,也可以通过修饰受体结合蛋白 (包括蛋白酶的剪切,基因的突变、重组和多拷贝等) 而适应全新的受体[12]。针对细菌中广泛存在的限制性修饰系统,噬菌体主要有 3种应对方式[44]:一是限制性内切酶识别位点的突变、低表达和空间阻遏;二是利用宿主的或自身携带的甲基化修饰酶对限制性酶切位点进行修饰;三是抑制宿主限制性修饰系统的活性。针对一些细菌中存在的流产感染系统,噬菌体可以通过某些特定基因的突变来避免激活该系统,也可以分泌抗毒性蛋白类似物,从而中和毒性蛋白的作用[45]。
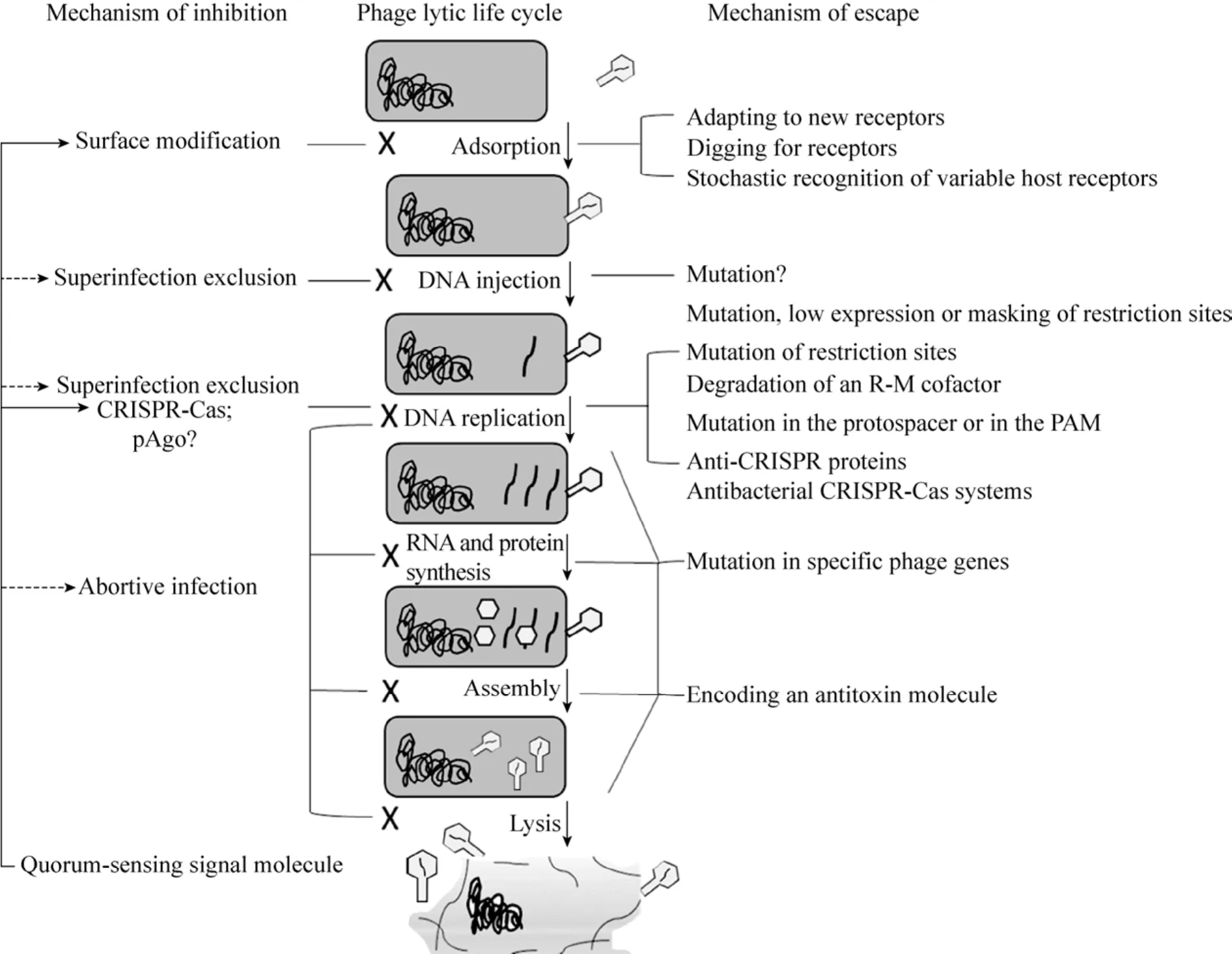
图1 细菌的抗性机制和噬菌体的逃逸机制[6,12,22,26]Fig. 1 The bacterial antiviral systems and the escape of bacteriophages[6,12,22,26]. Short-dash line and question mark indicate that they have not been proved.
针对细菌的CRISPR-Cas系统,目前发现了3种不同的噬菌体逃逸机制[12]。第一类是通过噬菌体前间隔序列或前间隔区邻接基序 (PAM) 的突变使CRISPR RNA (crRNA) 与噬菌体DNA无法配对,从而使CRISPR干扰失败[46-47]。这是一种广泛存在的、可针对各种CRISPR-Cas系统的噬菌体逃逸机制[48]。不过,即使原来的间隔序列失效了,CRISPR-Cas系统仍然可以继续从噬菌体DNA上获得新的间隔序列,从而重新对噬菌体产生抗性[8]。事实上,CRISPR-Cas系统可以从同一个噬菌体上获得多个间隔序列,从而对噬菌体产生更强的抗性。正是这种可叠加性赋予了CRISPR-Cas系统独特的优势。最新的研究也表明,当一个种群内存在足够多的针对一个噬菌体的间隔序列时,噬菌体并不能通过突变来逃逸[49]。此时,噬菌体或许可以利用第二类逃逸机制,即通过噬菌体编码的蛋白 (Anti-CRISPR蛋白) 干扰CRISPR-Cas系统的活性[50-52]。目前,已经发现了针对I-E、I-F和II-C型CRISPR-Cas系统的多个Anti-CRISPR蛋白[50,53-54]。其中针对I-F型的3类Anti-CRISPR蛋白有不同的作用机制[55-56],AcrF1和AcrF2干扰CRISPR-Cas复合物的形成,AcrF3干扰 Cas3的剪切活性,针对II-C型的Anti-CRISPR蛋白可以干扰Cas9的剪切活性[54]。第3种情况是有一些噬菌体可以劫持宿主的CRISPR-Cas系统以完成自身的繁殖[57]。如在霍乱弧菌噬菌体中发现的一个 CRISPR-Cas系统,其可以失活一个由噬菌体诱导的染色体岛元件编码的抗噬菌体系统,从而帮助噬菌体完成自身增殖[58]。
2.3 噬菌体的专一性
噬菌体吸附是其成功感染的第一步,噬菌体的专一性一般认为是由其吸附特异性决定的,而噬菌体尾部蛋白在这一过程中起着决定性作用[43,59]。有研究显示仅通过替换尾部蛋白基因就可以实现大肠杆菌、克雷伯杆菌、耶尔森氏杆菌噬菌体专一性的相互替换[60]。
传统的观点认为噬菌体具有高度的专一性,即一个噬菌体一般只能够感染某个特定种类的细菌甚至是某些特定的菌株[3]。但越来越多的研究显示噬菌体的敏感性和抗性不是绝对的,不是一个非此即彼的概念[61]。尤其是在自然环境中,更多的时候可能是通过噬菌体抗性水平的差异而不是抗性的有无来实现菌群的动态平衡[62]。宿主中存在的多种噬菌体的抗性机制可能是造成这种认识差异的原因[63],例如一个能够正常吸附的噬菌体可能由于其他抗性机制的存在而并不能正常繁殖,而在实验室中这种情况往往被误认为是噬菌体的宿主专一性导致的。
3 工业发酵中噬菌体感染防治策略
3.1 控制感染源
感染源的控制是预防噬菌体感染的第一步[21]。为了控制噬菌体的来源,首先要保证环境的清洁。工厂最好选在实验菌种丰度较低的环境,并对所有发酵相关设备和管道等进行灭菌处理,尤其要注意空气系统的灭菌处理,因为噬菌体体积小,常用的膜过滤系统并不能有效去除噬菌体。其次要建立噬菌体的日常检查制度,及时发现噬菌体感染,避免大规模暴发。内源性感染可能是在对工厂环境进行严格控制后仍然无法避免噬菌体感染的一个重要原因。尽管人们已经认识到内源性感染 (来源于细菌自身染色体上噬菌体的诱导) 是工业发酵过程中噬菌体感染的重要因素,但在实际生产过程中对发酵起始菌种中的内源性噬菌体仍然缺少关注。
3.2 菌株轮换
菌株轮换即通过不同噬菌体敏感类型菌株的轮换使用来降低噬菌体感染的风险和噬菌体感染带来的损失[6]。这是一种工业中常用的策略。不过,这一过程随机性很大,选育的抗噬菌体菌株的生产能力可能会降低或丢失。而且噬菌体很容易通过变异来适应之前的抗性菌。一个经典的例子是,在一个的丙酮丁醇工厂中,一年之内连续换了 12个菌株最终仍没能逃过噬菌体感染的命运[64]。
3.3 传统的基因工程策略
在噬菌体感染的各个阶段都可以采取相应的基因工程方法阻止噬菌体增殖[6]。例如,可以通过对宿主菌噬菌体受体的修饰来干扰吸附过程,也可以通过人工表达一些超感染排斥蛋白来阻止噬菌体DNA注入,还可以通过人工构建的流产感染系统去破坏噬菌体的增殖。在嗜热链球菌中利用高拷贝质粒表达一个超感染排斥蛋白 orf203可以使菌株的抗性水平显著增强(EOP≤10–3)[65]。在乳酸链球菌中表达一个自杀性的流产感染系统[66],当噬菌体感染时会激活该系统,从而使噬菌体感染失败,菌株抗性水平达到EOP≤10–4。在嗜热链球菌中也有研究采用反义RNA技术干扰噬菌体的翻译过程[67-68],依赖于所针对的基因,宿主的抗性水平提高到EOP=5×10–1到 2×10–3不等。不过以上这些策略都需要对噬菌体和宿主的相互作用关系有深入的认识,而目前人们仅对几种常见的噬菌体和宿主有一定的了解。另外,由于噬菌体基因组的高度可塑性和快速增殖的能力,噬菌体可以通过突变快速适应这些噬菌体抗性系统,这就给传统的噬菌体抗性菌株构建带来了巨大的 困难。
3.4 基于CRISPR-Cas系统的噬菌体防治策略
CRISPR-Cas系统是RNA引导的细菌获得性免疫系统,可用来对抗外来遗传物质的入侵[4]。仅需改变 20 bp左右的核酸序列就可以实现CRISPR-Cas系统的特异性切割,因此该系统可以方便地用来进行核酸修饰,目前基于这一系统的基因组编辑技术已在动物、植物、微生物和病毒等多种生命体中有了应用[69-70]。基于CRISPR-Cas系统的噬菌体抗性系统也有着极大的优势[33]。在大肠杆菌[71]和枯草芽孢杆菌[72]中,成功地利用外源CRISPR-Cas9的系统构建了噬菌体抗性菌株,它们的抗性水平分别提高到了 EOP=5×10–2和2×10–6。在嗜热链球菌中,利用天然的CRISPR-Cas系统,宿主的噬菌体抗性达到了EOP=10–4和10–5的水平,并且通过两个靶向gRNA序列的联用,抗性水平可进一步提高到 EOP=10–6到 10–7的水平[4]。另外,对获得的基于 CRISPR-Cas系统的抗性菌株的全基因组分析也表明,在噬菌体感染前后宿主基因组仅有几个单核苷酸突变和小片段缺失[73],这说明基于CRISPR-Cas的免疫对宿主基因组不会产生明显的影响。这对于构建基于CRISPR-Cas的噬菌体抗性菌株至关重要。正如对抗其他噬菌体抗性机制一样,噬菌体也可以通过快速的突变来逃避CRISPR-Cas系统的免疫作用[46-47]。不过最近的研究表明,只要靶向的gRNA序列多样性足够多,噬菌体就无法通过突变来逃避CRISPR-Cas系统的剪切作用[49]。
综上所述,基于CRISPR-Cas的噬菌体抗性系统有以下优点 (表1):1) 可理性设计,仅需要得到噬菌体的序列信息就可以理性设计噬菌体抗性菌;2) 操作简便,仅需改变20 bp的核酸序列就可以对特定序列实现特异性的剪切;3) 易开发广谱性的噬菌体抗性菌株,仅需对不同噬菌体的保守基因进行靶标设计就可以实现对多个噬菌体的免疫;4) 具有可叠加效应,噬菌体抗性水平可以通过靶向序列多样性的增加来增强;5) 基于 CRISPR-Cas的免疫对宿主基因组几乎没有影响,有利于保持菌株的发酵性能。
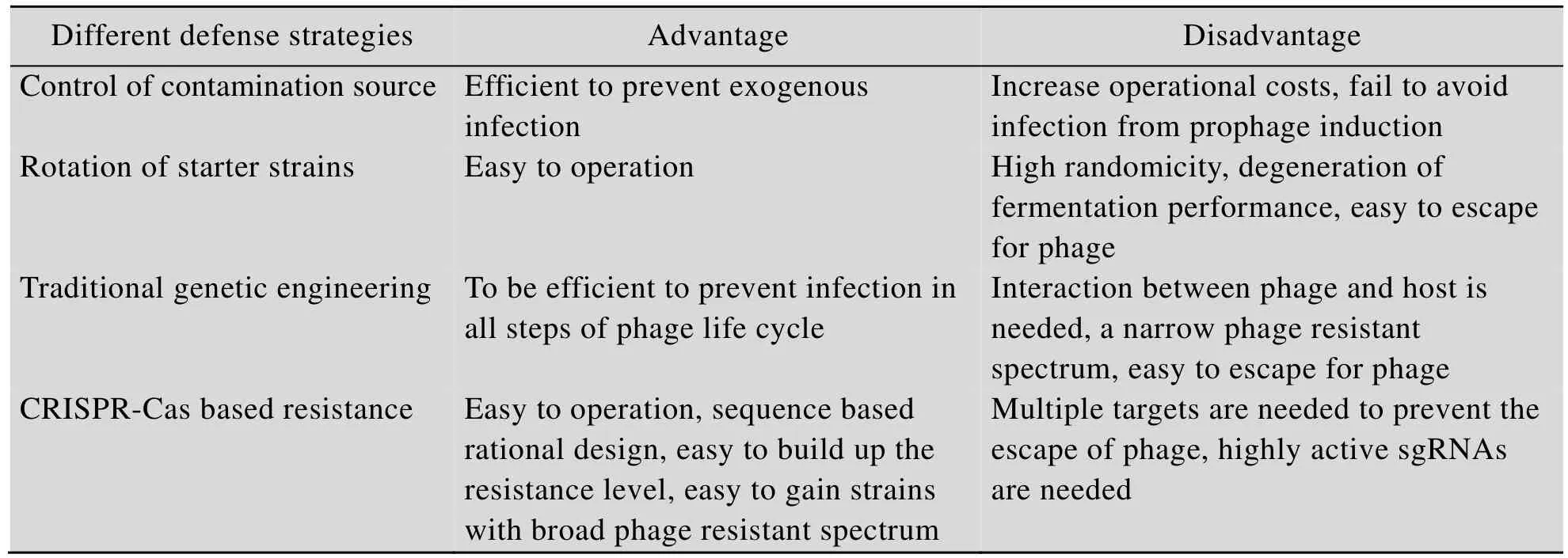
表1 不同噬菌体防治策略的比较Table 1 Different strategies to prevent phage infection
4 展望
在自然界中,噬菌体与宿主总是共同存在和进化的,两者之间存在着复杂的抗性和逃逸机制[28,31]。噬菌体感染是以细菌为基础的生物技术发展过程中的一个棘手的问题。深入理解噬菌体与宿主之间的相互作用关系是解决这一问题的关键。
近来,噬菌体基因组编辑技术的快速发展或许能帮助我们更好地认识噬菌体[74-75]。这些技术包括:1) 基于电穿孔 DNA的噬菌体重组工程(Bacteriophage recombineering of electroporated DNA,BRED)[76],其主要原理是将噬菌体 DNA和模板 DNA同时通过电穿孔技术导入宿主的感受态细胞,然后通过PCR等技术鉴定突变体;2) 基于酵母或细菌人工染色体的同源重组技术[60],其主要原理是将噬菌体基因组DNA插入到酵母人工染色体上,利用酵母的同源重组功能完成对噬菌体基因组的修饰,最后将含有噬菌体完整基因组的酵母人工染色体导入到宿主细胞完成噬菌体的繁殖;3) 基于CRISPR-Cas的基因组编辑技术[77-78],其主要原理是先构建一个基于CRISPR-Cas的噬菌体抗性菌株,并导入同源重组模板,噬菌体感染后会在CRISPR-Cas系统的作用下发生重组。最近,有研究者通过基于酵母人工染色体的同源重组技术对多个肠道菌属噬菌体 (包括大肠杆菌、克雷伯杆菌和耶尔森氏杆菌) 的尾部蛋白进行替换[60],成功实现了噬菌体特异性的替换。可以预见,噬菌体基因组编辑技术的快速发展必将极大地促进我们对噬菌体的理解,从而更好地防治噬菌体。
合成生物学是另一个可能极大改变我们对噬菌体认识的领域[79-80]。由于噬菌体的繁殖依赖于宿主的蛋白合成系统,一旦宿主的密码子和噬菌体的不匹配,噬菌体或将不能够成功增殖[81]。已有研究发现将大肠杆菌的所有终止密码子UAG替换成UAA可以增强宿主对T7噬菌体的抗性[82]。如果更大范围地改变宿主的密码子或许能从根本上避免噬菌体的感染[80,83]。合成生物学的快速发展使得成本越来越低[84],或许在不久的将来,人们就能够获得人工合成的免受噬菌体感染的全新细胞。
[1] Twort FW. An investigation on the nature of ultra-microscopic viruses by Twort FW, L.R.C.P.Lond., M.R.C.S. (From the Laboratories of the Brown Institution, London). Bacteriophage, 2014,1(3): 127–129.
[2] D’Herelle F. On an invisible microbe antagonistic toward dysenteric bacilli: brief note by Mr. F.D’Herelle, presented by Mr. Roux. 1917. Res Microbiol, 2007, 158(7): 553–554.
[3] Salmond GPC, Fineran PC. A century of the phage:past, present and future. Nat Rev Microbiol, 2015,13(12): 777–786.
[4] Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709–1712.
[5] Paez-Espino D, Eloe-Fadrosh EA, Pavlopoulos GA,et al. Uncovering Earth’s virome. Nature, 2016,536(7617): 425–430.
[6] Sturino JM, Klaenhammer TR. Engineered bacteriophage-defence systems in bioprocessing.Nat Rev Microbiol, 2006, 4(5): 395–404.
[7] Callanan MJ, Klaenhammer TR. Bacteriophages in industry//Encyclopedia of Life Sciences. Chichester:Wiley & Sons, Ltd, 2008.
[8] Mahony J, van Sinderen D. Novel strategies to prevent or exploit phages in fermentations, insights from phage-host interactions. Curr Opin Biotechnol,2015, 32: 8–13.
[9] Maniloff J. Bacteriophages//Encyclopedia of Life Sciences. Chichester: Wiley & Sons, Ltd, 2012.
[10] Pedulla ML, Ford ME, Houtz JM, et al. Origins of highly mosaic mycobacteriophage genomes. Cell,2003, 113(2): 171–182.
[11] Delbrück M. The burst size distribution in the growth of bacterial viruses (bacteriophages). J Bacteriol, 1945, 50(2): 131–135.
[12] Samson JE, Magadán AH, Sabri M, et al. Revenge of the phages: defeating bacterial defences. Nat Rev Microbiol, 2013, 11(10): 675–687.
[13] Samson JE, Moineau S. Bacteriophages in food fermentations: new frontiers in a continuous arms race. Annu Rev Food Sci Technol, 2013, 4:347–368.
[14] Jones DT, Shirley M, Wu XY, et al. Bacteriophage infections in the industrial acetone butanol (AB)fermentation process. J Mol Microbiol Biotechnol,2000, 2(1): 21–26.
[15] Yu ZN.Bacillus thuringiensis. Beijing: Science Press, 1990 (in Chinese).喻子牛. 苏云金杆菌. 北京: 科学出版社, 1990.
[16] Liao W, Song SY, Sun F, et al. Isolation,characterization and genome sequencing of phage MZTP02 fromBacillusthuringiensisMZ1. Arch Virol, 2008, 153(10): 1855–1865.
[17] Wu JX, Chen ZE, Zhang HZ. Studies on the effects of temperature and humidity on bacteriophage infection in fermentation ofBacillus thuringiensis.Chin J Biol Control, 1998, 14(3): 101–104 (in Chinese).吴继星, 陈在佴, 张汉珍. 苏云金杆菌发酵中环境温湿度对噬菌体污染影响的研究. 中国生物防治, 1998, 14(3): 101–104.
[18] Nan N, Cao F, Shen JT, et al. Characterization of a bacteriophage ofKlebsiellapneumoniaeproducing 1,3-propanediol. Acta Microbiol Sin, 2013, 53(9):943–949 (in Chinese).南南, 曹放, 沈俊涛, 等. 一株产1,3-丙二醇的克雷伯氏菌的噬菌体生物学特性. 微生物学报,2013, 53(9): 943–949.
[19] Shen JT, Zhou JJ, Fu HX, et al. AKlebsiella pneumoniaebacteriophage and its effect on 1,3-propanediol fermentation. Process Biochem,2016, 51(10): 1323–1330.
[20] Moineau S, Lévesque C. Control of bacteriophages in industrial fermentations//Kutter E, Sulakvelidze A. Bacteriophages: Biology and Applications. Boca Raton, FL: CRC Press, 2004.
[21] Łos M, Czyz A, Sell E, et al. Bacteriophage contamination: is there a simple method to reduce its deleterious effects in laboratory cultures and biotechnological factories? J Appl Genet, 2004,45(1): 111–120.
[22] Labrie SJ, Samson JE, Moineau S. Bacteriophage resistance mechanisms. Nat Rev Microbiol, 2010,8(5): 317–327.
[23] Shabbir MAB, Hao HH, Shabbir MZ, et al.Bacteria vs. bacteriophages: parallel evolution of immune arsenals. Front Microbiol, 2016, 7: 1292.[24] Diaz-Muñoz SL, Koskella B. Bacteria-phage interactions in natural environments. Adv Appl Microbiol, 2014, 89: 135–183.
[25] Cenens W, Mebrhatu MT, Makumi A, et al.Expression of a novel P22 ORFan gene reveals the phage carrier state inSalmonellatyphimurium.PLoS Genet, 2013, 9: e1003269.
[26] van Houte S, Buckling A, Westra ER. Evolutionary ecology of prokaryotic immune mechanisms.Microbiol Mol Biol Rev, 2016, 80(3): 745–763.
[27] Nechaev S, Severinov K. The elusive object of desire-interactions of bacteriophages and their hosts. Curr Opin Microbiol, 2008, 11(2): 186–193.
[28] Li TM, Du B. CRISPR-Cas system and coevolution of bacteria and phages. Hereditas (Beijing), 2011,33(3): 213–218 (in Chinese).李铁民, 杜波. CRISPR-Cas系统与细菌和噬菌体的共进化. 遗传, 2011, 33(3): 213–218.
[29] Lu MJ, Henning U. Superinfection exclusion by T-even-type coliphages. Trends Microbiol, 1994,2(4): 137–139.
[30] Tock MR, Dryden DTF. The biology of restriction and anti-restriction. Curr Opin Microbiol, 2005,8(4): 466–472.
[31] Chopin MC, Chopin A, Bidnenko E. Phage abortive infection in lactococci: variations on a theme. Curr Opin Microbiol, 2005, 8(4): 473–479.
[32] Dy RL, Przybilski R, Semeijn K, et al. A widespread bacteriophage abortive infection system functions through a type IV toxin-antitoxin mechanism. Nucleic Acids Res, 2014, 42(7):4590–4605.
[33] Barrangou R, Horvath P. CRISPR: new horizons in phage resistance and strain identification. Annu Rev Food Sci Technol, 2012, 3: 143–162.
[34] Hur JK, Olovnikov I, Aravin AA. Prokaryotic Argonautes defend genomes against invasive DNA.Trends Biochem Sci, 2014, 39(6): 257–259.
[35] Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol, 2015, 13(11): 722–736.
[36] Swarts DC, Makarova K, Wang YL, et al. The evolutionary journey of Argonaute proteins. Nat Struct Mol Biol, 2014, 21(9): 743–753.
[37] Debarbieux L. Bacterial sensing of bacteriophages in communities: the search for the Rosetta stone.Curr Opin Microbiol, 2014, 20: 125–130.
[38] Denes T, Wiedmann M. Environmental responses and phage susceptibility in foodborne pathogens:implications for improving applications in food safety. Curr Opin Biotech, 2014, 26: 45–49.
[39] Høyland-Kroghsbo NM, Maerkedahl RB,Svenningsen SL. A quorum-sensing-induced bacteriophage defense mechanism. mBio, 2013,4(1): e00362-12.
[40] Tan DM, Svenningsen SL, Middelboe M. Quorum sensing determines the choice of antiphage defense strategy inVibrio anguillarum. mBio, 2015, 6(3):e00627.
[41] Patterson AG, Jackson SA, Taylor C, et al. Quorum sensing controls adaptive immunity through the regulation of multiple CRISPR-Cas systems. Mol Cell, 2016, 64(6): 1102–1108.
[42] Høyland-Kroghsbo NM, Paczkowski J, Mukherjee S, et al. Quorum sensing controls thePseudomonas aeruginosaCRISPR-Cas adaptive immune system.Proc Natl Acad Sci USA, 2017, 114(1): 131–135.
[43] Moldovan R, Chapman-McQuiston E, Wu XL. On kinetics of phage adsorption. Biophys J, 2007,93(1): 303–315.
[44] Krüger DH, Bickle TA. Bacteriophage survival:multiple mechanisms for avoiding the deoxyribonucleic acid restriction systems of their hosts. Microbiol Rev, 1983, 47(3): 345–360.
[45] Sberro H, Leavitt A, Kiro R, et al. Discovery of functional toxin/antitoxin systems in bacteria by shotgun cloning. Mol Cell, 2013, 50(1): 136–148.
[46] Paez-Espino D, Sharon I, Morovic W, et al.CRISPR immunity drives rapid phage genome evolution inStreptococcusthermophilus. mBio,2015, 6(2): e00262–15.
[47] Levin BR, Moineau S, Bushman M, et al. The population and evolutionary dynamics of phage and bacteria with CRISPR-mediated immunity. PLoS Genet, 2013, 9(3): e1003312.
[48] Kennedy EM, Cullen BR. Bacterial CRISPR/Cas DNA endonucleases: a revolutionary technology that could dramatically impact viral research and treatment. Virology, 2015, 479–480: 213–220.
[49] van Houte S, Ekroth AKE, Broniewski JM, et al.The diversity-generating benefits of a prokaryotic adaptive immune system. Nature, 2016, 532(7599):385–388.
[50] Bondy-Denomy J, Pawluk A, Maxwell KL, et al.Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system. Nature, 2013, 493(7432):429–432.
[51] Maxwell KL. Phages fight back: inactivation of the CRISPR-Cas bacterial immune system by anti-CRISPR proteins. PLoS Pathog, 2016, 12(1):e1005282.
[52] Pawluk A, Staals RHJ, Taylor C, et al. Inactivation of CRISPR-Cas systems by anti-CRISPR proteins in diverse bacterial species. Nat Microbiol, 2016,1(8): 16085.
[53] Pawluk A, Bondy-Denomy J, Cheung VH, et al. A new group of phage anti-CRISPR genes inhibits the type I-E CRISPR-Cas system ofPseudomonas aeruginosa. mBio, 2014, 5(2): e00896.
[54] Pawluk A, Amrani N, Zhang Y, et al. Naturally occurring off-switches for CRISPR-Cas9. Cell,2016, 167(7): 1829–1838.e9.
[55] Bondy-Denomy J, Garcia B, Strum S, et al.Multiple mechanisms for CRISPR-Cas inhibition by anti-CRISPR proteins. Nature, 2015, 526(7571):136–139.
[56] Almendros C, Guzmán NM, García-Martínez J, et al. Anti-casspacers in orphan CRISPR4 arrays prevent uptake of active CRISPR-Cas I-F systems.Nat Microbiol, 2016, 1(8): 16081.
[57] Villion M, Moineau S. Virology: phages hijack a host’s defence. Nature, 2013, 494(7438): 433–434.
[58] Seed KD, Lazinski DW, Calderwood SB, et al. A bacteriophage encodes its own CRISPR/Cas adaptive response to evade host innate immunity.Nature, 2013, 494(7438): 489–491.
[59] Kutter E. Phage host range and efficiency of plating. Methods Mol Biol, 2009, 501: 141–149.
[60] Ando H, Lemire S, Pires DP, et al. Engineering modular viral scaffolds for targeted bacterial population editing. Cell Sys, 2015, 1(3): 187–196.
[61] Hyman P, Abedon ST. Bacteriophage host range and bacterial resistance. Adv Appl Microbiol, 2010,70: 217–248.
[62] Koskella B, Meaden S. Understanding bacteriophage specificity in natural microbial communities.Viruses, 2013, 5(3): 806–823.
[63] Sieber M, Gudelj I. Do-or-die life cycles and diverse post-infection resistance mechanisms limit the evolution of parasite host ranges. Ecol Lett,2014, 17(4): 491–498.
[64] Hongo M, Aono T, Murata A. Bacteriophages ofClostridiumsaccharoperbutylacetonicum: part IV.Serological characteristics of the twelve HM-phages.Agric Biol Chem, 1966, 30(4): 399–405.
[65] Bruttin A, Foley S, Brüssow H. DNA-binding activity of theStreptococcusthermophilusphage Sfi21 repressor. Virology, 2002, 303(1): 100–109.
[66] Djordjevic GM, O’Sullivan DJ, Walker SA, et al. A triggered-suicide system designed as a defense against bacteriophages. J Bacteriol, 1997, 179(21):6741–6748.
[67] Sturino JM, Klaenhammer TR. Expression of Antisense RNA Targeted againstStreptococcus thermophilusBacteriophages. Appl Environ Microbiol, 2002, 68(2): 588–596.
[68] Sturino JM, Klaenhammer TR. Antisense RNA targeting of primase interferes with bacteriophage replication inStreptococcusthermophilus. Appl Environ Microbiol, 2004, 70(3): 1735–1743.
[69] Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262–1278.
[70] Barrangou R, Doudna JA. Applications of CRISPR technologies in research and beyond. Nat Biotechnol, 2016, 34(9): 933–941.
[71] Sapranauskas R, Gasiunas G, Fremaux C, et al. TheStreptococcus thermophilusCRISPR/Cas system provides immunity inEscherichia coli. Nucleic Acids Res, 2011, 39(21): 9275–9282.
[72] Jakutyte-Giraitiene L, Gasiunas G. Design of a CRISPR-Cas system to increase resistance ofBacillussubtilisto bacteriophage SPP1. J Ind Microbiol Biotechnol, 2016, 43(8): 1183–1188.
[73] Barrangou R, Coûté-Monvoisin AC, Stahl B, et al.Genomic impact of CRISPR immunization against bacteriophages. Biochem Soc Trans, 2013, 41(6):1383–1391.
[74] Sagona AP, Grigonyte AM, MacDonald PR, et al.Genetically modified bacteriophages. Integr Biol(Camb), 2016, 8(4): 465–474.
[75] Pires DP, Cleto S, Sillankorva S, et al. Genetically engineered phages: a review of advances over the last decade. Microbiol Mol Biol Rev, 2016, 80(3):523–543.
[76] Marinelli LJ, Piuri M, Swigoňová Z, et al. BRED: a simple and powerful tool for constructing mutant and recombinant bacteriophage genomes. PLoS ONE, 2008, 3(12): e3957.
[77] Martel B, Moineau S. CRISPR-Cas: an efficient tool for genome engineering of virulent bacteriophages. Nucleic Acids Res, 2014, 42(14):9504–9513.
[78] Kiro R, Shitrit D, Qimron U. Efficient engineering of a bacteriophage genome using the type I-E CRISPR-Cas system. RNA Biol, 2013, 11(1):42–44.
[79] Lu TK, Koeris MS. The next generation of bacteriophage therapy. Curr Opin Microbiol, 2011,14(5): 524–531.
[80] Bohannon J. Synthetic biology. Mission possible:rewriting the genetic code. Science, 2016,353(6301): 739.
[81] Krakauer DC, Jansen VAA. Red queen dynamics of protein translation. J Theor Biol, 2002, 218(7):97–109.
[82] Lajoie MJ, Rovner AJ, Goodman DB, et al.Genomically recoded organisms expand biological functions. Science, 2013, 342(6156): 357–360.
[83] Ostrov N, Landon M, Guell M, et al. Design,synthesis, and testing toward a 57-codon genome.Science, 2016, 353(6301): 819–822.
[84] Smanski MJ, Zhou H, Claesen J, et al. Synthetic biology to access and expand nature’s chemical diversity. Nat Rev Microbiol, 2016, 14(3): 135–149.
