基于第二代测序技术兰州鲇线粒体基因组全序列测定与分析
2017-04-12连总强滚双宝李力张锋肖伟吴旭东
连总强滚双宝李 力张 锋肖 伟吴旭东
(1. 宁夏回族自治区水产研究所, 银川 750001; 2. 甘肃农业大学动物科学技术学院, 兰州 730070; 3. 宁夏渔业工程技术研究中心, 银川 750001)
基于第二代测序技术兰州鲇线粒体基因组全序列测定与分析
连总强1,2,3滚双宝2李 力1,3张 锋1,3肖 伟1,2,3吴旭东1,2,3
(1. 宁夏回族自治区水产研究所, 银川 750001; 2. 甘肃农业大学动物科学技术学院, 兰州 730070; 3. 宁夏渔业工程技术研究中心, 银川 750001)
研究采用高通量第二代测序技术, 构建获得兰州鲇(Silurus lanzhouensis)线粒体基因组全序列, 并对全序列特征和结构进行了分析。研究结果表明, 兰州鲇线粒体基因组全序列长度为16523 bp, 碱基组成具有高A+T低G+C含量的偏向性, 具有脊椎动物典型的结构组成。13个PCG基因中存在2种启动子(ATG、GTG)、3种终止子(TAG、TAA和T或TA)。除tRNA-Ser(AGN)基因二级结构中DHU臂缺失, 其余21个tRNA基因可折叠成典型三叶草结构。12S rRNA二级结构由45个茎环结构组成4个结构域, 16S rRNA由54个茎环结构组成6个结构域。含有关键序列标签的控制区(CR)可分为3个不同的结构域: 终止序列区(TAS1、TAS2)、中央保守区(CSB-F、CSB-E和CSB-D)和保守序列区(CSB1、CSB2和CSB3)。非编码区含有一段保守的控制轻链复制起始的序列区(OL)。基于线粒体基因组全序列和通用标签COX1基因标记可区分兰州鲇同其他鲇形目鱼类种质进化关系。
兰州鲇; 线粒体基因组; 结构特征; 系统发育
兰州鲇(Silurus lanzhouensis)又名黄河鲶, 隶属于鲇形目(Siluriformes)、鲇科(Siluridea)、鲇属(Silurus), 是我国黄河中上游特有的大型经济鱼类,具有较高营养价值和产业化养殖价值, 成为近年来技术研究与养殖开发领域的焦点。但由于过度捕捞、水利工程等因素, 兰州鲇野生种群数量日益减少, 已被《中国物种红色名录》列为濒危物种[1]。鉴于此, 农业部批准在宁夏回族自治区水产研究所科研基地建设国家级兰州鲇原种场, 项目单位先后攻克了兰州鲇人工驯养、人工繁育等关键技术并开展了相关的增殖保护工作, 目前正在开展良种选育工作。
迄今为止, 关于兰州鲇的研究主要集中于形态学[2]、生理生化学[3,4]和群体遗传学[5]等领域。此外, 王庆荣等[6]在北京某农贸市场采集来源于山东某养殖场样本并采用PCR扩增产物直接测序和引物行走法构建获得了兰州鲇线粒体基因组序列(GenBank: J F895472)。本课题组以兰州鲇原种为试验材料, 采用克隆测序的方式构建获得了线粒体细胞色素b (Cytb)基因全序列[7], 并与王庆荣等研究结果进行了鲇属鱼类系统进化关系的研究, 二者对兰州鲇分类存在一定分歧。
脊椎动物线粒体DNA(mtDNA)具有自我复制、转录和翻译功能, 突变率高, 进化速度快。通过对mtDNA的基因结构和序列特征分析, 可以获得丰富的遗传进化信息, 目前已广泛应用于动物系统发育、物种分类、群体遗传进化、种质鉴定等领域。鉴于此, 本研究在前人研究的基础上, 采集国家级兰州鲇原种场野生群体繁育的F1代群体样本,利用高通量第二代测序技术开展兰州鲇线粒体基因组全序列与结构特征研究, 进一步分析鲇科鱼类系统进化关系, 以期为兰州鲇物种分类、种质鉴定、良种选育提供更加可靠的技术依据。
1 材料与方法
1.1 实验材料
按照兰州鲇种质鉴定标准, 随机选取国家级兰州鲇原种场培育4冬龄的F1代兰州鲇3尾, 采集尾鳍去离子水冲洗后, 置于无水乙醇中-20℃保存。组织DNA的提取依据上海生工动物基因组DNA提取试剂盒说明进行, 核酸定量分析仪、1%琼脂糖凝胶电泳检测DNA浓度和完整性。
1.2 全基因组二代测序
混合经检测合格的3个DNA样品建立混合池,通过Covaris超声波破碎仪随机打断成长度为180—330 bp的片段, 构建兰州鲇小片段基因组DNA文库,进行Hiseq 2500二代测序。应用CLC Genomics Workbench v7.5软件进行raw data的质量修剪, 得到相同数量clean paired reads。
1.3 基因组组装与注释
以鲇形目鱼类线粒体全基因组序列为初始参照序列(表 1), 应用GENEIOUS R8 (Biomatters Ltd., Auckland, New Zealand)软件进行线粒体基因组注释。转运RNA (tRNAs)及核糖体RNA (rRNA) 注释应用在线软件MITOS Web Server (http://mitos.bioinf.uni-leipzig.de/index.py)[8]进行分析, 基于已有鲇形目鱼类线粒体基因组比对进行蛋白编码基因(PCGs)及D-loop注释。MEGA6.0软件进行鲇科鱼类线粒体基因组序列碱基组成特征、密码子使用情况及碱基偏移度分析。
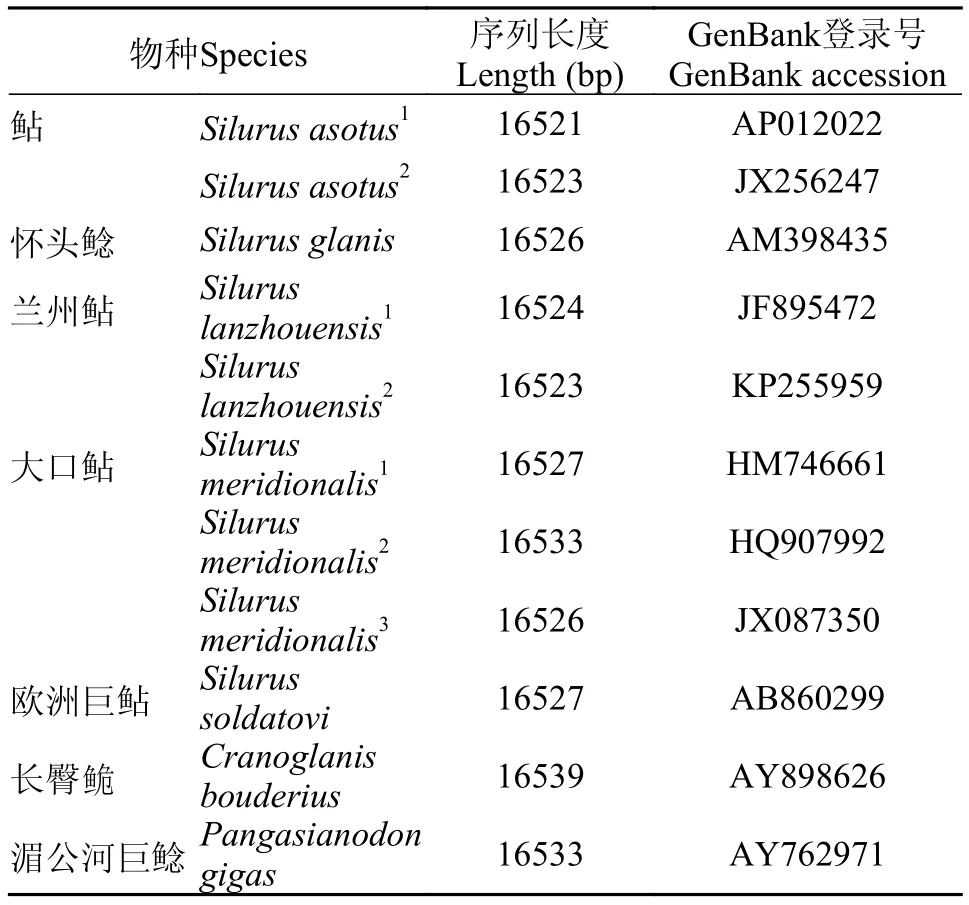
表 1 鲇形目鱼类线粒体系统发育分析基因组Tab. 1 The complete mitochondrial genomes used for phylogenetic analyses in this study
1.4 基因二级结构预测
兰州鲇线粒体tRNA基因定位采用在线tRNAscan-SE 1.21软件进行分析, tRNA基因二级结构预测使用RNA structure 5.3软件进行分析。参照黑尾地鸦(Podoces hendersoni) 12S rRNA[9]和麦穗鱼(Pseudorasbora parva)16S rRNA[10]基因二级结构, RNA-structure 4.6 和Rnaviz2.0.3[11]软件进行兰州鲇12S rRNA、16S rRNA基因二级结构预测。
1.5 鲇科鱼类系统进化树构建
2 结果
2.1 线粒体基因组的组成
本研究采用高通量第二代测序技术, 构建获得兰州鲇线粒体全基因组序列长度为16523 bp (Gen-Bank: KP255959), 包含有13个蛋白编码基因(PCG)、22个tRNA基因、2个rRNA基因和1个控制区(图1)。蛋白质编码基因、rRNA基因、tRNA基因和非编码区分别占整个线粒体长度的68.94%、9.41%、15.92%和5.77%。兰州鲇线粒体各基因大小、位点及排列顺序与大多数脊椎动物相一致, 除控制区序列长度呈现较大程度的差异、同源性相对较低外,蛋白质编码基因、rRNA基因、tRNA基因区段序列与本研究分析的鲇形目鱼类线粒体基因组序列长度差异较小, 同源性较高, 具体位点信息见表 2。

图 1 兰州鲇线粒体基因组结构Fig. 1 Schematic Map of the mitochondrial genome of Silurus lanzhouensis in this study
2.2 核苷酸组成
由表 3可以看出, 鲇形目鱼类基因组A+T含量(55.02%—56.49%)均高于G+C含量(43.51%—46.98%),核苷酸组成具有明显的AT偏向性, 其中本研究兰州鲇的A+T含量最高, 全基因组为56.49%, 蛋白质编码基因为56.36%, 转运tRNA基因为56.71%, 核糖rRNA基因为55.37%, 控制区更是高达61.08%。本研究中兰州鲇线粒体全基因组碱基含量由高到低依次为: A (30.41%)>C (27.75%)>T (26.08%)>G (15.76%), A+T skew碱基组成和G+C skew碱基组成分别为0.076和-0.276, 其他鲇形目鱼类及王庆荣所报道的兰州鲇线粒体基因组中腺嘌呤(A)和胞嘧啶(C)含量较本研究的更高(A+T skew: 0.089—0.096; G+C skew: -0.285— -0.280), 表明鲇形目鱼类线粒体基因组中含有较多的腺嘌呤(A)和胞嘧啶(C)。通过以上研究发现, 鲇形目鱼类各基因碱基组成表现出控制区含有相对较高的胸腺嘧啶(T)和鸟嘌呤(G), 而tRNA和rRNA基因具有相对较高腺嘌呤(A)和胞嘧啶(C)含量, 且本研究中兰州鲇除tRNA基因的腺嘌呤(A)和胞嘧啶(C)含量较其他种类高外,其余各基因及区段的均较低。
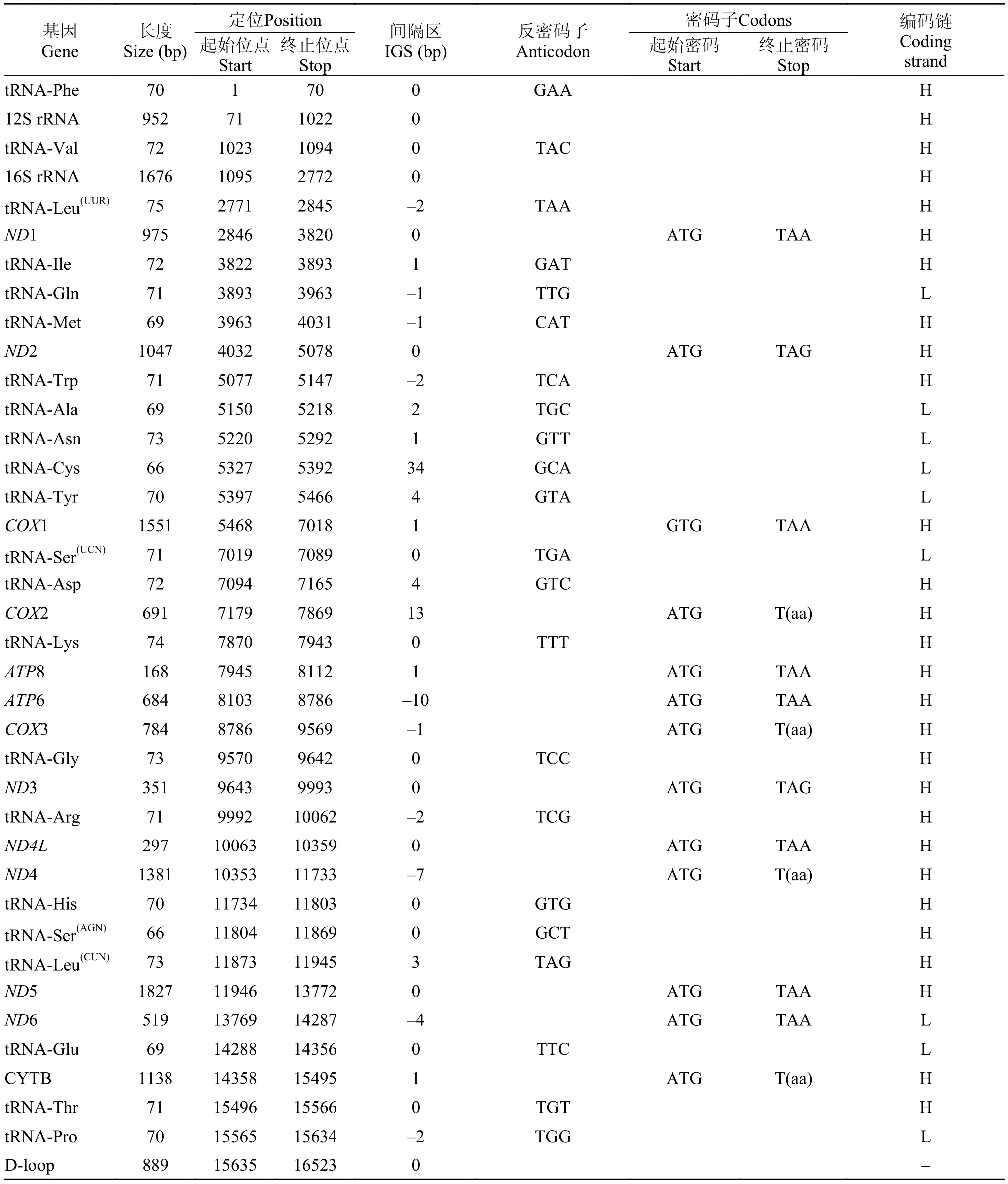
表 2 兰州鲇线粒体基因组注释结果Tab. 2 The results of the complete mitochondrial genome annotation for Silurus lanzhouensis

表 3 鲇属鱼类线粒体基因组碱基组成Tab. 3 Composition and skewness in the mitochondrial genomes of Silurus species
2.3 蛋白质编码基因(PCGs)
本研究中兰州鲇13个蛋白编码基因同大多数硬骨鱼类一样含有两种起始密码子(GTG、ATG),除COX1基因使用GTG作为起始密码子外, 其余12个基因均使用ATG作为起始密码子。相对于起始密码子的稳定性, 终止密码子在物种内和物种间变化较多。本研究中的兰州鲇终止密码子有3种(表 2), 其中TAA为ATP6、ND4L、COX1、ND1、ND5、ND6、和ATP8等7个基因的终止密码子, TAG为ND2和ND3基因的终止密码子, COX2、COX3、ND4和CYTB基因则使用不完全的T和TA作为终止密码子。
13个PCG基因序列总长度11391 bp, 除终止密码子外共有3768个密码子。应用MEGA6.0软件分析碱基使用频率, 密码子第一位点偏倚度最小, 第二位点偏倚度最大, 存在明显碱基偏向性, A、G含量分别为31.3%、9.4%, 以上碱基偏移现象也常见于其他脊椎动物。密码子平均使用频率和相对同义密码子平均使用频率分析结果表明, 兰州鲇13个PCG基因中存在32个偏好密码子(RSCU≥1), 其中以第三位点为A或C的密码子在编码同种氨基酸中具有较高的使用频率, 除密码子GAA (E)、CCA (P)、AGA (*)和CAC (H)、CUC (L)、AUC (I)、GUC (V)、CGC (R)、AGC (S)外, 密码子第3位为C、A碱基的密码子RSCU值均大于1, 密码子第三位点对A、C偏好性与蛋白质编码基因密码子的第三位点对A、C偏向性表现出正相关性(表 4)。
2.4 核糖RNA(rRNA)基因
本研究中兰州鲇12S rRNA基因和16S rRNA的序列长度分别为952和1676 bp (表 1)。采用同源序列对比分析法, 分别构建预测12S rRNA和16S rRNA基因二级结构。由图 2可见, 12S rRNA二级结构中包含有由45个茎环结构组成的第Ⅰ-第Ⅳ结构域, 其中第Ⅰ、第Ⅱ结构域为可变区, 第Ⅲ和第Ⅳ结构域为保守区。16S rRNA二级结构由54个茎环结构组成6个(Ⅲ-Ⅵ)结构域(图 3), 各个结构域之间由一段单链区分。其中第Ⅰ-第Ⅲ及第Ⅵ结构域为可变区, 第Ⅳ与第Ⅴ结构域为保守区。通过兰州鲇与麦穗鱼的碱基序列对比及二级结构分析发现,两个物种间的碱基突变、插入和缺失较多。在以上rRNA二级结构及基因序列对比分析中发现, 12S rRNA基因比16S rRNA基因更保守, 茎区碱基序列比环区更加高度保守。
2.5 转运RNA(tRNA)基因
通过对兰州鲇线粒体22个tRNA基因进行定位及二级结构预测分析, 结果表明22个tRNA基因分布于13个蛋白编码基因之间, 大小从66到75 bp不等, 总长度为1554 bp (图 1)。通过对比分析发现,兰州鲇tRNA基因中三对相邻的tRNA(如: tRNAIle与tRNA-Gln、tRNA-Gln与tRNA-Met、tRNAThr与tRNA-Pro)间存在1个或2个核苷酸相互折叠的现象, 这种现象也存在于其他鲇科鱼类中[14], 不同之处仅是折叠碱基数存在差异。22个tRNA基因中的14个由H-链编码, 另外8个由L-链编码(表 2)。所有tRNA基因序列的A+T碱基含量较控制区的低,较PCGs和rRNA基因序列的高。除tRNA-Ser(AGN)外, 其余tRNA基因均形成典型三叶草结构(图 4)。
2.6 控制区(CR)
富含A+T的控制区虽不编码蛋白质, 但在调控线粒体DNA复制和转录中具有重要作用。本研究在tRNA-Pro和tRNA-Phe基因之间识别出了由889 bp碱基序列组成的控制区, 该区域含有启动H-链复制的序列(图 1)。同其他鲇形目鱼类相比, 除了个别位点存在插入/缺失现象, 控制区相对保守, 序列长度和A+T含量在不同物种间无显著差异(表 3)。
同许多脊椎动物一样, 兰州鲇的控制区可分为三个结构域(图 5): ①终止序列区又称高变区(Hypervariable Domain), 在三个区域中变异最大, 长度为208 bp。兰州鲇终止序列区含有两个控制线粒体DNA复制终止功能的相关序列(TAS-1、TAS-2),该区段序列可由2对核心序列TACAT与其反向互补的ATGTA序列形成包含49个碱基的热稳定茎环结构。②中央保守区, 位于209nt—630 nt处, 该区域包含有3个保守序列, 分别是CSB-D、CSB-E和CSB-F。其中CSB-F位于289nt—316 nt处, 其关键序列为AGAGACCATC, 该序列被认为是区分终止
区和中央保守区的标志[15]。Lee等[16]所描述的CSBE中存在GTGGG-box已被研究者广泛认可, 本研究中识别出兰州鲇GTGGG-box位于352—377 nt, 识别关键序列为AGGGTCAGGGACAAACTTGTGG GGG。CSB-D位于控制区393—407 nt处, 在鲇形目鱼类中变异较大, 且鲇形目鱼类中识别的关键序列为TATTCTGGATCTG。③保守序列区, 在兰州鲇该区域识别出了3个保守序列(CSB1、CSB2和CSB3), 其中CSB1位于670—705 nt处, CSB2位于748—767 nt处, CSB3 位于793—810 nt处。据Broughton等[17]研究报道, 保守序列区中CSB-1保守性相对较低。通过与其他硬骨鱼类对比, 发现兰州鲇同大多数硬骨鱼类一样, 在该序列之后紧随一段TGCCCCC保守序列, 本研究识别的兰州鲇CSB-1的关键序列为ATTCCACGCGCATAAGGTTATTC TTTACTCCACATA。相比之下, CSB-2和CSB-3序列的保守性较CSB-1高, 二者在兰州鲇线粒体中的保守序列分别为: AAACCCCCCCTACCCCC和CAAACCC---AAA---CA。

表 4 兰州鲇13个蛋白质编码基因密码子平均使用频率Tab. 4 Total codon average usage in the thirteen protein-coding genes for S. lanzhouensis

图 2 兰州鲇12S rRNA基因二级结构Fig. 2 The secondary structures of 12S rRNA genes in the S. lanzhouensis mitogenome
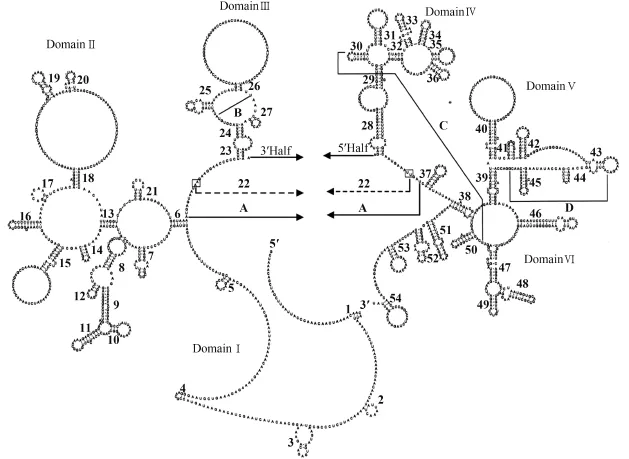
图 3 兰州鲇16S rRNA基因二级结构Fig. 3 The secondary structures of 16S rRNA genes in the S. lanzhouensis mitogenome
2.7 非编码区

图 4 兰州鲇tRNA-Ser(AGN)二级结构预测图Fig. 4 The secondary structures of the tRNA-Ser(AGN)genes in S. lanzhouensis mitogenome
脊椎动物线粒体基因组中的非编码区为调节线粒体DNA复制和转录的小片段, 主要分布于L-链复制起始区和tRNA基因之间。根据非编码区的结构组成和分布位置, 该区域亦可分为基因间隔序列区和基因序列重叠区。在兰州鲇线粒体该区域共识别出10处基因序列重叠区, 序列总长为32 bp, 最大碱基重叠数为10 bp, 位于ATP6 和ATP8基因之间, 最小碱基重叠数为1 bp。非编码区有11个间隔序列, 长度大小在1—34 bp。其中在tRNA-Asn和tRNA-Cys基因之间分布有最大的间隔序列(34 bp),并在该区域识别出了能够启动L-链复制的序列(OL),其在鲇形目鱼类中的关键序列为5′-GCCTG-3′, 该序列由10个碱基形成一个保守的茎环结构(图 6)。Shadel和Clayton[18]研究认为, 在脊椎动物线粒体基因组中, 该茎环结构主要调节L-链的复制。

图 5 兰州鲇线粒体控制区结构特征示意图Fig. 5 Schematic map characterizing of the control region of S. lanzhouensis
2.8 鲇科鱼类系统进化分析
COX1基因可作为通用标签进行物种有效分类[19]。本研究采用COX1基因序列进行了几种鲇形目鱼类NJ系统进化关系分析, 结果显示除部分节点拟合系数降低外, 基于几种鱼类COX1基因的系统进化关系具有与全基因组ML进化树分析相同的结果, 其中本研究中来源于国家级兰州鲇原种场的样本(KP255959)同已报道的兰州鲇(JF895472)仍分为两个系群(图 8)。此外, 通过本研究所得到的兰州鲶线粒体基因组与前人已发表的兰州鲇线粒体基因组(JF895472)序列比对分析, 两者间全基因组序列一致性百分比(Pairwise % Identity)为92.4%。其中, 通用标签COX1基因序列的同源性比例只有90.8%, K2P遗传距离为0.10。以上结果表明该核苷酸序列分化值明显大于常用的分子种划分阈值标准(0.03)[20]。综合兰州鲇传统分类学研究结果, 以及近8年本实验室对黄河兰州鲇种群资源调查结果,研究者普遍认为兰州鲇主要分布在黄河中上游水域, 而王庆容等报道的兰州鲇样本采自北京大学西门外农贸市场来源于山东省某养殖场的兰州鲇。在本研究的基础上, 我们推测已报道兰州鲇(JF895472)有可能是潜在的生物地理种群种或兰州鲇近缘种。
3 讨论
3.1 鱼类线粒体基因组组成
本研究发现兰州鲇线粒体基因组全序列具有与其他典型脊椎动物类似的基因组成和明显的AT偏向性。较高A+T含量和较低G+C含量的线粒体DNA特征, 在已报道的其他鲇形目鱼类中同样存在[13,21,22], 只是A+T含量或G+C含量大小因物种不同而有差异。目前已测定的大多数淡水鱼类的基因排列序列与其他脊椎动物相一致。大多数鱼类的L-链编码了8个tRNA (tRNA-Gln、tRNA-Ala、tRNA-Asn、tRNA-Cys、tRNA-Tyr、tRNA-Ser(UCN)、tRNA-Glu和tRNA-Pro)和ND6基因, 其余28个基因均由H-链编码, 这与本研究中兰州鲇的基因编码位置一致。

图 6 L-链复制起始区的茎环结构Fig. 6 Stem-loop structure of L-strand replication initiation region

图 7 鲇形目鱼类全线粒体基因组ML进化树Fig. 7 Phylogenetic tree based on the ML analysis of the siluriformes whole mitochondrial genome

图 8 鲇形目鱼类COX1基因NJ进化树Fig. 8 Phylogenetic tree based on the Neighbor-joining (NJ) analysis of the COX1 genes
3.2 蛋白质基因的起始和终止
兰州鲇13个蛋白编码基因中, 起始密码子与多数硬骨鱼类的结果相一致, 具有相对稳定的两种起始密码子(GTG、ATG)。相对于起始密码子的稳定性, 终止密码子在物种内和物种间变化较多。例如棒花鱼(Abbottina rivularis)[23]的COX1、ND4L、ND5、ATP6和ATP8基因以TAA作为终止密码子, ND1、ND2、ND6以TAG作为终止密码子, COX2、COX3、ND3、ND4和CYTB以不完全TA或T作为终止密码子。麦穗鱼(P. parva)[10]mtDNA的ATP6、COX1、ND2、ND4L、ND5、ND6基因终止密码子为TAA; ATP8和ND1基因终止密码子为TAG; CYTB、ND4、ND3、COX2、COX3基因以T作为不完全终止密码子。南方鲇(S. meridionalis)[24]CYTB、COX2、ND4、COX3基因以T和TA作为不完全终止密码子。本研究中兰州鲇PCGs中的7个基因(ATP6、ATP8、COX1、ND1、ND4L、ND5和ND6)的终止密码子是TAA, 2个基因(ND2和ND3)是TAG, 其余4个基因是T或TA。从以上研究结果可以看出, 不同鱼类的编码基因使用不同的终止密码子, 但均以TAA或TAG作为完全终止密码子, 以T和TA作为不完全终止密码子, 未发现特殊的密码子。脊椎动物线粒体PCGs中出现不完全终止密码子的现象较为常见, Ojala等[25]推测不完全密码子是由转录后mRNA3′端加入A残基所致。
3.3 tRNA基因和rRNA基因
据Lee等[26]报道, 12S rRNA基因序列长度主要由3′端一段长的插入缺失序列决定, 而16S rRNA则主要由5′端一段长的插入缺失序列决定。Sharma等[27]研究发现, rRNA基因的功能取决于其在蛋白质合成中的二级结构。在同麦穗鱼、黑尾地鸦及其他脊椎动物rRNA二级结构的对比分析中, 发现12S rRNA基因比16S rRNA基因更保守, 并且茎区碱基序列比环区更加高度保守, 同时我们推测兰州鲇同其他脊椎动物一样, 具有rRNA基因的二级结构比序列本身更高度保守的特征[28]。Dixon等[29]指出, rRNA独特的二级结构是核糖体重要功能和组成的一部分, 这种组成和功能的特征主要来自茎区和环区包含重要的亲缘信息。因此, rRNA基因已经被广泛地用于系统发生分析。本研究中兰州鲇22个tRNA基因中除tRNA-Ser(AGN)外, 其余均形成典型三叶草结构。硬骨鱼类tRNA-Ser(AGN)缺少二氢尿嘧啶臂(DHU臂)的现象较为多见, DHU臂的位置上仅形成一个单环结构。Cheng等[30]研究证明, 这种缺失DHU臂的tRNA可通过调整其结构形态和功能, 从而同典型三叶草结构的tRNA一样融入核糖体, 发挥其携带并转运氨基酸的功能。
3.4 非编码序列区(NR)
目前已报道的大多数淡水鱼类只有一个包含有控制区和L-链复制起始区的非编码区。鱼类的控制区包括终止序列区(TAS)、中央保守区(CD)、保守序列区(CSB)、H-链复制起始区(OH)[31]。本研究中在兰州鲇mtDNA的NR区仅发现TAS、CD和CSB区。在TAS区存在两对核心序列TACAT与其反向互补的ATGTA序列形成包含49个碱基的热稳定发夹结构, 参与线粒体基因组复制终止调控; AGAGACCATC这段基因序列作为CSB-F的关键序列, 在鲇形目鱼类间高度保守, 普遍认为是区分终止区和中央保守区的标志[15]; 已有研究报道发现在人[32]、虹鳟[33]等脊柱动物的L-链复制起始区中存在5′-GCCGG-3′关键序列, 且该序列在RNA、DNA合成中具有重要作用。本研究发现在鲇形目鱼类线粒体L-链复制起始区的关键序列为5′-GCCTG-3′,其在鲇形目鱼类中的碱基序列和在茎环中的位置均较保守, 推测兰州鲇5′-GCCTG-3′关键序列同其他脊椎动物5′-GCCGG-3′关键序列具有相同的功能。
3.5 系统进化分析
目前在鱼类线粒体的研究方面, 相关的基因或结构区域作为分子遗传标记广泛应用于系统发育、物种分类、种群鉴定等领域的研究中。例如:张燕等[34]利用线粒体DNA控制区有效地将中国鲿科鱼类进行了分类。周传江等[24]利用线粒体蛋白编码基因序列分析了南方鲇同其他35种鱼类的系统发育和分化时间, 阐明了鲇形目、鲤形目、脂鲤目、电鳗目的系统发育关系。综合利用线粒体16S rRNA和ND2基因标记, Near等[35]成功分析了南极鳕的演化发育关系。Phillip等[36]利用线粒体基因组全序列和控制区序列进行了虎鲸(Orcinus orca)地理种群和系统分类的研究, 发现基因组全序列较控制区更能准确的区分物种, 同时应用线粒体基因组全序列划分出了新的地理种群。近年来, 线粒体DNA的COX1基因由于长度和进化速率适中, 以及富含系统发育信息等特点, 被作为DNA通用标签广泛应用于种质鉴定、物种分类和系统进化领域研究。如: Hebert等[20]利用COX1基因作为DNA条形码较好的对200种亲缘关系较近的鳞翅目昆虫进行了系统进化关系的研究。Zhang[37]利用COX1基因有效地区分和分析了329种海洋鱼类的进化和分类关系。本研究分别应用鲇形目鱼类线粒体DNA全序列和COX1基因序列进行了兰州鲇同其他鲇形目鱼类的系统进化关系和物种鉴定分析, 结果表明以上两种标记均可有效分析鲇形目鱼类的系统进化与分类关系, 但鉴于鲇形目鱼类间的复杂性和较低的异质性, 尚需借鉴以上其他鱼类在线粒体系统进化分析的成功经验, 进一步深入开展鲇形目鱼类,尤其是鲇科鱼类的系统进化关系和物种分类鉴定的研究。
[1]Wang S, Xie Y. China Species Red List (Vol. Ⅱ Vertebrates Part 1) [M]. Beijing: Higher Education Press. 2009, 294—295 [汪松, 解焱. 中国物种红色名录(第二卷脊椎动物上册). 北京: 高等教育出版社. 2009, 294—295]
[2]Wu X D, Zhang Q, Zhao H X, et al. A new species of catfish in Ningxia——Silurus lanzhouensis and it’s intensive morphological description [J]. Freshwater Fisheries, 2006, 36(3): 26—29 [吴旭东, 张奇, 赵红雪, 等. 宁夏鲇属鱼类一新纪录种——兰州鲇形态学特征描述. 淡水渔业, 2006, 36(3): 26—29]
[3]Yang Y H, He Y L, Zhou J S, et al. Quality evaluation of the meat of Silurus lanzhouensis, Silurus asotus and Cyrinus carpio [J]. Acta Hydrobiologica Sinica, 2013, 30(1): 54—61 [杨元昊, 贺玉良, 周继术, 等. 兰州鲇与鲇、黄河鲤肌肉品质比较. 水生生物学报, 2013, 30(1): 54—61]
[4]Wu X D, Lian Z Q, Hou Y X, et al. Effects of arsenic on activities of the metabolic enzymes contents of RNA and protein in several tissues of Silurus lanzhouensis [J]. Acta Hydrobiologica Sinica, 2013, 37(6): 1078—1083 [吴旭东, 连总强, 侯玉霞, 等. 砷对兰州鲇组织中代谢酶活性及RNA和蛋白质含量影响. 水生生物学报, 2013, 37(6): 1078—1083]
[5]Wei D W, Lian Z Q, Wu X D, et al. Microsatellite enrichment by magnetic beads in Silurus lanzhouensis [J]. Acta Hydrobiologica Sinica, 2014, 38(4): 791—796 [魏大为,连总强, 吴旭东, 等. 磁珠富集法筛选兰州鲇微卫星分子标记. 水生生物学报, 2014, 38(4): 791—796]
[6]Wang Q R, Xu C, Xu C R, et al. Mitochondrial genome structure of Yellow river catfish (Silurus lanzhouensis) and phylogenetic analysis [J]. Acta Scientiarum Naturalium Universitatis Pekinensis, 2012, 48(3): 376—380 [王庆容, 许昌, 许崇任, 等. 兰州鲇线粒体基因组序列结构及系统发育分析. 北京大学学报, 2012, 48(3): 376—380]
[7]Wu X D, Xiao W, Lian Z Q. The cloning and sequence analysis of the mitochondrial cytochrome b gene of Silurus lanzhouensis [J]. Acta Hydrobiologica Sinica, 2014,38(4): 772—779 [吴旭东, 肖伟, 连总强. 兰州鲇线粒体Cytb基因的克隆与序列分析. 水生生物学报, 2014, 38(4): 772—779]
[8]Bernt M, Donath A, Jühling F, et al. MITOS: improved de novo metazoan mitochondrial genome annotation [J]. Molecular Phylogenetics and Evolution, 2013, 69(2): 313—319
[9]Ke Y, Huang Y, Lei F M. Sequencing and analysis of the complete mitochondrial genome of Podoces hendersoni (Ave, Corvidae) [J]. Hereditas, 2010, 32(9): 951—960 [柯杨, 黄原, 雷富民. 黑尾地鸦线粒体基因组序列测定与分析. 遗传, 2010, 32(9): 951—960]
[10]Chen T, Shi Y R, You P. Sequence and analysis of complete mitochondrial genome of Pseudorasbora parva [J]. Acta Zootaxonomica Sinica, 2012, 37(1): 10—19 [陈涛,史妍茹, 尤平. 麦穗鱼线粒体基因组序列测定及分析.动物分类学报, 2012, 37(1): 10—19]
[11]Peter D R, Jan W, Rupert D W. RnaViz2: an improved representation of RNA secondary structure [J]. Bioinformatics, 2003, 19(2): 299—300
[12]Peng Z G, Wang J, He S P. The complete mitochondrial genome of the helmet catfish Cranoglanis bouderius (Siluriformes: Cranoglanididae) and the phylogeny of otophysan fishes [J]. Gene, 2006, 376(2): 290—297
[13]Jondeung A, Sangthong P, Zardoya R. The complete mitochondrial DNA sequence of the Mekong giant catfish (Pangasianodon gigas), and the phylogenetic relationships among Siluriformes [J]. Gene, 2007, 387(1—2): 49—57
[14]Lee J S, Miya M, Lee Y S, et al. The complete mitochondrial genome of the self-fertilizing fish Rivulus marmoratus (Cyprinodontiformes, Rivulidae) and the first description of a duplication of a control region in fish [J]. Gene, 2001, 280(1—2): 1—7
[15]Zhao J L, Wang W W, Li S F, et al. Structure of mitochondrial DNA control region of the sinipercine fishes and their phylogentic relationship [J]. Acta Genetica Sinica, 2006, 33(9): 793—799
[16]Lee W J, Conroy J, Howell W H, et al. Structure and evolution of teleost mitochondrial control regions [J]. Journal of Molecular Evolution, 1995, 41(1): 54—66
[17]Broughton R E, Milam J E, Roe B A. The complete sequence of the Zebrafish (Danio rerio) mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA [J]. Genome Research, 2001, 11(11): 1958—1967
[18]Shadel G S, Clayton D A. Mitochondrial DNA maintenance in vertebrates [J]. Annual Review of Biochemistry, 1997, 66: 409—435
[19]Ward R D, Zemlak T S, Innes B H, et al. DNA barcoding Australia’s fish species [J]. Philosophical Transactions of the Royal Society Biological Sciences, 2005, 360(1462): 1847—1857
[20]Hebert P D, Cguinska A, Ball S L, et al. Biological identifications through DNA barcodes [J]. Proceedings of the Royal Society of London B, 2003, 270(1512): 313—321
[21]Wang K, Xu J, Cui J, et al. Complete mitochondrial genome of Northern Sheatfish (Silurus soldatovi) [J]. Mitochondrial DNA, 2015, 26(6): 891—892
[22]Nakatani M, Miya M, Mabuchi K, et al. Evolutionary history of Otophysi (Teleostei), a major clade of the modern freshwater fishes: Pangaean origin and Mesozoic radiation [J]. BMC Evolutionary Biology, 2011, 11: 177
[23]He H Y, Wang J, You P. The complete nucleotide sequence of mitochondrial genome of Abbottina rivularis (Cypriniformes Cyprinidae) [J]. Acta Zootaxonomica Sinica, 2013, 38(4): 695—704 [贺怀亚, 王佳, 尤平. 棒花鱼(鲤形目, 鲤科)全线粒体基因组测定与分析. 动物分类学报, 2013, 38(4): 695—704]
[24]Zhou C J, Wang X Z, Wang D Q, et al. The complete mitochondrial genome of the southern catfish (Silurus meridionalis) and divergence time estimations of Ostariophysan fishes [J]. Scientia Sinica Vitae, 2011, 41(2): 150—159 [周传江, 王绪祯, 汪登强, 等. 南方鲇线粒体基因组全序列与骨鳔鱼类分化时间的估算. 中国科学:生命科学, 2011, 41(2): 150—159]
[25]Ojala D, Montoya J, Attardi G. tRNA punctuation model of RNA processing in human mitochondria [J]. Nature, 1981, 290(5806): 470—474
[26]Lee W J, Kocher T D. Complete sequence of a sea lamprey (Petromyzon marinus) mitochondrial genome: early establishment of the vertebrate genome organization [J]. Genetics, 1995, 139(2): 873—887
[27]Sharma M R, Koc E C, Datta P P, et al. Structure of the mammalian mitochondrial ribosome reveals an expanded functional role for its component proteins [J]. Cell, 2003, 115(1): 97—108
[28]Springer M S, Douzery E. Secondary structure and patterns of evolution among mammalian mitochondrial 12S rRNA molecules [J]. Journal of Molecular Evolution, 1996, 43(4): 357—373
[29]Dixon M T, Hillis D M. Ribosomal RNA secondary structure: compensatory mutations and implications for phylogenetic analysis [J]. Molecular Biology and Evolution, 1993, 10(1): 256—267
[30]Cheng Y Z, Wang R X, Sun Y N, et al. The complete mitochondrial genome of the small yellow croaker and partitioned Bayesian analysis of Sciaenidae fish phylogeny [J].Genetics and Molecular Biology, 2012, 35(1): 191—199
[31]Guo X H, Liu S J, Liu Q, et al. New progresses on mitochondrial DNA in Fish [J]. Acta Genetica Sinica, 2004, 31(9): 983—1000 [郭新红, 刘少军, 刘巧, 等. 鱼类线粒体DNA研究新进展. 遗传学报, 2004, 31(9): 983—1000]
[32]Hixson J E, Wong T W, Clayton D A. Both the conserved stem-loop and divergent 5’-flanking sequence are required for initiation at the human mitochondrial origin of light-strand DNA replication [J]. Journal of Biological Chemistry, 1986, 261(5): 2384—2390
[33]Rafael Z, Amando G P, José M B. The complete nucleotide sequence of the mitochondrial DNA genome of the rainbow trout, Oncorhynchus mykiss [J]. Journal of Molecular Evolution, 1995, 41(6): 942—951
[34]Zhang Y, Zhang E, He S P. Studies on the structure of the control region of the bagridae in china and its phylogentic significance [J]. Acta Hydrobiologica Sinica, 2003, 27(5): 463—467 [张燕, 张鹗, 何舜平. 中国鲿科鱼类线粒体DNA控制区结构及其系统发育分析. 水生生物学报, 2003, 27(5): 463—467]
[35]Near T J, Pesavento J J, Cheng C H. Mitochondrial DNA, morphology, and the phylogenetic relationships of Antarctic icefishes (Notothenioidei: Channichthyidae) [J]. Molecular Phylogenetics and Evolution, 2003, 28(1): 87—98
[36]Phillip A M, Frederick I A, Andrew D F, et al. Complete mitochondrial genome phylogeographic analysis of killer whales (Orcinus orca) indicates multiple species [J]. Genome Research, 2010, 20(7): 908—916
[37]Zhang J B. Species identification of marine fishes in China with DNA barcoding [J]. Evidence-Based Complementary and Alternative Medicine, 2011, doi: 10. 1155/2011/978253
SEQUENCING AND ANALYSIS OF THE COMPLETE MITOCHONDRIAL GENOME OF SILURUS LANZHOUENSIS BASED ON NEXT GENERATION SEQUENCING TECHNOLOGIES
LIAN Zong-Qiang1,2,3, GUN Shuang-Bao2, LI Li1,3, ZHANG Feng1,3, XIAO Wei1,2,3and WU Xu-Dong1,2,3
(1. Ningxia Fisheries Research Institute, Yinchuan 750001, China; 2. College of Animal Science and Technology, Gansu Agricultural University, Lanzhou 730070, China; 3. Ningxia Engineering Research Center for Fisheries, Yinchuan 750001, China)
In this study, the high-throughput next-generation sequencing technology was used to build Lanzhou catfish (Silurus lanzhouensis) mitochondrial genome sequence, and the complete sequence and characteristics for mitochondrial genome has been analyzed. The main results were demonstrated in detail. The circular genome was 16523 bp in length with the typical mtDNA components, and the base composition showed a tendency of high A+T and low G+C. Two promoters (ATG, GTG) and three terminators (TAG, TAA and T or TA) were found in all 13 PCGs. Except tRNASer(AGN)gene showed the shortage of DHU, the other 21 genes of tRNA could be folded in clover shape. The 45 stemloop structures and 4 structural domains were identified in secondary structure of 12S rRNA, and 54 stem-loop structures and 6 structural domains were identified in 16S rRNA. Three different structural domains of termination sequence region (TAS1, TAS2), central conservative region (CSB-F, CSB-E and CSB-D) and conservative region (CSB1, CSB2 and CSB3) were divided in the controlled region that remains key sequence label. The non-code region has a conservative sequence to control light chain starting copy. The complete sequence of mitochondrial genome and general COX1 label could be used for the study of evolutionary relationship between S. lanzhouensis and other Siluriformes species.
Silurus lanzhouensis; Mitochondrial genome; Structure and characteristics; Phylogeny
Q173
A
1000-3207(2017)02-0334-12
10.7541/2017.41
2016-03-08;
2016-06-14
国家自然科学基金项目(31360633); 国家大宗淡水鱼产业技术体系项目(CARS-46-54); 宁夏对外科技合作项目资助 [Supported by the National Natural Science Foundation of China (31360633); National Technology System for Conventional Freshwater Fish Industries (CAR-46-54); the External Cooperation Projects of Ningxia, China]
连总强(1980—), 男, 甘肃靖远人; 博士, 高级工程师; 主要从事鱼类遗传育种与繁殖研究。E-mail: lianzq04@163.com
吴旭东(1967—), 男, 博士, 研究员; 主要从事鱼类遗传育种与繁殖研究。E-mail: amy95@126.com
