“中国春”小麦全基因组中特异SSR标记的发掘及其分布特征
2017-04-04崔婷王长彪韩斌赵兴华刘江任永康牛瑜琦唐朝晖
崔婷,王长彪,韩斌,赵兴华,刘江,任永康,牛瑜琦,唐朝晖
(1.山西大学生物工程学院,山西太原030006;2.山西省农业科学院生物技术研究中心,山西太原030031;3.山西省农业科学院作物科学研究所,山西太原030031)
“中国春”小麦全基因组中特异SSR标记的发掘及其分布特征
崔婷1,王长彪2,韩斌2,赵兴华2,刘江2,任永康3,牛瑜琦3,唐朝晖2
(1.山西大学生物工程学院,山西太原030006;2.山西省农业科学院生物技术研究中心,山西太原030031;3.山西省农业科学院作物科学研究所,山西太原030031)
SSR标记尤其是不同染色体上的单位点特异性SSR标记能为进行小麦基因组分子进化研究、遗传多样性分析、指纹图谱和遗传图谱构建、品种真实性鉴定等方面提供更加方便的工具。利用生物信息学技术从山西省农科院生物技术研究中心开发的295 267对“中国春”全基因组SSR分子标记中发掘得到了单位点标记102 287对,其中,不同染色体上特异标记80 374对;A,B,D这3个基因组上特异标记分别是26 738,31 707,21 929对,3个基因组共有的标记有3 293对;80 374对特异分子标记定位到“中国春”小麦全基因组上时,3B染色体定位的特异标记最多,达到5 756对,其次是2B染色体上,为5 680对,最少的是4D染色体上,为2 598对。对不同染色体开发的单位点SSR标记定位全基因组分析表明,定位到不同染色体上最多的标记来源主要集中在2B和5A染色体上。
SSR;小麦;特异标记;单位点标记;全基因组
小麦(Triticum aestivum L.)是人类食物中植物蛋白的主要来源,其植物蛋白的含量较玉米和水稻等主要作物都高。就全球范围而言,小麦在人类食用的主要粮食作物中的地位仅次于水稻。为了满足世界各国日益增加的小麦需求量,培育抗生物逆性强耐生物胁迫(白粉病、锈病等)和非生物胁迫(温度、寒冷、干旱、洪水等)以及在保护农业管理措施下能够维持产量和质量的优良小麦新品种势在必行[1-3]。与传统的既费人力又费物力的表型选择相比,从分子水平改良种质资源的方式逐步受到人们的重视[4]。在过去的几十年中,分子标记技术[5]在小麦研究中取得了很大的进展[6]。
SSR(Simple Sequence Repeats)标记是目前最常用的分子标记技术之一[7]。SSR即微卫星DNA(Microsatellites),是一类由几个核苷酸(一般为1~6个)[8]为重复单位组成的串联重复序列。由于每个SSR两侧的序列通常是保守性较强的单一序列,因而可以将微卫星侧翼的DNA片段克隆、测序,然后根据微卫星的侧翼序列就可以人工合成引物进行PCR扩增,从而将单个微卫星位点扩增出来。与其他分子标记相比,SSR标记具有以下优点:(1)数量丰富;(2)具有多等位基因;(3)呈共显性;(4)价格便宜,技术简便[9-10]。目前,该技术已广泛用于遗传图谱的构建、目标基因的标定、指纹图谱的绘制等研究方面。
开发单位点的特异性SSR标记[11],可以提高工作效率,为进一步研究基因组进化、遗传多样性分析、品种真实性鉴定等方面的工作奠定坚实的基础。我们利用生物信息学工具开发了基于“中国春”小麦的全基因组单位点的特异SSR标记,为进一步进行分子设计育种奠定基础。
1 材料和方法
1.1 材料
10 603 760条“中国春”小麦全基因组序列从URGI下载(https://urgi.versailles.inra.fr/download/iwgsc/)。数据公布更新时间为2014年9月6日。
1.2 方法
利用295 267对SSR标记[12]对小麦全基因组进行电子定位。具体参数为:re-PCR-Shash-file-n0-g0-r+。再利用在线软件Venn(http://bioinformatics. psb.ugent.be/webtools/Venn/)对不同基因组间定位结果进行韦恩作图,研究不同基因组之间的标记分布。
2 结果与分析
2.1 小麦不同基因组上单位点SSR分子标记分布
利用295 267对SSR标记对小麦全基因组进行电子定位,得到单位点标记(每个染色体上只有1个位点)102 287对,其中特异标记(只定位到1个染色体上,并只有1个位点)80 374对,80 374对标记中定位到A,B,D基因组上的分别是42 276,47 191,38 026对,这3个基因组上特异标记分别是26 738,31 707,21 929对,3个基因组共有的标记有3 293对。不同基因组两两间特有的共有标记也各有不同,A和B间有5 816对,A和D间有6 429,B和D间有6 375对(图1)。该定位结果是通过电子模拟定位的,严格按照无错配和无Gap进行,与当时开发标记用的参数[11]有差异,所以定位时没有把开发的标记全部定位上去,这样定位的结果更加准确,该部分结果能为进一步进行遗传多样性分析、基因组进化以及分子设计育种等方面奠定基础。
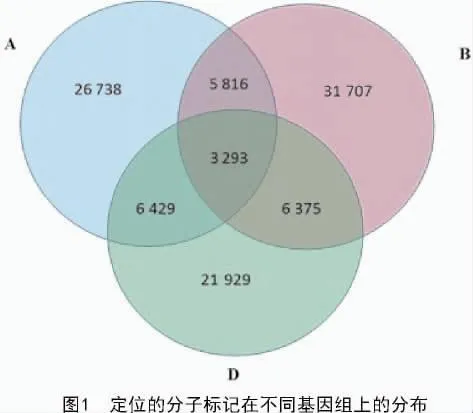
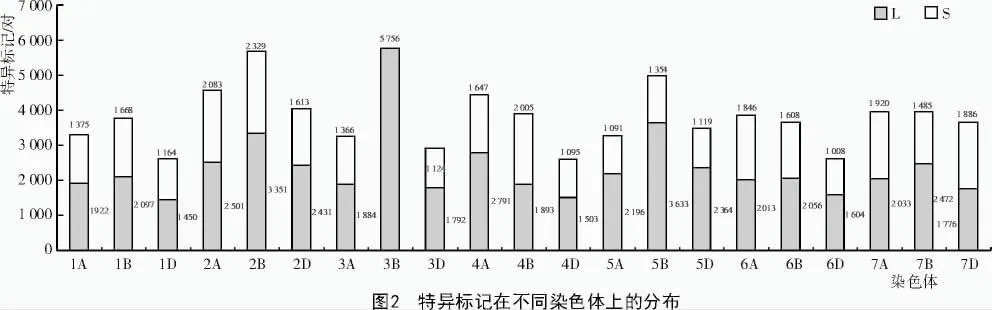
2.2 小麦不同染色体上特异SSR分子标记分布
对定位的不同基因组上的80 374对特异标记进行进一步定位分析,分别定位到不同染色体上的不同染色体臂上,其中,3B染色体定位的特异标记最多,达到5 756对,其次是2B染色体上,为5 680对,最少的是4D染色体上,为2 598对(图2)。由于3B染色体测序时,没有按照长臂(L)与短臂(S)分开,所以定位时没有分开长臂与短臂。
2.3 全基因组中不同染色体间标记的定位分析
利用“中国春”小麦全基因组中开发的102287对单位点SSR标记分别与每个染色体进行定位比较,结果显示,每个染色体上定位的单位点标记除了本染色体的标记外,还有别的染色体上开发的标记,说明小麦染色体间存在部分同源区段。而除了本染色体的标记外,定位到不同染色体上最多的标记来源主要集中在2B和5A染色体上(表1)。
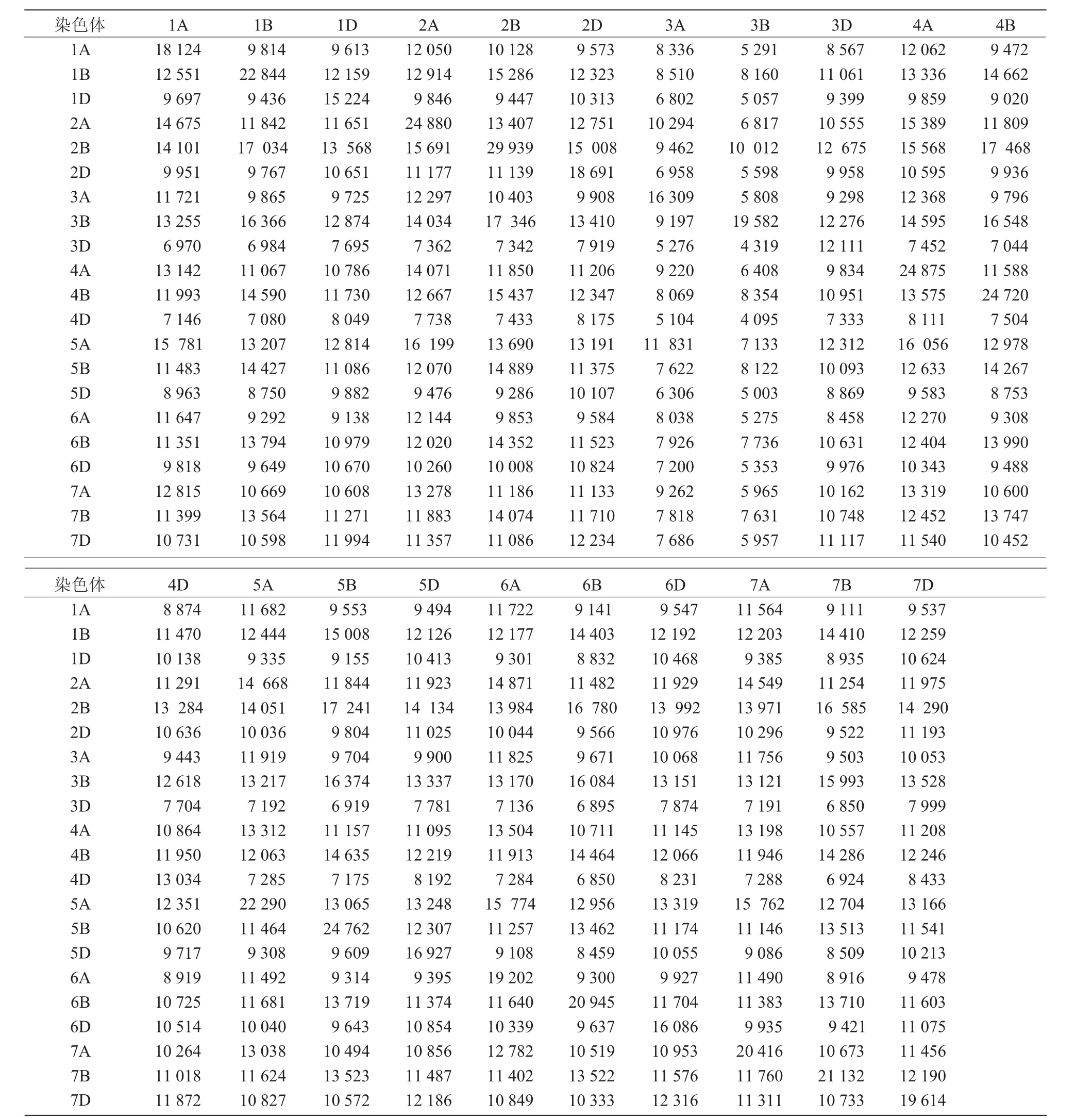
表1 不同染色体间单位点SSR标记定位结果
3 讨论与结论
SSR分子标记具有实用性强、检测快捷、重演性好和价格便宜等优点,成为分子育种较好的选择。此外,SSR标记较SNP标记应用广泛,主要是由于SSR标记能比二态性的SNP标记揭示更多的信息[12]。目前,已经超过4 000个小麦SSR标记被开发出来,并已运用到小麦遗传图谱的绘制工作中,这些分子标记在共线性图谱和比较作图以及加速分子标记密度的工作中将发挥积极的作用。此外,小麦染色体1AL和5DS上的SSR分子标记被相继开发出来[13-14]。LUCAS等[13]从小麦1AL染色体上鉴别出362个SSR标记和6 948个ISBP标记,然后对44个标记(8个SSR标记,26个ISBP和10个SSR及ISBP混合标记)进行了多态性检测,结果显示,23个(52.3%)标记是有用的。这些工作对于小麦染色体图谱的绘制和小麦分子标记辅助选择育种的后续研究将发挥重要的作用。
到目前为止,有大量的SSR分子标记在遗传多样性的检测、建立核心种质资源库、研究种群结构乃至定位目标和功能基因方面发挥着重要的作用[15]。很多EST-SSR分子标记被从EST序列中开发出来,用于检测小麦、大麦、玉米、水稻和高粱等谷类作物[16-17]的高度通用性。小麦全基因组序列草图的公布[18]加速了小麦种质进化的研究及育种工作。
从“中国春”小麦开发的单位点SSR标记尤其是80 374对特异性标记,对进一步研究小麦遗传多样性、基因组分子进化、指纹图谱构建、品种真实性检测以及分子设计育种等方面具有广阔的前景。
[1]韩斌,王长彪,任永康,等.小麦分子育种研究进展Ⅲ.与小麦主要农艺性状相关的功能基因[J].山西农业科学,2015,43(3):362-365.
[2]韩斌,王长彪,任永康,等.小麦分子育种研究进展Ⅱ.与小麦品质相关的功能基因[J].山西农业科学,2015,43(2):233-236.
[3]PAUX E,SOURDILLE P,MACKAY I,et al.Sequence-based marker development in wheat:Advances and applications tobreeding[J].BiotechnologyAdvances,2011,30(5):1071-1088.
[4]LANDJEVAS,KORZUNV,B魻RNER A.Molecular markers:actual and potential contributions to wheat genome characterization and breeding[J].Euphytica,2007,156(3):271-296.
[5]韩斌,王长彪,任永康,等.小麦分子育种研究进展IV.小麦相关分子标记[J].山西农业科学,2015,43(4):489-492.
[6]MARONE D,LAIDO G,GADALETA A,et al.A high-density consensus map of A and B wheat genomes[J].Theoretical and Applied Genetics,2012,125(8):1619-1638.
[7]李丽,王海岗,张晓丽,等.SSR分子标记在作物遗传育种中的应用[J].山西农业科学,2008,36(3):15-18.
[8]ASP T,FREI U K,DIDION T,et al.Frequency,type,and distribution ofEST-SSRs fromthree genotypes ofLolium perenne,and their conservation across orthologous sequences of Festuca arundinacea,Brachypodium distachyon,and Oryza sativa[J].BMCPlant Biology,2007,7:36.
[9]江汉民,文正华,刘莉莉,等.SSR分子标记鉴定青花菜杂交种‘津青一号’的纯度[J].天津农业科学,2016,22(12):57-59,63.
[10]刘何,辛艳.植物SSR分子标记技术的应用[J].天津农林科技,2015(5):34-37.
[11]HAN B,WANG C,TANG Z,et al.Genome-wide analysis of microsatellite markers based on sequenced database in Chinese springwheat(Triticum aestivum L.)[J].PLoS One,2015,10(11):e0141540.
[12]XU J,LIU L,XU Y,et al.Development and characterization of simple sequence repeat markers providing genome-wide coverage and high resolution in maize[J].DNA Research,2013,20(5):497-509.
[13]LUCAS S J,譒IMKOV魣H.Functional features of a single chromosome arm in wheat(1AL)determined from its structure[J].Functional&Integrative Genomics,2012,12(1):173-182.
[14]WANG Y,HAO C,ZHENG J,et al.A haplotype block associated with thousand kernel weight on chromosome 5DS in common wheat(Triticum aestivum L.)[J].Journal of Integrative Plant Biology,2015,57(8):662-672.
[15]YADAV H K,RANJAN A,ASIF M H,et al.EST-derived SSR markers in JatrophacurcasL.:development,characterization, polymorphism,and transferability across the species/genera[J]. Tree Genetics&Genomes,2011,7:207-219.
[16]TANG J,GAO L,CAO Y,et al.Homologous analysis of SSR-ESTs and transferability of wheat SSR-EST markers across barley,rice and maize[J].Euphytica,2006,151(1):87-93.
[17]LI L,WANG J,GUO Y,et al.Development of SSR markers from ESTs of gramineous species and their chromosome location on wheat[J].Progress in Natural Science,2008,18:1485-1490.
[18]MAYER KF,ROGERSJ,DOLE譕ELJ,et al.A chromosome-based draft sequence of the hexaploid bread wheat(Triticum aestivum)genome[J].Science,2014,345:1251788.
Excavation and Distribution Characteristics of Specific SSR Markers in Genome-wide of"Chinese Spring"Wheat
CUI Ting1,WANGChangbiao2,HANBin2,ZHAOXinghua2,LIUJiang2,RENYongkang3,NIUYuqi3,TANGZhaohui2
(1.College ofBioengineering,Shanxi University,Taiyuan 030006,China;2.Research Center ofBiotechnology,Shanxi AcademyofAgricultural Sciences,Taiyuan 030031,China;3.Institute ofCrop Sciences,Shanxi AcademyofAgricultural Sciences,Taiyuan 030031,China)
SSR markers,especially the specific SSR markers with a single locus on different chromosomes,can provide more convenient tools for research on genome molecular evolution,genetic diversity analysis,fingerprint and genetic map construction,varietal identification,and soon.This paper used bioinformatics technology todevelop 295 267 genome-wide SSR markers on"Chinese spring" wheat.80 374 SSR markers were specific markers with a single locus on the different chromosomes.The specific markers on A,B,D genome were 26 738,31 707,21 929,respectively and 3 293 markers were on all the three genome.Mapping the specific markers on "Chinese spring"wheat genome-wide,the chromosome 3B had the most markers(5 756),secondly 2B(5 680),the chromosome 4D had the least markers.The largest source ofmarkers localized on different chromosomes is concentrated on the 2Band 5Achromosomes.
SSR;wheat;specific markers;a single locus;genome-wide
S512.1
:A
:1002-2481(2017)06-0877-04
10.3969/j.issn.1002-2481.2017.06.03
2017-03-15
农业科技成果转化和示范推广项目(2016cgzh47);山西省农业科学院种业发展项目(2016zyzx50);山西省重点科研项目(201603D221025-2);山西省农业科学院重点研究项目(YGG624)
崔婷(1991-),女,山西襄垣人,在读硕士,研究方向:小麦遗传育种。唐朝晖为通信作者。
