基于小批量梯度下降的神经网络模型估算针叶林生物量
2017-03-27曾小强张化永
曾小强,徐 翔,张化永
(华北电力大学工程生态学与非线性科学研究中心,北京 102206)
陆地生态系统的碳循环是全球碳循环的重要组成部分,在延缓全球气候变暖过程中发挥着举足轻重的作用[1]。作为陆地生态系统的主体,森林的生物量占陆地植被总生物量的85%~90%,天然针叶林生物量是森林生物量的重要组成部分[2]。在《巴黎协定》的框架下,中国设定了四大减排目标之一:增加森林蓄积量和增加碳汇,到2030年中国森林蓄积量要比2005年增加45亿 m3。在这一目标下,衡量、报告、核算森林生物量、碳汇显得尤为重要。迄今为止,国内外对于生物量的测定仍然主要采用经典的手工方法,其工作量大、过程复杂、周期长、代表性差、测定技术没有形成体系,因此不能及时反映大面积宏观生态系统的动态变化及生态环境状况,无法满足现实中的需要[3]。随着“3S”技术的不断发展,基于遥感技术对植被生物量的研究越来越受到学者的广泛关注[4]。
光学遥感数据能够反映植被冠层的光谱信息[5]。因此国内外学者通过光学遥感数据的光谱信息和植被指数等建立与森林生物量的相关模型来反演森林生物量。Blackard等人通过研究MODIS遥感数据、地面覆盖数据等与实测生物量数据之间的关系,绘制美国森林生物量分布图[6];Gong等人通过结合Landsat叶面积指数与GLAS传感器提取的树高数据估算了加利福尼亚森林生物量[7]。王新云等基于极化雷达数据和光学遥感数据等多源遥感数据估算了宁夏荒漠半荒漠草原草地生物量[8]。郭志华等基于TM数据的波段线性和非线性组合,运用逐步回归方法分别估算了针叶林与阔叶林材积[9]。
遥感技术的发展为大尺度森林生物量估算与动态变化的研究提供了方便经济的方法[10]。遥感已成功应用于大区域森林制图、灾害监测,但快速地建立一个广泛通用的跨越时空的精确模型还有很大的局限:大多数生物量估测研究的是实测生物量与遥感数据之间的线性关系,忽略了对非线性森林生物量的研究[11]。而人工神经网络作为近年来兴起的优秀非线性模型,有很强的非线性拟合能力,可映射任意复杂的非线性关系,而且学习规则简单,便于计算机实现,采用并行分布处理方法,使得快速进行大量运算成为可能,能够同时处理定量、定性数据,具有很强的鲁棒性、记忆能力、快速学习能力、非线性映射能力以及强大的自学习能力[12]。
而常用的B-P神经网络训练算法是随机梯度下降法、批量梯度下降,这些训练算法收敛速度很慢,不适合做大范围快速建模:
1)批量梯度下降法(BGD)
由于需要计算整个数据集的梯度以仅执行一次更新,批量梯度下降可能非常慢,对于不适合内存的数据集是棘手的。
2)随机梯度下降法(SGD)
批量梯度下降为大型数据集执行冗余计算,因为它在每次参数更新之前重新计算类似示例的梯度。SGD通过每次执行一次更新来消除这种冗余。SGD相对BGD来说要快很多,但是也存在一些问题,由于单个样本的训练可能会带来很多噪声,使得SGD并不是每次迭代都向着整体最优化方向,因此在刚开始训练时可能收敛得很快,但是训练一段时间后就会变得很慢。在此基础上又提出了小批量梯度下降法,它是每次从样本中随机抽取一小批进行训练,而不是一组。
3)小批量梯度下降法(MBGD)
它充分利用上面2种方法的优点,对每一小批量样本执行更新,小批量梯度下降减小参数更新的方差,使得最终达到稳定的收敛。以往生物量反演模型研究往往忽略了森林覆盖率对生物量反演模型的影响,而本研究基于小批量梯度下降的BP神经网络算法,根据MODIS遥感影像、地面调查数据、地形数据以及森林覆盖率,建立生物量反演模型,寻求快速、准确的生物量遥感估测方法。
1 研究区域
研究区为美国北卡罗莱州国家森林(34°39′~34°25′N, 76°43′~80°11′W)。西部山区属大陆性气候,东南地区属亚热带气候。生长期:沿海为275 d,山区175 d。年均温:东部19°C,中部16°C。山区13°C。7、8月多雨,10、11月最干燥。年均降雨量:沿海地区117~137 cm,西部山区100~200 cm。由于气温、降水变化跨度大,土壤类型丰富,森林的树种丰富,主要有针叶树和阔叶树。主要树种包括火炬松、长叶松、南方松、橡树(图1)。
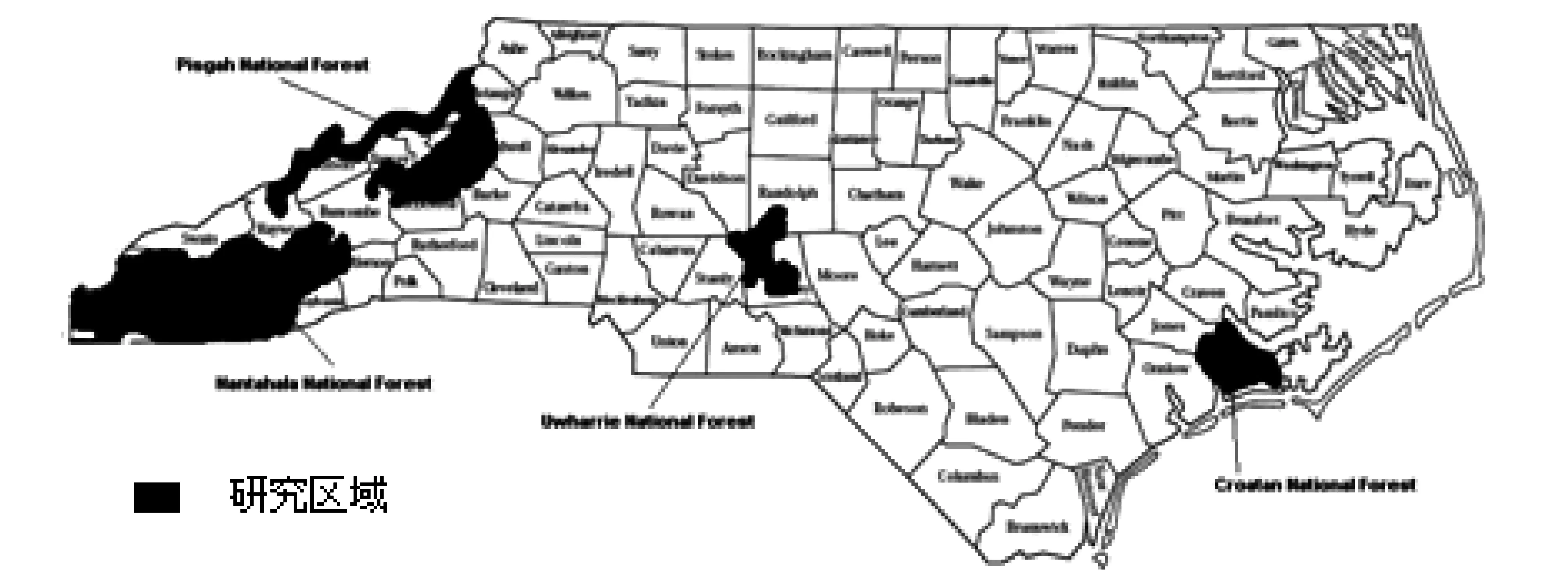
图1 北卡罗莱州国家森林分布Fig.1 Distribution of national forest parks inNorth Carolina
2 方法
2.1 数据
选取MODIS遥感影像波段灰度值(B1~B7)、归一化植被指数(NDVI)、增强型植被指数(EVI)、植被冠层覆盖率(COD)、海拔(DEM)、坡度(SLOPE)、坡向(ASPECT)、土地覆盖类型(LandCover)共14个变量作为森林生物量遥感模型的自变量。卫星影像和土地覆盖类型数据来源于MODIS传感器[13]。植被冠层覆盖率数据来源于基于Landsat传感器数据生成的国家土地覆盖数据集[14]。海拔数据来源于航天飞机雷达地形测绘计划(SRTM),坡度、坡向数据基于海拔数据生成。上述数据有着不同的空间分辨率,对于连续性数据通过双线性插值方法重采样;对于离散型数据通过最邻近法重采样,将这些数据集调整成统一的500 m空间分辨率。
来源于MODIS传感器的自变量数据包括所有的地面反射光谱波段(MOD09A1)、2种植被指数(MOD13A1)和土地覆盖类型,地面发射光谱波段数据是8天合成数据,空间分辨率为500 m,采集时间为2002-03-06。16天合成的植被指数数据(VI)的空间分辨率为250 m,采集时间与地面反射数据一致。MOD09 八天影像合成采用最小蓝光原则来选择最清晰的图像;MOD13 植被指数数据合成算法是首先选择云量少的情况,对双向反射分布函数(BRDF)的像素级拟合,然后计算每个波段的地面反射率来计算植被指数。如果在合成期内少于5个像素是清晰的,则该算法基于视角选择一个清晰的像素,否则选择差异植被指数(NDVI)最大的像素[15];土地覆盖类型数据基于10年(2001—2010)的MODIS土地覆盖类型数据(MCD12Q1)合成[16],此数据的空间分辨率为500 m。
森林冠层覆盖率基于ETM+传感器30 m影像,利用回归树建立森林冠层与遥感影像经验模型并利用模型外推[17]。该模型在3个验证区域相关系数分别高达89%、85%、87%,可见该数据集准确性有一定的保证。由于空间分辨率为30 m,需用双线性重采样法调整空间分辨率为500 m。
来源于航天飞机雷达地形测绘使命(SRTM)系统制成的全球海拔数据(DEM)SRTM3 SRTM, 此数据空间分辨率也是30 m,同样需要基于双线性重采样调整空间分辨率。利用此数据提取坡度(SLOPE)和坡向(ASPECT)数据。
森林生物量训练数据与验证数据是基于美国森林蓄积和分析项目(FIA)提供的树种、树高、胸径等样地数据,通过相对生长法推算出森林生物量。
2.2 模型方法
利用ArcGIS软件将地面反射率各波段、植被指数、土地覆盖类型等模型自变量栅格数据转换投影,转换到统一的投影系统(USA Contiguous Albers Equal Area Conic);然后,重采样调整空间分辨率为500 m;再将这些栅格数据进行空间合并,即根据空间位置将这些栅格数据表合并,提取有地面样方空间位置落入的栅格;最后,筛选出栅格的土地覆盖类型为常绿针叶林的栅格作为模型的训练和验证数据。
本研究采用B-P神经网络构建森林生物量遥感模型。常用的标准B-P神经网络的传递函数是Sigmoid型可微函数,可以实现输入和输出间的任意非线性映射。但是标准B-P算法常用的优化算法是批量梯度下降和随机梯度下降,实现权矢量的更新。而小批量梯度下降在收敛速度和收敛到全局最优解的表现上优于前两者,因此本文采取小批量梯度优化算法来训练B-P神经网络。
因为神经网络模型对不相关的因子比较敏感,如果对于模型原始数据不加以筛选,会降低模型的预测能力,故先对原始数据进行皮尔逊相关性检验。用SPSS软件分别对每个自变量与生物量进行皮尔逊检验,筛选出与生物量显著相关的因子作为模型的因子。对进入模型的因子再进行数据归一化预处理,即:y=(x-min)/( max - min ),其中min为x的最小值,max为x的最大值,输入向量为x,归一化后的输出向量为Y,将数据归一化到[0,1]。
2.2.1 基于小批量梯度下降的B-P神经网络森林生物量遥感模型的建立
为了提高建立一个广泛通用的跨越时空的生物估测模型的速度和精确度,满足模型的应用需求,本研究主要是从模型方法的选择、训练算法的优化、自变量的选择来构建生物量估测模型。图2为构建B-P神经网络森林生物量模型的步骤。
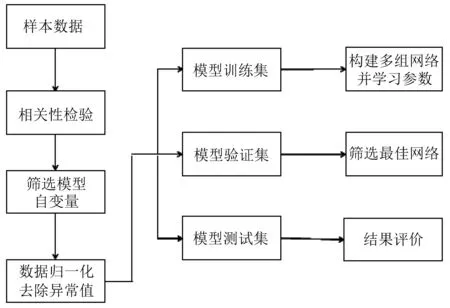
图2 构建B-P神经网络森林生物量模型的步骤Fig.2 Establishment procedure of BP neural networkbiomass model
由于不同数据分辨率不同,而这些数据都是栅格形式,需要利用ArcGIS重采样将其转化成同一分辨率。本研究采用的是双线性插值法,重采样后产生部分异常数据[6],需要根据范文义[26]文中方法(通过设置检验异常样本的标准化残差阈值)来删除异常样本点。
2.2.2 建立网络结构
由于单个隐藏层的B-P神经网络已经具备强大的非线性拟合能力,在此主要研究单隐藏层模型(输入层神经元个数和输出层神经元个数是确定的,故只需设置隐藏层个数即可)。对于单隐藏层神经网络来说,隐藏层神经元个数越多,模型拟合非线性能力越强。本研究探索满足精度要求前提下,尽可能地减少隐藏层神经元个数以降低模型计算复杂度,设置了一组网络结构,其隐藏层神经元个数分别为(20,50,100,150,200,500)。将数据集随机按60%、20%、20%比例分为训练集、测试集和验证集,分别在训练集上训练不同网络结构的神经网络模型,然后在测试集上测试模型的均方误差,选择较低的均方误差中神经元个数较少的网络结构,最后在验证集上评价B-P神经网络模型的表现。
不同网络结构神经网络模型分别在测试集上进行模型验证,评价各模型的性能,即计算模型训练时间和模型在测试集上的均方误差。
3 结果与讨论
3.1 单因素相关性分析
用SPSS软件分别对每个自变量与生物量进行皮尔逊检验,结果如表1。可以看出,遥感反射波段B1、B2、B3、B4、B7,植被指数NDVI、EVI,坡度、海拔、森林覆盖率通过Pearson相关性检验。
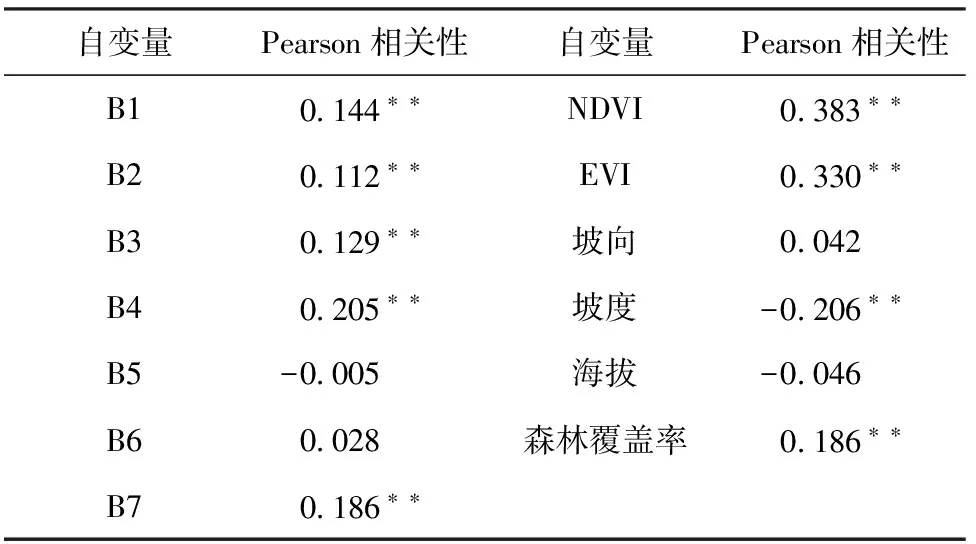
表1 Pearson相关性检验结果
注:**表示在0 .01水平(双侧)上显著相关。
3.2 筛选网络结构
神经网络的训练方法为小批量梯度下降法,批量个数为5,学习速率为0.01,网络训练次数为 1 000。5个不同的网络结构的测试集均方误差与训练时间结果如表2。随着隐藏层神经元个数的增加,模型的均方误差逐步减小。当隐藏层神经元个数增加到200个时,再增加神经元个数,均方误差变化很小,基本上趋于稳定,故选择隐藏层神经元个数为200作为模型最终的隐藏层神经元个数。
3.3 3种模型在同一测试集上预测结果
3.3.1 小批量梯度下降的神经网络
为了评价模型建模过程中的过拟合和欠拟合现象,对模型结果进行验证必不可少。随机选择了未参与模型训练和测试的样地数据65个,用实测数据作为样地的真实值来验证模型,交叉验证集中样地的反演预测值与真实值对比(图3)。可以看出,图中各点都分布在1∶1线附近,R2=0.835。使用单一日期的被动光学遥感数据建立的生物量经验模型的最显著限制是在高生物量区域的数据饱和问题[18,19],这与图3的规律一致。
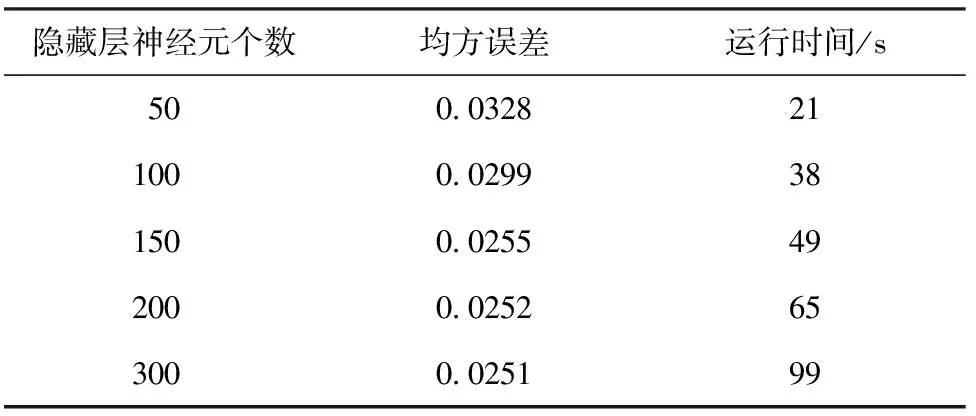
表2 不同网络结构模型的性能
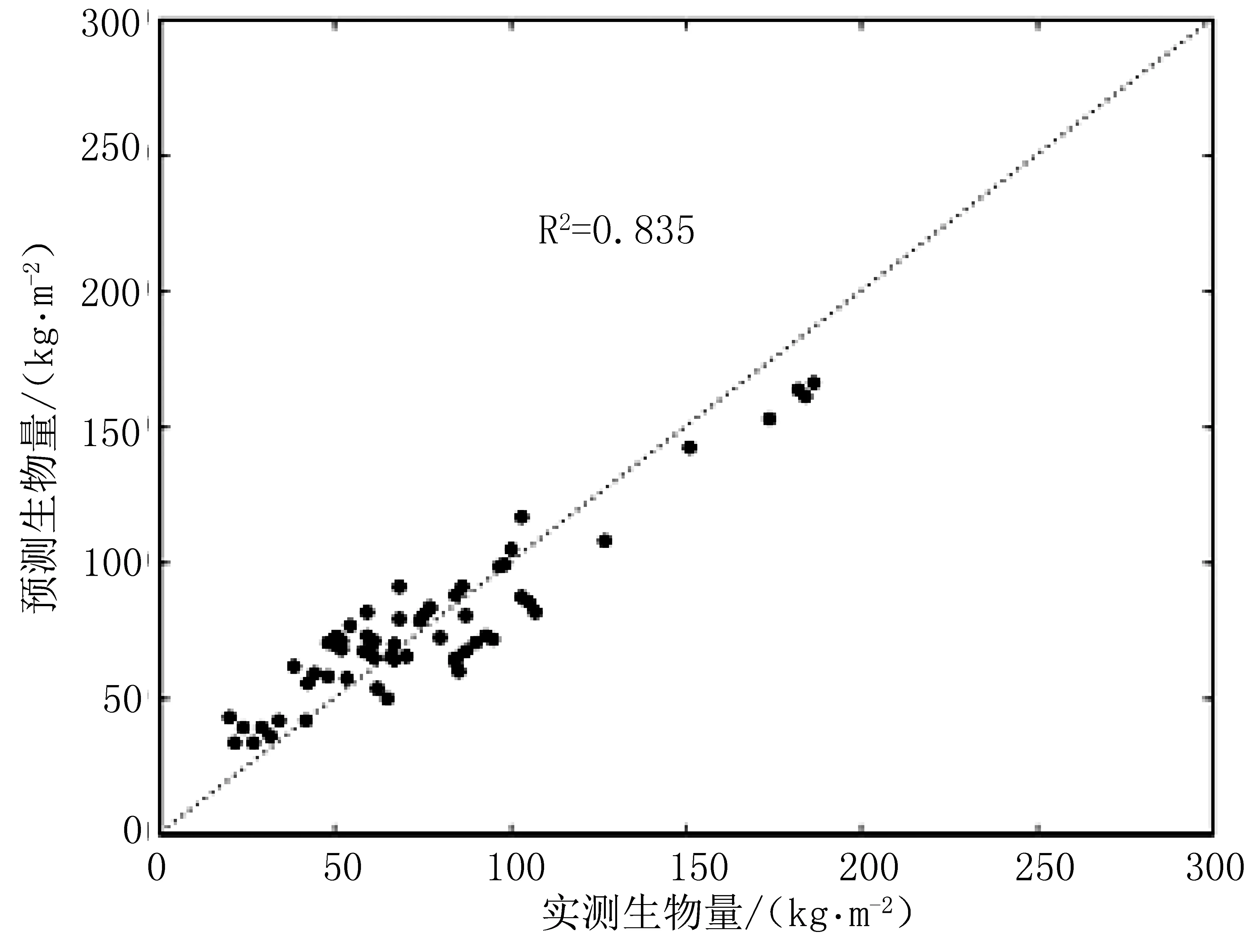
图3 小批量神经网络模型交叉验证集中样地的预测生物量与真实生物量的对比Fig.3 Comparison between predicted biomass andmeasured biomass in cross-validation concentrated samplesof small batch neural network
3.3.2 逐步回归
用传统的逐步回归方法建模,模型结果见表3。线性模型的调整R2仅有0.419,前文MODIS各波段皮尔逊相关系数都小于0.4,说明在原始波段数据基础上,增加植被指数数据和地形数据能明显提高模型的准确性,这与很多研究结果一致[20,21]。但海拔数据并没有进入模型,这与Ohmann 和Gregory[22]的研究结果相矛盾,他们的研究区域海拔梯度变化很大,对生物量影响比较大,本研究区域海拔梯度变化较小,对生物量的影响也较小。
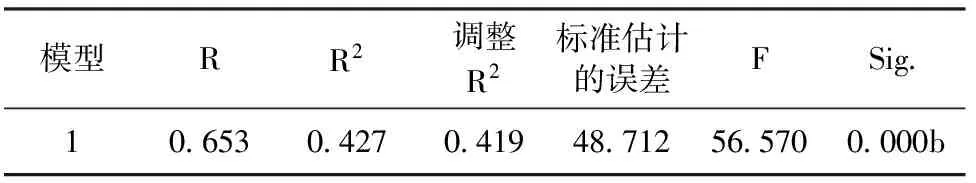
表3 逐步回归模型结果
3.3.3 Erf-BP
基于范文义[26]文章方法建立模型,模型结果如图4、表4。
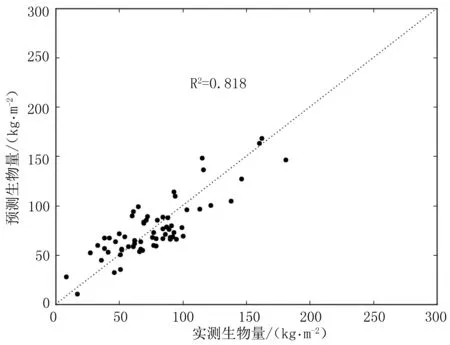
图4 Erf-BP交叉验证集中样地的预测生物量与真实生物量的对比Fig.4 Comparison between predicted biomass andmeasured biomass in cross-validation concentrated samplesof Erf-BP
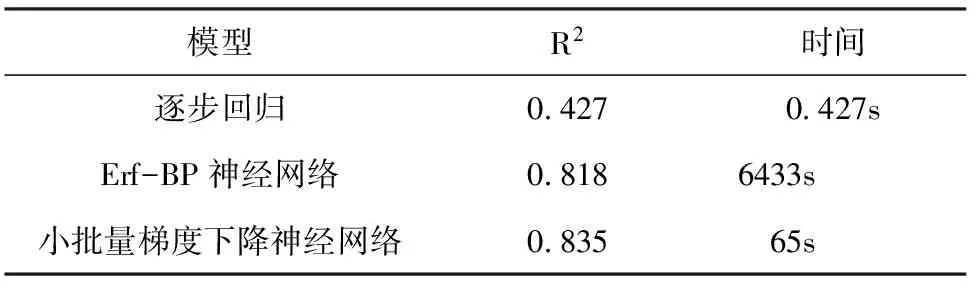
模型R2时间逐步回归04270427sErf-BP神经网络08186433s小批量梯度下降神经网络083565s
3.4 三种模型对比分析
可以看出,基于逐步回归方法建立的模型训练速度最快,但是模型的精度最低,而小批量梯度下降神经网络模型在收敛速度和精度上都好于Erf-BP神经网路,说明此模型在北卡罗莱州国家森林数据上更适用。
综合比较近些年相关文献(表5),本研究的验证集R2=0.835,与近年的文献比较,R2比较接近。同时,本研究验证数据有65个,是这些相关文献中较多的,更能说明模型预测的准确性。与王立海[23]的文献对比,可以发现,本研究隐藏层神经元个数远小于其隐藏层神经元个数,也即是说本研究神经网络结构相对简单;本研究验证集上的R2略微低于其R2,不过也保持较高精确度;本研究的训练速度很快,仅仅只有65 s,而王的训练时间为12 h。考虑到不同计算机计算速度的差别和神经网络结构复杂程度不同给训练速度造成影响,在一定程度上,可以说明本研究学习算法的速度优越性,在实时监测森林生物量有一定的应用潜力。但是,在不同的森林条件下,关于哪种统计模型是最稳健并没有达到共识[24],这往往是由于模型参数变量不同,研究的是特定的森林系统、特定的环境,大气、太阳角度、图像采集时的植被物候条件都外生地影响着模型的结果[25]。

表5 相关文献对比
4 结语
本研究初步探究了利用样地实测生物量和MODIS卫星遥感图像波段数据、2种植被指数、植被覆盖率、地形数据、地面覆盖类型等数据,通过基于小批量梯度递减的B-P神经网络方法估算了天然针叶林地上生物量。结果表明,基于小批量梯度递减的B-P神经网络方法显著地提高模型收敛速度的同时,模型反演预测值逼近真实值,保证了较高的准确度。B-P神经网络模型很好地刻画了地上生物量与遥感数据、植被覆盖率、地形数据间的非线性关系。
由于森林结构参数(树高、冠层面积、林龄等)能显著地提高模型的准确性[20,28],而结构参数(树高、冠层面积)往往通过激光雷达等遥感数据提取,拟在今后工作中通过激光雷达数据引入森林结构参数。另外,由于本研究不同栅格数据的分辨率不同,采用的是双线性插值法重采样调整到同一分辨率,这给模型带来了一定的不确定性,需要进一步研究不同重采样方法和分辨率大小给模型带来的影响。
[1] 周广胜.全球碳循环[M].北京:气象出版社,2003.
[2] 张建设,王刚.植物生物量研究综述[J].四川林业科技,2014,35(1):44-48.
[3] 陶波,葛全胜,李克让,等.陆地生态系统碳循环研究进展[J].地理研究,2001,20(5):564-575.
[4] 张慧芳,张晓丽,黄瑜.遥感技术支持下的森林生物量研究进展[J].世界林业研究,2007,20(4):30-34.
[5] 杨可明,郭达志,陈云浩.高光谱植被遥感数据光谱特征分析[J].计算机工程与应用,2006,42(31):213-215.
[6] BLACKARD J A, FINCO M V, HELMER E H, et al. Mapping US forest biomass using nationwide forest inventory data and moderate resolution information[J]. Remote sensing of Environment,2008,112(4):1658-1677.
[7] ZHANG G, GANGULY S, NEMANI R R, et al. Estimation of forest aboveground biomass in California using canopy height and leaf area index estimated from satellite data[J]. Remote Sensing of Environment,2014,151:44-56.
[8] 王新云,郭艺歌,何杰.基于多源遥感数据的草地生物量估算方法[J].农业工程学报,2014,30(11):159-166.
[9] 郭志华,彭少麟,王伯荪.利用TM数据提取粤西地区的森林生物量[J].生态学报,2002,22(11):1832-1839.
[10] 汤旭光,刘殿伟,王宗明,等.森林地上生物量遥感估算研究进展[J].生态学杂志,2012,31(5):1311-1318.
[11] 王淑君,管东生.神经网络模型森林生物量遥感估测方法的研究[J].生态环境学报,2007,16(1):108-111.
[12] 阎平凡,张长水.人工神经网络与模拟进化计算-第2版[M].北京:清华大学出版社,2005.
[13] JUSTICE C O, GIGLIO L, KORONTZI S, et al. The MODIS fire products[J]. Remote Sensing of Environment,2002,83(1):244-262.
[14] VOGELMANN J E, HOWARD S M, YANG L, et al. Completion of the 1990s National Land Cover Data Set for the conterminous United States from Landsat Thematic Mapper data and ancillary data sources[J]. Photogrammetric Engineering and Remote Sensing,2001,67(6).
[15] HUETE A, DIDAN K, MIURA T, et al. Overview of the radiometric and biophysical performance of the MODIS vegetation indices[J]. Remote sensing of environment,2002,83(1):195-213.
[16] BROXTON P D, ZENG X, Sulla-Menashe D, et al. A global land cover climatology using MODIS data[J]. Journal of Applied Meteorology and Climatology,2014,53(6):1593-1605.
[17] HUANG C, YANG L, WYLIE B K, et al. A strategy for estimating tree canopy density using Landsat 7 ETM+ and high resolution images over large areas[Z]. 2001.
[18] STEININGER M K. Satellite estimation of tropical secondary forest above-ground biomass: data from Brazil and Bolivia[J]. International Journal of Remote Sensing,2000,21(6-7):1139-1157.
[19] TURNER D P, COHEN W B, KENNDY R E, et al. Relationships between leaf area index and Landsat TM spectral vegetation indices across three temperate zone sites[J].Remote sensing of environment,1999,70(1):52-68.
[20] HALL R J, SKAKUN R S, ARSENAULT E J, et al. Modeling forest stand structure attributes using Landsat ETM+ data: Application to mapping of aboveground biomass and stand volume[J]. Forest ecology and management,2006,225(1):378-390.
[21] FOODY G M, BOYD D S, CUTLER M E J. Predictive relations of tropical forest biomass from Landsat TM data and their transferability between regions[J]. Remote Sensing of Environment,2003,85(4):463-474.
[22] OHMANN J L, GREGORY M J. Predictive mapping of forest composition and structure with direct gradient analysis and nearest-neighbor imputation in coastal Oregon, USA[J]. Canadian Journal of Forest Research,2002,32(4):725-741.
[23] 王立海,邢艳秋.基于人工神经网络的天然林生物量遥感估测[J].应用生态学报,2008,19(2):261-266.
[24] LU D. The potential and challenge of remote sensing‐based biomass estimation[J]. International journal of remote sensing,2006,27(7):1297-1328.
[25] POWELL S L, COBEN W B, HEALEY S P, et al. Quantification of live aboveground forest biomass dynamics with Landsat time-series and field inventory data: A comparison of empirical modeling approaches[J]. Remote Sensing of Environment,2010,114(5):1053-1068.
[26] 范文义,张海玉,于颖,等.三种森林生物量估测模型的比较分析[J].植物生态学报,2011,35(4):402-410.
[27] 李丹丹,冯仲科,汪笑安,等.BP神经网络反演森林生物量模型研究[J].林业调查规划,2013,38(1):5-8.
[28] ZHENG D, RADEMACHER J, CHEN J, et al. Estimating aboveground biomass using Landsat 7 ETM+ data across a managed landscape in northern Wisconsin, USA[J]. Remote sensing of environment,2004,93(3):402-411.
