陆地棉微管结合蛋白CLASP家族基因的鉴定及表达分析
2017-03-16朱守鸿赵兰杰刘永昌李艳军张新宇
朱守鸿 赵兰杰 刘永昌李艳军 张新宇 孙 杰
石河子大学农学院 / 新疆生产建设兵团绿洲生态农业重点实验室, 新疆石河子 832003
陆地棉微管结合蛋白CLASP家族基因的鉴定及表达分析
朱守鸿 赵兰杰 刘永昌*李艳军 张新宇 孙 杰
石河子大学农学院 / 新疆生产建设兵团绿洲生态农业重点实验室, 新疆石河子 832003
CLASP蛋白(CLIP-associated protein, CLASP)是一种调节微管结构与功能的微管结合蛋白, 在植物生长发育及形态建成过程中起着至关重要的作用。本研究利用生物信息学的方法, 在陆地棉基因组数据库中鉴定出 6个编码CLASP蛋白的基因。多重序列比对及系统进化树分析表明, 陆地棉CLASPs可分为2个亚家族(I和II), 亚家族I含有HEAT重复结构域和典型的CLASP-N端结构域, 亚家族II只含有CLASP-N端结构域。实时荧光定量PCR结果表明, 6个 CLASPs基因在棉花各组织中均有表达, 但表达模式各不相同, 其中 CotAD_63740 (GenBank登录号为KX881965)和 CotAD_04861 (GenBank登录号为 KX881961)在纤维中优势表达, CotAD_48232 (GenBank登录号为KX881962)、CotAD_48665 (GenBank登录号为 KX881963)、CotAD_55570 (GenBank登录号为 KX881964)和CotAD_68468 (GenBank登录号为KX881966)在茎中优势表达, 表达模式的不同表明CLASPs在棉花不同组织中功能不同。本研究为深入研究棉花CLASPs的功能奠定了基础。
陆地棉; 微管结合蛋白; 微管; 表达分析
在真核生物中, 微管和微管结合蛋白在组织微管阵列和空间上控制细胞的生长和发育[1-3]。目前,已经发现多类微管结合蛋白家族, 这些家族的微管结合蛋白通过多种功能影响微管组织, 包括调节微管动态特性、影响微管之间的相互作用、微管切割和γ-微管蛋白依靠微管分支等[2-5]。
CLASP蛋白是一种调节微管结构与功能的微管结合蛋白[6-12], 能结合微管和细胞质连接蛋白, 由于结合在微管的正端, 也被称为正端示踪蛋白[13-15]。CLASP蛋白是微管结合蛋白家族ORBIT/MAST/ CLASP成员之一[16-17], 含有典型的CLASP-N端保守结构域, 有些CLASP蛋白还含有HEAT重复结构域。在真核生物中, CLASP通过这些功能域调节微管动态活性。
植物中CLASP的功能研究鲜有报道, 大多来自模式植物拟南芥。拟南芥基因组中只有1个CLASP基因[7]。功能缺失突变体 clasp-1表现出株型矮小,主根生长发育缓慢, 侧根增多, 下胚轴短粗, 叶表皮毛分支数目减少, 花序短而粗, 果荚短小等不良性状[7-8,10]。在 PLETHORA-生长素-MAP65-CLASP模型中, CIASP通过定位在细胞的上下两面调控微管列阵的方向从横向排列变为纵向排列, 促使拟南芥根细胞从垂周分裂转变成平周分裂[18]。表明CLASP在植物细胞形态建成和细胞分裂过程中具有重要的作用[7-8,18]。当用微管解聚药剂氨磺乐灵分别处理野生型拟南芥和突变体clasp-1时, 突变体的下胚轴及根生长缓慢, 根毛异常肿胀, 表明AtCLASP促使微管成束抵抗氨磺乐灵解聚, 促进微管稳定性[7-8,18]。同时, CLASP还促进微管到细胞皮层与质膜结合,在细胞边缘抑制微管解聚, 具有稳定皮层微管的功能[7,9,19-21]。有研究表明, GSK3β (glycogen synthase kinase 3β)激酶通过磷酸化作用调节 CLASP蛋白与微管的结合, 从而影响细胞内微管局部的稳定性[22]。因此, CLASP参与调节微管结构与功能, 在细胞形成、形态建成等方面起着关键作用。
迄今, 在陆地棉中仅克隆了 1个 CLASP基因GhCLASP1 (GenBank登录号为KP742966), 其蛋白定位于细胞膜[23], 对其功能没有进一步的报道。本研究利用拟南芥 AtCLASP蛋白(GenBank登录号为AAM13846)为查询序列, 从陆地棉基因组数据库[24-25]查找棉花 CLASPs基因, 并对其进行生物信息学和表达模式分析, 为进一步明确 CLASPs家族基因在棉花生长发育中的功能奠定基础。
1 材料与方法
1.1 试验材料
陆地棉(Gossypium hirsutum L.)品种新陆早33由石河子大学棉花研究所提供, 种植于石河子大学农学院试验站, 常规田间管理。在棉花盛花期挂牌标记当日开花花蕾, 分别摘取6、12、18和24 DPA (days post anthesis, 开花后天数)的棉铃, 室内剥取纤维样品。取棉花种子, 剥去种皮后用0.1% HgCl2浸泡10 min, 灭菌水冲洗3~4次, 播于1/2 MS培养基上, 28℃暗培养4 d, 用Hoagland营养液培养至2片真叶时,取幼苗的根、茎、叶。花从田间植株摘取。上述材料经液氮速冻后保存于–80℃冰箱, 用于RNA的提取。
1.2 陆地棉总RNA提取及cDNA第一链合成
采用 CTAB法提取棉花样品总 RNA[26], 用DNase I处理后, 利用ND1000核酸-蛋白测定仪测定OD260和OD280值, 计算RNA的浓度与纯度。用1.2%琼脂糖凝胶电泳检测RNA的完整性后保存于–80℃备用。按照Reverse Transcriptase M-MLV (RNase H-)说明书合成cDNA的第1链。
1.3 陆地棉CLASPs基因鉴定与生物信息学分析
用 Perl解析四倍体陆地棉基因组数据库中的gff3文件, 获得CLASP基因家族成员的染色体长度,利用MapInspect软件绘制CLASPs基因的染色体物理分布图。用 ProtParam 软件在线(http://web. expasy.org/protparam/)分析CLASPs蛋白质的基本理化性质; SOPMA软件(https://npsa-prabi.ibcp.fr/cgi-bin/ npsa_automat.pl?page=/NPSA/npsa_sopma.html)在线预测和分析 CLASPs蛋白质的二级结构组成形式; EBL-EBI (http://www.ebi.ac.uk/interpro/search/sequencesearch)和NCBI的BlastP分析CLASPs蛋白质保守区; IBS 2.0软件绘制CLASPs蛋白结构域图; WOLF PSORT (http://psort.hgc.jp/)和 TMHMM 软件对CLASPs蛋白进行信号肽、跨膜区及亚细胞定位分析; 采用DNAMAN和ClustalX对CLASPs蛋白CLASP-N结构域进行多重序列比对; 利用 MEGA 6.0软件构建系统进化树, 分析 CLASPs蛋白进化关系。
1.4 陆地棉CLASPs基因在棉花不同组织中的表达分析
以拟南芥AtCLASP蛋白作为探针序列, 与四倍体陆地棉的基因组数据库序列进行Blast比对(http:// cgp.genomics.org.cn/page/species/index.jsp), 获得同源性较高的陆地棉 CLASPs基因。根据陆地棉CLASPs基因的cDNA序列, 用Primer3设计基因特异引物(表1)。以根、茎、叶、花和不同发育时期的棉纤维cDNA为模板, 利用qRT-PCR方法, 检测陆地棉CLASPs基因的表达情况。在LightCycler 480上进行实时荧光定量PCR, 内参基因为UBQ7, 程序为94℃预变性1 min, 95变性15 s, 60℃退火20 s, 72℃延伸30 s, 45个循环。重复3次试验, 每次试验3个重复, 按照2–ΔΔCT法分析数据。
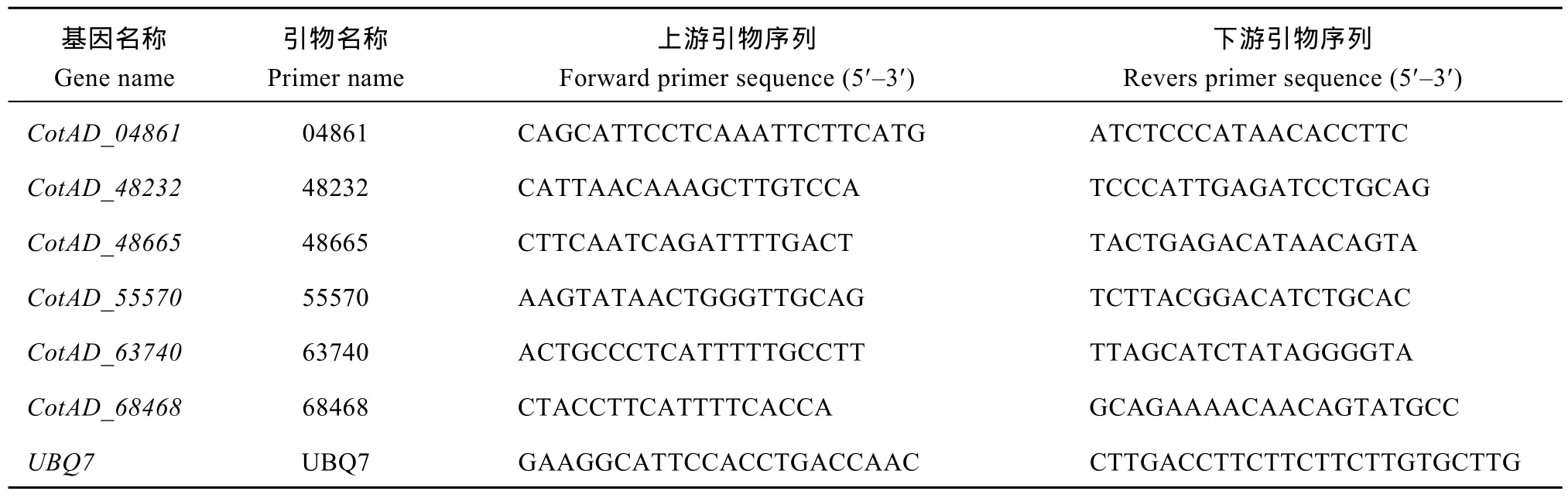
表1 qPCR实验中所用的引物Table 1 The primers used in the qPCR experiment
2 结果与分析
2.1 陆地棉CLASPs基因的序列分析
利用拟南芥AtCLASP蛋白作为探针, 在四倍体陆地棉基因组数据库(http://cgp.genomics.org.cn/ page/species/index.jsp)中比对, 鉴定获得6个与其同源性较高的陆地棉 CLASPs基因, 基因序列号分别为 CotAD_04861、CotAD_48232、CotAD_48665、CotAD_55570、CotAD_63740和CotAD_68468 (GenBank登录号依次为KX881961至KX881966)。基因组序列长度(与 CDS相对应基因组的长度)为 8812~12 183 bp, 编码区长度为 3975~4371 bp, 编码蛋白序列长度为1264~1456个氨基酸(表2)。
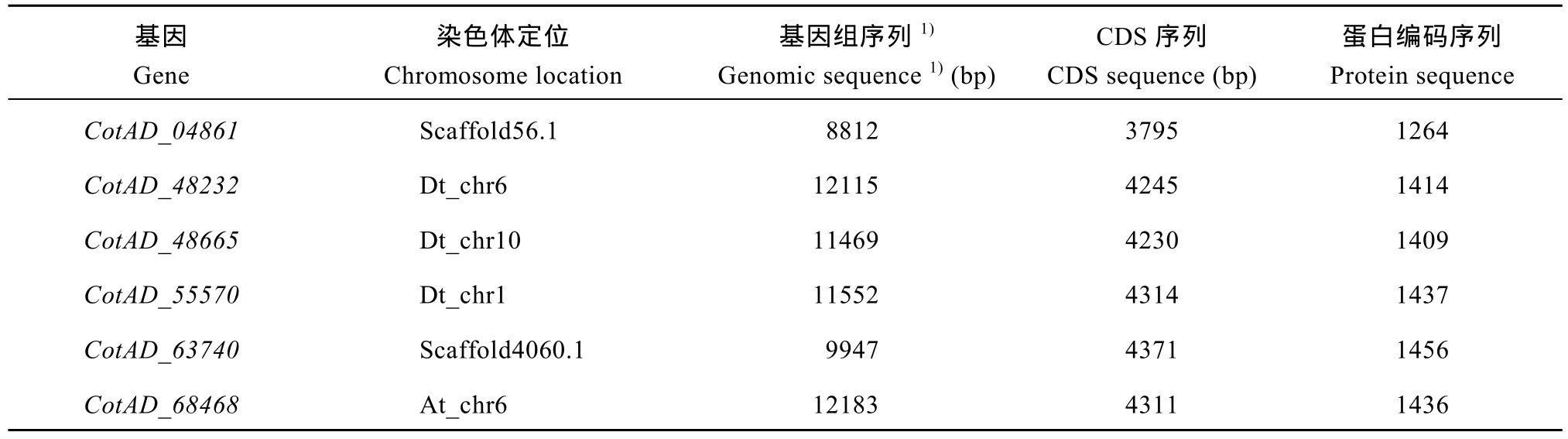
表2 陆地棉CLASPs基因序列信息Table 2 Sequence information of GhCLASPs genes in genome database of Gossypium hirsutum
利用http://cgp.genomics.org.cn/page/species/index. jsp网站进行Blast比对发现CotAD_04861、CotAD_ 48665、CotAD_55570和 CotAD_63740与拟南芥AtCLASP蛋白序列一致性较高, 分别为73%、66%、68%和69%。CotAD_68468和CotAD_48232与拟南芥 AtCLASP蛋白序列一致性较低, 分别为 50%和51% (表3)。根据与拟南芥AtCLASP蛋白序列的一致性, 将CotAD_04861、CotAD_48665、CotAD_55570和CotAD_63740分为一类亚家族蛋白, 命名为I类亚家族蛋白, 将CotAD_68468和CotAD_48232分为另一类亚家族蛋白, 命名为II类亚家族蛋白。
2.2 陆地棉CLASPs基因编码蛋白的生物信息学分析
四倍体陆地棉基因组数据库中鉴定出的 6个CLASPs基因家族成员分布在4条染色体和2条Scaffold上, 分别为 D01、A06、D06、D10、Scaffold56.1和Scaffold4060.1 (图1)。在陆地棉的A亚组和D亚组中只有一对共线性同源 CLASP基因, 分别为 CotAD_ 68468和CotAD_48232, 分布在A06和D06染色体上。
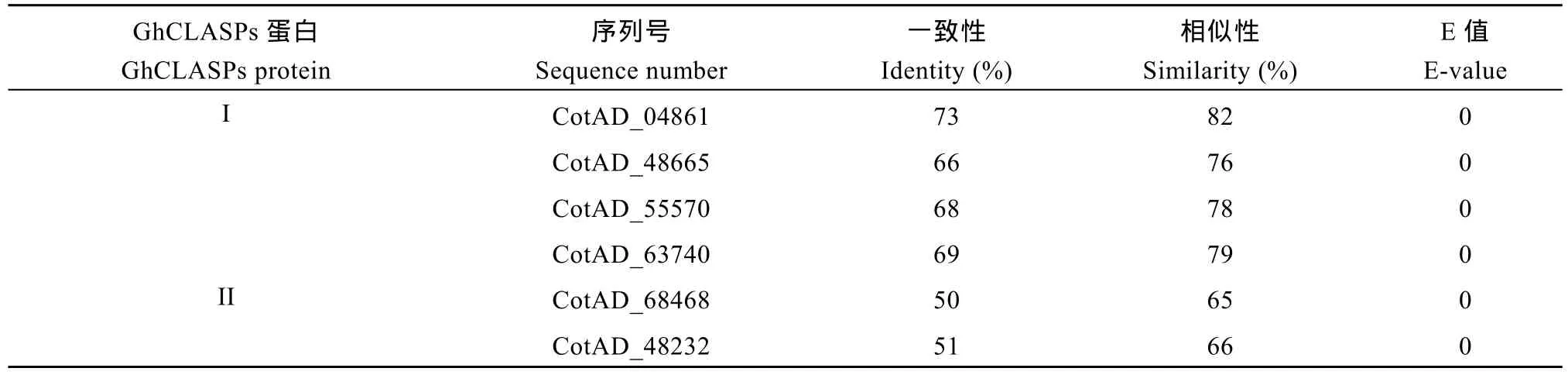
表3 陆地棉CLASPs蛋白与拟南芥AtCLASP氨基酸序列一致性分析Table 3 Identity analysis of amino acid sequence of GhCLASPs proteins and AtCLASP
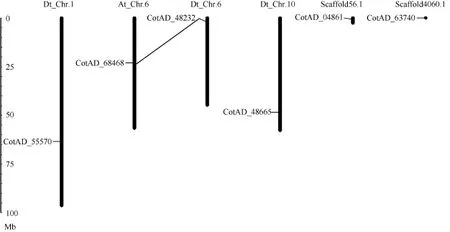
图1 陆地棉亚基因组上GhCLASPs基因的分布Fig. 1 Mapping of the GhCLASPs genes on upland cotton subgenomes线条代表共线性同源基因。Lines represent collinear homologous genes.
利用ProtParam预测分析6个陆地棉CLASPs蛋白的基本理化性质(表4)表明, 这些蛋白的分子量为139.9~61.1 kD, 理论等电点为6.40~6.92, 负电荷的氨基酸残基数(Asp+Glu)为 152~171, 正电荷的氨基酸残基数(Arg+Lys)为 144~165, 脂肪系数为91.24~96.82, 总平均亲水性为 0.155~0.226, 不稳定系数为44.52~48.08, 均为亲水性不稳定蛋白质。
利用SOPMA在线预测分析(表5)表明, 这6个陆地棉 CLASPs蛋白的二级结构都是以 α-螺旋、延伸直链、β-转角和无规则卷曲 4种结构形式组成。其中以 α-螺旋为主, 超过 45%, 而延伸直链、β-转角和无规则卷曲少于 35%。可见, α-螺旋是6个陆地棉CLASPs蛋白二级结构的主要组成部分。
6个陆地棉CLASPs蛋白均具有2个与微管结合的典型 CLASP-N 端结构域(图 2)。其中, CotAD_04861、CotAD_63740和CotAD_55570含有2个 HEAT重复结构域, CotAD_48665只含有一个HEAT重复结构域, CotAD_48232和 CotAD_68468不含有HEAT重复结构域。这些HEAT重复结构域和CLASP-N端结构域是CLASPs蛋白行使其正常功能所必需的。
6个陆地棉CLASPs均没有信号肽和跨膜区, 其中CotAD_63740定位在线粒体膜上, 其他的陆地棉CLASPs蛋白定位在细胞膜上(表 5)。由此可见, 这些蛋白都具有膜定位信号, 由于定位在不同的膜上,故推测 CLASPs蛋白具有不同的功能, 且某些成员具有特殊功能。
2.3 陆地棉CLASPs蛋白进化关系
利用 DNAMAN和 ClustalX将 6个陆地棉CLASPs蛋白的 CLASP-N端结构域与已经报道的AtCLASP和GhCLASP1蛋白的CLASP-N端结构域进行多重序列比较(图 3), 结果表明, 其同源区域主要集中在2个串联的CLASP-N端结构域。



图2 陆地棉CLASPs蛋白与AtCLASP蛋白结构比较Fig. 2 GhCLASPs proteins and AtCLASP protein structure黑色和斜线区域分别表示蛋白的CLASP-N端结构域和HEAT重复结构域。Black blocks and that filled with oblique lines represent the CLASP-N terminal and HEAT repeat domain of protein, respectively.
系统进化树表明, CotAD_04861、CotAD_ 48665、CotAD_55570和 CotAD_63740聚为一类, CotAD_68468和CotAD_48232及GhCLASP1聚为一类。进一步明确了6个CLASPs蛋白在CLASP家族中的类别(图4)。
2.4 陆地棉CLASPs基因的表达模式分析
6个陆地棉CLASPs基因在根、茎、叶、花及棉纤维中均能表达, 但表达模式各不相同(图 5)。CotAD_48232、CotAD_48665、CotAD_55570 和CotAD_68468在茎中优势表达, 其中 CotAD_48232和 CotAD_68468在叶中表达量也较高, CotAD_48665和CotAD_55570在根中表达量也较高。CotAD_63740在茎中表达量较高, CotAD_04861在茎和花中表达量较高。CotAD_63740、CotAD_48232和CotAD_68468在纤维中的表达量呈现先升高再降低的趋势, CotAD_04861、CotAD_48665和CotAD_ 55570在纤维中的表达呈现上升趋势。CotAD_55570在6DPA表达量较高, CotAD_63740、CotAD_04861、CotAD_48232、CotAD_48665和 CotAD_68468在24DPA纤维中表达量明显较高, 但在其他时期表达量均较低。CotAD_48232、CotAD_48665、CotAD_ 55570和CotAD_68468在纤维中的表达量明显低于在其他器官中, 因此, CotAD_48232、CotAD_48665、CotAD_55570和CotAD_68468在纤维中均没有表达优势, 6个陆地棉CLASPs基因中只有CotAD_ 63740和CotAD_04861在纤维中优势表达, 明显高于在其他器官中, 且主要在纤维发育的中后期表达, 其表达量随着纤维的发育逐渐升高, 暗示这些基因在棉纤维发育过程中发挥着重要的作用。
3 讨论
本研究共鉴定出6个陆地棉CLASPs基因成员,其编码的蛋白长度各异, 均具典型的CLASP-N端结构域, 有的成员还具有HEAT重复结构域。HEAT结构域能够形成螺旋结构, 调节蛋白之间的相互作用,参与胞内物质的转运[27-28]。CLASP-N端结构域是很保守的结构域, 不但是微管结合的部位, 还具有调节动态微管的稳定性[29-30]。由于CLASP功能域的不同, 表明陆地棉 CLASPs蛋白成员间可能存在功能上的差异; 蛋白理化性质预测结果表明, 6个陆地棉CLASPs蛋白都为偏酸性, 并且都是亲水性不稳定蛋白, 相似的理化性质预示各个成员间可能具有某些相似的功能。系统进化树将 6个陆地棉 CLASPs蛋白分为2个亚类。CotAD_04861、CotAD_48665、CotAD_55570和 CotAD_63740属于 I类, CotAD_68468和CotAD_48232属于II类。陆地棉中已报道的GhCLASP1属于CLASPs II类亚家族成员,与 CotAD_48232同源性最高, 在同一个分支, 表明四倍体陆地棉不同品种间的核酸序列之间存在一些差异。由于四倍体陆地棉基因组A亚组和D亚组的基因存在很高的共线性[24-25], 6个陆地棉CLASPs基因成员仅有 1对共线性基因(CotAD_68468和 CotAD_ 48232)。系统进化树中CotAD_04861、CotAD_48665、CotAD_55570和CotAD_63740聚为一类, 推测分布在2条Scaffold上的CotAD_04861和CotAD_63740可能与CotAD_48665和CotAD_55570也存在共线性关系。CotAD_04861和CotAD_63740具体分布在哪条染色体上, 还有待进一步研究。
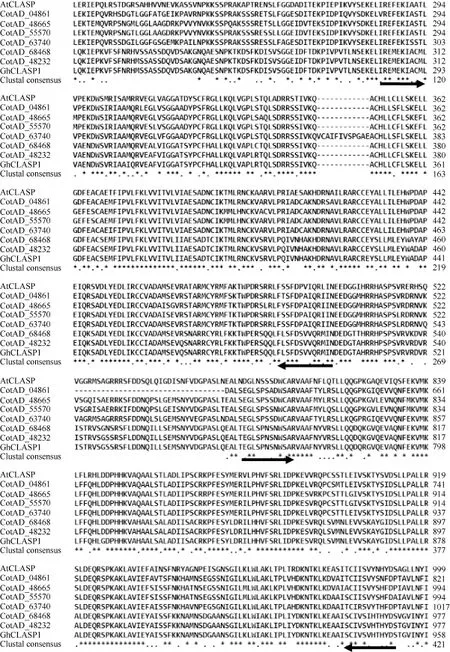
图3 陆地棉CLASPs蛋白与拟南芥AtCLASP蛋白的CLASP-N端结构域多重序列比对Fig. 3 Amino acid sequences alignment of GhCLASPs proteins and AtCLASP protein黑色箭头表示2个CLASP-N端结构域。所用植物CLASP来自拟南芥(AAM13846)、陆地棉(CotAD_48232、CotAD_48665、CotAD_04861、CotAD_68468、CotAD_63740和CotAD_55570)和GhCLASP1(AKA60051)。The black arrows show two CLASP-N terminal domains of protein. The plant CLASP proteins used are from Arabidopsis thaliana(AAM13846), Gossypium hirsutum (CotAD_48232, CotAD_48665, CotAD_04861, CotAD_68468, CotAD_63740, and CotAD_55570), and GhCLASP1 (AKA60051).
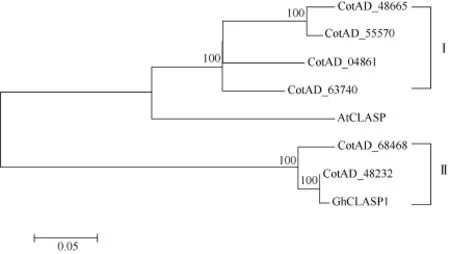
图4 陆地棉CLASPs蛋白与拟南芥AtCLASP蛋白的系统进化树Fig. 4 Phylogenic relationship of GhCLASPs proteins and AtCLASP proteins系统树各分支上数字是BootStrap 1000次循环检验的置信度; 标尺代表遗传距离。所用植物CLASP来自拟南芥(AAM13846)、陆地棉(CotAD_48232、CotAD_48665、CotAD_04861、CotAD_68468、CotAD_63740和CotAD_55570)和GhCLASP1(AKA60051)。The numbers on the tree branches represent bootstrap confidence values as BootStrap is 1000; the scale bar represents genetic distance.The plant CLASP proteins used are from Arabidopsis thaliana (AAM13846), Gossypium hirsutum (CotAD_48232, CotAD_48665, CotAD_04861, CotAD_68468, CotAD_63740, and CotAD_55570), and GhCLASP1 (AKA60051).
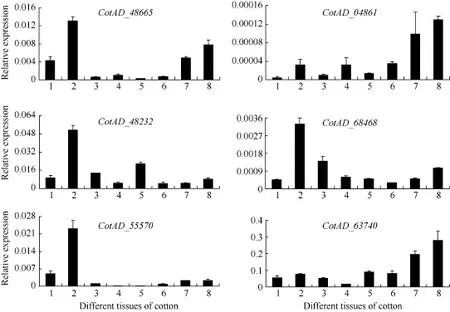
图5 CLASPs基因在棉花不同组织中的qRT-PCR分析Fig. 5 Quantitative Real-time PCR analyses of GhCLASPs genes expression in different tissues in cotton1: 根; 2: 茎; 3: 叶; 4: 花; 5~8: 开花后6、12、18和24 d纤维。1: root; 2: stem; 3: leaf; 4: flower; 5–8: fiber cells at 6, 12, 18, and 24 DPA, respectively.
6个陆地棉CLASPs基因在棉花各组织中均有表达, 但表达模式各不相同, 其中, CotAD_ 48232、CotAD_48665、CotAD_55570和CotAD_ 68468在茎中优势表达, 尤其是 CotAD_55570在茎中高表达,均高于根、叶、花和不同发育时期的表达, 推测这些基因可能参与茎部细胞内物质的运输。有研究表明, 在拟南芥中由于AtCLASP功能缺失抑制了生长素等物质的运输, 使拟南芥clasp-1表现出和植物缺乏生长素相似的表型, 如株型矮小, 根发生很多分支, 植株发育缓慢, 荚果短小[10,31-33]。微管是植物物质运输的通道, 棉花中CLASP怎样调控微管参与细胞内物质的运输, 还有待进一步研究。CotAD_63740和CotAD_04861在纤维中优势表达, 主要在纤维发育的中后期表达, 尤其是在24DPA纤维中表达量最高。该时期正是纤维细胞发育的次生壁加厚期[34-35],暗示这2个基因可能参与棉纤维次生壁的发育。在棉花纤维细胞壁次生加厚过程中, 周质微管数量显著增加, 其排列方向与新形成的纤维层的方向一致[36], 而棉纤维细胞次生壁的合成与周质微管的排列模式相关[37-38], 因此推测这 2个基因可能通过调控微管动态重组及排列方向的变化来影响棉纤维发育。所以, CotAD_63740和CotAD_04861基因具有进一步研究的价值, 可将分子生物学和遗传学方法运用到解析棉花纤维生长发育调控机制研究中, 并可将其选为目标基因转化棉花改良棉花纤维的品质。
陆地棉中已报道的GhCLASP1属于CLASPs蛋白II类亚家族成员, 被定位于细胞膜[23]。本研究中预测到5个陆地棉CLASPs蛋白被定位在细胞膜上,推测它们可能与已报道的GhCLASP1存在功能冗余,与细胞内物质的运输相关。而CotAD_63740被定位在线粒体膜上, 由于细胞中线粒体分布方向和微管一致, 线粒体在细胞质中向功能旺盛的区域迁移是以微管为导轨的[39-40], 所以CotAD_63740可能通过调控微管来影响纤维细胞中线粒体的结构、分布或功能, 参与纤维发育调控。
4 结论
从陆地棉中鉴定到 6个 CLASPs基因, 分别是CotAD_04861、CotAD_48232、CotAD_48665、CotAD_ 55570、CotAD_63740和CotAD_68468。它们编码的蛋白在 1264~1456个氨基酸残基之间, 均含有典型的CLASP-N端结构域和微管结合位点, 分为2个(I和 II)亚类。它们在棉花各组织中均有表达, 表达模式各不相同, 其中CotAD_63740和CotAD_04861在纤维中优势表达, 在纤维发育过程中可能发挥着重要作用。
[1] Sedbrook J C. MAPs in plant cells: delineating microtubule growth dynamics and organization. Curr Opin Plant Boil, 2004, 7: 632–640
[2] Hamada T. Microtubule-associated proteins in higher plants. J Plant Res, 2007, 120: 79–98
[3] Hamada T. Microtubule organization and microtubule-associated proteins in plant cells. Int Rev Cel Mol Biol, 2014, 312: 1–52
[4] Tian J, Han L B, Feng Z D, Wang G D, Liu W W, Ma Y P, Yu Y J, Kong Z S. Orchestration of microtubules and the actin cytoskeleton in trichome cell shape determination by a plant-unique kinesin. eLife, 2015, 4: e09351
[5] Kong Z S, Ioki M, Braybrook S, Li S D, Ye Z H, Lee Y R J, Hotta T, Chang A, Tian J, Wang G D, Liu B. Kinesin-4 functions in vesicular transport on cortical microtubules and regulates cell wall mechanics during cell elongation in plants. Mol Plant, 2015, 8: 1011–1023
[6] Akhmanova A, Hoogenraad C C, Drabek K, Stepanova T, Dortland B, Verkerk T, Vermeulen W, Burgering B M, Zeeuw C I D, Grosveld F, Galjart N. CLASPs are CLIP-115 and -170 associating proteins involved in the regional regulation of microtubule dynamics in motile fibroblasts. Cell, 2001, 104: 923–935
[7] Ambrose J C, Shoji T, Kotzer A M, Pighin J A, Wasteneys G O. The Arabidopsis CLASP gene encodes a microtubule-associated protein involved in cell expansion and division. Plant Cell, 2007, 19: 2763–2775
[8] Kirik V, Herrmann U, Parupalli C, Sedbrook J C, Ehrhardt D W, Hülskamp M. CLASP localizes in two discrete patterns on cortical microtubules and is required for cell morphogenesis and cell division in Arabidopsis. J Cell Sci, 2007, 120: 4416–4425
[9] Ambrose C, Allard J F, Cytrynbaum E N. A CLASP-modulated cell edge barrier mechanism drives cell-wide cortical microtubule organization in Arabidopsis. Nat Commun, 2011, 2: 430
[10] Ambrose C, Ruan Y, Gardiner J. CLASP interacts with sorting Nexin 1 to link microtubules and auxin transport via PIN2 recycling in Arabidopsis thaliana. Dev Cell, 2013, 24: 649–659
[11] Gardiner J. The evolution and diversification of plant microtubule-associated proteins. Plant J, 2013, 75: 219–229
[12] Pietra S, Gustavsson A, Kiefer C, Kalmbach L, Horstedt P, Ikeda Y, Stepanova A N, Alonso J M, Grebe M. Arabidopsis SABRE and CLASP interact to stabilize cell division plane orientation and planar polarity. Nat Commun, 2013, 4: 2779
[13] Carvalho P, Tirnauer J S, Pellman D. Surfing on microtubule ends. Trends Cell Biol, 2003, 13: 229–237
[14] Mimori-Kiyosue Y, Tsukita S. “Search-and-capture” of microtubules through plus-end-binding proteins (+TIPs). J Biochem, 2003, 134: 321–326
[15] Akhmanova A, Hoogenraad C C. Microtubule plus-end-tracking proteins: mechanisms and functions. Curr Opin Cell Biol, 2005, 17: 47–54
[16] Inoue Y H, Avides M C, Shiraki M, Deak P, Yamaguchi M, Nishimoto Y, Matsukage A. Glover D M. Orbit, a novel microtubule-associated protein essential for mitosis in Drosophila melanogaster. J Cell Biol, 2000, 149: 153–166
[17] Lemos C L, Sampaio P, Maiato H, Costa M, Omelyanchuk L V, Liberal V, Sunkel C E. Mast, a conserved microtubule associated protein required for bipolar mitotic spindle organization. EMBO J, 2000, 19: 3668–3682
[18] Dhonukshe P, Weits D A, Cruz-Ramirez A, Deinum E E, Tindemans S H, Kakar K, Prasad K, Maehoenen A P, Ambrose C, Sasabe M, Wachsmann G, Luijten M, Bennett T, Machida Y, Heidstra R, Wasteneys G, Mulder B M, Scheres B. A PLETHOR-auxin transcription module controls cell division plane rotation through MAP65 and CLASP. Cell, 2012, 149: 383–396
[19] Ambrose J C, Wasteneys G O. CLASP modulates microtubule-cortex interaction during self-organization of acentrosomal microtubules. Mol Biol Cell, 2008, 19: 4730–4737
[20] Jawdat A B, Fred C. Regulation of microtubule dynamics by TOG-domain proteins XMAP215/Dis1 and CLASP. Trends Cell Biol, 2011, 21: 604–614
[21] Maiato H, Khodjakov A, Rieder C L. Drosophila CLASP is required for the incorporation of microtubule subunits into fluxing kinetochore fibres. Nat Cell Biol, 2004, 7: 42–47
[22] Wittman T, Waterman-storer C M. Spatial regulation of CLASPaffinity for microtubules by Rac1 and GSK3 beta in migrating epithelial cells. J Cell Biol, 2005, 169: 929–939
[23] 朱守鸿, 薛飞, 赵兰杰, 刘永昌, 李艳军, 熊显鹏, 孙杰. 棉花微管结合蛋白基因 GhCLASP1的克隆与表达分析. 西北植物学报, 2015, 35: 1941–1948 Zhu S H, Xue F, Zhao L J, Liu Y C, Li Y J, Xiong X P, Sun J. Cloning and expression analysis of GhCLASP1 gene in Gossypium hirsutum L. Acta Bot Boreali-Occident Sin, 2015, 35: 1941–1948 (in Chinese with English abstract)
[24] Li F, Fan G, Lu C, Xiao G, Zou C, Kohel R J, Ma Z, Shang H, Ma X, Wu J, Liang X, Huang G, Percy R G, Liu K, Yang W, Chen W, Du X, Shi C, Yuan Y, Ye W, Liu X, Zhang X, Liu W, Wei H, Wei S, Huang G , Zhang X, Zhu S, Zhang H, Sun F, Wang X, Liang J, Wang J, He Q, Huang L, Wang J, Cui J, Song G, Wang K, Xu X, Yu J Z, Zhu Y, Yu S. Genome sequence of cultivated upland cotton (Gossypium hirsutum TM-1) provides insights into genome evolution. Nat Biotechnol, 2015, 33: 524–530
[25] Zhang T, Hu Y, Jiang W, Fang L, Guan X, Chen J, Zhang J, Saski C A, Scheffler B E, Stelly D M, Hulse-Kemp A M, Wan Q, Liu B, Liu C, Wang S, Pan M, Wang Y, Wang D, Ye W, Chang L, Zhang W, Song Q, Kirkbride R C, Chen X, Dennis E, Llewellyn D J, Peterson D G, Thaxton P, Jones D C, Wang Q, Xu X, Zhang H, Wu H, Zhou L, Mei G, Chen S, Tian Y, Xiang D, Li X, Ding J, Zuo Q, Tao L, Liu Y, Li J, Lin Y, Hui Y, Cao Z, Cai C, Zhu X, Jiang Z, Zhou B, Guo W, Li R, Chen Z J. Sequencing of allotetraploid cotton (Gossypium hirsutum L. acc. TM-1) provides a resource for fiber improvement. Nat Biotechnol, 2015, 33: 531–537
[26] 蒋建雄, 张天真. 利用 CTAB/酸酚法提取棉花组织总 RNA.棉花学报, 2003, 15: 166–167 Jiang J X, Zhang T Z. Extraction of total RNA in cotton tissues with CTAB-acidic phenolic method. Cotton Sci, 2003, 15: 166–167 (in Chinese with English abstract)
[27] Andrade M A, Bork P. HEAT repeats in the Huntington’s disease protein. Nat Genet, 1995, 11: 115–116
[28] Groves M R, Hanlon N, Turowski P, Hemmings B A, Barford D. The structure of the protein phosphatase 2A PR65/A subunit reveals the conformation of its 15 tandemly repeated HEAT motifs. Can Metall Q, 1999, 96: 99–110
[29] Grallert A, Beuter C, Craven R A, Wilks D, Grallert A, Fleig U, Beuter C, Hagan L M. S. pombe CLASP needs dynein, not EB1 or CLIP170, to induce microtubule instability and slows polymerization rates at cell tips in a dynein-dependent manner. Genes Dev, 2006, 20: 2421–2436
[30] Pereira A L, Pereira A J, Maia A R, Drabek K, Sayas C L, Hergert P J, Faria M L, Matos I, Duque C, Stepanova T, Rieder C L, Earnshaw W C, Galjart N, Maiato H, Bloom K. Mammalian CLASP1 and CLASP2 cooperate to ensure mitotic fidelity by regulating spindle and kinetochore function. Mol Biol Cell, 2006, 17: 4526–4542
[31] Brandizzi F, Wasteneys G O. Cytoskeleton-dependent endomembrane organization in plant cells: an emerging role for microtubules. Plant J, 2013, 75: 339–349
[32] Kakar K, Zhang H T, Scheres B, Dhonukshe P. CLASP-mediated cortical microtubule organization guides PIN polarization axis. Nature, 2013, 495: 529–533
[33] Zhang C, Raikhel N V, Hicks G R. CLASPing microtubules and auxin transport. Dev Cell, 2013, 24: 569–571
[34] Basra A S, Malik C P. Development of the cotton fiber. Int Rev Cytol, 1984, 89: 65–113
[35] Kim H J, Triplett B A. Cotton fiber growth in planta and in vitro: models for plant cell elongation and cell wall biogenesis. Plant Physiol, 2001, 127: 1361–1366
[36] Yatsu L Y, Jacks T J. An ultrastructural study of the relationship between microtubules and microfibrils in cotton (Gossypium hirsutum L.) cell wall reversals. Am J Bot, 1981, 68: 771–777
[37] Dixit R, Cyr R. Encounters between dynamic cortical microtubules promoter ordering of the cortical array through angledependent modifications of microtubule behavior. Plant Cell, 2004, 16: 3274–3284
[38] Dixit R, Cyr R. The cortical microtubule array: from dynamics to organization. Plant Cell, 2004, 16: 2546–2552
[39] Nangaku M, Sato-Yoshitake R, Okada Y, Noda Y, Takemura R, Yamazaki H, Hirokawa N. KIF1B, novel microtubule plus end-directed monomeric motor protein for transport of mitochondria. Cell, 1994, 79: 1209–1220
[40] Yaffe M P, Stuurman N, Vale R D. Mitochondrial positioning in fission yeast is driven by association with dynamic microtubules and mitotic spindle poles. Proc Natl Acad Sci USA, 2003, 100: 11424–11428
Identification and Expression Analysis of Microtubule Binding Protein CLASP Family Genes in Gossypium hirsutum L.
ZHU Shou-Hong, ZHAO Lan-Jie, LIU Yong-Chang*, LI Yan-Jun, ZHANG Xin-Yu, and SUN Jie
College of Agronomy, Shihezi University / Key Laboratory of Oasis Eco-agriculture of Xinjiang Production and Construction Corps, Shihezi 832003, China
CLIP-Associated Proteins (CLASPs) are microtubule associated proteins (MAPS) which are regulators of microtubule structure and function, and play a key role in plant growth and morphogenesis. In this study, six CLASPs genes were preliminarily identified in Gossypium hirsutum L. by bioinformatics. Based on multiple sequence alignment and phylogenetic tree analysis, we divided the six CLASPs proteins into I and II subfamilies. The subfamily I has HEAT domain and CLASP-N structure; and the subfamily II only has CLASP-N structure. Real-time quantitative reverse transcription PCR (qRT-PCR) revealed that GhCLASPs genes expressed in root, stem, leaf, petal and fiber at different developmental periods, and there existed different expression patterns. CotAD_63740 (GenBank accession No. KX881965) and CotAD_04861 (GenBank accession No. KX881961) were preferentially expressed in fiber; CotAD_48232 (GenBank accession No. KX881962), CotAD_48665 (GenBank accession No. KX881963), CotAD_55570 (GenBank accession No. KX881964), and CotAD_68468 (GenBank accession No. KX881966) were preferentially expressed in stem. Different expression patterns suggest that they may play different roles during cotton development. The results will be helpful for the further analysis of CLASPs proteins in cotton.
Gossypium hirsutum L.; Microtubule binding protein; Microtubule; Expression analysis
10.3724/SP.J.1006.2017.00389
本研究由国家自然科学基金项目(U1128301), 新疆生产建设兵团博士资金专项(2013BB003)和石河子大学高层次人才启动项目(RCZX201218)资助。
This study was support by the National Natural Science Foundation of China (U1128301), the Special Research Funds for Doctor of Xinjiang Production and Constraction Crops (2013BB003), and the Startup Project for Outstanding Scholars of Shihezi University (RCZX201218).
*通讯作者(Corresponding author): 刘永昌, E-mail: liuyongchang2003@126.com, Tel: 18799292449
联系方式: E-mail: zhushouhong2014@126.com, Tel: 13565543505
稿日期): 2016-04-04; Accepted(接受日期): 2016-09-18; Published online(
日期): 2016-10-09.
URL: http://www.cnki.net/kcms/detail/11.1809.S.20161009.1404.004.html
