羊口疮病毒QD/2015株B2L基因克隆及生物信息学分析
2017-03-16刘春羽范俊豪王海峰牛云雷张七斤
李 智,仲 亮,张 静,刘春羽,范俊豪,王海峰,牛云雷,张七斤
(1. 内蒙古农业大学兽医学院,内蒙古呼和浩特 010018;2. 巴林左旗动物疫病预防控制中心,内蒙古赤峰 025450)
羊口疮病毒QD/2015株B2L基因克隆及生物信息学分析
李 智1,仲 亮1,张 静1,刘春羽1,范俊豪1,王海峰1,牛云雷2,张七斤1
(1. 内蒙古农业大学兽医学院,内蒙古呼和浩特 010018;2. 巴林左旗动物疫病预防控制中心,内蒙古赤峰 025450)
为分析羊口疮病毒(ORFV/QD/2015株)B2L基因的分子特征,预测其编码蛋白的生物学功能,对其B2L基因进行PCR扩增、克隆及序列测定,应用生物信息学相关软件及方法,对扩增所得的基因进行序列分析,并对其编码蛋白的二级结构、细胞抗原表位、三级结构、跨膜结构域和信号肽等进行预测和分析。结果显示:ORFV/QD/2015株B2L基因序列长1 137 bp,编码379个氨基酸;该毒株与其他12株羊口疮病毒参考株的B2L基因核苷酸序列同源性为96.8%~99.7%,氨基酸序列同源性为96.8%~99.2%。系统进化分析显示,ORFV/ QD/2015株与2015年分离到的ORFV/ShaanXi/2015/China株亲缘关系最近;B2L基因编码蛋白二级结构以α-螺旋区域和β-折叠区域所占比例较大,预测此蛋白可能存在7个细胞优势抗原表位,无跨膜区域,无信号肽区域;三级结构呈弯曲状螺旋结构。
羊口疮病毒;B2L基因;生物信息学分析;蛋白结构预测
羊口疮(ORF)是由羊口疮病毒(ORFV)引起的山羊和绵羊的一种急性、接触性传染病,人也可感染。本病以羊口唇等处的皮肤和粘膜形成红斑、丘疹、脓疮、溃疡和结痂等为主要特征[1]。ORFV全基因组大小为135~139 kb,是双链线性DNA病毒,基因组包含131个基因,其B2L基因位于基因组的左末端。ORFV在宿主细胞内增值时,其B2L基因在病毒DNA尚未复制时,便大量转录翻译为42 KDa大小的蛋白。该蛋白是ORFV外表囊膜的主要成分,可强烈刺激宿主免疫系统产生免疫应答[2]。目前国内关于ORFV B2L基因的生物信息学结构预测分析的报道较少。通过本试验可以了解ORFV/QD/2015流行株的生物信息学特征,充实ORFV分子流行病学资料,为下一步进行B2L基因的表达奠定基础。
1 材料与方法
1.1 病毒
羊口疮病毒株由涂明亮2015年从青岛市分离到,命名为ORFV/QD/2015。通过病毒滴度测定,其TCID50为10-5.5/0.1 mL。毒株由内蒙古农业大学传染病实验室保存。
1.2 细胞与菌株
DH5α感受态细胞购自天根生化科技(北京)有限公司。
1.3 主要试剂
2×Easy Taq SuperMix,购自北京全式金生物技术有限公司;氨苄青霉素(Ampicillin),购自AMERSCO公司;DL2000、PMDTM18-T Vector Cloning Kit,购自宝生物(大连)工程有限公司;病毒基因组DNA/RNA提取试剂盒、凝胶回收试剂盒、质粒小提试剂盒,购自天根生化科技(北京)有限公司。其他试剂均为标准化学分析纯试剂。
1.4 主要设备
22331型PCR仪, 购 自德国Eppendorf公司;水平电泳仪BG-Power600i,购自BAYGENE公司;Syngene G:BOX凝胶成像仪,购自英国SYNGENE公司;恒温水浴锅,购自北京市长风仪器仪表公司;5417R型低温高速离心机,购自德国Eppendorf公司;恒温振荡培养箱(HZC-250),购自培英仪器公司。
1.5 引物设计与合成
根据GenBank中的ORFV B2L基因序列,利用primer 5.0,在基因保守区设计特异性引物[3]。引物由生工生物工程(上海)有限公司合成(表1)。

表1 B2l基因引物序列及产物大小
1.6 PCR扩增
采用病毒基因组DNA提取试剂盒,提取ORFV/QD/2015株全基因组,以提取的ORFV基因组为模板,用生理盐水作为阴性对照,用设计的B2L特异性引物对其进行PCR扩增。PCR扩增反应体系(25 µL):2×Taq PCR Master Mix 12.5 µL,上游引物(10 µmol/L)1.0 µL,下游引物(10 µmol/L)1.0 µL, 模 板 DNA 1.0 µL,RNase-Free ddH2O 9.5 µL。PCR扩增反应参数:94 ℃预变性5 min,94 ℃变性30 s,53 ℃退火45 s,72 ℃延伸30 s,35个循环,72 ℃终延伸10 min。将扩增得到的产物用1%琼脂糖凝胶电泳检测。
1.7 基因克隆及序列分析
回收纯化的目的DNA片段,将其连接至PMD18-T载体上,然后转化到大肠杆菌DH5α感受态中;利用含氨苄青霉素抗性的LB平板,筛选阳性重组质粒;提取重组质粒,进行PCR和质粒双酶切鉴定;用1%琼脂糖凝胶电泳检测将鉴定为阳性的重组质粒命名为PMD18-T-B2L,送北京华大生物工程有限公司测序。参考GenBank上公布的ORFV B2L基因序列,利用DNAStar软件中的MegAlign程序,分析测序结果,构建进化树[4]。ORFV参考毒株信息见表2。
1.8 生物信息学分析
运用DNAStar软件中的Protean程序,使用Garnier-Robson、Chou-Fasman两种方法,分析预测ORFV/QD/2015株B2L基因编码蛋白的二级结构[5-6];运用DNAStar软件中的Protean程序,分析预测该蛋白细胞抗原表位参数;运用SWISSMODEL预测该蛋白的三级结构;采用SignalP 4.1预测该蛋白信号肽;运用TMHMM Server 2.0 预测该蛋白跨膜结构域[7]。
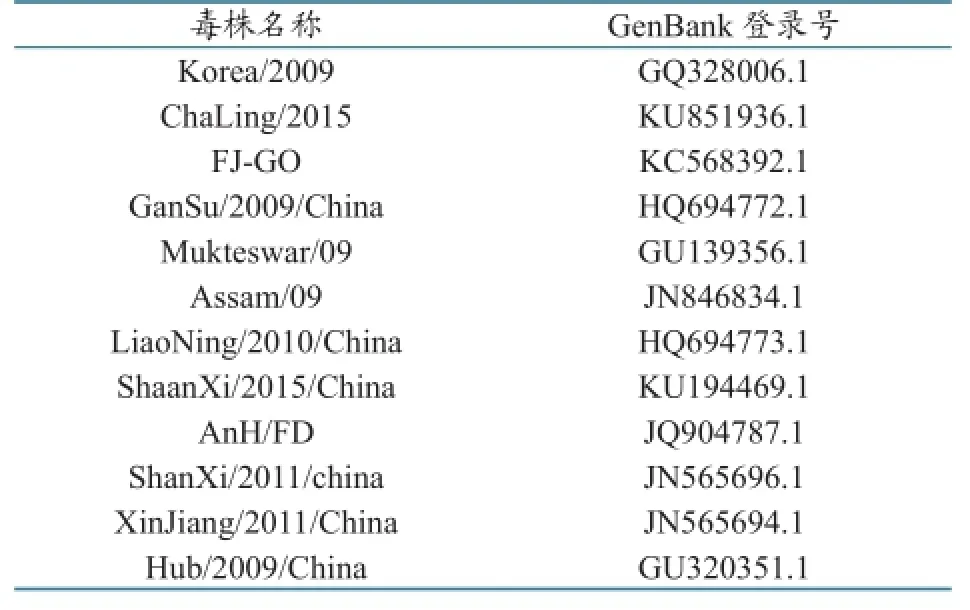
表2 GenBank中ORFV参考毒株信息
2 结果与分析
2.1 PCR扩增
以提取的ORFV/QD/2015株DNA为模板,进行B2L基因特异性PCR扩增,然后经1%琼脂糖凝胶电泳分析,得到与预期相符、大小为1 137 bp的条带(图1)。
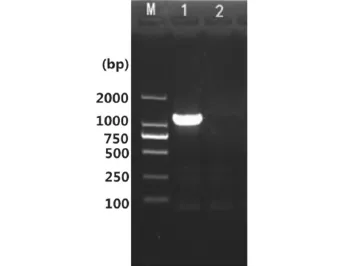
图1 ORFV/QD/2015株B2L基因PCR扩增产物电泳
2.2 测序结果
对质粒PCR和质粒双酶切鉴定为阳性的质粒进行测序,将测序结果进行拼接,其核苷酸序列见图2。由图2可知,B2L基因序列长为1 137 bp,可编码379个氨基酸。

图2 ORFV/QD/2015株B2L基因测序结果
2.3 ORFV/QD/2015株B2L基因核苷酸序列同源性分析
运用Clustal W法比对分析ORFV/QD/2015株B2L基因核苷酸序列与GenBank上公布的12株ORFV参考毒株B2L基因的核苷酸序列。结果表明,ORFV/QD/2015株B2L基因与其他12株ORFV参考株B2L基因的核苷酸序列同源性为96.8%~99.7%,其中与陕西省2015年分离到的毒株同源性高达99.7%(图3)。
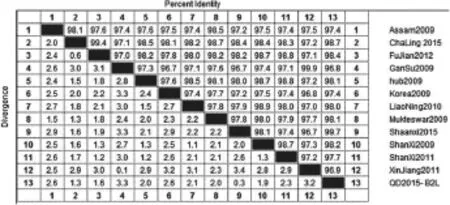
图3 ORFV/QD/2015株B2L基因的核苷酸同源性分析
2.4 ORFV/QD/2015株B2L基因系统进化树分析
将ORFV/QD/2015株B2L基因核苷酸序列与GenBank上公布的12株ORFV参考株B2L基因的核苷酸序列构建进化树。结果表明,ORFV/ QD/2015株与陕西省2015年分离到的病毒株处于同一遗传进化分支,亲缘性最近(图4)。
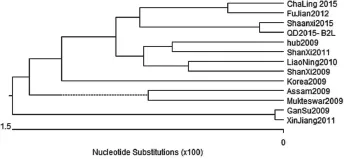
图4 ORFV/QD/2015株B2L基因核苷酸序列系统进化树
2.5 ORFV/QD/2015株B2L基因氨基酸序列同源性分析
对ORFV/QD/2015株B2L基因氨基酸序列与GenBank上公布的12株ORFV参考株B2L基因氨基酸序列进行比对分析。结果表明,ORFV/ QD/2015株B2L基因氨基酸序列与其他12株ORFV参考株的B2L基因氨基酸序列同源性为96.8%~99.2%,其中与陕西省2015年分离到的和福建省2012年分离到的毒株B2L基因氨基酸序列同源性高达99.2%(图5)。

图5 ORFV/QD/2015株B2L基因氨基酸同源性分析
2.6 ORFV/QD/2015株B2L基因编码蛋白的二级结构预测
运用DNAStar程序中的Protean软件,使用Garnier-Robson和Chou-Fasman两种方法,分析ORFV/QD/2015株B2L基因编码蛋白的二级结构,结果显示,不同预测方法对蛋白二级结构的分析结果不完全相同。β-折叠和β-转角等特定结构的数目和所处位置有较大差异,Garnier-Robson方法分析该蛋白β-转角较少,并且几乎没有无规则卷区。结合两种算法,该蛋白α-螺旋位于18~28、37~47、64~76、92~99、155~160、218~229、244~248、256~266、314~318、329~337和357~371等区间,β-折叠位于14~17、34~37、48~50、80~83、106~108、128~132、139~146、147~150、166~173、179~187、191~195、200~206、211~215、235~239、241~244、252~256、270~278、297~309和342~349等区间,β-转角位于4~6、9~11、29~31、124~128、136~138、161~165、196~200和338~340等区间(图6)。

图6 ORFV/QD/2015株B2L基因编码蛋白二级结构预测与分析
2.7 ORFV/QD/2015株B2L基因编码蛋白细胞抗原表位参数预测
运用DNAStar程序中的Protean软件,综合分析预测ORFV/QD/2015株B2L基因编码蛋白细胞抗原表位参数。采用Kyte-D00little法分析亲水性,Karplus-Schulz法分析柔韧性,Jameson-Wolf法分析抗原指数,Emini法分析氨基酸表面可及性,结果显示,31~32、58~67、71~72、87~96、98~99、101~109、111~113、128~129、149~167、190~198、206~210、212~213、216~219、222~233、279~284、310~311、314~319、337~345、352~353、355~358和366~376位 氨 基酸亲水性较高;28~32、43~46、58~64、66~69、75~78、86~95、106~116、134~146、151~154、174~176、187~191、197~201、201~210、217~221、228~234、279~284、311~321、352~360和367~373位氨基酸柔韧性较高;9~15、30~32、42~45、57~79、86~98、107~116、150~154、160~165、172~178、206~210、217~223、226~233、265~271、278~285、291~297、309~321、323~329、338~342和 351~378位 氨基酸抗原指数较高;43~44、58~60、87~94、151~153、161~166、206~208、217~218、279~282、315~318、340~342和367~369位氨基酸表面可及性较高(图7)。

图7 ORFV/QD/2015株B2L基因编码蛋白细胞表位参数预测
运用DNAStar程序中的Protean软件,分析比较不同参考株之间的B2L基因编码蛋白的抗原指数。结果显示,ORFV/QD/2015株与5株参考毒株的B2L基因编码蛋白抗原指数较高的氨基酸位点基本一致(图8)。
2.8 ORFV/QD/2015株B2L基因编码蛋白的三级结构预测
利用SWISS-MODEL(http://swissmodel.expasy.org/interactive)预测ORFV/QD/2015株B2L基因编码蛋白的三级结构,结果显示该蛋白可能呈弯曲状螺旋结构(图9)。
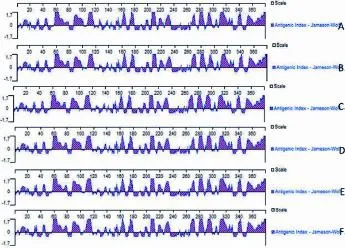
图8 ORFV不同参考株间抗原指数比较
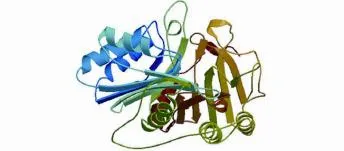
图9 ORFV/QD/2015 株B2L基因编码蛋白三级结构预测
2.9 ORFV/QD/2015株B2L基因编码蛋白的信号肽预测
采用SignalP 4.1(http://www.chs.dtu.dk/services/ SignalP/)预测ORFV/QD/2015株B2L基因编码蛋白信号肽,结果显示该蛋白无信号肽(图10)。
2.10 ORFV/QD/2015株B2L基因编码的蛋白的跨膜结构域预测
运 用 TMHMM Server V2.0(http://www.cbs. dtu.dk/services/TMHMM/)预测ORFV/QD/2015株B2L基因编码蛋白跨膜结构域,结果显示该蛋白并无跨膜结构域(图11)。
3 讨论
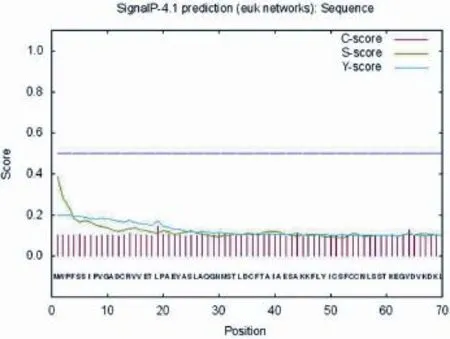
图10 ORFV/QD/2015株B2L基因编码蛋白信号肽预测
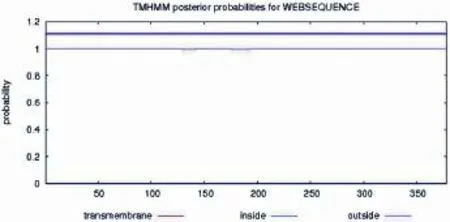
图11 ORFV/QD/2015株B2L基因编码蛋白跨膜结构域预测
目前,ORF广泛流行于全球各羊养殖国家或地区,且发生频率和流行范围呈逐年上升趋势。我国虽是农牧业大国,但由于对ORF缺乏关注,很多科研机构没有对该病开展理论研究,市场需求也较小,因此缺少相关ORF检测试剂盒。本研究通过对涂明亮分离到的ORFV/QD/2015株的B2L基因进行生物信息学研究,为进一步对该基因的表达和诊断试剂盒制备奠定了基础,同时也了解了当前ORFV/QD/2015株的生物信息学特征。
本研究将ORFV/QD/2015株B2L基因核苷酸序列和氨基酸序列与GenBank上公布的12株ORFV参考株B2L基因核苷酸序列和氨基酸序列分别进行比对分析发现:ORFV/QD/2015株B2L基因与其他12株参考株的B2L基因核苷酸序列同源性为96.8%~99.7%。其中,与2015年陕西省分离到的羊口疮病毒B2L基因核苷酸序列同源性高达99.7%;与其他12株参考株B2L基因的氨基酸序列同源性为96.8%~99.2%,其中与2015年陕西省和2012年福建省分离到的羊口疮病毒B2L基因氨基酸序列同源性高达99.2%。从进化树上可以看出,ORFV/QD/2015株与2015年陕西省分离到的ORFV处于同一遗传进化分支,因此认为涂明亮分离到的ORFV/QD/2015株与陕西株的亲缘性最近。
蛋白的二级结构中的α-螺旋和β-折叠的化学键能较高,能牢固地维持蛋白的高级结构,通常处于内部且不易变形,不利于抗原抗体的结合,而转角和无规则卷曲的区域结构比较松散,稳定性差,易发生扭曲变构,多处于蛋白质分子表面,容易与抗体分子接近及结合[7-8]。该蛋白α-螺旋和β-折叠所占比例较大,几乎没有无规则卷曲,所以该蛋白是否有良好的反应原性有待验证。该蛋白无跨膜结构域和信号肽是非分泌性蛋白,因此预测蛋白的生物学特征时,通过单一参数的确定是不准确的,还需要同时考虑亲水性、表面可及性、抗原指数和柔韧性等参数[9-10]。本研究经综合考虑,选取亲水性高、柔韧性好、表面可及性大、抗原指数高的区域作为候选抗原表位,并兼顾二级结构各参数,比较了不同参考株B2L基因编码蛋白的抗原指数,最终推测出ORFV/QD/2015株B2L基因编码蛋白的细胞优势抗原表位区域为58~69、86~95、205~211、216~219、279~284、311~321和352~378位氨基酸[11-12],认为该蛋白有一定的免疫原性。这些抗原优势表位区域的确定,为以后表达该蛋白提供了参考,但其蛋白免疫原性和反应原性如何,还需要通过实验进一步验证[13]。
[1] 李旭东. 羊传染性脓疱实时荧光定量PCR方法的建立[D]. 呼和浩特:内蒙古农业大学,2015.
[2] 涂明亮. 羊口疮病毒生物学特性研究[D]. 呼和浩特:内蒙古农业大学,2016.
[3[ 李金明. 实时荧光PCR技术[M]. 北京:人民军医出版社,2011:4-5.
[4] 安维雪. 羊痘病毒内蒙古分离株生物学特性的研究[D].呼和浩特:内蒙古农业大学,2016.
[5] 吴健敏,吕茂民,阳玉彪,等. 猪内源性反转录病毒囊膜蛋白基因的克隆及其蛋白二级结构和B细胞表位预测[J].畜牧兽医学报,2007,38(4):412-416.
[6] KARPLUS P A,SCHULTZ G E. Prediction of chain fl exibility in proteins [J]. Immunology,1985,72(2):212.
[7] 张海,王开功,文明,等.牛乳头状瘤病毒贵州株L1基因克隆及生物信息学分析[J]. 中国畜牧兽医,2016,43(11):2834-2843.
[8] 黄莉,谢丽基,邓显文,等. 禽呼肠孤病毒σB和σC蛋白二级结构及其细胞抗原表位预测[J]. 中国畜牧兽医,2016,43(11):2880-2885.
[9] 马凡舒,张蕾,王洋. B细胞抗原表位预测方法的研究进展[J]. 中国兽医杂志,2016,43(1):63-67.
[10] 温立斌,何孔旺,杨汉春,等. 类猪圆环病毒因子P1 VP2蛋白二级结构与B细胞表位预测[J]. 华北农学报,2009,24(5):45-49.
[11] 王光祥,尚佑军,吕占禄,等. 羊口疮病毒B2L蛋白二级结构及其细胞表位的预测[J].中国兽医科学,2014,42(11):1133-1138.
[12] POLANCO C,BUHSE T,UVERSKY V N. Structure and function relationships of proteins based on polar pro fi le:A review[J]. Acta Biochim Pol,2016,63(2):229-233.
[13] 杜杰,尹惠琼,韦平,等. 蓝舌病病毒VP7蛋白的B细胞表位预测[J]. 中国动物传染病学报,2013,21(1):29-36.
(责任编辑:朱迪国)
Cloning and Bioinformatic Analysis of B2L Gene of Orf Virus QD/2015 Strain
Li Zhi1,Zhong Liang1,Zhang Jing1,Liu Chunyu1,Fan Junhao1,Wang Haifeng1,Niu Yunlei2,Zhang Qijin1
(1. College of Veterinary Medicine,Inner Mongolia Agricultural University,Hohhot,Inner Mongolia 010018;2. Baarin Left Banner Animal Disease Prevention and Control Center,Chifeng,Inner Mongolia 025450)
In order to analyze molecular characteristics of B2L gene of Orf virus(ORFV/QD/2015) strain and to predict biological function of B2L protein. In this study,the B2L gene was ampli fi ed,cloned and sequenced. Using related bio-informatics softwares and methods,the amplified sequence was analyzed and B2L protein's secondary structure,tertiary structure,B-cell preponderant epitope,conserved domain,transmembrane domain and signal peptide were also predicted and analyzed. Results showed that the length of B2L gene was 1 137 bp,encoding 379 amino acids. The B2L gene of ORFV/QD/2015 strain shared amino acid identities of 96.8%~99.2% and nucleotide identities of 96.8%~99.7%,respectively,compared with other 12 ORFV reference strains. Phylogenetic analysis indicated a closest relationship between ORFV/QD/2015 strain and ORFV/ShaanXi/2015/China strain,the latter was isolated in 2015. Prediction of secondary structure of B2L protein indicated α-helix and β-sheet took up a large proportion.,and B2L protein possibly contained 7 potential antigen epitopes. However,no transmembrane domain or signal peptide was identi fi ed. The tertiary structure of B2L protein was curved spiral structure.
ORFV;B2L gene;bioinformatics analysis;prediction of protein structure
S852.65
B
1005-944X(2017)03-0091-06
10.3969/j.issn.1005-944X.2017.03.024
