广西猕猴中发现GⅡ.17型诺如病毒及其全基因组序列分析
2017-03-10信云云敖元云李利利虞结梅李金松林琳张兵
信云云 敖元云 李利利 虞结梅 李金松 林琳 张兵
410003 长沙,湖南师范大学附属第一医院儿童医学中心(信云云、张兵);100052 北京,中国疾病预防控制中心病毒病预防控制所腹泻室(敖元云、李利利、虞结梅、李金松);250014 济南,山东省疾病预防控制中心艾滋病防治所(林琳)信云云、敖元云对本文同等贡献
诺如病毒(Norovirus)是一种无包膜、单股正链RNA病毒,属于杯状病毒科诺如病毒属,在各个年龄段的人群中均可引起散发或暴发的非细菌性胃肠炎。该病毒的传播途径较广,主要包括粪—口途径,也包括水源、食源及呕吐物的气溶胶途径。大多数与诺如病毒有关的胃肠炎均发生在一些相对封闭的环境中,例如学校、幼儿园、餐厅、疗养院以及医院等[1-2]。该病毒的基因组是由三个开放阅读框(Open reading frames, ORFs)以及两端的5′/3′非编码区(UTR)组成的,ORF1和ORF3分别编码非结构蛋白(Non-structural protein)和一些次要结构蛋白(Viral protein 2, VP2),ORF2主要编码结构蛋白(Viral protein 1, VP1),即衣壳蛋白[3-5]。根据诺如病毒衣壳区核苷酸序列的不同,诺如病毒可分为7个基因组(Genogroups, G),GⅠ~GⅦ,其中基因组GⅠ包含9种基因型(Genotype),GⅡ包含22种基因型[6]。在这些基因组中,GⅠ、GⅡ和GⅣ以感染人类为主。自2002年以来,GⅡ.4诺如病毒一直在全球的暴发流行中占优势地位[7],但从2014年底至2015年初,GⅡ.17型诺如病毒变异株明显增加,流行范围涉及中国多个省市及一些东南亚国家,并取代GⅡ.4型的位置而成为了这些国家和地区导致人类非细菌性胃肠炎暴发流行的优势病毒株[8-15]。有研究显示在不同种类的动物体内,例如牛、猪、狗、鼠、狮子有自然感染的诺如病毒变异株或者重组株,表明诺如病毒可能有一条向人类传播的动物源性传播途径,并由此推断被病毒感染的人类也可将其传染给家畜,并可能引发动物胃肠炎的暴发[16-21]。2016年Farkas等首次报道了猕猴体内存在自然感染的GⅠ、GⅡ及GⅣ型诺如病毒[22]。随后谭明等在猕猴粪便中发现自然感染的GⅡ.17型诺如病毒[23],并认为猕猴可能是诺如病毒的自然储存宿主,人诺如病毒感染猕猴后因基因重组或突变可能出现新的病毒变异株。但关于人猕猴间诺如病毒感染的机制、特征及传播等特点的研究还非常缺乏。本研究采用建立好的HISEQ高通量测序技术,在广西龙虎山的猕猴粪便中发现了GⅡ.17型诺如病毒,命名为GX213,测定和分析了该型诺如病毒的全基因组序列,对其在猕猴群中的检出状况进行了调查研究。
1 材料与方法
1.1标本来源和处理本研究的标本来源于2015年3~8月收集的我国广西省龙虎山自然保护区野生猕猴的粪便标本。对于龙虎山四个猕猴群,每个猕猴群每次采集5份粪便标本,采集20次,每个猴群100份,共采集400份标本。收集的全部标本需低温送至中国疾病预防控制中心病毒病预防控制所,立即保存于﹣70 ℃冰箱待用。阳性标本是利用建立好的HISEQ高通量测序技术发现的。在二级生物安全柜中取黄豆大小(约1 g)的粪便至1.5 ml EP管中,加入1 ml PBS处理液,制成10%的粪便悬液,6 868×g,4 ℃离心5 min,取140 μl上清液,存于-20 ℃冰箱待用。
1.2所用试剂核酸提取试剂盒(QIAamp Viral RNA Mini Kit)和一步法RT-PCR试剂盒(Qiagen OneStep RT-PCR Kit)购于德国Qiagen公司;SuperscriptⅢ反转录酶,RNA酶抑制剂,dNTP购自于美国Invitrogen公司;Ex-Taq DNA聚合酶购自日本TaKaRa公司;1-5TM 2×High-Fidelity Master Mix购自于北京擎科新业生物技术有限公司,引物合成及DNA序列测定由北京擎科新业生物技术有限公司完成;3′-5′ exo-Klenow试剂盒购于美国New England Biolabs公司。
1.3高通量测序方法首先对上述粪便标本进行过滤和核酸酶消化处理,并使用QIAamp Viral RNA Mini Kit提取病毒总核酸;其次使用随机引物将RNA反转录成cDNA,再用exo-Klenow DNA聚合酶合成双链DNA,经琼脂糖凝胶电泳鉴定PCR产物,对300~500和500~800 bp区间的弥散带进行纯化回收;然后将回收的DNA片段用超声波打断,分别使用Klenow酶和dATP将片段两端修复,并在末端加“A”后,将其与接头Solexa连接,根据接头上的引物进行PCR扩增进而构建DNA文库。最后将DNA文库与病毒寡核苷酸探针库结合,进入Illumina测序。测序获得的读长(Reads)经BOWTIE 2和PRINSEQ等软件除去低质量和重复的序列,使用SOAPdenovo2软件将序列拼接成重叠序列(Contigs)。进一步通过Blastn和Blastx在GenBank中进行核苷酸序列比对,同时将E值≤10e-5的序列定为有意义的序列,之后去掉人、细菌、真菌等相关序列,利用MEGAN软件对病毒序列进行属种分类。
1.4所用软件利用Primer Premier 5.0设计引物,利用Clustalx进行多序列比对,利用DNAStra中的SeqMan软件进行序列拼接,利用MEGA 6.0软件中Clustal W软件进行序列的多重比对分析,采用邻接法(Neighbor-Joining method)构建系统进化树,并进行Bootstrap 500检验,以及利用GeneDoc比对分析VP1区氨基酸序列的变异情况。用于序列进化分析的参考株来自于GenBank中不同年代、不同国家和地区的诺如病毒株,其中包括14株人GⅡ.17型诺如病毒株和1株猴GⅡ.17型诺如病毒株,外群为GI.1型诺如病毒。
1.5特定病毒核酸提取及cDNA的合成使用QIAamp Viral RNA Mini Kit提取上述粪便悬液上清中的病毒RNA,以RNA为模板,用特异性引物VN3T20进行反转录,获得cDNA。反应体系及条件如下:RNA 9.5 μl,dNTP(10 mmol/L) 1 μl,VN3T20(20 μmol/L)2.5 μl,条件:65 ℃ 5 min, 冰浴5 min;然后再加以下体系:5 × First Stand Buffer 4 μl,DTT 1 μl,SuperscriptⅢ反转录酶2 μl,RNase Inhibitor 1 μl。反应条件: 42 ℃ 90 min延伸,99 ℃ 5 min灭活。
1.6阳性标本的验证前期处理的粪便标本,经HISEQ高通量测序平台测序,产生的10条序列被注释到人GⅡ.17型诺如病毒。本研究利用文献中提及的筛查GⅡ型诺如病毒的特异性引物G2SKR和MON431[24],用一步法反转录聚合酶链反应(OneStep RT-PCR)进行阳性标本的确认及筛查,共检测出2份阳性标本。反应体系:5 × Qiagen RT-PCR buffer 5 μl,dNTP(10 mmol/L) 1 μl,Enzyme Mix (RT and Taq) 1 μl,MON431(50 μmol/L) 0.5 μl,G2SKR(50 μmol/L) 0.5 μl,RNase Inhibitor 1 μl,Rnase free water 11 μl,RNA 5 μl;反应条件:42 ℃ 30 min,95 ℃ 15 min,(95 ℃ 1 min, 50 ℃ 1 min, 72 ℃ 1 min)×40,72 ℃ 10 min,4 ℃保存。
1.7病毒基因组的扩增利用文献[25,26]中已知的引物Ⅱ.17-2F、Ⅱ.17-2R、Ⅱ.17-4F、Ⅱ.17-4R,VN3T20、COG2F、G2SKF及根据诺如病毒参考株(GenBank ID:KU757050)设计的特异性引物扩增病毒的3个ORFs和2个末端区。扩增出的PCR产物经1.5%琼脂糖凝胶电泳鉴定,将检测到的与预期片段大小相符的PCR产物送北京擎科新业生物技术有限公司测序。实验中所用的引物序列见表1。
2 结果
2.1GX213的发现及检测本研究通过HISEQ高通量测序技术发现广西猕猴粪便中存在有GⅡ.17型诺如病毒基因序列。利用特异性引物G2SKR和MON431对阳性标本进行确认,同时对其进行流行病学调查。PCR扩增出约570 bp左右的ORF1和ORF2重叠区,即RdRp区与VP1区的交接区。扩增出的PCR片段经Blastn比对分析发现病毒株属于GⅡ.17型诺如病毒。本研究共400份粪便标本,经筛查后发现2份阳性标本,阳性率为0.5%。
2.2GX213的全基因序列扩增和拼接本研究选择其中1份阳性粪便标本进行该病毒全基因序列的扩增,命名为GX213。经相应的特异性引物扩增后分别获得了5个特异性PCR片段,长度分别为1 684 nt、1 511 nt、1 323 nt、1 483 nt和2 500 nt,利用DNAStar中的SeqMan软件将分段获得的基因片段进行序列拼接后得到病毒基因组的全长序列(7 565 nt)。经和其他GⅡ.17型诺如病毒序列比对发现,GX213可分为3个ORFs、5′和3′非编码区(UTR)和PloyA尾:ORF1(10~5 112 nt)、ORF2(5 093~6 715 nt)和ORF3(6 715~7 494 nt),其中ORF1与ORF2之间重叠20 bp,ORF2和ORF3之间重叠1 bp。此序列已被NCBI的GenBank数据库所收录(GenBank ID:MF918359)。

表1 GⅡ.17诺如病毒基因序列验证和全基因组扩增所用的引物
注:A,扩增GⅡ.17型诺如病毒PCR片段所用引物;B,验证阳性标本所用引物
Note: A, The primers used for amplification of sequence of GⅡ.17 norovirus gene; B, The primers used for confirmation the positive samples for GⅡ.17 norovirus
2.3GX213的序列分析将获得的GX213病毒株的全基因组序列进行核苷酸序列的Blast比对分析,结果显示,其与引起2014年至2015年亚洲部分地区暴发的新型人GⅡ.17型诺如病毒变异株CUHK-NS-613病毒株(GenBank ID:KU561248)同源性最高,为99.5%;分别将ORF1、ORF2、ORF3区核苷酸和氨基酸序列与GenBank中其他诺如病毒参考株进行多比对分析,发现ORF1和ORF3都与CUHK-NS-613同源性最高(分别为99.5%,99.4%;99.5%,99.2%),而在ORF2区,其与人GⅡ.17型诺如病毒变异株CUHK-NS-491毒株(GenBank ID:KP698928)具有最高的核苷酸同源性(99.4%),氨基酸序列同源性高达99.8%;其中,完整的P区与CUHK-NS-491毒株同样具有99.8%的核苷酸与氨基酸同源性,而对P2亚区的氨基酸序列比对发现,其与CUHK-NS-491株同源性为100%。
2.4GX213的遗传进化树分析使用MEGA 6.0软件中邻接(Neibor-joining)法将GX213株与已报道的其他人GⅡ.17型诺如病毒和猴GⅡ.17型诺如病毒参考株在RdRp区核苷酸序列和完整的VP1区氨基酸序列区绘制系统进化树,如图1所示。从RdRp进化树中可以看出,GX213株和CUHK-NS-613病毒株在同一个亚枝,亲缘关系最近;在VP1区进化树中,GX213株与CUHK-NS-491株的亲缘关系最近,与首次报道的猕猴GⅡ.17型诺如病毒KM1509毒株(GenBank ID:KX356908)次之。从两个进化树均可以看出,GⅡ.17诺如病毒进一步被分为3个亚群(Ⅰ~ Ⅲ),而根据VP1区序列的不同,Ⅲ亚群又分为Earlier GⅡ.17 2013-2014和Novel GⅡ.17 Kawasaki 2014群。GX213与之前报道的猴GⅡ.17型诺如病毒均属于Ⅲ亚群中Novel GⅡ.17 Kawasaki 2014群,与2014年至2015年暴发的人GⅡ.17型诺如病毒变异株进化关系最相近。
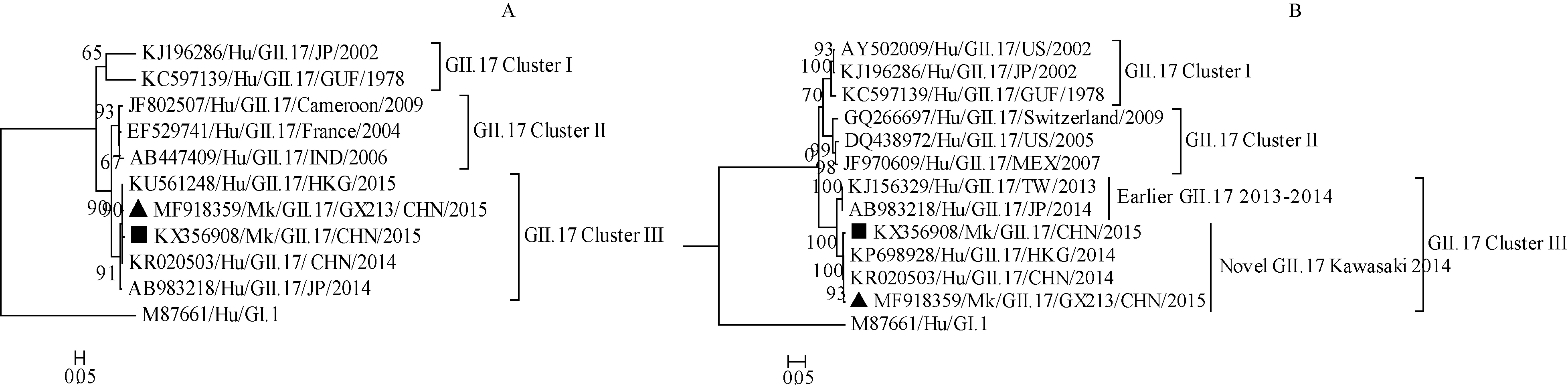
A GX213 RdRp区核苷酸进化树;B GX213 VP1区氨基酸进化树注:图1中,■标注的是已报道的猕猴GⅡ.17型诺如病毒[23];▲标注的是本研究中的诺如病毒GX213图1 诺如病毒GX213系统进化树A, Phylogenetic tree for RdRp nt of GX213;B, Phylogenetic tree for VP1 aa of GX213Note: In figure 1, black square indicates the monkey GⅡ.17 NoV reported[23]; black triangle indicates norovirus GX213 from this studyFig.1 Phylogenetic tree of norovirus GX213
2.5GX213的VP1区氨基酸序列分析将GX213株与2014年至2015年在亚洲地区引起暴发的人GⅡ.17型诺如病毒和首次在猕猴体内发现的猴GⅡ.17型诺如病毒参考株衣壳蛋白区氨基酸序列进行比对分析,结果显示,与之前预测的人GⅡ.17型诺如病毒变异株的抗原位点(Epitope A、D和E)和人组织血型抗原(HBGAs)结合位点(Site I、Ⅱ和Ⅲ)相比[15],GX213病毒株在相应位点没有氨基酸的突变,仅在245的位置(P1.1区)由脯氨酸(P)变成了丝氨酸(S);与KM1509病毒株的几个突变位点相比,GX213也没有氨基酸变化。同时发现,与GX213进化关系亲近的Novel GⅡ.17 Kawasaki 2014群病毒株比Ⅲ亚群中Earlier GⅡ.17 2013-2014群病毒株VP1区多2个氨基酸,比Ⅱ亚群病毒少1个氨基酸,而与Ⅰ亚群病毒VP1区氨基酸长度相同,这与之前文献中的结果相符合[27]。
3 讨论
1972年,美国科学家Kapikian等首次利用免疫电子显微镜(Immunoelectron Microscope,IEM)技术在一位暴发性急性胃肠炎患者的粪便标本中发现了一种小型病毒,将其命名为诺瓦克病毒[28]。随着分子诊断技术水平的发展和应用,发现诺如病毒目前已成为引起各个年龄段人群非细菌性胃肠炎暴发的主要病原体。诺如病毒自然宿主包括人及牛、猪、狗、鼠等动物。2014年至2015年期间,GⅡ.17型诺如病毒取代GⅡ.4型诺如病毒成为亚洲部分地区诺如病毒暴发的主要流行病毒株[8-14],故有必要对该型诺如病毒进行监测和溯源。谭明等在猕猴体内首次发现有GⅡ.17型诺如病毒的存在,并将其感染实验猴,证实了自然条件下猕猴可感染诺如病毒,并推断猕猴可能作为诺如病毒的储存宿主[23],但其致病机制、宿主特异性及跨物种传播等特点仍并不清楚。因此,不同种群猕猴体内的GⅡ.17型诺如病毒的发现具有重要的意义。本研究利用HISEQ高通测序技术在广西龙虎山猕猴粪便标本中发现了GⅡ.17型诺如病毒基因序列,对其中一份阳性标本中该型病毒的全基因组序列进行扩增测序和遗传进化分析,其结果丰富了野生动物中诺如病毒基因库。
人诺如病毒VP1区的P2亚区为高突变区,其可以与人组织血型抗原(HBGAs)结合,这种相互结合可能增加宿主对诺如病毒的易感性。研究表明,由于GⅡ.17型诺如病毒变异株的出现,人群可能对其变得更加易感,进而导致了该型变异株的暴发流行,其原因可能是该型病毒抗原位点、HBGAs结合位点及其附近氨基酸突变所致。由此可见,VP1区的氨基酸序列分析对发现新型变异株极其重要[15, 27, 29-30]。谭明等对发现的猴GⅡ.17型诺如病毒进行序列分析,发现P2高突变区有3个氨基酸突变位点,同时功能性试验显示该型诺如病毒与HBGAs弱结合,推测猴GⅡ.17型诺如病毒可能来源于人GⅡ.17型诺如病毒,而此突变可能是因为猴GⅡ.17型诺如病毒对新宿主(猕猴)的适应而导致的[23]。本研究中诺如病毒GX213与2014年至2015年引起亚洲部分地区暴发的人GⅡ.17型诺如病毒的同源性最高,而且GX213毒株的VP1区与其具有99.8%的氨基酸序列同源性,在预测的抗原位点和HBGAs结合位点及其附近均没有氨基酸突变,仅P1.1区存在一个氨基酸位点的突变,推测本研究中猴GⅡ.17型诺如病毒来源于2014年至2015年引起人群暴发的人GⅡ.17型诺如病毒变异株,提示GⅡ.17型诺如病毒为人猕猴共患病毒。
本研究的GX213在猕猴中阳性检出率为0.5%(2/400),显示出较低的阳性率,可能与低病毒载量或者标本代表性不足有关,需要继续扩大标本量或者选择对不同地方以及不同猴类标本进行筛查。但考虑到GX213病毒株VP1区抗原位点和HBGAs结合位点没有突变,推测阳性率低的一个重要原因可能是人GⅡ.17型诺如病毒在猕猴体内尚不能很好适应,亦或仅为一过性感染。
本研究结果支持Farkas及谭明等[22-23]的研究结果,猕猴可能为诺如病毒的自然储存宿主,但有待于更多的研究积累;本研究结果也提示若对猕猴群乃至整个猴群诺如病毒的流行变异情况进行监测,有可能发现引起暴发和流行的新型诺如病毒变异株。
[1] Robilotti E, Deresinski S,Pinsky BA. Norovirus[J]. Clin Microbiol Rev, 2015. 28(1):134-164.doi:10.1128/CMR00075-14.
[2] Zheng DP, Ando T, Fankhauser RL, et al. Norovirus classification and proposed strain nomenclature[J]. Virology, 2006. 346(2):312-323.doi:10.1016/j.virol.2005.11.015.
[3] Clarke IN,PR L. Organization and Expression of Calicivirus Genes[J]. J Infect Dis, 2000. 181:S309-316.doi:10.1086/315575.
[4] Jiang X, Wang M, Graham DY, et al. Expression, self-assembly, and antigenicity of the Norwalk virus capsid protein. [J]. J Virol, 1992. 66:6527-6532.
[5] Glass PJ, White LJ, Ball JM, et al. Norwalk virus open reading frame 3 encodes a minor structural protein.[J]. J Virol, 2000. 74:6581-6591.
[6] Vinje J. Advances in laboratory methods for detection and typing of norovirus[J]. J Clin Microbiol, 2015, 53(2):373-381.doi:10.1128/JCM.01535-14.
[7] Bok K, Abente EJ, Realpe-Quintero M, et al. Evolutionary dynamics of GⅡ.4 noroviruses over a 34-year period[J]. J Virol, 2009. 83(22):11890-11901.doi:10.1128/JUI.11864-09.
[8] Gao Z, Liu B, Huo D, et al. Increased norovirus activity was associated with a novel norovirus GⅡ.17 variant in Beijing, China during winter 2014-2015[J]. BMC Infect Dis, 2015,15:574.doi:10.1186/s12879-015-1315-z.
[9] de Graaf M, van Beek J, Vennema H, et al. Emergence of a novel GⅡ.17 norovirus - End of the GⅡ.4 era?[J]. Euro Surveill, 2015,20(26):21178.
[10] Fu J, Ai J, Jin M, et al. Emergence of a new GⅡ.17 norovirus variant in patients with acute gastroenteritis in Jiangsu, China, September 2014 to March 2015[J]. Eur Surveill, 2015,20(24):21157.
[11] Parra GI,Green KY. Genome of Emerging Norovirus GⅡ.17, United States, 2014[J]. Emerg Infect Dis, 2015,21(8):1477-1479.doi:10.3201/edi2108.150652.
[12] Matsushima Y, Ishikawa M, Shimizu T, et al. Genetic analyses of GⅡ.17 norovirus strains in diarrheal disease outbreaks from December 2014 to March 2015 in Japan reveal a novel polymerase sequence and amino acid substitutions in the capsid region[J]. Eur Surveill, 2015,20(26):21173.
[13] Lu J, Sun L, Fang L, et al. Gastroenteritis Outbreaks Caused by Norovirus GⅡ.17, Guangdong Province, China, 2014-2015[J]. Emerg Infect Dis, 2015,21(7):1240-1242.doi:10.3201/eid2017.150226.
[14] Lee C C, Feng Y, Chen S Y, et al. Emerging norovirus GⅡ.17 in Taiwan[J]. Clin Infect Dis, 2015,61(11):1762-1764.doi:10.1093/cid/civ674.
[15] Jin M, Zhou YK, Xie HP, et al. Characterization of the new GⅡ.17 norovirus variant that emerged recently as the predominant strain in China[J]. J Gen Virol, 2016,97(10):2620-2632.doi:10.1099/jgv.0.000582.
[16] Mattison K, Shukla A, Cook A, et al. Human noroviruses in swine and cattle[J]. Emerg Infect Dis, 2007,13(8):1184-1188.doi:10.3201/eid1308.070005.
[17] Martella V, Lorusso E, Decaro N, et al. Detection and molecular characterization of a canine norovirus[J]. Emerg Infect Dis, 2008,14(8):1306-1308.doi:10.3201/eid1408.080062.
[18] Martella V, Campolo M, Lorusso E, et al. Norovirus in captive lion cub (Panthera leo)[J]. Emerg Infect Dis, 2007,13(7):1071-1073.doi:10.3201/eid1307.070268.
[19] Mesquita JR, Barclay L, Nascimento MS, et al. Novel norovirus in dogs with diarrhea[J]. Emerg Infect Dis, 2010,16(6):980-982.doi:10.3201/eid1606.091861.
[20] Silva PF, Alfieri AF, Barry AF, et al. High frequency of porcine norovirus infection in finisher units of Brazilian pig-production systems[J]. Trop Anim Health Prod, 2015,47(1):237-241.doi:10.1007/s11250-014-0685-3.
[21] Karst SM, Wobus CE, Lay M, et al. STAT1-dependent innate immunity to a Norwalk-like virus[J]. Science, 2003,299(5612):1575-1578.doi:10.1126/science.1077905.
[22] Farkas T. Natural Norovirus Infections in Rhesus Macaques[J]. Emerg Infect Dis, 2016,22(7):1272-1274.doi:10.3201/eid2207.151740.
[23] He Z, Liu B, Tao Y, et al. Norovirus GⅡ.17 Natural Infections in Rhesus Monkeys, China[J]. Emerg Infect Dis, 2017,23(2):316-319.doi:10.3201/eid2302.161077.
[24] Vicentini F, Denadai W, Gomes Y M, et al. Molecular characterization of noroviruses and HBGA from infected Quilombola children in Espirito Santo State, Brazil[J]. PLoS One, 2013,8(7):e69348.doi:10.1371/journal.pone.0069348.
[25] Xue L, Cai W, Wu Q, et al. Direct sequencing and analysis of the genomes of newly emerging GⅡ.17 norovirus strains in South China[J]. J Appl Microbiol, 2016,120(4):1130-1135.doi:10.1111/jam.13052.
[26] Katayama K, Shirato-Horikoshi H, Kojima S, et al. Phylogenetic Analysis of the Complete Genome of 18 Norwalk-like Viruses[J]. Virology, 2002,299(2):225-239.doi:10.1006/viro.2002.1568.
[27] Singh BK, Koromyslova A, Hefele L, et al. Structural Evolution of the Emerging 2014-2015 GⅡ.17 Noroviruses[J]. J Virol, 2015,90(5):2710-2715.doi:10.1128/JVI.03119-15.
[28] Kapikian A Z, Wyatt R G, Dolin R, et al. Visualization by Immune Electron Microscopy of a 27-nm Particle Associated with Acute Infectious Nonbacterial Gastroenteritis[J]. J Virol, 1972,10(5):1075-1081.
[29] Chan M C, Lee N, Hung TN, et al. Rapid emergence and predominance of a broadly recognizing and fast-evolving norovirus GⅡ.17 variant in late 2014[J]. Nat Commun, 2015,6:10061.doi:10.1038/ncomms10061.
[30] Zhang X F, Huang Q, Long Y, et al. An outbreak caused by GⅡ.17 norovirus with a wide spectrum of HBGA-associated susceptibility[J]. Sci Rep, 2015,5:17687.doi:10.1038/srep17687.
