多房棘球绦虫亮氨酰氨肽酶抗原表位的生物信息学预测#
2017-03-08杨宝良洛桑达哇刘文磊庞明泉阳丹才让王海久樊海宁
杨宝良,洛桑达哇,刘文磊,庞明泉,4,阳丹才让,4,王海久,4*,汤 锋,樊海宁,4***
(1.青海大学附属医院肝胆胰外科,青海 西宁 810001;2.青海大学高原人畜共患病研究所,青海 西宁 810001;3.青海大学高原医学研究中心,青海 西宁 810001;4.青海省包虫病研究重点实验室,青海 西宁 810001;5.青海红十字医院耳鼻咽喉-睡眠医学-甲状腺外科,青海 西宁 810000)
多房棘球蚴病(alveolar echinococcosis,AE)是由多房棘球绦虫(Echinococcus multilocularis)感染人体引起的一种严重危害人类健康的人兽共患寄生虫病,主要流行于我国的青海、四川、西藏三省交界区。多房棘球绦虫在肝脏内呈侵蚀性肿瘤样生长,又被称之为“虫癌”。目前对于多房棘球蚴病缺乏有效的早期预防措施。因此,研制有效的抗多房棘球绦虫疫苗尤为重要。
免疫预防与血清学诊断是诊断防治各种流行性疾病方法中比较有效的措施。作为免疫预防的主要研究方向,目前多表位疫苗已被应用于抗感染、抗肿瘤发生等免疫预防相关研究中[1]。与传统疫苗相比,多表位疫苗安全、稳定、特异性高,可被具有多种遗传背景的MHC分子识别并结合、抗原呈递高效的优势,可有效应对病原微生物的变异和免疫反应中的诸多不利因素[2]。然而寻找具有良好免疫原性的抗原位点是当前多表位疫苗研究的难点[3]。
亮氨酰氨肽酶(leucyl aminopeptidase,LAP)是一类从蛋白质或肽链的N端选择性切割氨基酸残基的外肽酶的总称,具有维持细胞的正常生理功能的重要作用,具有较强的免疫原性,其氨肽酶M17家族(m17lap)是目前寄生虫免疫学研究的热点,其种属特异性较高,与其他种属来源的血清很少发生交叉反应。因而不同来源的m17lap已被作为多种寄生虫特异性诊断及药物靶向治疗的研究重点。在多房棘球绦虫相关蛋白的优势抗原表位,尤其是多表位疫苗的研究中,LAP从未被报道过[4-13]。
本研究主要通过生物信息学软件SOPMA分析多房棘球绦虫亮氨酰氨肽酶的蛋白质二级结构特征。进一步通过生物信息学软件IEDB和Syfpeithi预测分析多房棘球绦虫亮氨酰氨肽酶潜在的T细胞优势抗原表位,使用生物信息学软件Bcepred和ABCpred预测分析多房棘球绦虫亮氨酰氨肽酶潜在的B细胞优势抗原表位。为后续多房棘球绦虫相关亮氨酰氨肽酶的血清学检测、免疫预防及疫苗研发奠定基础。
1 材料与方法
1.1 获取多房棘球绦虫亮氨酰氨肽酶的氨基酸序列
从Gen Bank中(http://www.ncbi.nlm.nih.gov/genbank/)获得多房棘球绦虫亮氨酰氨肽酶氨基酸序列。
1.2 预测多房棘球绦虫亮氨酰氨肽酶的蛋白质二级结构特征
使用生物信息学软件SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)分析多房棘球绦虫亮氨酰氨肽酶的蛋白质二级结构特征。输入1.1中从Gen Bank中获得的多房棘球绦虫亮氨酰氨肽酶的全部氨基酸序列,设置参数(均为默认值),分析其蛋白二级结构特征。
1.3 预测多房棘球绦虫亮氨酰氨肽酶的T细胞抗原表位
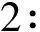
1.4 预测多房棘球绦虫亮氨酰氨肽酶的B细胞抗原表位
使用生物信息学软件Bcepred(http://www.imtech.res.in/raghava/bcepred/bcepred_ submission.html)、ABCpred(http://www.imtech.res.in/raghava/abcpred/ABC_submission. html)预测分析多房棘球绦虫亮氨酰氨肽酶潜在的B细胞抗原表位。输入1.1中从Gen Bank中获得的多房棘球绦虫亮氨酰氨肽酶的全部氨基酸序列,设置Bcepred参数(均为默认值)。设置ABCpred的抗原表位长度为10/12/14/16,余为默认值。
2 结果
2.1 多房棘球绦虫亮氨酰氨肽酶的氨基酸序列
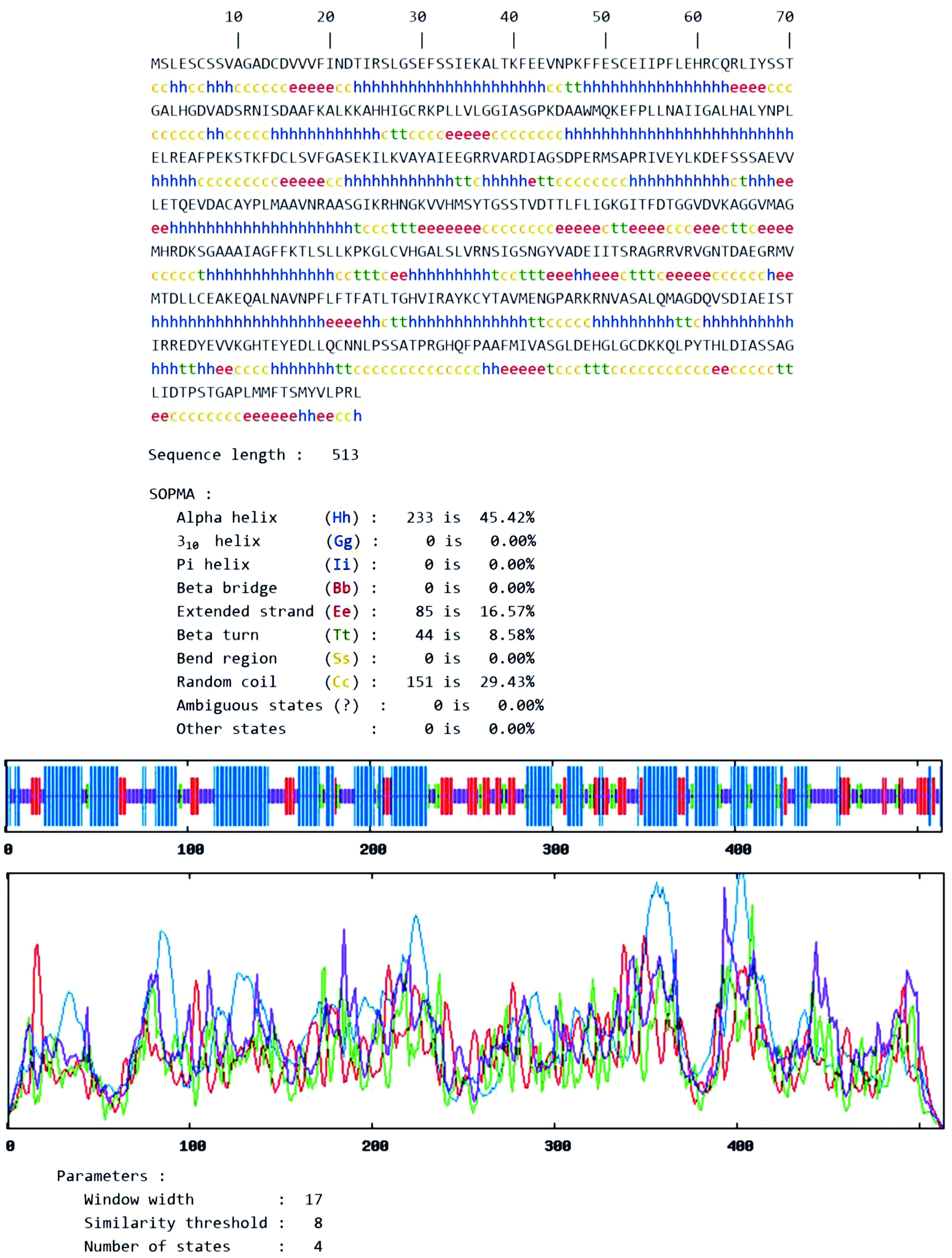
从Gen Bank中获得多房棘球绦虫亮氨酰氨肽酶的全部氨基酸序列(表1),多房棘球绦虫亮氨酰氨肽酶由513个氨基酸组成(Gen Bank:CDS42600.1)
表1多房棘球绦虫亮氨酰氨肽酶的氨基酸序列

Table 1 Amino acid sequence of the EmLAP
2.2 多房棘球绦虫亮氨酰氨肽酶的蛋白质二级结构特征预测结果
使用生物信息学软件SOPMA对多房棘球绦虫亮氨酰氨肽酶的蛋白质二级结构进行分析(图1),其中α螺旋占比为45.42%、β折叠占比为16.57%、β转角占比为8.58%、无规卷曲占比为29.43%。
2.3 多房棘球绦虫亮氨酰氨肽酶的T细胞抗原表位预测结果
使用生物信息学软件IEDB预测分析多房棘球绦虫亮氨酰氨肽酶潜在的T细胞抗原表位(表2),将分值较高的20个位点区域作为被预测的潜在抗原表位,分别是L59-67、L392-400、L392-403、L75-84、L233-240、L228-240、L230-240、L331-344、L333-346、L445-454、L395-402、L174-186、L227-239、L231-243、L331-343、L332-344、L334-346、L388-400、L391-403、L392-404。使用生物信息学软件Syfpeithi预测分析多房棘球绦虫亮氨酰氨肽酶潜在的T细胞抗原表位(表3),将分值较高的20个位点区域作为被预测的潜在抗原表位,分别是L128-136、L125-133、L19-27、L290-298、L160-168、L300-308、L299-308、L354-363、L65-73、L132-140、L362-370、L124-133、L305-314、L406-415、L311-319、L482-491、L26-34、L404-412、L64-73、L127-136。结合亮氨酰氨肽酶的蛋白质二级结构特征,综合分析生物信息学软件IEDB、Syfpeithi的预测结果,预测出亮氨酰氨肽酶具有9个T细胞潜在优势抗原表位,分别为L65-73、L75-84、L174-186、L233-240、L301-308、L333-343、L392-400、L445-454、L482-490。
蓝色:α螺旋;红色:β折叠;绿色:β转角;紫色:无规卷曲
图1多房棘球绦虫亮氨酰氨肽酶的蛋白质二级结构预测分析图
表2多房棘球绦虫亮氨酰氨肽酶的T细胞抗原表位IEBD预测结果
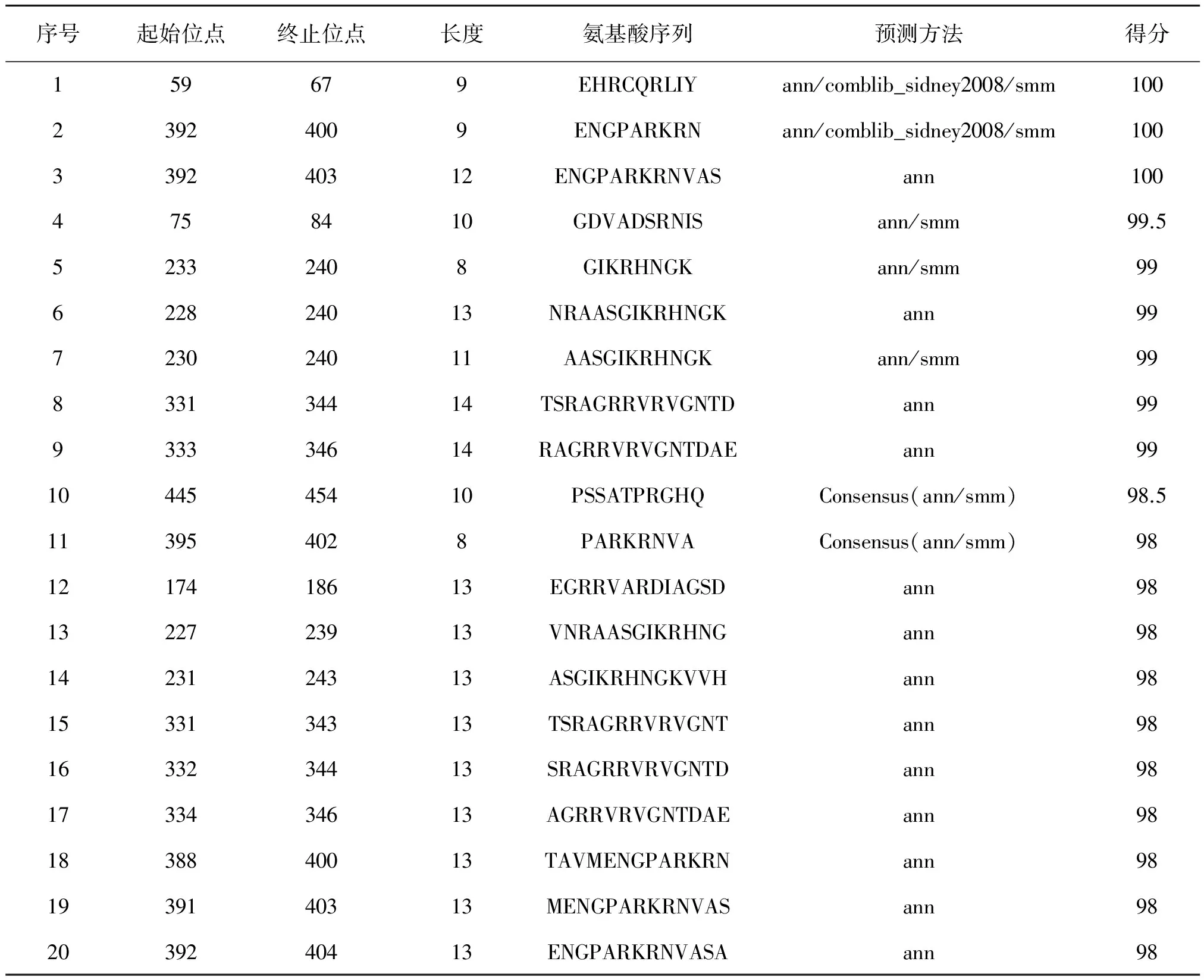
Table 2 Analysis of the T-cell epitopes of EmLAP using IEDB online prediction software
表3多房棘球绦虫亮氨酰氨肽酶的T细胞抗原表位Syfpeithi预测结果
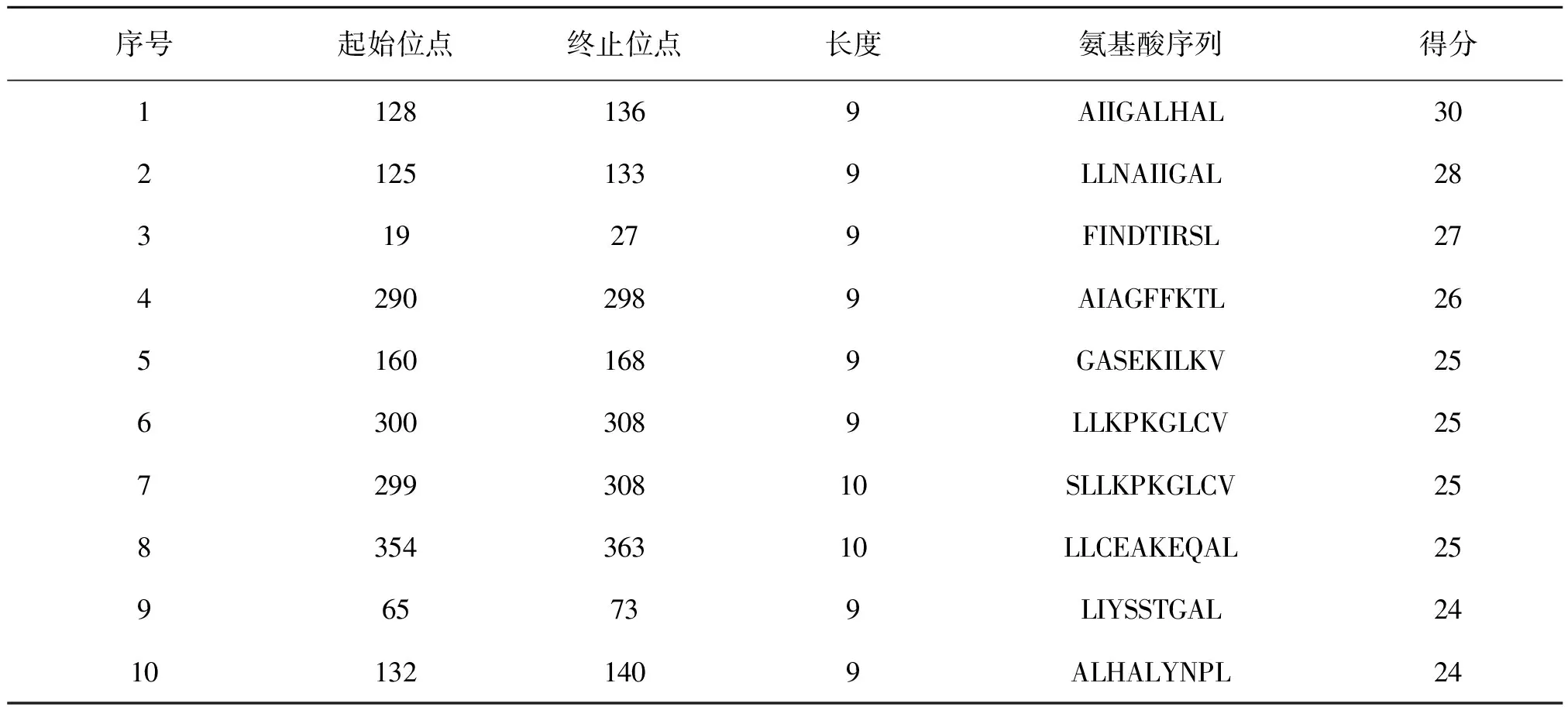
Table 3 Analysis of the T-cell epitopes of EmLAP using Syfpeithi online prediction software
续表:

序号起始位点终止位点长度氨基酸序列得分113623709ALNAVNPFL241212413310PLLNAIIGAL241330531410GLCVHGALSL241440641510QMAGDQVSDI24153113199ALSLVRNSI231648249110HLDIASSAGL231726349SLGSEFSSI22184044129ALQMAGDQV2219647310RLIYSSTGAL222012713610NAIIGALHAL22
2.4 多房棘球绦虫亮氨酰氨肽酶的B细胞抗原表位预测结果
使用生物信息学软件Becpred预测分析多房棘球绦虫亮氨酰氨肽酶潜在的B细胞抗原表位(图2),对其亲水性、弹性、抗原倾向性、抗原暴露表面积进行分析,其中亲水性较强的位点区域为L74-85(HGDVADSRNISD)、L110-117(ASGPKDAA)、L148-154(EKSTKFD)、L183-191(AGSDPERMS)、L200-206(KDEFSSS)、L212-218(ETQEVDA)、L244-254(SYTGSSTVDTT)、L281-289(HRDKSGAAA)、L330-335(TSRAGRR)、L339-348(VGNTDAEGR)、L356-362(CEAKEQA)、L392-398(ENGPARK)、L408-414(AGDQVSD)、L430-437(KGHTEYED)、L446-452(SSATPRG)、L493-499(DTPSTGA);弹性较强的位点区域为L22-31(DTIRSLGSEF)、L76-83(DVADSRNI)、L146-154(EAFPEKSTK)、L172-178(YAIEEGR)、L183-190(DIAGSDP)、L200-207(EYLKDEFS)、L235-240(SGIKRHN)、L244-250(VHMSYTG)、L281-288(AGMHRDKS)、L315-321(SLVRNSI)、L330-337(EIITSRAG)、L393-400(MENGPARK)、L445-451(NLPSSAT)、L471-477(HGLGCDK)、L492-499(GLIDTPS);抗原倾向性较强的位点区域为L146-154(FPEKSTKFD)、L234-241(IKRHNGKV)、L393-401(NGPARKRNV)、L421-428(IRREDYEV)、L473-481(CDKKQLPYT);抗原暴露表面积较强的位点区域为L4-9(LESCSSV)、L13-22(DCDVVVFIND)、L51-68(SCEIIPFLEHRCQRLIYS)、L98-109(GCRKPLLVLGGI)、L152-160(KFDCLSVFG)、L193-199(PRIVEYL)、L208-216(EVVLETQEV)、L240-247(KVVHMSYT)、L252-258(VDTTLFL)、L295-310(FKTLSLLKPKGLCVHG)、L350-356(VMTDLLC)、L426-432(YEVVKGH)、L435-444(YEDLLQCNNL)、L477-484(QLPYTHLD)、L504-513(FTSMYVLPRL)(表4)。使用生物信息学软件ABCpred预测分析多房棘球绦虫亮氨酰氨肽酶潜在的B细胞抗原表位(表5)。从抗原表位长度为10、12、14、16中分别选取前10名(含并列)作为潜在优势表位,分别为L237-246、L117-126、L58-67、L245-254、L148-157、L197-206、L176-185、L205-214、L281-290、L292-301、L40-49、L303-312、L134-143,L317-328、L38-49、L107-118、L295-306、L195-206、L335-346、L74-85、L144-155、L111-122、L101-112、L158-169、L129-140、L65-76、L230-241、L78-89、L373-384、L289-300、L27-38、L200-211、L164-175、L118-129,L349-362、L397-410、L125-138、L97-110、L213-226、L156-169、L10-23、L326-339、L224-237、L52-65、L39-52、L295-308、L385-398、L195-208、L359-372、L18-31,L489-504、L256-271、L31-46、L207-222、L458-473、L274-289、L464-479、L431-446、L326-341、L51-66、L181-196、L389-404、L415-430、L378-393、L7-22、L497-512、L317-332、L106-121。结合亮氨酰氨肽酶的蛋白质二级结构特征,使用生物信息学软件Becpred、ABCpred预测结果,预测出亮氨酰氨肽酶具有20个B细胞潜在优势抗原表位,分别为L13-22、L39-47、L75-84、L98-110、L146-154、L174-186、L183-191、L233-240、L247-254、L281-288、L295-306、L319-328、L330-337、L339-347、L392-400、L397-410、L421-428、L464-473、L493-504、L504-512。
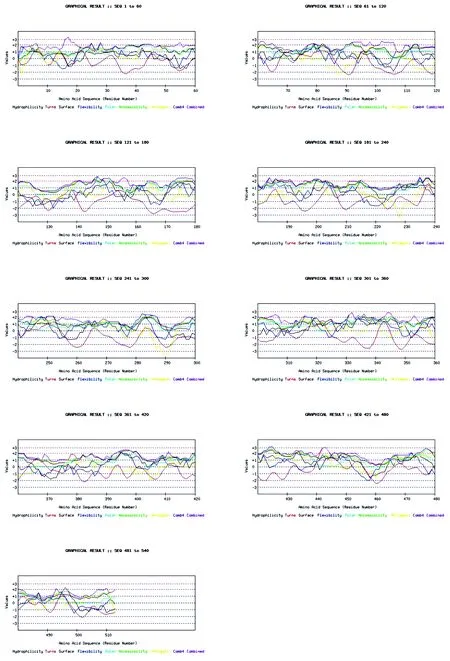
黑色:亲水性;蓝色:弹性;黄色:抗原倾向性;棕色:抗原暴露表面积
图2多房棘球绦虫亮氨酰氨肽酶的B细胞抗原表位Becpred预测结果
Figure2AnalysisoftheBcellepitopesofEmLAPusingBecpredonlinepredictionsoftware
表4多房棘球绦虫亮氨酰氨肽酶的B细胞抗原表位Becpred预测结果
*:划线区域为预测表位的氨基酸残基.
表5多房棘球绦虫亮氨酰氨肽酶的B细胞抗原表位ABCpred预测结果
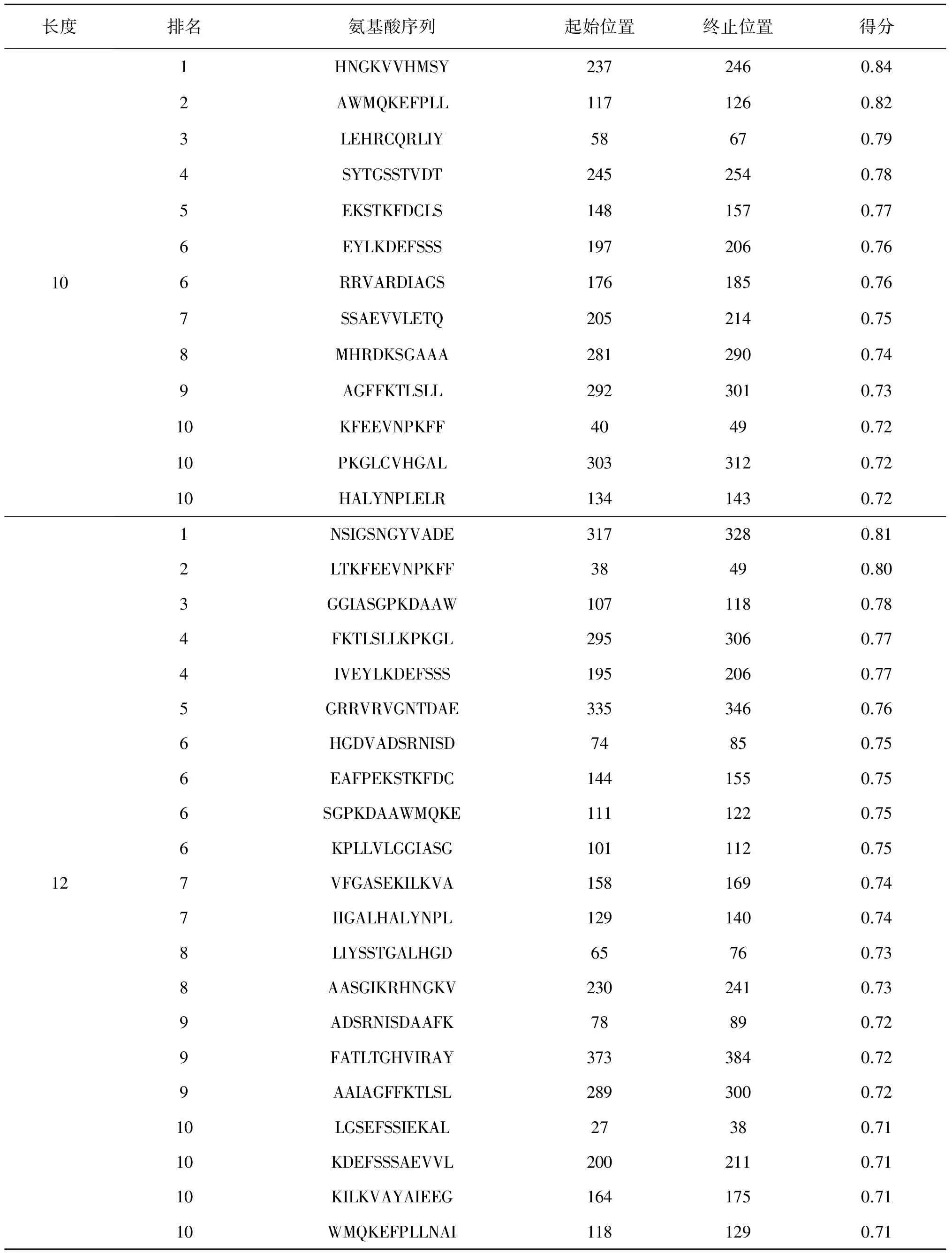
Table 5 Analysis of the B cell epitopes of EmLAP using ABCpredonline prediction software
续表:
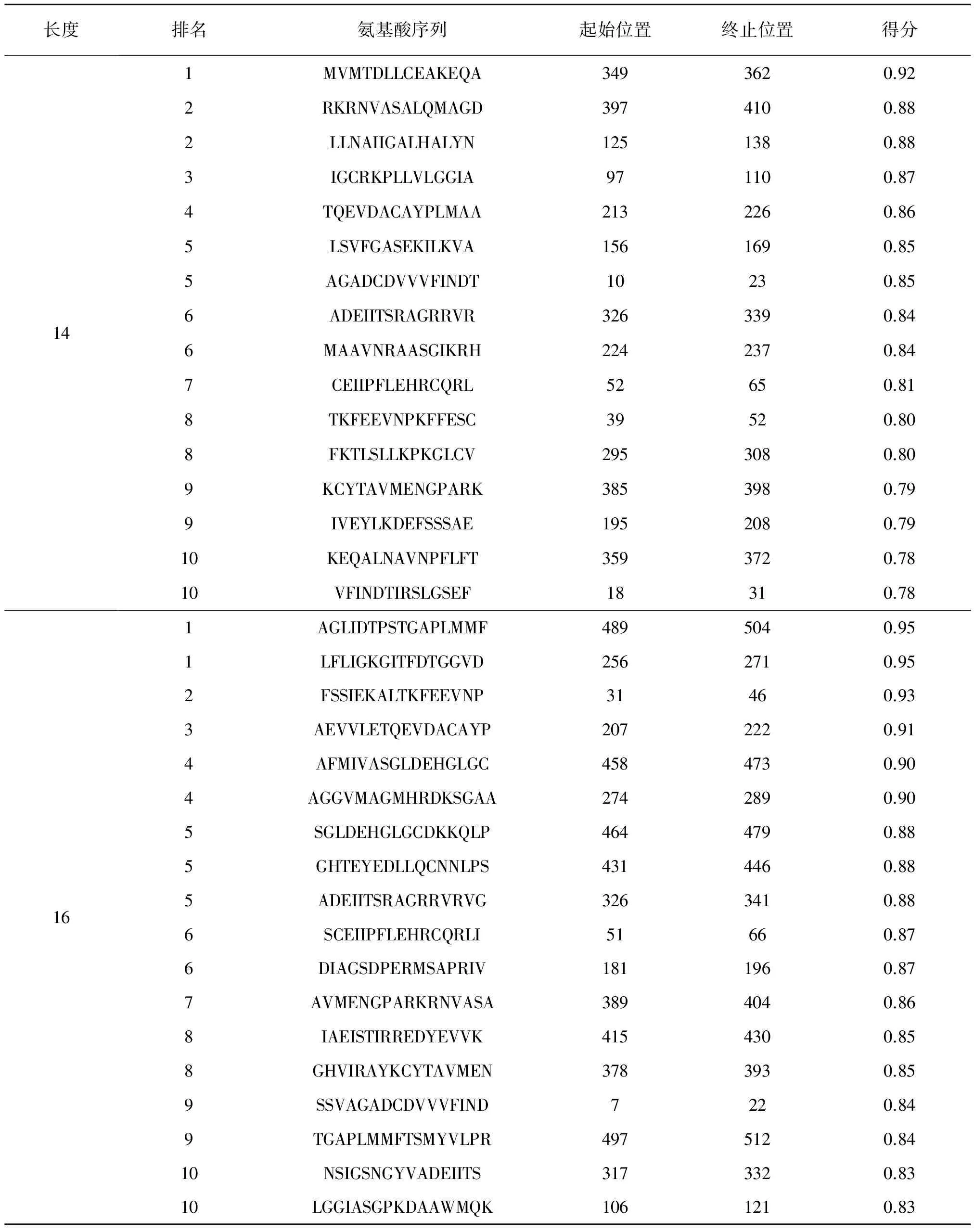
长度排名氨基酸序列起始位置终止位置得分141MVMTDLLCEAKEQA3493620.922RKRNVASALQMAGD3974100.882LLNAIIGALHALYN1251380.883IGCRKPLLVLGGIA971100.874TQEVDACAYPLMAA2132260.865LSVFGASEKILKVA1561690.855AGADCDVVVFINDT10230.856ADEIITSRAGRRVR3263390.846MAAVNRAASGIKRH2242370.847CEIIPFLEHRCQRL52650.818TKFEEVNPKFFESC39520.808FKTLSLLKPKGLCV2953080.809KCYTAVMENGPARK3853980.799IVEYLKDEFSSSAE1952080.7910KEQALNAVNPFLFT3593720.7810VFINDTIRSLGSEF18310.78161AGLIDTPSTGAPLMMF4895040.951LFLIGKGITFDTGGVD2562710.952FSSIEKALTKFEEVNP31460.933AEVVLETQEVDACAYP2072220.914AFMIVASGLDEHGLGC4584730.904AGGVMAGMHRDKSGAA2742890.905SGLDEHGLGCDKKQLP4644790.885GHTEYEDLLQCNNLPS4314460.885ADEIITSRAGRRVRVG3263410.886SCEIIPFLEHRCQRLI51660.876DIAGSDPERMSAPRIV1811960.877AVMENGPARKRNVASA3894040.868IAEISTIRREDYEVVK4154300.858GHVIRAYKCYTAVMEN3783930.859SSVAGADCDVVVFIND7220.849TGAPLMMFTSMYVLPR4975120.8410NSIGSNGYVADEIITS3173320.8310LGGIASGPKDAAWMQK1061210.83
3 讨论
多表位疫苗指同时携带多个目标抗原相关的以及辅助性的表位的疫苗(常为T细胞、B细胞组合),相关表位的预测及筛选是多表位疫苗研究的重点。使用生物信息学软件对棘球绦虫抗原蛋白优势抗原表位进行预测分析,已成为棘球绦虫相关多表位疫苗研究的热点。马秀敏等[14]使用Predict Protein软件预测了细粒棘球绦虫AgB1抗原蛋白的二级结构;使用Bcepred、ABCpred、IEDB、syfpeithi四种软件预测出了细粒棘球绦虫AgB1抗原蛋白的4个B细胞表位及4个T细胞表位。刘学磊等[15]使用SOMPA软件预测分析了细粒棘球绦虫AgB5抗原蛋白的二级结构;使用LEPS、ABCpred、IEDB、syfpeithi四种软件预测出了细粒棘球绦虫AgB5抗原蛋白的3个B细胞表位及4个T细胞表位。李玉娇等[16]使用SOMPA软件预测分析了细粒棘球绦虫Eg95抗原蛋白的二级结构,使用DNAstar、IEDB、syfpeithi三种软件分别预测出了细粒棘球绦虫Eg95抗原蛋白的7个B细胞表位及7个T细胞表位。蛋白质的二级结构是其表位预测的重要参数,β转角为凸出结构,多出现在蛋白质抗原表面,有利于与抗体结合,无规卷曲同样位于蛋白质的表面,容易与抗体结合,因此β转角和无规卷曲较有可能成为抗原表位;而α螺旋和β折叠在维持蛋白质的二级结构稳定中起重要作用,结构规则且不易变形,通常位于该蛋白质的内部,较难结合抗体,一般不作为抗原表位。本研究通过使用软件SOPMA对多房棘球绦虫亮氨酰氨肽酶的蛋白质二级结构进行分析表明,多房棘球绦虫亮氨酰氨肽酶具有多个形成抗原表位的β转角及无规卷曲区域,可作为多房棘球绦虫多表位疫苗研究的候选分子。
使用生物信息学软件预测B细胞线性表位主要基于蛋白氨基酸残基的理化性质。天然状态下,蛋白质的疏水性氨基酸被包埋于蛋白内部,而亲水性氨基酸一般位于蛋白表面,较易与抗体结合,可作为抗原表位[18],由于抗原抗体结合是一个嵌合过程,蛋白构象会发生变化,弹性参数较高的氨基酸残基易发生弯曲和折叠,弹性参数较高的氨基酸残基最有可能成为抗原表位[19]。抗原倾向性反映了抗原潜在的免疫原区,潜在的免疫原区最有可能出现具有抗原倾向的表位。抗原暴露表面积主要是通过分析反映氨基酸残基在蛋白质的外层中的分布,即溶剂分子接触抗原氨基酸的可能性,间接反映与抗体的结合能力[20]。本研究使用Becpred、ABCpred两种生物信息学软件对多房棘球绦虫亮氨酰氨肽酶的B细胞表位进行预测分析,两种不同软件的预测结果同样有诸多重复,结合亮氨酰氨肽酶的蛋白质二级结构特征,预测出多房棘球绦虫亮氨酰氨肽酶具有20个B细胞潜在优势抗原表位。
本研究使用多房棘球绦虫亮氨酰氨肽酶作为研究对象,因其在血吸虫、阿米巴原虫、华支睾吸虫、巴贝虫、疟原虫、弓形虫等多种寄生虫的虫体及分泌物中存在,具有较强的免疫原性、较高的种属特异性等优点。杨祖婷[21]等已利用生物信息学软件及PCR技术得到曼氏迭宫绦虫亮氨酸氨基肽酶(SmLAP)的基因及其编码蛋白的结构,并通过原核表达方法得到单一组分的SmLAP重组蛋白,而多房棘球绦虫亮氨酰氨肽酶相关研究仍未被报道。本研究的主要目的是通过使用生物信息学软件SOPMA分析多房棘球绦虫亮氨酰氨肽酶的蛋白质二级结构特征,通过使用生物信息学软件IEDB和Syfpeithi预测分析出多房棘球绦虫亮氨酰氨肽酶潜在的T细胞优势抗原表位,使用生物信息学软件Bcepred和ABCpred预测分析多房棘球绦虫亮氨酰氨肽酶潜在的B细胞优势抗原表位。结果表明多房棘球绦虫亮氨酰氨肽酶具有9个T细胞潜在优势抗原表位、20个B细胞潜在优势抗原表位。并且其中4个同时具有B/T双细胞表位,分别为L75-84、L174-186、L233-240、L392-400。
本研究可为后续多房棘球绦虫相关亮氨酰氨肽酶的血清学检测、免疫预防及多表位疫苗研发奠定理论基础。
[1]Hazama S,Maeda K,Oka M.Epitope peptide vaccine with oncoantigen for cancer and its biomarker[J].Nihon Rinsho,2012,70:2189-2193.
[2]MELOEN R H,CASALK J I.Synthetic peptide vaccines:suc-cess at last[J].Vaccine,1995,13(10):885-886.
[3]You L,Brusic V,Gallagher M ,et al.Using Gaussian process with test rejection to detect T-cell epitopes in pathogen genomes[J].IEEE/ACM Trans Comput Biol Bioinform,2010,7(4):741-751.
[4]Chaudhary M,Singh V,Anvikar AR,et al.Screening and In Vitro Evaluation of Potential Plasmodium falciparum Leucyl Aminopeptidase Inhibitors[J].Curr Comput Aided Drug Des,2016,12(4):282-293.
[5]Lee YR,Na BK,Moon EK,et al.Essential Role for an M17 Leucine Aminopeptidase in Encystation of Acanthamoeba castellanii[J].PLoS One,2015,10(6):e0129884.
[6]Aboge GO,Cao S,Terkawi MA,et al.Molecular Characterization of Babesia bovis M17 Leucine Aminopeptidase and Inhibition of Babesia Growth by Bestatin[J].J Parasitol,2015,101(5):536-541.
[7]Zheng J,Jia H,Zheng Y,et al.Knockout of leucine aminopeptidase in Toxoplasma gondii using CRISPR/Cas9[J].Int J Parasitol,2015,45(2-3):141-148.
[8]Qu H,Xu Y,Sun H,et al.Systemic and local mucosal immune responses induced by orally delivered Bacillus subtilis spore expressing leucine aminopeptidase 2 of Clonorchis sinensis[J].Parasitol Res,2014,113(8):3095-3103.
[9]Changklungmoa N,Chaithirayanon K,Kueakhai P,et al.Molecular cloning and characterization of leucine aminopeptidase from Fasciola gigantica[J].Exp Parasitol,2012,31(3):283-291.
[10]Kang JM,Ju HL,Ju JW,et al.Comparative biochemical and functional properties of two leucine aminopeptidases of Clonorchis sinensis[J].Mol Biochem Parasitol,2012,182(1-2):17-26.
[11]Faustina HL,Luo QL,Zhong ZR,et al.Evaluation of recombinant SjLAP and SjFBPA in detecting antibodies to Schistosoma japonicum[J].Zhongguo Ji Sheng Chong Xue Yu Ji Sheng Chong Bing Za Zhi,2011,29(5):339-347.
[12]Lee JY,Song SM,Seok JW,et al.M17 leucine aminopeptidase of the human malaria parasite Plasmodium vivax[J].Mol Biochem Parasitol,2010,170(1):45-48.
[13]Maric S,Donnelly SM,Robinson MW,et al.The M17 leucine aminopeptidase of the malaria parasite Plasmodium falciparum:importance of active site metal ions in the binding of substrates and inhibitors[J].Biochemistry,2009,48(23):5435-5439.
[14]马秀敏,胡晓安,阿尔孜古丽·吐尔逊,等.细粒棘球绦虫AgB1抗原表位的生物信息学预测[J].科技导报,2013,31(27):27-30.
[15]刘学磊,刘玉梅,曹文艳,等.细粒棘球绦虫AgB5抗原表位的生物信息学预测[J].中国病原生物学杂志,2015,10(1):38-41.
[16]李玉娇,王晶,赵慧,等.细粒棘球绦虫Eg95抗原表位的生物信息学预测[J].中国人兽共患病学报,2011,27(10):892-900.
[17]Yan C,Wang R,Li J,et al.HLA-A gene polymorphism de-fined by high-resolution sequence-based typing in 161Northern Chinese Han people[J].Gen Prot Bioinform,2003,1(4):304-309.
[18]Zhang W,Liu J,Zhao M,et al.Predicting linear B-cell epitopes by using sequence-derived structural and physico-chemical features[J].Int J Data Min Bioinform,2012,6(5):557-569.
[19]Mahdavi M,Mohabatkar H,Keyhanfar M,et al.Linearand conformational B cell epitope prediction of the HER 2 ECD-subdomain III by in silico methods[J].Asian Pac J CancerPrev,2012,13(7):3053-3059.
[20]Xue M,Shi X,Zhang J,et al.Identification of a con-served B cell epitope on reticuloendotheliosis virus envelope pro-tein by screening a phage-displayed random peptide library[J].PLo S One,2012,7(11):e49842.
[21]杨祖婷,陈立强,符瑞佳,等.曼氏迭宫绦虫亮氨酸氨基肽酶(SmLAP)的生物信息学分析克隆及表达[J].中国病原生物学杂志,2016,11(11):1004-1009.
