基于PCR-DGGE技术辅助筛选氨氮降解菌株
2017-03-06张智超吴逸飞姚晓红汤江武葛向阳
张智超,孙 宏,吴逸飞,王 新,姚晓红,柳 永,汤江武,*,葛向阳,*
(1.华中农业大学 微生物学国家重点实验室,湖北 武汉 430030; 2.浙江省农业科学院 植物保护与微生物研究所,浙江 杭州 310021)
基于PCR-DGGE技术辅助筛选氨氮降解菌株
张智超1,孙 宏2,吴逸飞2,王 新2,姚晓红2,柳 永2,汤江武2,*,葛向阳1,*
(1.华中农业大学 微生物学国家重点实验室,湖北 武汉 430030; 2.浙江省农业科学院 植物保护与微生物研究所,浙江 杭州 310021)
将PCR-变性梯度凝胶电泳(DGGE)与传统梯度浓度富集手段相结合,从不同氨氮浓度养殖污水中筛选高活性氨氮降解菌株。结果表明,经过不同氨氮浓度的驯化,初筛得到22株菌株,分别测定其氨氮降解能力,最终得到3株氨氮降解能力较强的菌株,分别命名为ZZC-3、ZZC-4和ZZC-14。经16S rDNA鉴定后,分别为戈登氏菌属、反硝化菌属和红球菌属细菌。PCR-DGGE结果表明,反硝化杆菌属的微生物在富集液中的含量最高,因此选择ZZC-4为目标菌株。经过污水降解效果验证,添加ZZC-4菌株的处理在144 h后水中氨氮和化学需氧量降解率分别达到90.8%和94.7%,显示该菌株具有良好的应用开发前景。
PCR-DGGE;氨氮降解;反硝化菌属
1 材料与方法
1.1 污水来源与配比
活性污泥采集于以活性污泥法处理污水并稳定运行的污水处理厂。养殖污水采集自金华市金帆生猪养殖场,原水氨氮含量829.3 mg·L-1,化学需氧量(COD)13 410 mg·L-1。配置氨氮含量分别为100、300、500、700 mg·L-1的驯化梯度污水,其配比分别为:10 mL 活性污泥+12.5 mL养殖污水+77.5 mL纯净水,10 mL 活性污泥+37.5 mL养殖污水+52.5 mL纯净水,10 mL 活性污泥+62.5 mL养殖污水+27.5 mL纯净水,10 mL 活性污泥+87.5 mL养殖污水+2.5 mL纯净水。
1.2 污泥驯化与氨氮降解菌初步筛选
驯化按照以下2种方式进行。
方式1(水平驯化):取10 mL活性污泥分别接入到所设置的驯化梯度污水中,于160 r·min-1、30 ℃下培养5 d,取出100 μL培养液涂布LB固体平板,37 ℃培养48 h,挑取单菌落,进行3次划线分离纯化后,保藏于新鲜的LB斜面。
方式2(垂直驯化):取10 mL活性污泥先接入到含氨氮100 mg·L-1的污水中,于160 r·min-1、30 ℃下培养5 d后转接至下一驯化梯度,直至700 mg·L-1的驯化浓度培养完毕,取出100 μL培养液涂布LB平板, 37 ℃培养48 h,挑取单菌落,进行3次划线分离纯化后,保藏于新鲜的LB斜面。
同时将上述每个驯化梯度的污泥悬浮液于5 000 r·min-1离心5 min,保存至-20 ℃,用于提取总DNA。
1.3 菌株氨氮降解性能的测定
将获得的初筛菌株接一环到LB液体培养基中,培养48 h后,取出2 mL培养液于5 000 r·min-1离心5 min,以无菌水洗涤菌体3次,最后加入500 μL无菌水混匀备用。
配制模拟污水,配方为:氯化铵8 g,无水乙酸钠2 g,七水合硫酸镁0.05 g,磷酸氢二钾0.2 g,氯化钠0.12 g,四水合硫酸锰0.01 g,硫酸亚铁0.01 g,水1 L,调节pH至7.2,121 ℃灭菌30 min后备用,其氨氮含量为1 033.6 mg·L-1。
将获得的菌悬液取500 μL接入到100 mL模拟污水中。每个菌株设置3个平行,于160 r·min-1、30 ℃条件下培养144 h,每隔48 h测定一次各供试模拟污水中的氨氮含量,并将具有较优氨氮降解效果的菌株接种于LB斜面,4 ℃条件下保存。
1.4 菌种鉴定
菌株DNA提取按照BioFlux细菌基因组抽提试剂盒(Cat.No:BSC12S1)说明书的步骤进行。PCR体系所用引物为27F(5’-AGAGTTTGATCCTGGCTCAG-3’)和1492R(5’-CTGAGCCAGGATCAAACTCT-3’)。PCR体系包括:10×transtaq buffer 2.5 μL(含有15 mmol·L-1的Mg2+),dNTP(2.5 mmol·L-1)2 μL,上下游引物(10 μmmol·L-1)各0.4 μL,DNA模板1 μL(约30 ng),用ddH2O补至25 μL。PCR程序:94 ℃ 4 min;94 ℃ 1 min,55 ℃ 1 min,72 ℃ 1 min,31个循环;72 ℃ 5 min。扩增产物经1.5%(m/V)琼脂糖凝胶电泳检测后,送交生工生物工程(上海)有限责任公司测序。
1.5 污泥样本的PCR-DGGE分析
取出1.2节所保藏的活性污泥样本,以MOBIO Powersoil试剂盒(Cat.No:12888-50)提取总DNA,并扩增16S rDNA的V3区。所用引物为GC-341F(5’-CTACGGGAGGCAGCAG-3’)、517R(5’-ATTACCGCGGCTGCTGG-3’)。PCR体系同1.4节所述。PCR程序为:94 ℃ 4 min;94 ℃ 1 min,55 ℃45 s,72 ℃ 50 s,32个循环;72 ℃ 8 min。PCR产物经1.5%(m/V)琼脂糖凝胶电泳检测后,根据文献的报道进行DGGE,分析其中细菌群落的变化。
1.6 DGGE条带的测序分析
选取驯化结束时DGGE泳道中较亮的条带放入无菌水中,4 ℃过夜后,以浸泡胶块的无菌水为模板进行PCR扩增。扩增反应体系和程序同1.5节。扩增产物(约200 bp)经1.5%(m/V)琼脂糖凝胶电泳检测后,送交生工生物工程(上海)有限责任公司测序。
1.7 菌株氨氮与COD降解能力验证
将所获得的复筛菌株接入LB液体培养基中,于30 ℃、160 r·min-1摇床培养2 d。取2 mL培养液, 5 000 r·min-1离心5 min后弃上清。无菌水洗涤3次后,取500 μL菌液接入至100 mL 模拟污水中(氨氮含量为1 027.3 mg·L-1,COD含量为1 545 mg·L-1),30 ℃、160 r·min-1振荡培养144 h,每隔48 h测定一次模拟污水中的氨氮和COD含量。
2 结果与分析
2.1 氨氮降解菌株的初筛分离与鉴定
通过2种方式的污水梯度驯化,共挑取22株菌株,依次命名为ZZC-1至ZZC-22。其氨氮降解能力检测结果如表1所示。可以看出,有3株菌具有较强的氨氮降解能力,分别为ZZC-3、ZZC-4和ZZC-14。其中:ZZC-3和ZZC-4培养96 h后水中氨氮含量最低,氨氮降解率分别达到97.2%和97.8%;ZZC-14菌株在培养144 h后水中氨氮含量最低,氨氮降解率达到94.4%。
对ZZC-3、ZZC-4和ZZC-14菌株进行16S r-DNA测序后发现,3株菌的PCR产物长度均为1 500 bp左右(图1)。将测序结果在GenBank中进行序列比对分析,ZZC-3属于戈登氏菌属(Gordoniasp.),ZZC-4属于反硝化菌属(Denitrobactersp.),ZZC-14属于红球菌属(Rhodococcussp.),相似度均达到99%。
2.2 驯化污泥样品的PCR-DGGE分析
对驯化富集过程中的污泥样本提取总DNA后进行PCR-DGGE分析,结果如图2所示。6号和5号条带所对应的菌株随着驯化氨氮浓度的增加也逐渐增加。
表1 模拟污水中不同培养时间菌株对氨氮的降解效果
Table 1 Ammonia degradation rate of selected strains after different incubation time
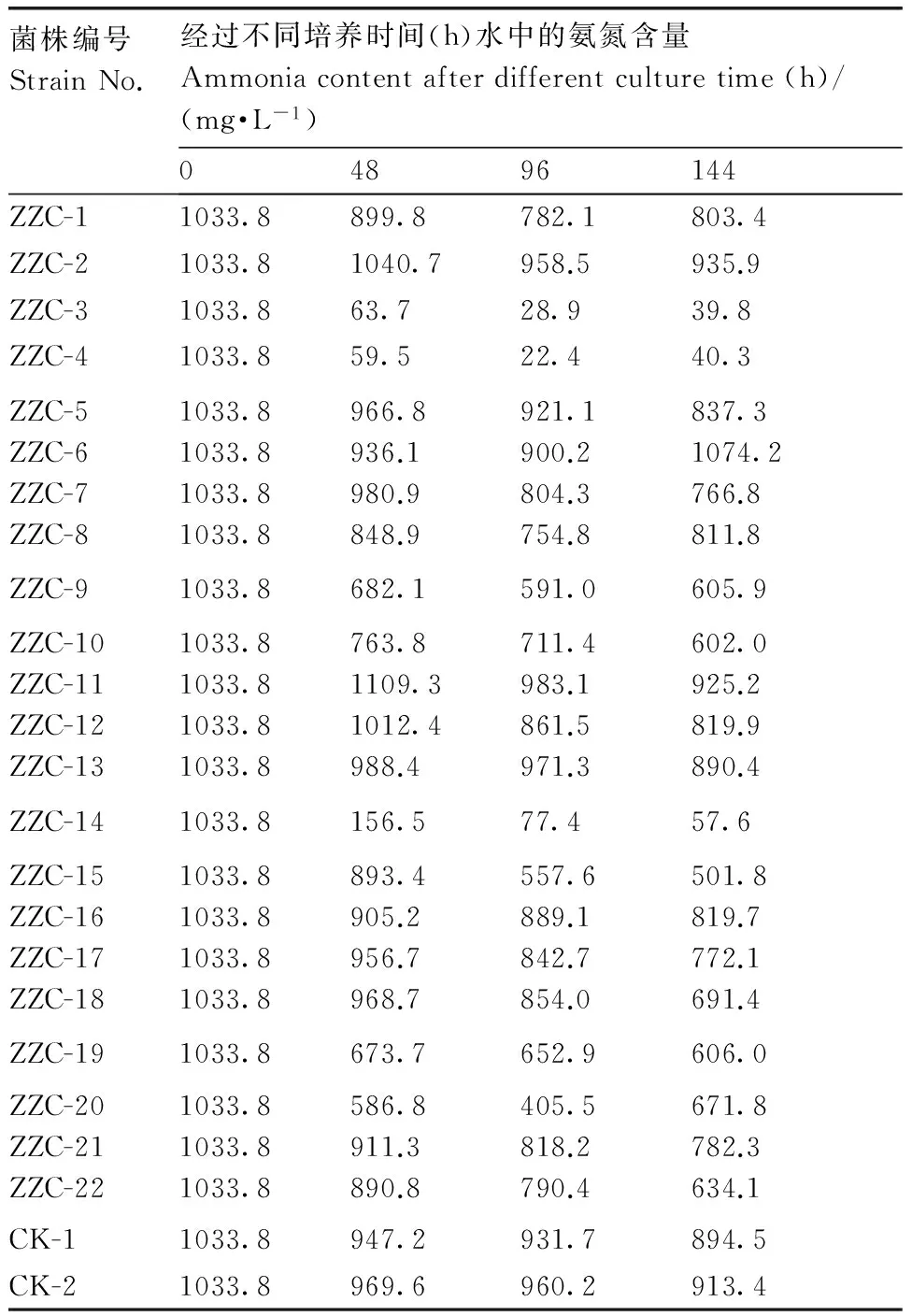
菌株编号StrainNo.经过不同培养时间(h)水中的氨氮含量Ammoniacontentafterdifferentculturetime(h)/(mg·L-1)04896144ZZC-11033.8899.8782.1803.4ZZC-21033.81040.7958.5935.9ZZC-31033.863.728.939.8ZZC-41033.859.522.440.3ZZC-5ZZC-6ZZC-7ZZC-81033.81033.81033.81033.8966.8936.1980.9848.9921.1900.2804.3754.8837.31074.2766.8811.8ZZC-91033.8682.1591.0605.9ZZC-10ZZC-11ZZC-12ZZC-131033.81033.81033.81033.8763.81109.31012.4988.4711.4983.1861.5971.3602.0925.2819.9890.4ZZC-141033.8156.577.457.6ZZC-15ZZC-16ZZC-17ZZC-181033.81033.81033.81033.8893.4905.2956.7968.7557.6889.1842.7854.0501.8819.7772.1691.4ZZC-191033.8673.7652.9606.0ZZC-20ZZC-21ZZC-221033.81033.81033.8586.8911.3890.8405.5818.2790.4671.8782.3634.1CK-11033.8947.2931.7894.5CK-21033.8969.6960.2913.4
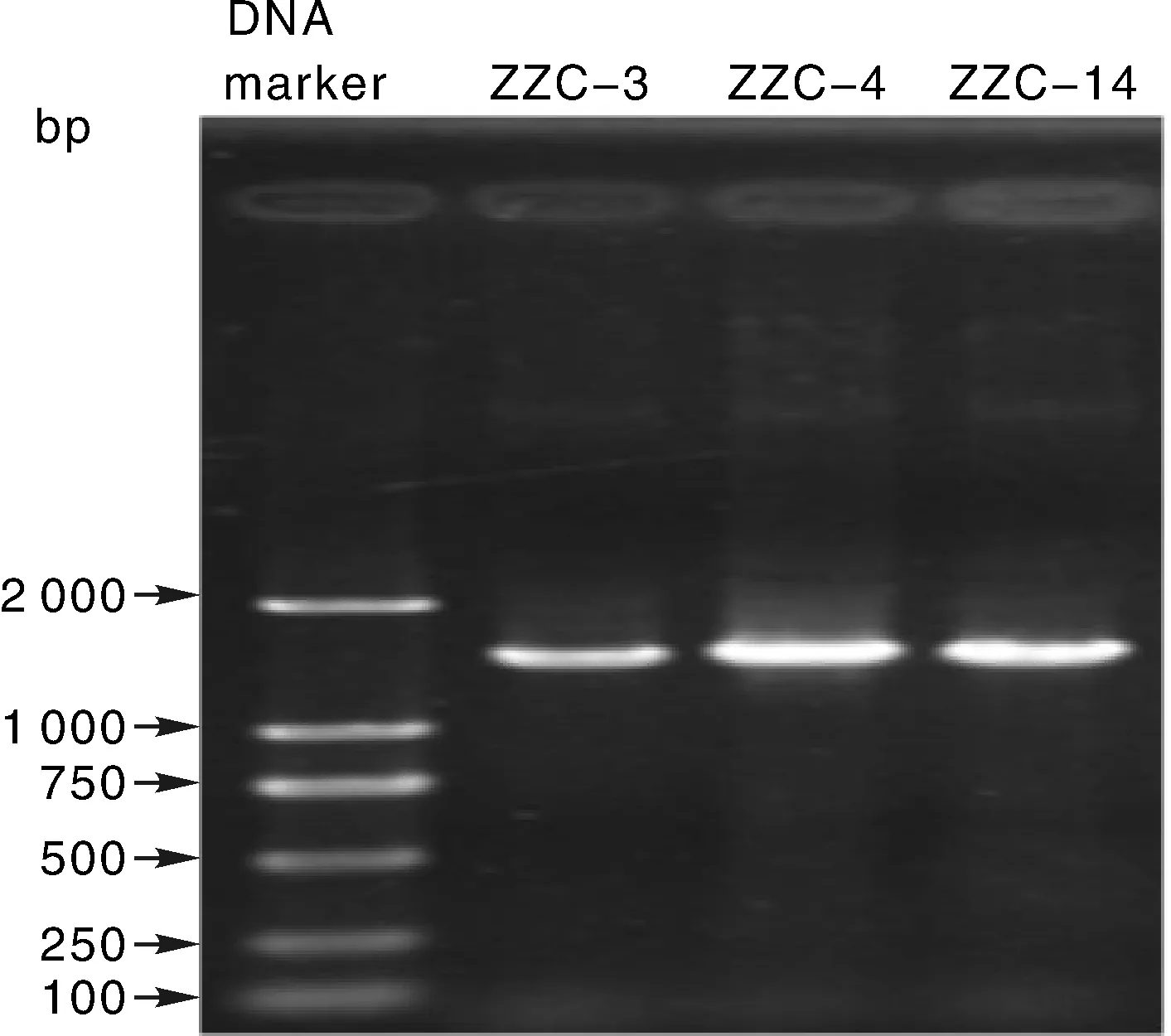
图1 所选菌株的16S rDNA PCR结果Fig.1 16Sr DNA PCR results of the selected strains
2.3 DGGE条带的测序鉴定
将图2所示的7条条带(长度约200 bp)切取、测序,序列在NCBI上进行比对后,结果如表2所示。条带1号、3号、6号和7号分别属于假单胞菌(Pseudomonassp.)、溶杆菌属(Lysobactersp.)、反硝化菌属(Denitrobactersp.)及产微球茎菌属(Microbulbifermarinus);条带2号、4号和5号则属于未培养菌(uncultured bacterium)。
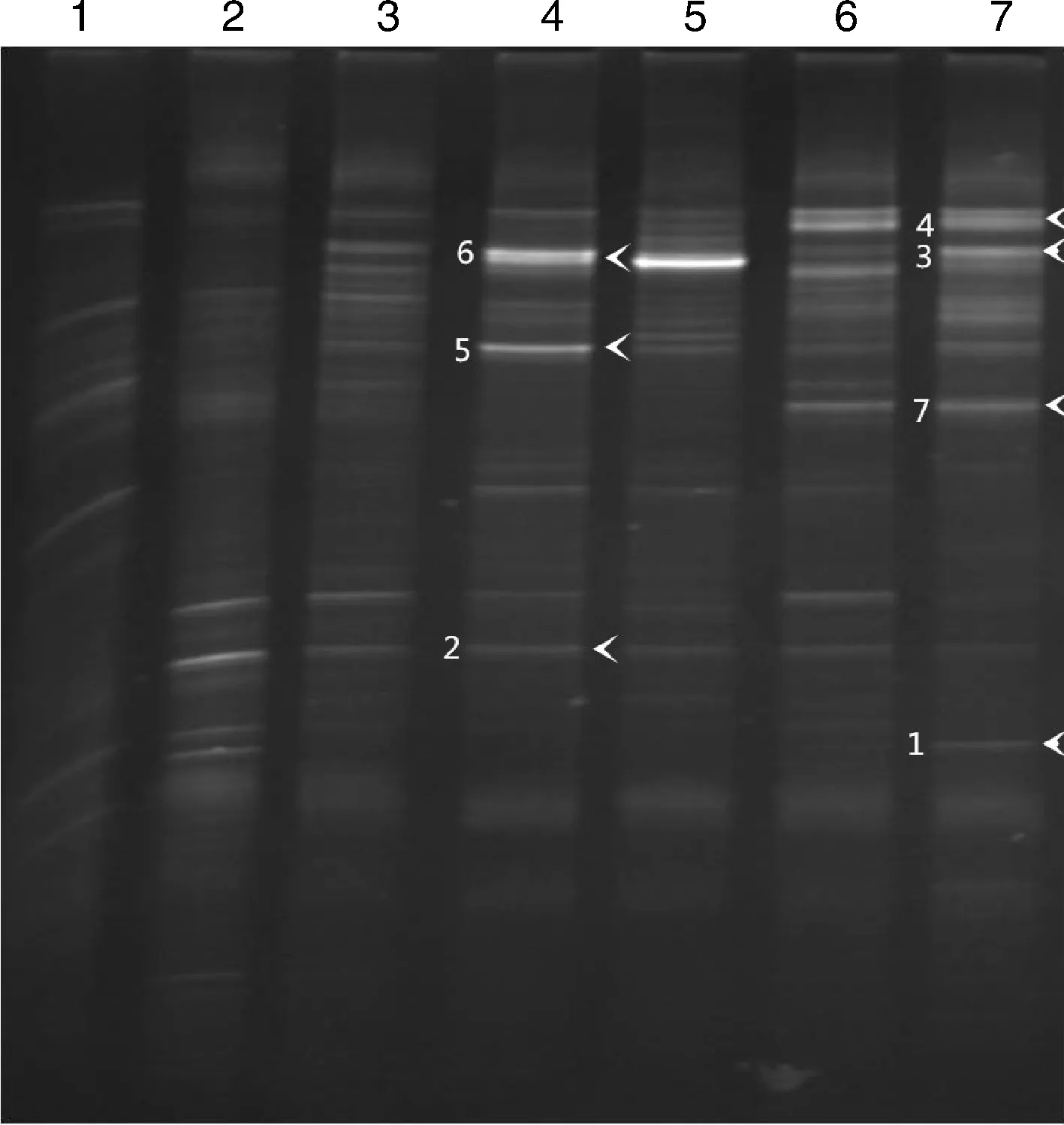
1~4泳道为污泥水平驯化试验结果,其中1、2、3和4泳道氨氮驯化浓度分别为100、300、500和700 mg·L-1;5~7泳道为污泥垂直驯化试验结果,其中5、6和7泳道氨氮驯化浓度分别为300、500和700 mg·L-1。图中标记1~7条带为切胶位点Lane 1-4 showed the results of level acclimation, in which the ammonia concentration of lane 1, 2, 3, 4 was 100, 300, 500 and 700 mg·L-1, respectively. Lane 5-7 showed the results of vertical acclimation, in which the ammonia concentration of lane 5, 6, 7 was 300, 500 and 700 mg·L-1, respectively. Band 1-7 showed the cutting sites图2 水平以及垂直梯度浓度氨氮污水驯化活性污泥DGGE指纹图谱Fig.2 DGGE fingerprint picture of strain acclimation with gradient concentrations of ammonia nitrogen
根据图2可知,反硝化菌属在氨氮驯化终浓度的活性污泥中为优势菌株,因此选择ZZC-4作为后续的主要研究菌株。
2.4 ZZC-4菌株降解氨氮能力复测
由图3可知,氨氮和COD在ZZC-4培养48 h内降解迅速,之后降解速率趋于平稳。培养144 h后,菌株ZZC-4对模拟污水中氨氮和COD的降解率达到最高,分别为90.8%和94.7%。
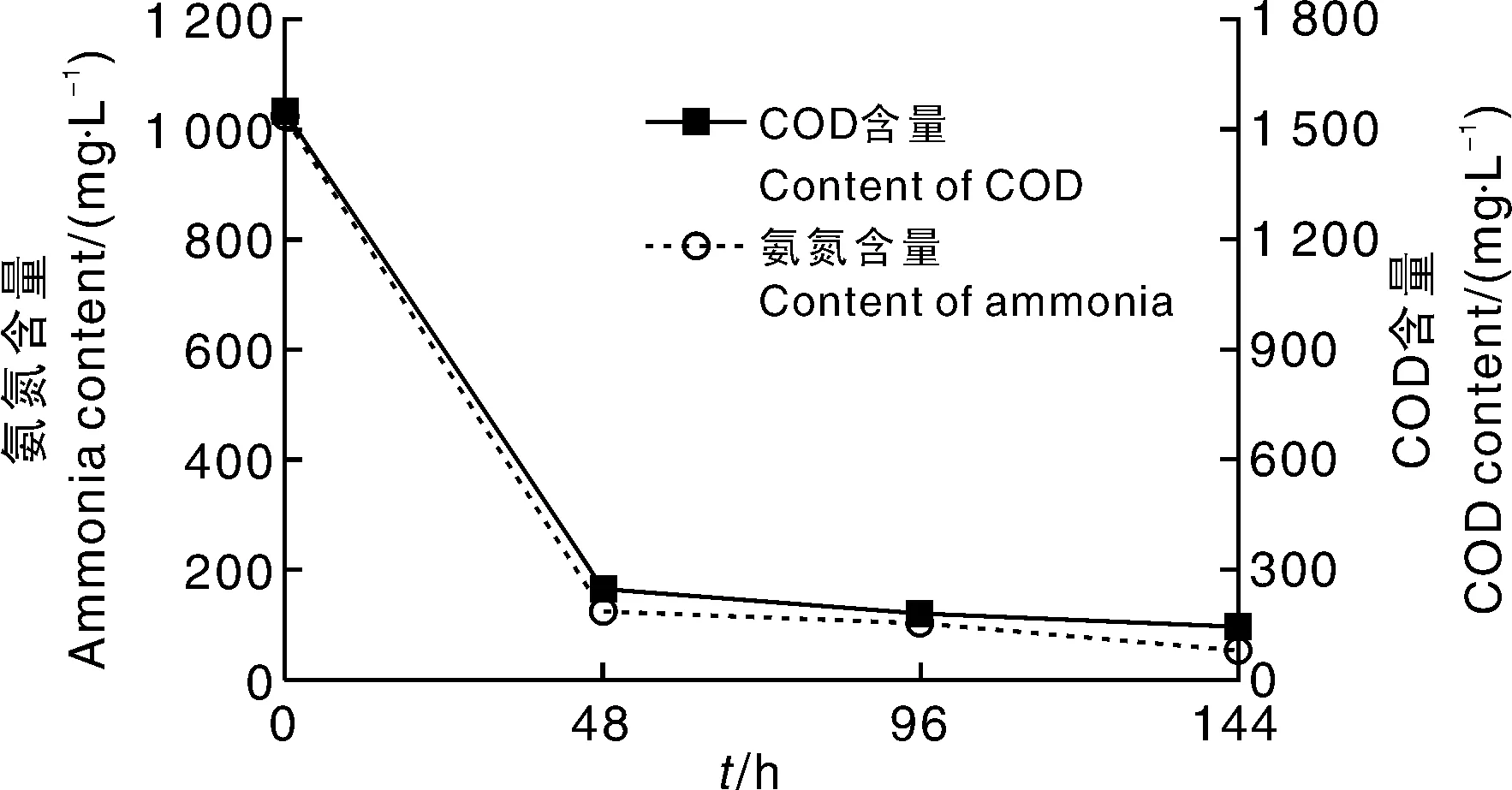
图3 ZZC-4菌株降解COD和氨氮的能力Fig.3 Degradation of ammonia nitrogen and COD by ZZC-4 strain
表2 DGGE条带测序结果
Table 2 Sequencing and analysis result of DGGE band

条带号BandNo.近亲种属与NCBI登录号ClosestrelativeandNCBIaccessionnumber相似度Identity/%1Pseudomonassp.154B7_12AR2A(KU644293.1)952UnculturedbacteriumcloneBR2-20(JQ855798.1)923Lysobactersp.FJY8(KR698371.1)964UnculturedsludgebacteriumcloneASB92(FJ947148.1)1005UnculturedbacteriumcloneI3Q1XXJ01A92AS(KP954248.1)986Denitrobactersp.BBTR53(DQ337593.1)997MicrobulbifermarinusstrainHQB734(KT758589.1)94
3 讨论
经过不同浓度氨氮污水驯化,本研究获得3株降解氨氮能力较高的菌株,其对氨氮的降解率均达到95%以上。目前,常用的筛选氨氮降解菌的方法一般有3种:传统培养法,改变培养条件法和指示剂法。胡君利等[6]比较了筛选氨氮降解菌的方法后发现,通过平板筛选往往无法得到目的菌株。改变培养条件是根据所要筛选的菌株生长特性,设计、优化一种或多种因素,使目的菌株迅速成为培养基中的优势菌株。对于氨氮降解菌来说,温度、溶氧、pH、游离氨浓度等条件的改变都有可能使氨氮降解菌成为混合培养物中的优势菌株,从而更易被分离[7]。此外,也有研究通过在培养基中添加指示剂,来推测目的菌株的存在[8]。但上述方法均存在选择性不强、工作量大的问题。本研究采取活性污泥进行梯度驯化,同时采用PCR-DGGE指纹图谱技术辅助分析驯化时活性污泥中细菌丰度的变化。试验表明,既具有较强氨氮降解能力,同时又是驯化活性污泥中的优势菌的只有反硝化菌,因而可初步选择反硝化菌为目标菌。DGGE胶图中其他割取条带所对应的可培养优势菌株还包括假单胞菌属、溶杆菌属等。根据文献报道,溶杆菌和假单胞菌在污水处理中也均可不同程度地降解氨氮[9-10]。此外,本试验中水平和垂直驯化之间的反硝化菌含量有差异。这可能是由于垂直驯化时活性污泥需不断转接,培养周期较长而导致反硝化菌优势丧失引起,需进一步研究。结果提示,PCR-DGGE技术可作为平板筛选的一种辅助手段,在处理养殖污水的活性污泥中获得功能优势菌。
本研究中,3株分离得到的氨氮降解菌经过测序后分别属于戈登氏菌属、反硝化菌属和红球菌属。目前尚无这3属细菌有氨氧化作用的报道。细菌降解氨氮通常有2条途径,即同化吸收和氨氧化作用,其中氨氧化作用又分为自养氨氧化与厌氧氨氧化。陈元芬等[11]发现,自养氨氧化菌株或菌群在富含氨氮的水体中会迅速降解氨氮并积累亚硝酸盐;喻其林等[12]发现,能进行厌氧氨氧化的菌株在培养过程中能同时利用氨氮和亚硝酸盐,从而达到降解氨氮的效果。此外,部分微生物也能够吸收环境中的氨氮来进行自身物质的合成[13]。本研究曾尝试对ZZC-4菌株的培养液进行亚硝酸盐和硝酸盐检测,但发现两者均处于极低水平,且氨氮浓度已经大幅下降,故推测该菌株可能采用同化利用氨氮的方式降解污水中的氨氮,但其具体机制需要进一步验证。
[1] KENNISH M J. Environmental threats and environmental future of estuaries[J].EnvironmentalConservation, 2002, 29(1):78-107.
[2] RABALAIS N N, TURNER R E, SCAVIA D. Beyond science into policy: Gulf of Mexico hypoxia and the Mississippi river[J].BioScience, 2002, 52:129-142.
[3] 刘茹, 宋英豪, 王焕升. 畜禽养殖污水污染治理现状及发展趋势[J]. 环境保护科学, 2012, 38(4):19-21. LIU R, SONG Y H, WANG H S. Current situation and development trend of livestock and poultry wastewater treatment[J].EnvironmentalProtectionScience, 2012, 38(4): 19-21. (in Chinese with English abstract)
[4] 杨义飞, 陈双双, 赵飞飞. SBR中活性污泥培养驯化的研究[J]. 环境科学与管理, 2011, 36(7):102-104. YANG Y F, CHEN S S, ZHAO F F. Research on cultivation of activated sludge by sequencing batch reactor (SBR)[J].EnvironmentalScienceandManagement, 2011, 36(7): 102-104. (in Chinese with English abstract)
[5] 钟成华, 李杰, 邓春光. 人工湿地废水处理中氮、磷去除机理研究[J]. 重庆建筑大学学报, 2008, 30(4):141-146. ZHONG C H, LI J, DENG C G. Advances in the study of nitrogen and phosphorus removal mechanisms in constructed wetlands[J].JournalofChongqingJianzhuUniversity, 2008, 30(4): 141-146. (in Chinese with English abstract)
[6] 胡君利, 林先贵, 褚海燕, 等. 土壤氨氧化细菌的分离方法研究[J]. 土壤, 2005, 37(5):569-571. HU J L, LIN X G, CHU H Y, et al. Isolation of soil ammonia-oxidizing bacteria[J].Soils, 2005, 37(5):569-571. (in Chinese with English abstract)
[7] 王新, 姚晓红, 吴逸飞, 等. 氨氧化细菌的筛选鉴定及其最适反应条件研究[J]. 浙江农业学报, 2015, 27(6): 1042-1047. WANG X, YAO X H, WU Y F, et al. Screening and identification of ammonia oxidizing bacteria and optimization of ammoxidation conditions[J].ActaAgriculturaeZhejiangensis, 2015, 27(6): 1042-1047. (in Chinese with English abstract)
[8] 李焕, 汪群慧, 孙晓红, 等. 高效氨氧化菌群富集、驯化及其动态变化规律分析[J]. 环境工程学报, 2012, 6(5):1548-1552. LI H, WANG Q H, SUN X H, et al. Domesticate and enrichment of high concentration of ammonia oxidation bacteria and its dynamic change analysis[J].ChineseJournalofEnvironmentalEngineering, 2012, 6(5): 1548-1552. (in Chinese with English abstract)
[9] 宋协法, 李华龙. 养殖污水中主要分解氨氮微生物的分离鉴定[J]. 渔业现代化, 2013, 40(1):1-6. SONG X F, LI H L. Isolation and identification of nitrifying bacteria from cultivation wastewater[J].FisheryModernization, 2013, 40(1): 1-6. (in Chinese with English abstract)
[10] 王丽华, 吕铮, 郝春博, 等. 某石油污染场地地下水中降解菌群落结构研究[J]. 环境科学与技术, 2013, 36(7): 1-8. WANG L H, LYU Z, HAO C B, et al. Degrading bacteria community structure in groundwater of a petroleum-contaminated site[J].EnvironmentalScience&Technology, 2013,36(7):1-8. (in Chinese with English abstract)
[11] 陈元芬, 李大平, 张杰, 等. 自养混合菌群氨氧化特征及结构分析[J]. 四川大学学报(自然科学版), 2009, 46(4):1193-1198. CHEN Y F, LI D P, ZHANG J, et al. Ammoxidation characteristics and composition analysis of autotrophic mixed bacteria[J].JournalofSichuanUniversity(NaturalScienceEdition), 2009, 46(4): 1193-1198. (in Chinese with English abstract)
[12] 喻其林, 陈旭, 黄明媛, 等. 一株异养氨氧化青霉(Penicilliumsp.)特性研究[J]. 中国农业科学, 2010, 43(5):986-992.
YU Q L, CHEN X, HUANG M Y, et al. Study on the characteristics of a strain ofPenicilliumsp. with the capacity of heterotrophic ammonium oxidation[J].ScientiaAgriculturaSinica, 2010, 43(5): 986-992. (in Chinese with English abstract)
[13] 李谷, 吴振斌, 侯燕松, 等. 养殖水体氮的生物转化及其相关微生物研究进展[J]. 中国生态农业学报, 2006, 14(1):11-15. LI G, WU Z B, HOU Y S, et al. Nitrogen biotransformation and its relative microorganism in aquaculture waters[J].ChineseJournalofEco-Agriculture, 2006, 14(1): 11-15. (in Chinese with English abstract)
(责任编辑 高 峻)
PCR-DGGE assisted selection of ammonia degrading bacteria
ZHANG Zhichao1, SUN Hong2, WU Yifei2, WANG Xin2, YAO Xiaohong2, LIU Yong2, TANG Jiangwu2,*, GE Xiangyang1,*
(1.StateKeyLaboratoryofMicrobiology,HuazhongAgriculturalUniversity,Wuhan430030,China;2.InstituteofPlantProtectionandMicrobiology,ZhejiangAcademyofAgriculturalSciences,Hangzhou310021,China)
In the present study, strains able to efficiently degrade ammonia in livestock waste water were screened with traditional enrichment selection method combined with PCR-denaturing gradient gel electrophoresis (DGGE). Twenty-two strains were screened out after domestication under different ammonia concentrations. Among them, 3 strains exhibited relatively higher ammonia degradation abilities, and were named as ZZC-3, ZZC-4 and ZZC-14, respectively, which were identified asGordoniasp.,Denitrobactersp. andRhodococcussp. by 16S rDNA sequence analysis. The PCR-DGGE results confirmed that theDenitrobactersp. had the highest concentrations in the enriched solution of waste water. Therefore, strain ZZC-4 was selected as the target strain. After incubation for 144 h, the treatment with strain ZZC-4 reached the highest degradation efficiency, which was 90.8% for ammonia nitrogen and 94.7% for COD, respectively.
PCR-DGGE; ammonia degradation;Denitrobactersp.
10.3969/j.issn.1004-1524.2017.02.15
2016-05-30
浙江省重点研发计划项目(2015C03013);浙江省农科院青年人才培养项目
张智超(1989—),男,河南驻马店人,硕士研究生,从事环境污染处理方面的研究。E-mail: 116113258@qq.com
*通信作者,葛向阳,E-mail: gxy@mail.hzau.edu.cn;汤江武,E-mail: tangjiangwu@gmail.com
X703
A
1004-1524(2017)02-0286-06
