荞麦脱落酸不敏感基因序列分析
2017-03-06梁成刚陈晴晴石桃雄陈其皎孟子烨陈庆富
汪 燕,刘 瑶,梁成刚,*,陈晴晴,石桃雄,陈其皎,孟子烨,陈庆富
(1.贵州师范大学 荞麦产业技术研究中心,贵州 贵阳550001; 2.陕西米仓山国家级自然保护区管理局,陕西 汉中 723500)
荞麦脱落酸不敏感基因序列分析
汪 燕1,刘 瑶2,梁成刚1,*,陈晴晴1,石桃雄1,陈其皎1,孟子烨1,陈庆富1
(1.贵州师范大学 荞麦产业技术研究中心,贵州 贵阳550001; 2.陕西米仓山国家级自然保护区管理局,陕西 汉中 723500)
脱落酸不敏感基因(ABA-insensitive, ABI)是调控植物逆境胁迫应答、种子脱落和休眠等的一类转录因子。生物信息学分析发现,普通荞麦ABI(CL38501)基因cDNA全长为963 bp,编码320个氨基酸,含有典型的bZIP结构域和ABI5家族保守区Ⅱ,推测该基因可能编码ABI5家族蛋白。与野生种质相比,花蕾期和开花期栽培品种ABA信号转导途径中绝大多数基因表达量上调,其中,ABI基因表达量分别上调4.42和3.66倍。对9个栽培品种和5份野生种质的ABI基因进行PCR扩增,发现14份材料的405个位点中多态位点为30个,9个栽培品种间仅含1个多态位点,5个野生种质间含有7个多态位点,说明普通荞麦ABI基因序列高度保守。栽培品种与野生种质ABI序列存在11个共同氨基酸差异位点,蛋白构象预测发现第32位点和第60位点氨基酸发生置换导致蛋白构象发生变化,表明其可能与栽培驯化过程中荞麦丧失休眠与落粒特性相关。
普通荞麦;脱落酸不敏感基因;序列分析;蛋白结构
荞麦(Fagopyrum)起源于中国西南地区,包含普通荞麦(F.esculentum)和鞑靼荞麦(F.tatricum)两个主要的栽培种[1-2]。荞麦的营养成分丰富、赖氨酸含量高、且富含黄酮类化合物,特别是芦丁,具有降血糖、降血脂和降血压等疗效[1-4]。早在1946年,Couch等就提出将荞麦作为芦丁提取的主要来源作物[5]。普通荞麦(common buckwheat),又名甜荞,是我国重要的小宗作物。在历史上,荞麦一直被用作救灾粮食作物,具有生育期短、耐逆性强等特性,尤其是在抗寒、抗旱、抗病和耐贫瘠等方面表现较好[1-3]。野甜荞(F.esculentumssp.ancestralis)与甜荞相似,且能与甜荞杂交结实,因此被认为是甜荞的祖先[1-2]。不过,野甜荞具有强烈的种子休眠特性,而且种子成熟过程中果实易脱落,这是利用野甜荞与栽培甜荞进行杂交育种所必须克服的两个表型特征[1,3]。
脱落酸是植物生长过程中的重要信号分子,对种子休眠与果实脱落具有重要调控作用[6-9]。植物脱落酸不敏感(abscisic acid insensitive,ABI)家族蛋白是调控ABA信号途径的重要转录调控因子[10-11]。通过对拟南芥等模式植物的研究发现,ABI1、ABI2属于PP2C(protein phosphatase 2C)家族成员,abi1和abi2突变体都表现出对ABA高度敏感,证实ABI1、ABI2蛋白在ABA信号转导途径中起负调控作用[12-13]。ABI3、ABI4和ABI5则属于bZIPs(basic leucine zippers)家族成员,ABI3蛋白参与调控储藏物质的积累、种子干燥性以及诱导种子休眠;ABI4通过诱导ABA途径相关基因表达,参与调控植物的生长和发育;ABI5则是ABA信号转导的关键保守靶位点,调控ABA信号转导和逆境胁迫反应,同时,ABI3还可与ABI5相互作用,激活ABA响应元件(ABRE/RY)介导的基因转录过程[14-16]。目前,尚无有关荞麦ABI基因的相关研究报道。因此,本试验通过对转录组基因测序所得的数据库进行筛选,获得荞麦ABI的Unigene序列,设计特异性引物,进行PCR扩增、测序,分析普通荞麦栽培品种与野生种质间基因序列的差异,以期为荞麦ABI基因功能研究与荞麦种子休眠、落粒以及起源与进化等相关研究提供基础。
1 材料与方法
1.1 试验材料
试验选用9个普通荞麦栽培品种与5个具有休眠性和落粒性的野生种质(贵州师范大学荞麦产业技术研究中心提供)为材料(表1)。苗期对植株进行取样,用液氮迅速冷却后储存于-80 ℃冰箱,供样品DNA的提取。花蕾期和开花期对植株花梗进行取样,用液氮迅速冷却后储存于-80 ℃冰箱,供样品RNA的提取。
1.2 转录组测序
使用多糖多酚植物总RNA提取试剂盒(北京百泰克生物技术有限公司)对花蕾期和开花期植株样品进行RNA提取。将质量检查合格的RNA样品送由百迈客生物科技有限公司进行转录组测序。RNA提取操作步骤、转录组的测序与组装参考石桃雄等[17]的方法。
1.3 PCR扩增与测序
对转录组基因测序所得到数据库进行Unigene筛选,获得荞麦ABI的Unigene序列片段。利用软件Primer5.0设计荞麦ABIUnigene的正向特异性引物5’-TGTGGGGAAACCATTGAGTAG-3’和反向特异性引物5’-CATGTTTTGCTGTGGATGATG-3’。改良CTAB法提取样品的DNA,操作步骤参考梁成刚等[18]方法。PCR反应体系体积为60 μL (包含30.0 μL 2×TaqPCR Master Mix、9.0 μL 40 ng·μL-1DNA模板、6.0 μL 5 μmol·L-1引物和9.0 μL ddH2O)。PCR扩增反应设置: 94 ℃预变性10 min;94 ℃变性60 s,55.6 ℃退火45 s,72 ℃延伸1 min,循环35次;72 ℃延伸10 min。PCR扩增结束后,将产物置于琼脂糖凝胶电泳检测后送由生工生物工程(上海)有限公司测序。
表1 试验材料
Table 1 The materials used in this study
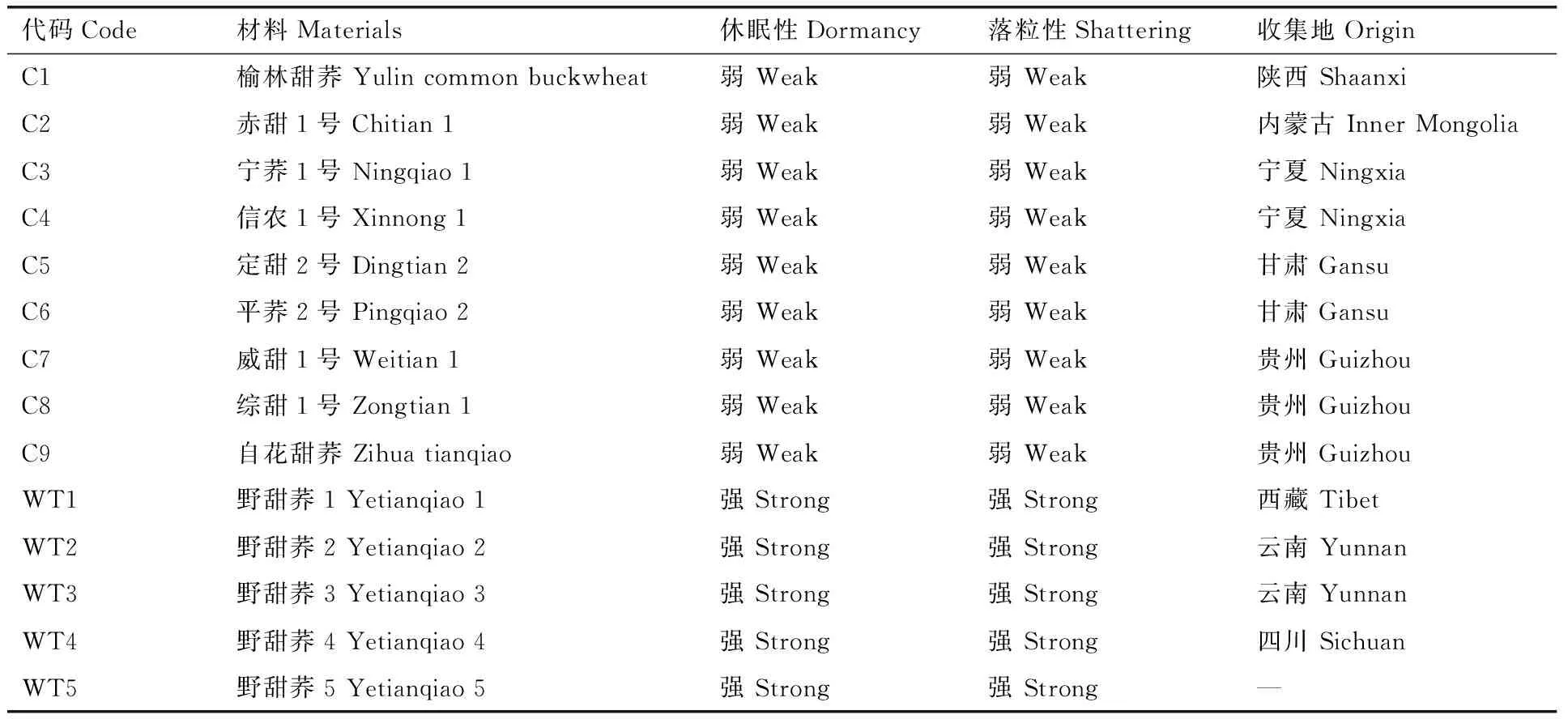
代码Code材料Materials休眠性Dormancy落粒性Shattering收集地OriginC1榆林甜荞Yulincommonbuckwheat弱Weak弱Weak陕西ShaanxiC2赤甜1号Chitian1弱Weak弱Weak内蒙古InnerMongoliaC3宁荞1号Ningqiao1弱Weak弱Weak宁夏NingxiaC4信农1号Xinnong1弱Weak弱Weak宁夏NingxiaC5定甜2号Dingtian2弱Weak弱Weak甘肃GansuC6平荞2号Pingqiao2弱Weak弱Weak甘肃GansuC7威甜1号Weitian1弱Weak弱Weak贵州GuizhouC8综甜1号Zongtian1弱Weak弱Weak贵州GuizhouC9自花甜荞Zihuatianqiao弱Weak弱Weak贵州GuizhouWT1野甜荞1Yetianqiao1强Strong强Strong西藏TibetWT2野甜荞2Yetianqiao2强Strong强Strong云南YunnanWT3野甜荞3Yetianqiao3强Strong强Strong云南YunnanWT4野甜荞4Yetianqiao4强Strong强Strong四川SichuanWT5野甜荞5Yetianqiao5强Strong强Strong—
“—”代表采集地不详。
— represents the origin is unknown.
1.4 序列分析与进化树的构建
使用网络数据库(http://blast.ncbi.nlm.nih.gov/)的Nucleotide BLAST功能进行序列的在线比对。将序列对齐分值大于100,序列同一性大于65%的基因序列定义为高度同源。使用DNAsp5软件进行序列差异分析,使用MEGA5.05软件进行ClustalW序列比对分析和聚类分析,使用网页(https://swissmodel.expasy.org/interactive)的Swiss-model功能进行蛋白结构预测。
2 结果与分析
2.1 荞麦ABI基因序列
对荞麦转录组基因测序构建的数据库进行Unigene筛选,获得荞麦ABI的Unigene序列(CL38501)。生物信息学分析发现,该基因cDNA序列全长963 bp,其中碱基A占32.2%、T占23.7%、G占25.4%、C占18.7%,编码320个氨基酸。从图1可知,cDNA编码的氨基酸具有典型的bZIP结构域。在线BLASTp比对结果发现,甜荞ABI蛋白片段序列与葡萄Vitisvinifera等数十个物种、上百个ABI家族蛋白片段序列相似度高于65%,属于高度同源。同时,从图1还可以发现,cDNA编码的氨基酸还具有ABI5家族保守区Ⅱ(CRⅡ),因此,推测该Unigene可能编码ABI5家族蛋白。
2.2 荞麦ABI基因序列的PCR扩增
提取普通荞麦9个栽培品种和5个野生种质植株的DNA,设计ABI基因的特征引物,进行PCR扩增。通过琼脂糖凝胶电泳检测发现,14个供试材料中PCR扩增得到的产物在约400~500 bp处产生特异性条带;测序结果发现,C1中产生的特异条带序列片段为442 bp(图2-A)。在荞麦转录组基因测序(登录号:SRS1350375)的数据库中对C1测序获得片段进行Nucleotide blast对比分析,结果发现该片段部分序列与荞麦ABI基因cDNA片段部分序列的相似性达到99.5%(图2-B),说明PCR扩增产物即为荞麦ABI基因片段。利用MEGA5.05软件对该片段的蛋白序列进行系统进化分析,从聚类图可知,荞麦ABI序列与葡萄Vitisvinifera、苹果Malusdomestica、梅花Prunusmume、欧洲草莓Fragariavesca、麻风树Jatrophacurcas、胡杨Populuseuphratica等ABI序列差异较小,被聚在一起,亲缘关系较近(图3)。

单下划线标记氨基酸序列为ABI5家族蛋白保守区Ⅱ;双下划线标记氨基酸序列为bZIP结构域Single underline marks for the amino acid sequence of conservative region Ⅱ of ABI5 family; Double underline marks for the amino acid sequence of bZIP domain图1 荞麦ABI Unigene的cDNA序列及其编码的氨基酸序列Fig.1 The cDNA and its coding amino acid sequence of buckwheat ABI Unigene
2.3 普通荞麦ABI基因序列差异分析
利用MEGA5.05软件中ClustalW对14个供试材料扩增得到的ABI基因片段序列进行多重比较分析,去掉序列两端缺失导致排列不整齐的位点,对其中405个位点进行多态性统计,结果发现在14个供试材料中该基因序列片段的不变位点为375个,多态位点为30个(包含简约信息位点数27个)。其中,在9个栽培品种间多态位点仅为1个,无简约信息位点;5个野生材料间多态位点为7个(包含简约信息位点数1个)。
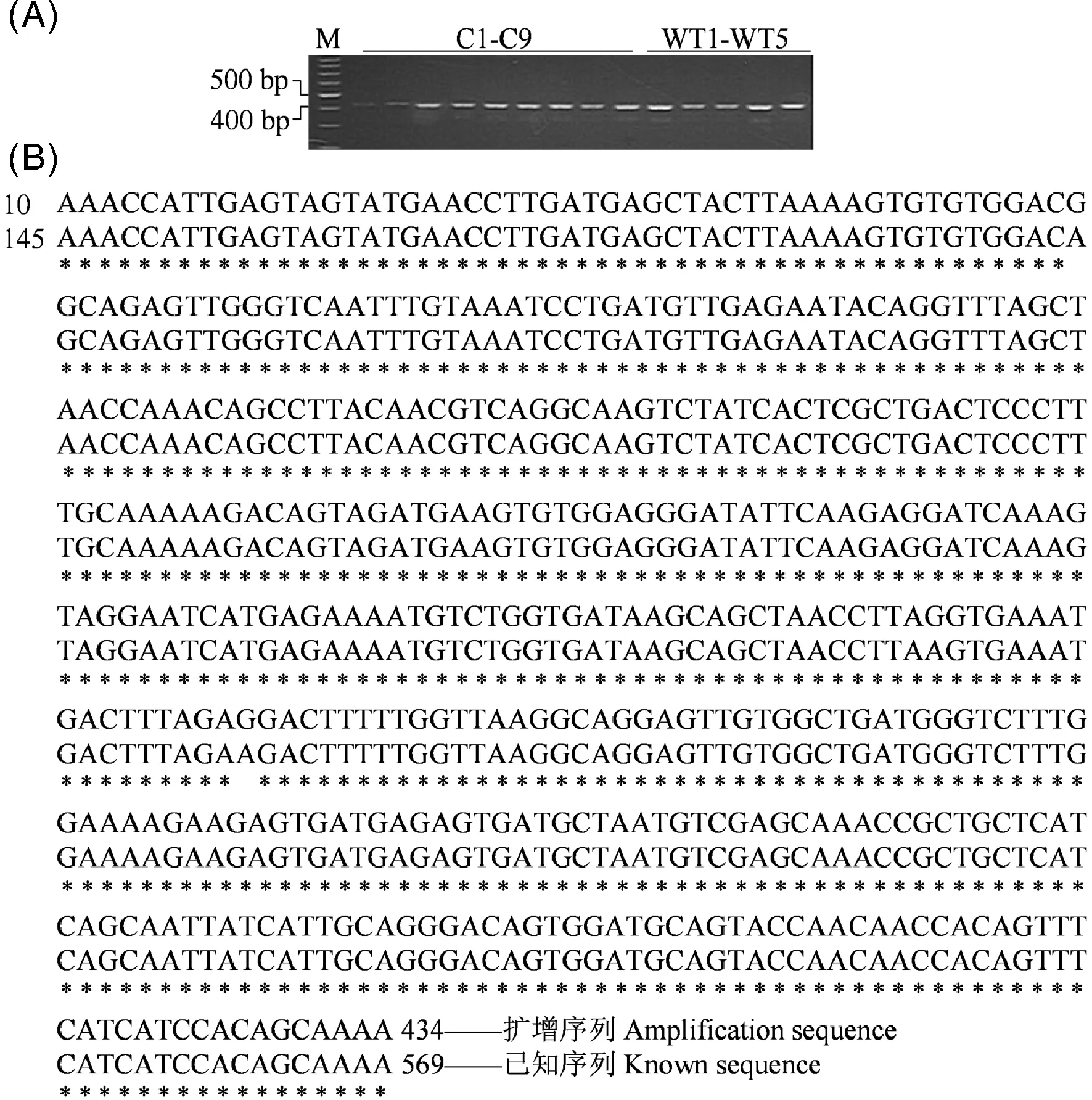
M: Marker图2 普通荞麦PCR扩增产物(A)及其序列比对分析(B)Fig.2 The PCR amplification product (A) of common buckwheat DNA and its sequence analysis (B)
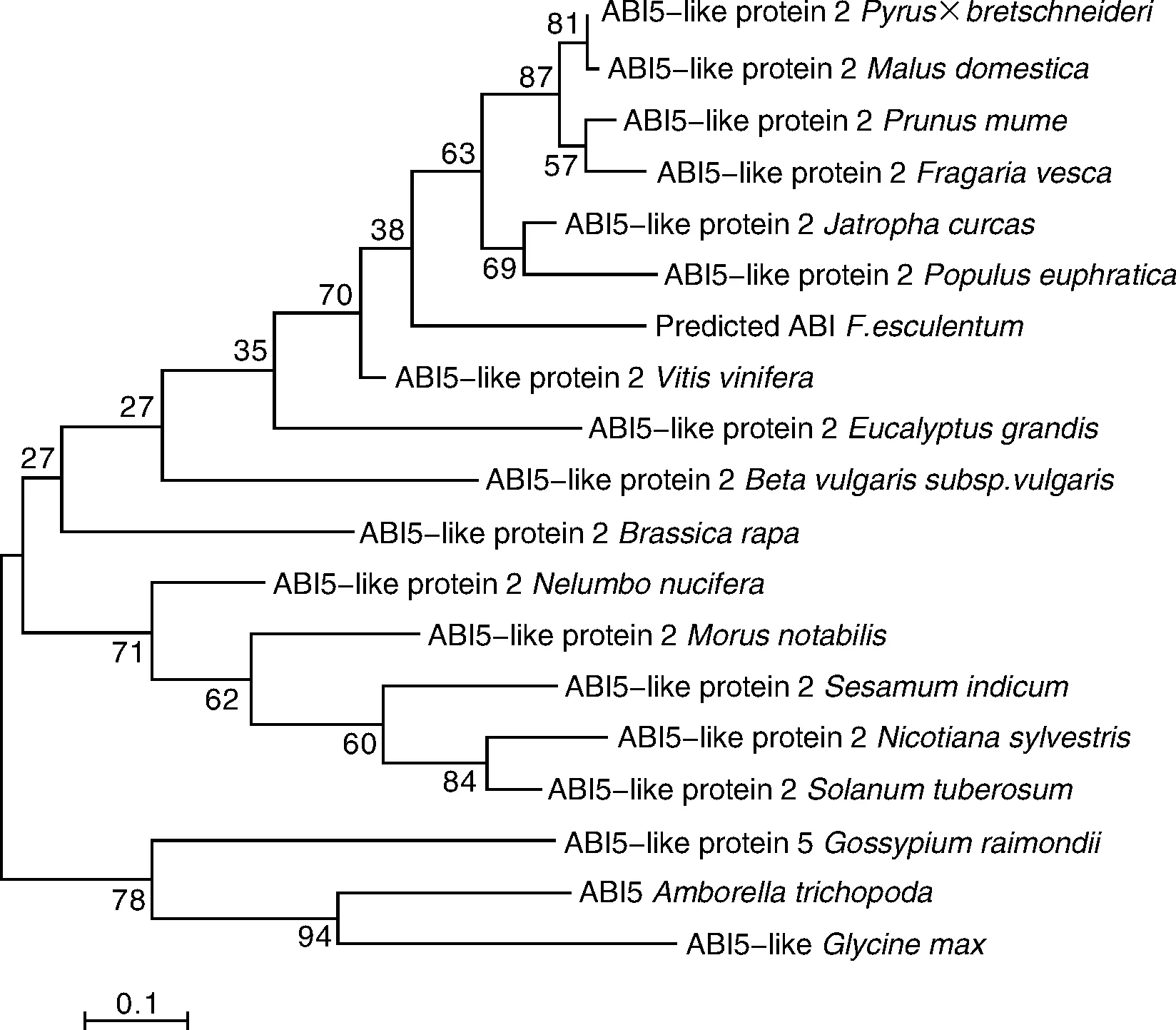
图3 不同植物ABI氨基酸序列的聚类分析Fig.3 Phylogenetic tree of ABI amino acid sequence from different plant species
2.4 普通荞麦ABI蛋白序列差异分析
利用ClustalW软件对14个供试材料扩增得到ABI片段序列开放阅读框(ORF)编码的ABI氨基酸序列进行比较分析,结果发现9个普通荞麦栽培品种的135个氨基酸位点中仅含1个差异位点,高度保守;5个野生种质含有4个差异位点,高度保守;栽培品种与野生种质间存在15个差异氨基酸位点,其中11个为共同差异位点(表2)。
表2 普通荞麦栽培品种与野生种质间脱落酸不敏感蛋白的共同氨基酸差异位点
Table 2 The differential loci of amino acid sites of ABI between cultivar and wild-type of common buckwheat
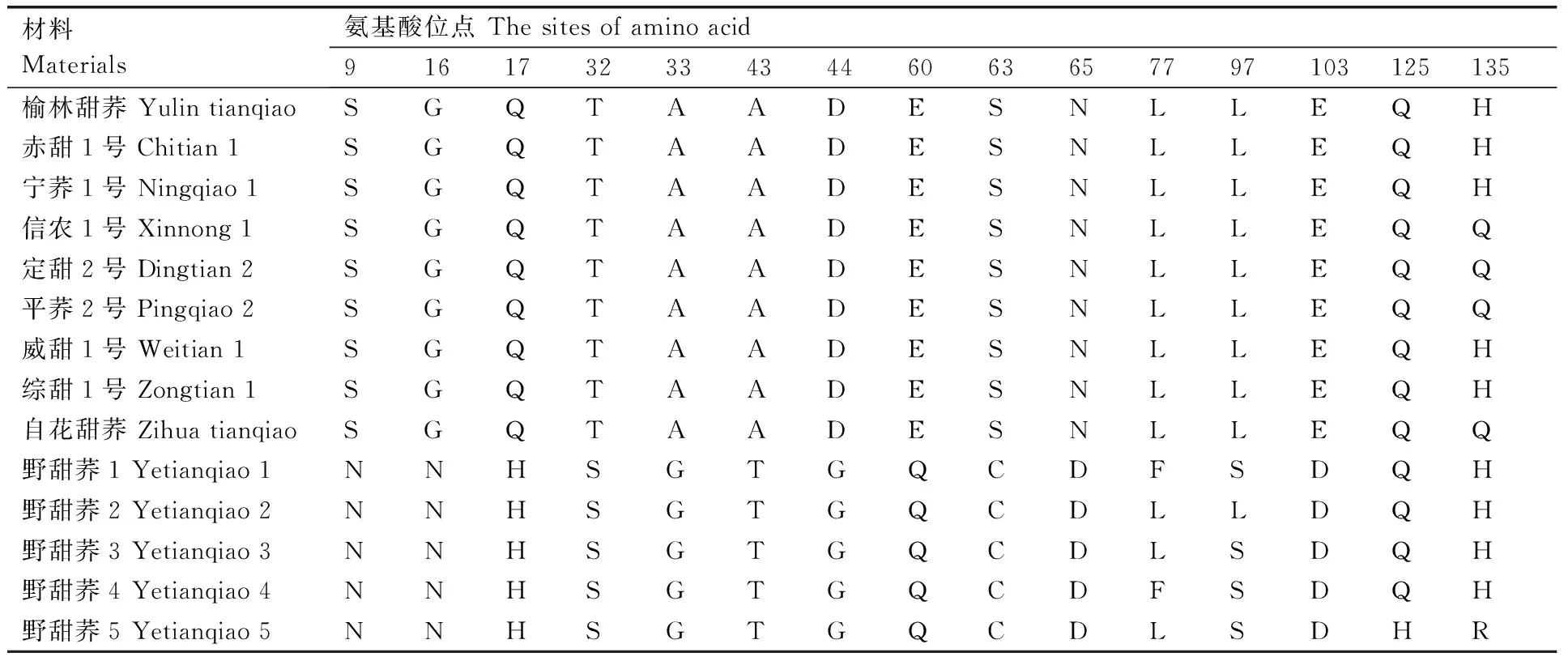
材料Materials氨基酸位点Thesitesofaminoacid91617323343446063657797103125135榆林甜荞YulintianqiaoSGQTAADESNLLEQH赤甜1号Chitian1SGQTAADESNLLEQH宁荞1号Ningqiao1SGQTAADESNLLEQH信农1号Xinnong1SGQTAADESNLLEQQ定甜2号Dingtian2SGQTAADESNLLEQQ平荞2号Pingqiao2SGQTAADESNLLEQQ威甜1号Weitian1SGQTAADESNLLEQH综甜1号Zongtian1SGQTAADESNLLEQH自花甜荞ZihuatianqiaoSGQTAADESNLLEQQ野甜荞1Yetianqiao1NNHSGTGQCDFSDQH野甜荞2Yetianqiao2NNHSGTGQCDLLDQH野甜荞3Yetianqiao3NNHSGTGQCDLSDQH野甜荞4Yetianqiao4NNHSGTGQCDFSDQH野甜荞5Yetianqiao5NNHSGTGQCDLSDHR
利用网页(https://swissmodel.expasy.org/interactive)的Swiss-model对扩增得到的ABI蛋白片段进行蛋白质结构在线预测,结果发现,栽培品种和野生种质间ORF蛋白结构存在明显的构象差异。由图4可知,栽培品种和野生种质间存在的11个氨基酸差异位点导致ORF区域蛋白的1个片段,β-片层和α-螺旋结构发生改变(图4-A、B)。进一步对11个氨基酸差异位点发生单位点置换后进行蛋白质结构预测,结果发现其中9个位点的置换对蛋白空间结构没有影响,但ORF第32位点T→S和第60位点E→Q发生转换导致蛋白结构域发生构象变化(图4-A、C、D)。
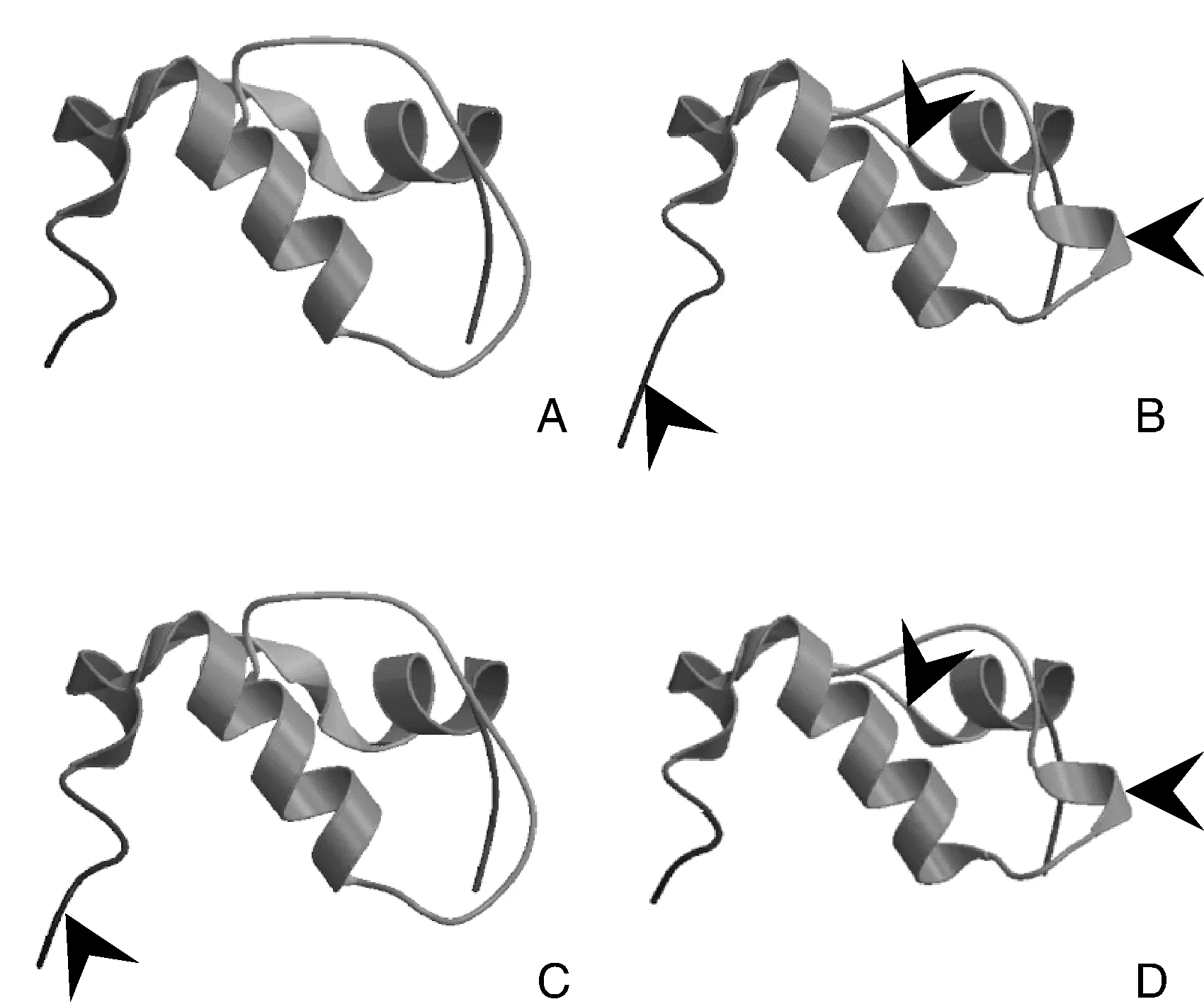
栽培品种(A)、野生种质(B)、栽培品种ABI蛋白第32位点T→S发生转换(C)和第60位点E→Q发生转换(D)的结构域;箭头指向结构与栽培品种ABI蛋白结构发生改变的区域The protein conformation of cultivar (A), wild germplasm (B), the amino acid transformation at the 32nd (C) and 60th (D) sites of ABI. Arrows point to the changed protein conformation of ABI, as compared with that of cultivar图4 普通荞麦ABI蛋白片段结构域分析Fig.4 Analysis of the domain of ABI protein fragment of common buckwheat
2.5 普通荞麦ABI基因表达差异分析
对栽培品种与野生种质的转录组基因测序结果进行分析发现,花蕾期和开花期栽培品种ABA信号途径中PYR/PYL、PP2C、SnRK2和ABF家族绝大多数基因较野生种质表达量上调(图5-A、B)。其中,花蕾期栽培品种ABI(CL38501)基因较野生种质表达量上调4.42倍,开花期较野生种质表达量上调3.66倍。
3 讨论
植物ABA信号途径在调控逆境胁迫应答、种子脱落和休眠等方面具有重要作用[9,19-20]。研究发现,ABA 能够与 PYR/PYL受体作用,通过负调控作用调节蛋白磷酸酶PP2C活性,再调节下游SnRK2蛋白激酶活性,进而调节ABF蛋白活性[21-22]。ABI属于ABF类家族蛋白,是调控ABA信号途径的重要转录调控因子。ABI家族蛋白中ABI3、ABI4和ABI5属于bZIP家族成员。本研究发现,荞麦ABI的Unigene(CL38501)
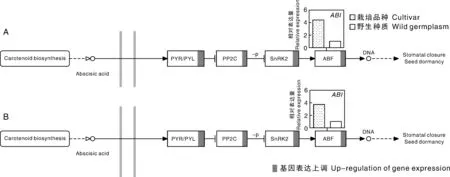
图5 花蕾期(A)和开花期(B)栽培品种与野生种质ABA信号途径中基因的相对表达图Fig.5 The relative expression map of genes involved in the ABA signal pathway of cultivar and wild-type of common buckwheat in the bud stage (A) and flowering stage (B)
序列cDNA全长为963 bp,编码320个氨基酸,含有典型的bZIP结构域和ABI5家族保守区Ⅱ(图1),因此,推测该基因可能编码ABI5家族蛋白。
苏亮等[23]对ABI5蛋白序列进行聚类分析,发现水稻OsABI5-1和OsABI5-2序列高度保守,被聚在一起,玉米ZmABI5与高粱SbABI5聚在一起,其亲缘关系近。本试验通过PCR扩增获得荞麦ABI基因片段,序列分析发现5个野生种质405个位点中含有7个多态位点,9个栽培品种仅含1个多态位点,14份材料共含有30个多态位点,说明栽培驯化过程中该基因序列高度保守。聚类分析发现,荞麦ABI序列与葡萄、苹果、梅花、草莓、麻风树、胡杨等ABI5-Like2序列差异较小,被聚在一起,推测荞麦ABI基因可能编码ABI5-Like2蛋白。
普通荞麦栽培品种是由野甜荞长期栽培驯化而来,现已不具备种子休眠与落粒等性状[1]。对拟南芥、水稻和小麦等的研究发现,ABI5对种子成熟、休眠、萌发、幼苗生长和逆境应答等方面均有重要调控作用[8,24-25]。Yan等[26]研究发现,玉米叶片ZmABI5在盐、水杨酸、脱落酸、高温和低温处理下表达量上调,在干旱和伤害处理下表达量下调;但玉米根ZmABI5在干旱、伤害、高温和低温处理下表达量上调,在盐、水杨酸和脱落酸处理下表达量下调,说明ABI5调控ABA信号途径的机制较为复杂。苏亮等[23]利用cDNA-SRAP转录组分析证实ZmABI5基因与玉米磷胁迫反应有关。花蕾期和开花期对普通荞麦栽培品种与野生种质进行转录组测序发现,栽培品种ABA信号转导途径中PYR/PYL、PP2C、SnRK2和ABF基因家族绝大多数基因表达量均上调。其中,ABI(CL38501)基因表达量较野生种质植株分别上调4.42和3.66倍。对荞麦ABI蛋白片段进行序列分析发现,14个材料间存在15个差异氨基酸位点,而栽培品种和野生种质间存在11个共同差异氨基酸位点(表2),栽培驯化对这11个差异氨基酸位点的选择具有高度一致性。蛋白结构预测发现,栽培品种和野生种质间蛋白构象存在明显差异。进一步分析发现,构象差异是由第32位点T→S和第60位点E→Q发生转换导致(图4-A、C、D)的,表明其可能与栽培驯化过程中甜荞丧失休眠与落粒特性相关。本试验结果为进一步研究荞麦ABI基因功能以及栽培进化提供了理论基础。
[1] 陈庆富. 荞麦属植物科学[M]. 北京: 科学出版社, 2012: 5-55.
[2] WEISSKOPF A, FULLER D Q. Buckwheat: Origins and development [M]// SMITH C. Encyclopedia of global archaeology. New York: Springer, 2014: 1025-1028.
[3] AHMED A, KHALID N, AHMAD A, et al. Phytochemicals and biofunctional properties of buckwheat: a review[J].TheJournalofAgriculturalScience, 2014, 152(03): 349-369.
[4] JACQUEMART A L, LEDENT J F O, QUINET M, et al. Is buckwheat (FagopyrumesculentumMoench) still a valuable crop today?[J].EuropeanJournalofPlantScienceandBiotechnology, 2012, 6(S2): 1-10.
[5] COUCH J F, NAGHSKI J, KREWSON C F. Buckwheat as a source of rutin[J].Science, 1946, 103(2668): 197-198.
[6] CHENG Z J, ZHAO X Y, SHAO X X, et al. Abscisic acid regulates early seed development inArabidopsisby ABI5-mediated transcription ofSHORTHYPOCOTYLUNDERBLUE1[J].ThePlantCell, 2014, 26(3): 1053-1068.
[7] FINKELSTEIN R R. Abscisic acid-insensitive mutations provide evidence for stage-specific signal pathways regulating expression of anArabidopsislate embryo genesis-abundant (lea) gene[J].MolecularandGeneralGenetics, 1993, 238(3): 401-408.
[8] ANTONI R, RODRIGUEZ L, GONZALEZ-GUZMAN M, et al. News on ABA transport, protein degradation, and ABFs/WRKYs in ABA signalling[J].CurrentOpinioninPlantBiology, 2011, 14(5): 547-553.
[9] KASHIWAKURA Y I, KOBAYASHI D, JIKUMARU Y, et al. Highly sprouting tolerant wheat grain exhibits extreme dormancy and cold imbibition resistant accumulation of abscisic acid[J].PlantandCellPhysiology, 2016, 57(4):715-732.
[10] KOORNNEEF M, REULING G, KARSSEN C M. The isolation and characterization of abscisic acid-insensitive mutants ofArabidopsisthaliana[J].PhysiologiaPlantarum, 1984, 61(3): 377-383.
[11] FINKELSTEIN R R, SOMERVILLE C R. Three classes of abscisic acid (ABA)-insensitive mutations ofArabidopsisdefine genes that control overlapping subsets of ABA responses[J].PlantPhysiology, 1990, 94(3): 1172-1179.
[12] MERLOT S, GOSTI F, GUERRIER D, et al. The ABI1 and ABI2 protein phosphatases 2C act in a negative feedback regulatory loop of the abscisic acid signalling pathway[J].ThePlantJournal, 2001, 25(3): 295-303.
[13] LEUNG J, MERLOT S, GIRAUDAT J. The ArabidopsisABSCISICACID-INSENSITIVE2(ABI2) andABI1 genes encode homologous protein phosphatases 2C involved in abscisic acid signal transduction[J].ThePlantCell, 1997, 9(5): 759-771.
[14] REEVES W M, LYNCH T J, MOBIN R, et al. Direct targets of the transcription factors ABA-Insensitive (ABI)4 and ABI5 reveal synergistic action by ABI4 and several bZIP ABA response factors[J].PlantMolecularBiology, 2011, 75(4-5): 347-363.
[15] FENG C, CHEN Y, WANG C, et al. Arabidopsis RAV1 transcription factor, phosphorylated by SnRK2 kinases, regulates the expression ofABI3,ABI4, andABI5 during seed germination and early seedling development[J].ThePlantJournal, 2014, 80(4): 654-668.
[16] MITTAL A, GAMPALA S S, RITCHIE G L, et al. Related to ABA-Insensitive3 (ABI3)/Viviparous1 and AtABI5 transcription factor coexpression in cotton enhances drought stress adaptation[J].PlantBiotechnologyJournal, 2014, 12(5): 578-589.
[17] 石桃雄,黎瑞源,郭菊卉,等. 基于微卫星标记普通荞麦种子序列表达标签的开发[J]. 贵州农业科学. 2014, 42(3): 1-5. SHI T X, NI R Y, GUO J H, et al. Development of SSR molecular markers based on expressed sequence tags from seeds ofFagopyrumesculentum[J].GuizhouAgriculturalSciences, 2014, 42(3): 1-5. (in Chinese with English abstract)
[18] 梁成刚,陈晴晴,石桃雄,等. 普通荞麦种内丝裂原活化蛋白激酶序列分析[J]. 华南农业大学学报. 2016, 37(4): 90-96. LIANG C G, CHEN Q Q, SHI T X, et al. Sequence analysis of mitogen-activated protein kinases of common buckwheat,Fagopyrumesculentum[J].JournalofSouthChinaAgriculturalUniversity, 2016, 37(4): 90-96. (in Chinese with English abstract)
[19] HAYASHI K, KINOSHITA T. Plant signaling: abscisic acid receptor hole-in-one [J].NatureChemicalBiology, 2014, 10(6): 414-415.
[20] GOMEZ-CADENAS A, VIVES V, ZANDALINAS S I, et al. Abscisic acid: a versatile phytohormone in plant signaling and beyond[J].CurrentProteinandPeptideScience, 2015, 16(5): 413-434.
[21] MELCHER K, NG L M, ZHOU X E, et al. A gate-latch-lock mechanism for hormone signaling by abscisic acid receptors[J].Nature, 2009, 462(7273): 602-608.
[22] RAGHAVENDRA A S, GONUGUNTA V K, CHRISTMANN A, et al. ABA perception and signaling[J].TrendsinPlantScience, 2010, 15(7): 395-401.
[23] 苏亮,白建荣,王秀红,等. 玉米低磷胁迫诱导的ZmABI5基因的克隆及表达分析[J]. 山西农业科学, 2015, 43(04): 393-396. SU L, BAI J R, WANG X H, et al. Cloning and expression analysis ofZmABI5 gene induced by low phosphate stress in Maize[J].JournalofShanxiAgriculturalSciences, 2015, 43(04): 393-396. (in Chinese with English abstract)
[24] LOPEZ MOLINA L, MONGRAND S, MCLACHLIN D T, et al. ABI5 acts downstream of ABI3 to execute an ABA-dependent growth arrest during germination[J].ThePlantJournal, 2002, 32(3): 317-328.
[25] CARLES C, BIES ETHEVE N, ASPART L, et al. Regulation ofArabidopsisthalianaEmgenes: role of ABI5[J].ThePlantJournal, 2002, 30(3): 373-383.
[26] YAN F, DENG W, WANG X, et al. Maize (ZeamaysL.) homologue ofABA-insensitive(ABI)5 gene plays a negative regulatory role in abiotic stresses response[J].PlantGrowthRegulation, 2012, 68(3): 383-393.
(责任编辑 张 韵)
Sequence analysis of ABA-insensitive gene of common buckwheat,Fagopyrumesculentum
WANG Yan1, LIU Yao2, LIANG Chenggang1,*, CHEN Qingqing1, SHI Taoxiong1, CHEN Qijiao1, MENG Ziye1, CHEN Qingfu1
(1.ResearchCenterofBuckwheatIndustryTechnology,GuizhouNormalUniversity,Guiyang550001,China; 2.ShaanxiMicangshanNationalNatureReserve,Hanzhong723500,China)
ABA-insensitive (ABI) is a kind of transcription factors that regulates plant stress response, seed shattering and dormancy. Bioinformatic analysis indicated that the cDNA sequence of common buckwheatABI(CL38501) gene contained 963 bp, coded 320 amino acids including the bZIP domain and the conserved domain Ⅱ of ABI5, suggesting that it might encode the ABI5 family protein. The major genes involve in ABA signal transduction pathway were up-regulated in cultivar includingABIthat was up-regulated by 4.42-fold and 3.66-fold at bud stage and flowering stage, respectively, as compared with the wild germplasm of common buckwheat. A total of 405 sites inABIgene including 30 polymorphic sites were identified among 9 cultivars and 5 wild germplasms of common buckwheat. Only 1 polymorphic site was identified among 9 cultivars, whereas 7 polymorphic sites were identified in 5 wild germplasms. These results indicated that theABIsequence of common buckwheat was highly conserved. A total of 11 consistent polymorphic amino acid sites were identified among cultivar and wild germplasm. The change of protein conformation was found between cultivar and wild germplasm of common buckwheat induced by the amino acid transformation at the 32nd and 60th sites of ABI, implying that it might be associated with the loss of seed shattering and dormancy during the long domestication process.
common buckwheat; ABA-insensitive gene; sequence analysis; protein structure
10.3969/j.issn.1004-1524.2017.02.02
2016-07-29
贵州省科技厅合作计划项目(黔科合LH字[2015]7770号);贵州师范大学资助博士科研项目(0516029);贵州省“三区”人才工作专项经费(0515075);国家自然科学基金面上项目(31471562);国家燕麦荞麦现代农业产业技术体系专项资金(CARS-08-A4);贵州省荞麦工程技术研究中心项目(黔科合农G字[2015]4003号)
汪燕(1985—),女,重庆人,博士,助理研究员,从事作物遗传育种与分子机制等相关研究。E-mail: yanwanguf@163.com
*通信作者,梁成刚,E-mail: jesselcg@163.com
S517;Q941+.2
A
1004-1524(2017)02-0186-07
