牛BBOX1基因3′UTR变异体的克隆及其多态性分析
2017-03-01宋雨霏辛友志崔建伟周国利
宋雨霏,郭 彦,辛友志,崔建伟,周国利
(聊城大学 生命科学学院,山东 聊城 252059)
牛BBOX1基因3′UTR变异体的克隆及其多态性分析
宋雨霏,郭 彦,辛友志,崔建伟,周国利*
(聊城大学 生命科学学院,山东 聊城 252059)
为鉴定牛BBOX1基因选择性多聚腺苷酸化的不同变异体及其3′UTR的多态性,采用3′RACE的方法检测出BBOX1基因3个不同3′UTR全长的APA变异体,短APA变异体3′ UTR长度为229 bp,长APA变异体3′UTR长度分别为664 bp和678 bp。利用SSCP与测序相结合的方法,在BBOX1基因的3′UTR中检测到10个多态性,其中7个为SNP,分别为c.12T>C、c.100A>G、c.241T>C、c.480T>C、c.557T>C、c.606T>C和c.650T>C;3个为插入或缺失突变,分别为c.28_29insC、c.434_435delGT和c.512_513insTGC。SNP c.650T>C定位在Poly(A)加尾信号PAS3中,使加尾信号序列AAUAAA突变成AACAAA,导致3′ UTR长度为678 bp的变异体出现。
牛;BBOX1基因; 选择性多聚腺苷酸化; 多态性; 3′ UTR
左旋肉碱是体内脂肪代谢必需的内在物质,其具有促进脂肪酸的β-氧化、降低血清胆固醇及甘油三酯的含量、提高机体耐受力等重要生理功能[1]。还可以和线粒体内的短链酰基(乙酰、丙酰、支链酰等)结合,形成酰-肉碱排出细胞外,从而起到调节线粒体内酰基CoA与CoA-SH的比例的作用,并为细胞质中脂肪酸合成提供乙酰基原料[2]。因此,左旋肉碱对动物的生长发育、繁殖、健康具有重要的作用[3]。左旋肉碱主要是在5种酶的作用下,以赖氨酸和蛋氨酸为原料合成[4]。在左旋肉碱的生物合成途径中,最后一步反应由γ-丁酰甜菜碱羟化酶(gamma-butyrobetaine hydroxylase 1,BBOX1)催化完成[5]。研究报道,不同饮食可以改变BBOX1基因的表达水平和酶的活性,从而影响肉毒碱的利用。增加脂肪酸利用将会相应地提高左旋肉碱合成相关酶的含量及左旋肉碱的水平。禁食时机体将优先利用脂肪酸作为能量来源,所以会引起BBOX1基因 mRNA水平、BBOX1酶活性和左旋肉碱含量的上升[6]。
人和鼠的BBOX1基因的cDNA翻译区已被克隆,在这2个物种中,开放阅读框(ORF)长度为1 161 bp,编码387个氨基酸[7-8]。通过3′RACE方法,已经鉴定出人、小鼠的BBOX1基因存在不同长度的3′UTR选择性多聚腺苷酸化(alternative polyadenylation,APA)变异体[9-10]。小鼠高脂肪饲喂后,BBOX1基因的近端多聚腺苷酸加尾信号的变异体的mRNA表达量增加2.1倍,从而导致BBOX1活性和左旋肉碱产量的提高[10]。目前,关于牛的BBOX1基因的APA变异体及该基因遗传变异的研究尚未见报道。为此,鉴定了牛BBOX1基因的APA变异体,并检测该基因3′UTR的多态性,以期为该基因在牛肉质性状及脂肪沉积中的功能研究奠定分子生物学基础。
1 材料和方法
1.1 供试动物
供试鲁西黄牛和草原红牛分别来自山东省聊城市张炉集镇牛场和吉林省农业科学院实验牛场。
1.2 主要试剂
SMARTTMRACE cDNA 扩增试剂盒和Advantage®2 PCR 试剂盒购自宝生物工程(大连)有限公司;pGM Simple T Fast克隆试剂盒购自天根生化科技(北京)有限公司;动物组织基因组DNA提取试剂盒、琼脂糖凝胶DNA回收试剂盒、Trizol、TaqDNA 聚合酶、dNTPs、EDTA、SDS、氯仿、去离子甲酰胺、丙烯酰胺、甲叉双丙烯酰胺、琼脂糖均购自生工生物工程(上海)股份有限公司;其他常规试剂均为分析纯。
1.3 组织样品采集、DNA及总RNA的提取
鲁西黄牛和草原红牛各30头,用打耳器采集耳组织样品,将其放入75%乙醇中带回实验室,-20 ℃保存。用动物组织基因组DNA抽取试剂盒提取耳组织的基因组DNA,溶于TE缓冲液中,-20 ℃保存。在山东省聊城市张炉集镇屠宰厂,采集3头鲁西黄牛的肝脏组织,屠宰后20 min内采集肝脏组织并迅速投到液氮中带回实验室,利用Trizol试剂提取总RNA,用1%的琼脂糖凝胶电泳检测总RNA的完整性。
1.4 3′ RACE鉴定BBOX1基因的APA变异体
根据NCBI中电子克隆的牛BBOX1基因(NM_001101881.2)的mRNA序列,利用Oligo 7.0软件设计1条3′RACE基因特异性引物,用于扩增BBOX1基因的不同长度的APA剪切变异体。引物序列为5′-GGCTTATGCTGACTGGGATGTG-3′。利用方法1.3中提取的肝脏组织的总RNA为材料进行3′RACE,操作按试剂盒说明书进行。3′ RACE的产物用2%的琼脂糖凝胶电泳检测,然后用凝胶DNA回收试剂盒纯化回收,将回收的产物进行TA克隆,获得的阳性克隆由生工生物工程(上海)股份有限公司进行双向测序,测序结果采用DNAStar 5.0进行序列分析。
1.5 牛BBOX1基因3′ UTR的多态性检测
根据NCBI中牛BBOX1基因(NM_001101881.2)的mRNA序列设计2对引物,用于扩增BBOX1基因3′UTR区域。F1为5′-ATGCTGACTGGGATGTGG-3′,R1为5′-GGGCAAAAGAGAGTTCAGGAT-3′;F2为5′-GAAATGAATCCGCCACAGGTAT-3′,R2为5′-TTGGTGAGGGCTGGAAAT-3′。PCR 扩增反应体系为25 μL,包括50 ng基因组DNA,10 pmol/L 的上、下游引物,200 μmol/L 的dNTPs,1.5 mmol/L MgCl2,1.5 UTaq酶。样品经94 ℃变性3 min后,按下列程序进行扩增:94 ℃变性30 s,59 ℃退火30 s,最后72 ℃延伸30 s,33个循环,最后72 ℃延伸7 min。PCR产物用1%琼脂糖凝胶电泳检测。
利用SSCP的方法鉴定BBOX1基因3′UTR区的遗传变异。取2 μL PCR 产物和5 μL上样缓冲液[98%甲酰胺、0.025%溴酚蓝、0.025%二甲苯氰、10 mmol/L EDTA(pH值8.0)、10%甘油]置于PCR 管中,离心混匀,98 ℃变性10 min,迅速插入冰中,放置10 min,使之保持变性状态。样品用12%非变性聚丙烯酰胺凝胶电泳进行检测。电泳结束后,进行银染显带。用凝胶成像系统拍照,进行基因型分析并记录结果。然后分别选择2个不同电泳条带的纯合基因型个体的PCR产物进行克隆测序,用NCBI 中的BLAST程序及DNAStar软件比较它们之间的核苷酸变异。应用RegRNA 2.0程序(http://regrna2.mbc.nctu.edu.tw/)预测BBOX1基因3′UTR区的miRNA结合位点。
2 结果与分析
2.1 牛BBOX1基因APA变异体
牛BBOX1基因3′ RACE的产物通过2%的琼脂糖凝胶电泳检测,获得了条带清晰、特异性好的2条带,片段长度与预期的相符(图1)。进一步将大小不同的2个3′ RACE产物进行克隆测序验证,结果表明,3′ RACE产物是BBOX1基因3′ UTR的不同APA变异体,且发现了3个不同长度的APA变异体(图2)。通过分析发现,变异体Ⅰ利用的是近端加尾信号PAS1,而变异体Ⅱ和Ⅲ分别利用远端加尾信号PAS3和PAS4。加尾信号PAS2功能未知,需进一步研究。以加poly(A)位点为终点,短的APA变异体3′ UTR长度为229 bp(变异体Ⅰ),加尾信号与其加尾位点之间为15 bp,较长的APA变异体3′ UTR长度为664 bp(变异体Ⅱ)和678 bp(变异体Ⅲ),加尾信号PAS3、PAS4与各自的加尾位点之间的距离分别是11 bp和7 bp。而且,PAS3中的碱基T突变为碱基C(c.650T>C),导致变异体Ⅲ的出现。
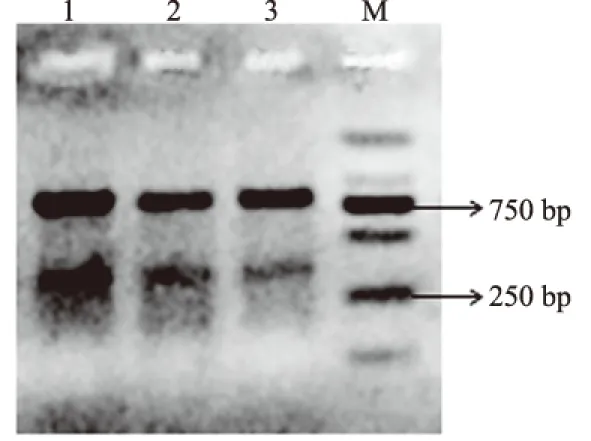
M.DL2000 DNA Marker; 1—3.3′ RACE扩增产物图1 鲁西黄牛BBOX1基因3′ RACE产物的琼脂糖凝胶电泳结果

带有双下划线的“TGA”为终止密码子,终止密码子后的第1个碱基记为“+1”;矩形方框内的“AATAAA”为多聚腺苷酸加尾信号,依次为PAS1、PAS2、PAS3和PAS4;带单下划线的碱基位置为pol(A)加尾位点,并代表不同的剪切变异体,依次为变异体Ⅰ、Ⅱ、Ⅲ图2 牛BBOX1基因不同多聚腺苷酸化位点的3′ UTR序列
2.2 牛BBOX1基因3′UTR的SNPs鉴定
在鲁西黄牛和草原红牛的研究群体中,通过SSCP与测序相结合的方法,在BBOX1基因3′UTR中共检测到10个多态性(表1),其中7个为SNPs,分别为c.12T>C、c.100A>G、c.241T>C、c.480T>C、c.557T>C、c.606T>C和c.650T>C;3个为插入或缺失突变,分别为c.28_29insC、c.434_435delGT和c.512_513insTGC。SNP c.650T>C落在Poly(A)加尾信号PAS3中,使加尾信号序列AAUAAA突变成AACAAA。这些多态性都能造成某种限制性核酸内切酶酶切位点缺失。对BBOX1基因3′UTR区的miRNA结合位点进行预测发现,bta-miR-2451、bta-miR-1343和bta-miR-2454分别定位在c.12T>C、c.512_513insTGC、c.557T>C突变区域内。
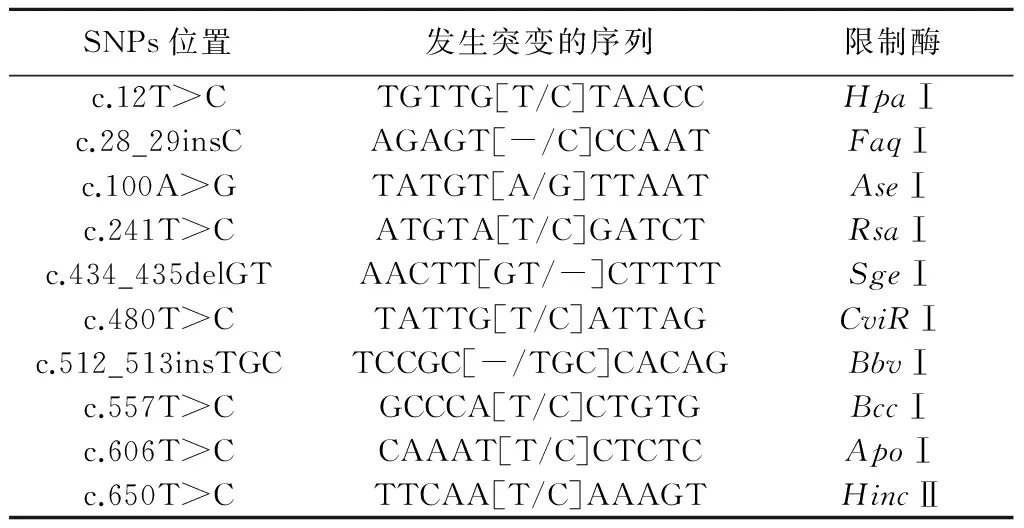
表1 牛BBOX1基因3′ UTR区的遗传变异
注:SNPs位置以mRNA序列(NM_001101881.2)为参考序列,以终止密码子下游的第1个碱基定为“1”。
3 结论与讨论
多聚腺苷酸化位点的选择决定着mRNA的3′UTR的长度,3′UTR调控序列的呈现或缺失最终可能会影响蛋白质表达水平。mRNA 3′UTR的不同长度可能会导致mRNA不同的稳定性或翻译能力。更长的3′UTR包含更多的与miRNA和/或RNA结合蛋白等结合的顺式作用元件,从而影响mRNA的稳定性、定位和翻译[11-12]。研究表明,大约有54%的人基因和52%的小鼠基因存在APA[13-14]。本研究鉴定了3个不同长度的3′ UTR,每个多聚腺苷酸化位点前都包含至少1个加尾信号(AATAAA),但在加尾过程中具体使用的加尾信号需进一步研究确定。一般情况下,加尾信号位于切割位点上游10~30 bp。由此推测,这些加尾信号可能都起到一定的作用。许多加尾信号下游的切割位点是不精确的,且常包含着多个切割位点。这样的情况在22%的啮齿动物转录本中也有发现,而且在最近的研究中发现,大约47%的鼠科动物基因中存在这种异质性[15]。目前,在人类中已经发现有2个不同的3′ UTR变异体,1个长的和1个短的3′ UTR变异体,每个加尾信号后的切割位点也不精确,存在多个切割位点[9]。在小鼠中鉴定出5个不同长度的3′UTR变异体,4个较短的3′ UTR变异体,1个长的3′UTR变异体,其中最短的3′UTR变异体包含2个加尾切割位点[10]。本研究通过3′ RACE试验,在牛中鉴定了3个不同长度的3′ UTR变异体,1个短的和2个长的3′UTR变异体,都与人类的相似性较高,而与小鼠的差别较大。
SNPs是指在基因组上单个核苷酸的变异,包括转换、颠换、缺失和插入,但通常所说的SNPs并不包括后2种情况。SNPs形成的遗传标记,其数量很多,多态性丰富。SNPs 是指变异频率大于1%的单核苷酸变异,在人类基因组中大概每1 000个碱基就有1个SNP。因此,SNPs成为第3代遗传标志,人体许多表型差异、对药物或疾病的易感性等都可能与SNPs有关[16-18]。本研究在牛BBOX1基因的3′UTR中检测到了10个多态性,如此高的多态性数量可能与3′UTR区变异程度较高有关。其中,c.650T>C定位在Poly(A)加尾信号中,使加尾信号序列AAUAAA突变成AACAAA,推测该SNP可能对APA的形成及基因的表达有影响。另外,bta-miR-2451、bta-miR-1343和bta-miR-2454分别位于c.12T>C、c.512_513insTGC、c.557T>C突变区域内,推测这些突变位点可能是成因性突变,对基因的表达调控及基因的功能有重要影响。通过对牛BBOX1基因的APA变异体的鉴定及3′UTR的SNPs检测,可以为牛BBOX1基因的功能研究奠定基础,也为牛肉质及脂肪沉积的研究提供了分子生物学素材。
[1] Le Borgne F,Ben Mohamed A,Logerot M,etal.Changes in carnitine octanoyltransferase activity inducealteration in fatty acid metabolism[J].Biochemical and Biophysical Research Communications,2011,409(4):699-704.
[2] Ramsay R R.The role of the carnitine system in peroxisomal fatty acid oxidation[J].American Journal of the Medical Sciences,1999,318(1):28-35.
[3] Vaz F M,Wanders R J.Carnitine biosynthesis in mammals[J].Biochemical Journal,2002,361(Pt 3):417-429.
[4] Tanphaichitr V,Broquist H P.Site of carnitine biosynthesis in rat[J].Journal of Nutrition,1974,104(12):1669-1673.
[5] Hausinger R P.Fe(II)/alpha-ketoglutarate-dependent hydroxylases and related enzymes[J].Critical Reviews in Biochemistry and Molecular Biology,2004,39(1):21-68.
[6] Vlies N V,Ferdinandusse S,Turkenburg M,etal.PPAR alpha-activation results in enhanced carnitine biosynthesis and OCTN2-mediated hepatic carnitine accumulation[J].Biochimica Et Biophysic Acta,2007,1767(9):1134-1142.
[7] Vaz F M,VanGool S,Ofman R,etal.Carnitine biosynthesis: Identification of the cDNA encoding human gamma-butyrobetaine hydroxylase[J].Biochemical and Biophysical Research Communications,1998,250(2):506-510.
[8] Galland S,Le Borgne F,Bouchard F,etal.Molecular cloning and characterization of the cDNA encoding the rat liver gamma-butyrobetaine hydroxylase[J].Biochimica Et Biophysica Acta,1999,1441(1):85-92.
[9] Rigault C,Le Borgne F,Demarquoy J.Genomic structure,alternative maturation and tissue expression of the humanBBOX1 gene[J].Biochimica Et Biophysica Acta,2006,1761(12):1469-1481.
[10] Rigault C,Le Borgne F,Tazir B,etal.A high-fat diet increases L-carnitine synthesis through a differential maturation of theBbox1 mRNAs[J].Biochimica Et Biophysica Acta,2013,1831(2):370-377.
[11] Akman H B,Erson-Bensan A E.Alternative polyadenylation and its impact on cellular processes[J].MicroRNA,2014,3(1):2-9.
[12] Sandberg R,Neilson J R,Sarma A,etal.Proliferating cells express mRNAs with shortened 3′ untranslated regions and fewer microRNA target sites[J].Science,2008,320:1643-1647.
[13] Shepard P J,Choi E A,Lu J,etal.Complex and dynamic landscape of RNA polyadenylation revealed by PAS-Seq[J].RNA,2011,17(4):761-772.
[14] Tian B,Hu J,Zhang H B,etal.A large-scale analysis of mRNA polyadenylation of human and mouse genes[J].Nucleic Acids Research,2005,33(1):201-212.
[15] Mandel C R,Kaneko S,Zhang H,etal.Polyadenylation factor CPSF-73 is the pre-mRNA 3′-end-processing endonuclease[J].Nature,2006,444:953-956.
[16] Allais S,Journaux L,Levéziel H,etal.Effects of polymorphisms in the calpastatin and μ-calpain genes on meat tenderness in 3 French beef breeds[J].Journal of Animal Science,2011,89(1):1-11.
[17] Capon F,Allen M H,Ameen M,etal.A synonymous SNP of the corneodesmosin gene leads to increased mRNA stability and demonstrates association with psoriasis across diverse ethnic groups[J].Human Molecular Genetics,2004,13(20):2361-2368.
[18] Kimchi-Sarfaty C,Oh J M,Kim I W,etal.A “silen” polymorphism in theMDR1 gene changes substrate specificity[J].Science,2007,315(5811):525-528.
Cloning of 3′UTR Isoforms and Polymorphisms in Bovine BBOX1 Gene
SONG Yufei,GUO Yan,XIN Youzhi,CUI Jianwei,ZHOU Guoli*
(College of Life Science,Liaocheng University,Liaocheng 252059,China)
The objective of this study was to identify isoforms of 3′UTR and polymorphisms in 3′UTR regions of bovineBBOX1 gene.Three different alternative polyadenylation(APA) isoforms of 3′UTR in theBBOX1 gene were identified by 3′RACE.Their lengths were 229,664,678 bp,respectively.Ten polymorphisms loci were identified in 3′UTR ofBBOX1 gene by single strand conformation polymorphism(SSCP) and sequencing.There were seven SNPs,including c.12T>C,c.100A>G,c.241T>C,c.480T>C,c.557T>C,c.606T>C,and c.650T>C.Three were deletion/insertion mutations,including c.28_29insC,c.434_435delGT,and c.512_513insTGC.SNP c.650T>C was located in polyadenylation signals sequence PAS3.Moreover,the mutation changed AAUAAA into AACAAA,resulting in presence of 678 bp isform of 3′UTR in bovineBBOX1 gene.
cattle;BBOX1 gene; APA; polymorphisms; 3′UTR
2016-08-20
国家自然科学基金项目(31571274);山东省自然科学基金项目(ZR2015CM025);聊城大学博士启动基金项目(31805)
宋雨霏(1993-),女,山东菏泽人,在读硕士研究生,研究方向:生物化学与分子生物学。 E-mail:448953745@qq.com
*通讯作者:周国利(1975-),男,内蒙古赤峰人,副教授,博士,主要从事动物遗传与功能基因遗传调控研究。 E-mail:glzhou1975@163.com
S813.2
A
1004-3268(2017)02-0120-04
