雌蕊缺失茶树花3个发育期的数字基因表达谱分析
2017-02-22李梅陈林波田易萍夏丽飞宋维希梁名志江昌俊
李梅,陈林波,田易萍,夏丽飞,宋维希,梁名志,江昌俊
雌蕊缺失茶树花3个发育期的数字基因表达谱分析
李梅1,陈林波1,田易萍1,夏丽飞1,宋维希1,梁名志1,江昌俊2*
1. 云南省农业科学院茶叶研究所云南省茶树种质资源创新与配套栽培技术工程研究中心,云南勐海 666201; 2. 安徽农业大学茶树生物学与资源利用国家重点实验室,安徽合肥 230036
以雌蕊缺失茶树花为试材,利用转录组、数字基因表达谱技术,研究了雌蕊缺失茶树花的花芽、花蕾、花3个时期相关基因的表达规律。结果发现,雌蕊缺失茶树花生长发育过程中进行着各种旺盛的生物合成和代谢活动。生长素信号转导途径的6个基因和ABCDE类识别基因的A类、C类和E类可能与茶树花的雌蕊缺失和雄蕊发育密切相关,并且基因调控过程较复杂;基因中和下调可能调控了C类和E类基因,从而导致雌蕊的缺失;KNOX家族中,未检测到KNOXⅡ的同源基因,KNOX I类同源基因下调表达和缺失可能减弱了茶树花雌蕊心皮的启动和雌蕊边缘组织的生长。可以看出,本研究通过数字基因表达谱分析,初步了解了雌蕊缺失茶树花发育前后的网络途径,为后期对茶树花雌蕊缺失和雄蕊发育的研究,探明茶树花不育和性别决定基因的分子机制提供理论依据。
茶树花;雌蕊缺失;数字基因表达谱;转录组;花器官调控
茶树是重要的叶用经济作物,茶树栽培的目的是收获其营养器官——芽叶。因此,茶树枝叶生长繁茂是茶园高产、稳产的前提。茶树的生长过程中包括营养生长和生殖生长,其中生殖生长期较长,从当年的花芽分化和花器官发育,到下一年的茶果生长成熟,要经历1年半的时间,在此期间将消耗大量的营养物质,从而影响了茶叶的营养生长[1]。花的发育过程中,花芽的形成意味着生殖生长的开始。植物雌蕊发育正常与否是影响植物生殖生长的重要因素,因此,深入研究雌蕊缺失的分子遗传学,对于揭示植物花器官性别分化、发育的分子机理,以及在生产实践中利用雌性不育来提高产量具有重要的理论和实践意义。
近年来,以雌性不育突变体植株为材料,应用遗传学方法与分子生物学技术来研究雌配子体及胚珠发育的调控机制取得了重要进展。研究大都以拟南芥、金鱼草、矮牵牛、油松、大豆、甘蓝型油菜、小麦和水稻等模式植物为材料,但以雌蕊缺失为材料的研究相对甚少。吴剑锋等[2]基于转录组水平上分析芜菁雌蕊退化突变和其野生型植株的开放花,获得了152个差异表达基因,其中9个显著差异基因不但在雌蕊发育等生殖发育过程起重要作用,还参与了植物营养生长的生理生化过程。萝卜中,败育花蕾与正常花蕾相比有221条差异表达的转录片段,在败育的萝卜花蕾中发现液泡加工酶(Vacuolar processing enzyme,)表达量较高,研究者进而在拟南芥中高表达,发现拟南芥花芽败育[3-4]。拟南芥中,生长素响应因子(Auxin response factor,)的一个调控因子突变导致雌蕊的背腹结构混乱[5]。研究()家族对雌蕊发育影响,发现突变导致第4轮花器官缺失,另外,的一个负调控因子的缺失产生更多的花器官,主要是花萼和花瓣,也有额外的心皮和雄蕊[6]。在茶树中,宋维希等[7]初步分析了雌蕊缺失茶树的农艺性状与品质特征。但雌蕊缺失茶树花分子机理的研究知之甚微,有待进一步探索。
茶树是多年生常绿木本植物,茶树花属完全花,两性花,由花托、花萼、花冠、雄蕊群和雌蕊组成。云南省农业科学院茶叶研究所收集保存了1株特异花资源,该材料为天然的雌蕊缺失突变体,与正常的茶树花相比,该突变体花无雌蕊、无子房的单性雄花,是一种功能的性器官[8]。单性花其性别分化更彻底,是研究其发育、性别分化和性别决定机制的理想材料。因此本研究利用新一代高通量测序技术对雌蕊缺失茶树花的花芽、花蕾、花3个阶段进行数字基因表达谱分析,获得大量与雌蕊缺失和雄花发育相关的差异基因及生物学通路,为后期深入研究茶树花性别分化和性别决定机制提供理论依据。
1 材料与方法
1.1 试验材料
选用云南省农业科学院茶叶研究所国家大叶种茶树资源圃保存的20年树龄的雌蕊缺失茶树为材料,2015年10月20号分别采摘花芽(初期)、花蕾(中期)、花(含苞待放),利用液氮迅速固样,并进行总RNA的提取。
1.2 RNA的提取与检测
RNA的提取选用CTAB-LiC1方法[9-10],分别提取花芽、花蕾、花的总RNA,采用2%的琼脂糖凝胶电泳分析RNA降解程度以及是否有污染,利用Nanodrop检测RNA的纯度,利用Qubit对RNA浓度进行定量,以及利用Agilent 2100精确检测RNA的完整性。
1.3 文库构建与测序数据分析
检测合格的总RNA样品送北京诺禾致源生物信息科技有限公司,进行mRNA富集、双链cDNA的合成和文库构建,转录组测序采用Illumina HiSeq 2000平台进行。利用RSEM软件的bowtie2将Trinity拼接得到的转录组作为ref(参考序列),将每个样品clean reads(对原始数据进行过滤后,剔除了低质量数据的剩余数据)往ref上做mapping(比对)。采用TMM对read count数据进行标准化处理,再利用DEGseq技术分别对花芽与花蕾、花芽与花、花蕾与花进行差异分析,差异基因筛选条件为qvalue<0.005且log2(fold change)>1。差异基因的表达量按照FPKM法(Reads per kb per million reads)计算。筛选阈值<0.005及log2(fold change)>1;对于差异基因,如果基因的log2(fold change)>0,则认为该差异基因是上调,反之,则认为该差异基因是下调。
2 结果与分析
2.1 雌蕊缺失茶树花3个发育期基因表达差异分析
对花芽、花蕾、花3个发育期文库基因表达互相比对发现,花蕾—花芽上调基因有688条,下调有606条,涉及的代谢通路有247条。花—花蕾上调基因有909条,下调有713条,涉及的代谢通路有230条;花—花芽上调基因有1 555条,下调有1 490条,差异基因涉及的代谢通路有287条(表1)。同时对差异表达基因进行维恩作图,发现在花芽中特异表达基因有128条,在花蕾中特异的有165条,在花中特异的有1 227条(图1)。说明随着花芽的发育,越来越多的unigene(取每条基因中最长的转录本作为该基因的代表,称为unigene)基因被启动,有些基因启动后有沉默,同时花在不同的发育时期有特异基因的表达。
2.2 雌蕊缺失茶树花不同发育期基因通路显著性富集分析
对雌蕊缺失茶树花的花芽、花蕾和花3个时期的差异基因进行代谢通路(Pathway)显著性富集分析,以FDR≤0.05的Pathway定义为在差异表达基因中显著富集的Pathway。
分析结果表明,花蕾—花芽的差异表达上调的基因中涉及的通路主要集中在植物激素信号转导、光合作用、苯丙素的生物合成、光合作用-天线;下调的基因中涉及的通路主要集中在核糖体、淀粉和蔗糖代谢、戊糖和葡萄糖醛酸转换。说明在花芽初期植物激素信号转导途径为上调差异基因富集的通路,对花器官分化以及发育起着重要的调控作用。
花—花蕾的差异表达上调的基因中涉及的通路主要集中在苯丙素的生物合成、类黄酮生物合成、苯丙氨酸代谢、光合作用、DNA复制;下调的基因中涉及的通路主要集中在淀粉和蔗糖代谢、戊糖、葡萄糖醛酸转换、α-亚麻酸的代谢、类胡萝卜素生物合成。说明花蕾期为生物合成和代谢活动的旺盛期,包括次生代谢物黄酮类、氨基酸等的生物合成等,为后期发育积累能量与物质。
花—花芽的差异表达上调的基因中涉及的通路主要集中在碳代谢作用、淀粉和蔗糖代谢、糖酵解途径、戊糖和葡萄糖醛酸转换、柠檬酸循环、脂肪酸代谢;下调的基因中涉及的通路主要集中在苯丙素的生物合成、光合作用、植物激素信号转导、细胞周期。说明花期比花芽初期的呼吸代谢和非氧化糖代谢增强,但是光合作用、次生代谢等减弱。
从以上3组各通路的富集结果可以看出,雌蕊缺失茶树花发育初期表现为花的分化与发育,花蕾期表现为次生代谢物合成的旺盛期,后期表现为呼吸代谢和碳水化合物的积累。
2.3 雌蕊发育相关基因分析
从雌蕊缺失茶树花3个不同时期花芽转录组数据中挖掘出差异表达基因34个。其中生长素信号途径的有生长素载体(Auxin resistant,)、运输抑制剂响应蛋白(Transport inhibitor response protein 1,)、生长素/吲哚乙酸蛋白(Auxin/Indole-acetic acids protein,)转录因子家族、家族、(Gretchen hagen 3)和(Small auxin up RNA);与花器官发育相关的ABCDE类识别基因,A类基因有(APETALA1)、(APETALA2),B类基因有(PISTILLATA),C类基因有、、,D类基因有,E类基因有(SEPALLATA1);WUS基因有、、;以及(KNOTTEDⅠ-like homeobox)类基因。
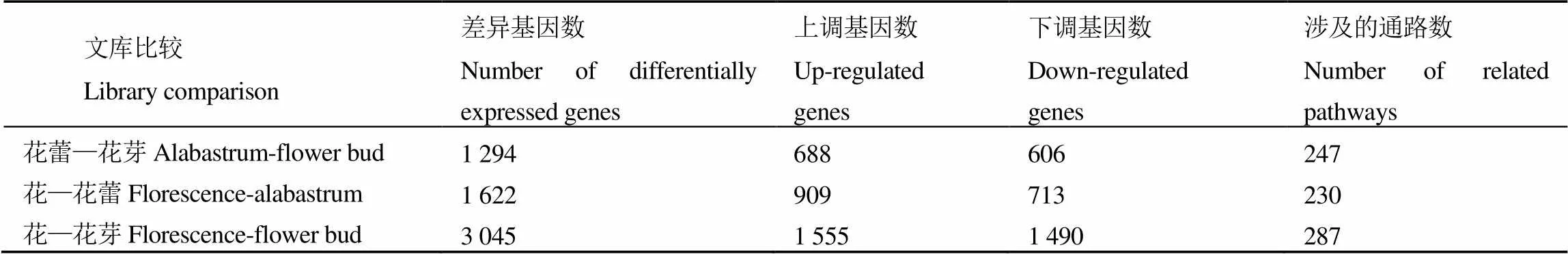
表1 雌蕊缺失茶树花3个发育阶段差异表达基因统计
注:花蕾—花芽:花蕾与花芽对比,花—花蕾:花与花蕾对比,花—花芽:花与花芽对比。下同。
Note: Alabastrum-flower bud: compare comparison between alabastrum to and flower bud, florescence-alabastrum: comparison betweencompare florescence to and alabastrum, florescence - flower bud: comparison betweencompare florescence to and flower bud. The same below.

注:HY:花芽,HL:花蕾,HF:花。Note: HY: flower bud, HL: alabastrum, HF: florescence.
2.3.1 生长素转导途径关键基因表达模式分析
生长素参与植物生长和发育诸多过程,它的极性运输、信号转导对花的形态形成起着关键作用[11-12]。雌蕊缺失花3个时期的转录组数据中发现共有133条unigene,其中有18条差异基因,8条差异基因上调表达,10条差异基因下调表达。从转录组中确定了生长素调控途径关键的几个基因,并对3个不同发育时期的表达量进行了比较,采用DESeq进行基因差异表达分析,筛选阈值为qvalue <0.005且log2(fold change) > 1(表2)。
对表2数据结果分析得出,生长素信号转导途径中,生长素载体的差异基因在雌蕊缺失花的3个时期均上调表达;生长素受体和生长素响应因子的差异基因在雌蕊缺失花的3个时期主要为下调表达;转录因子家族同源基因中c59897_g2和c96532_g3在开花的3个时期主要为下调表达,而c84220_g1和c90409_g2呈上调表达;同源基因c91582_g1表达量在花蕾—花芽期间表达下调,而花—花蕾表达量又明显上调,而c78585_g1和c91671_g1基因在花发育的3个时期表达量逐渐下调;植物生长素应答基因家族的c80304_g1在花芽初期表达量最高,在花蕾和花中的表达量都较低,c76568_g1、c78437_g1、c82050_g1基因的表达量均随着花的发育明显升高。

表2 生长素转导途径关键基因表达模式
2.3.2 ABCDE模型关键基因表达模式分析
花器官的形成与ABCDE类基因密切相关,其中A类基因单独控制萼片的形成;A类和B类基因联合控制花瓣的发育;B类和C类基因共同决定雄蕊的形成;C类基因单独调控心皮的形成;A类基因和C类基因相互抑制,A类基因抑制C类基因在萼片和花瓣中表达,而C类基因抑制A类基因在雄蕊和心皮中表达[13-14];D类基因与胚珠和种子的发育相关[15];E类基因决定着萼片的特性,它与ABC类联合表达可以使叶片转化为完整的花器官[16]。在雌蕊缺失茶树花3个发育期的转录组中共发现34条unigene,其中有9条差异表达基因,花—花芽期5条为上调表达,3条为下调表达,1条花蕾—花芽为上调,花—花蕾为下调(表3)。
对表3数据分析得出,在雌蕊缺失茶树花的花芽、花蕾和花3个发育期的转录组中,A类的差异基因为下调表达;花—花芽期C类和E类的差异基因呈上调表达;B类和D类基因未出现差异表达的基因。
2.3.3基因表达模式分析
基因是第三、第四轮花器官决定基因(C和E类基因)的主要调控因子,它的缺失会对第三、第四轮花器官的发育产生影响[17-19]。在雌蕊缺失茶树花3个发育期的转录组中共发现16条unigene,有3条差异基因,均为下调表达(表4)。
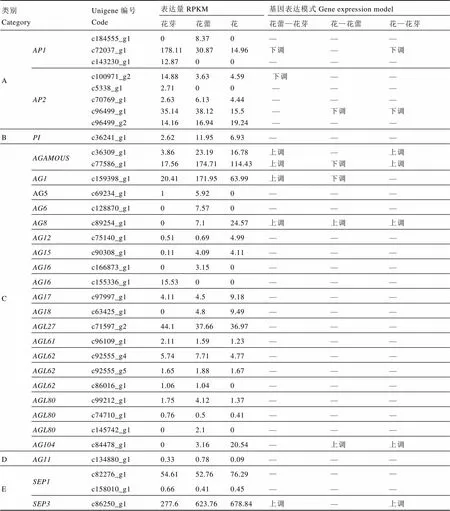
表3 ABCDE模型关键基因表达模式
从表4数据分析得出,在雌蕊缺失茶树花的花芽、花蕾和花3个发育期的转录组中,基因在花的3个发育时期表达量一直降低;的同源基因c92094_g2表达量在花蕾期下调较为明显,在花中的表达量与花蕾中保持一致,同源c60584_g1的表达量在花芽和花蕾中表达量相差不大,而在花中明显降低,其他同源基因无明显差异;、、、、、在3个发育期无明显差异。
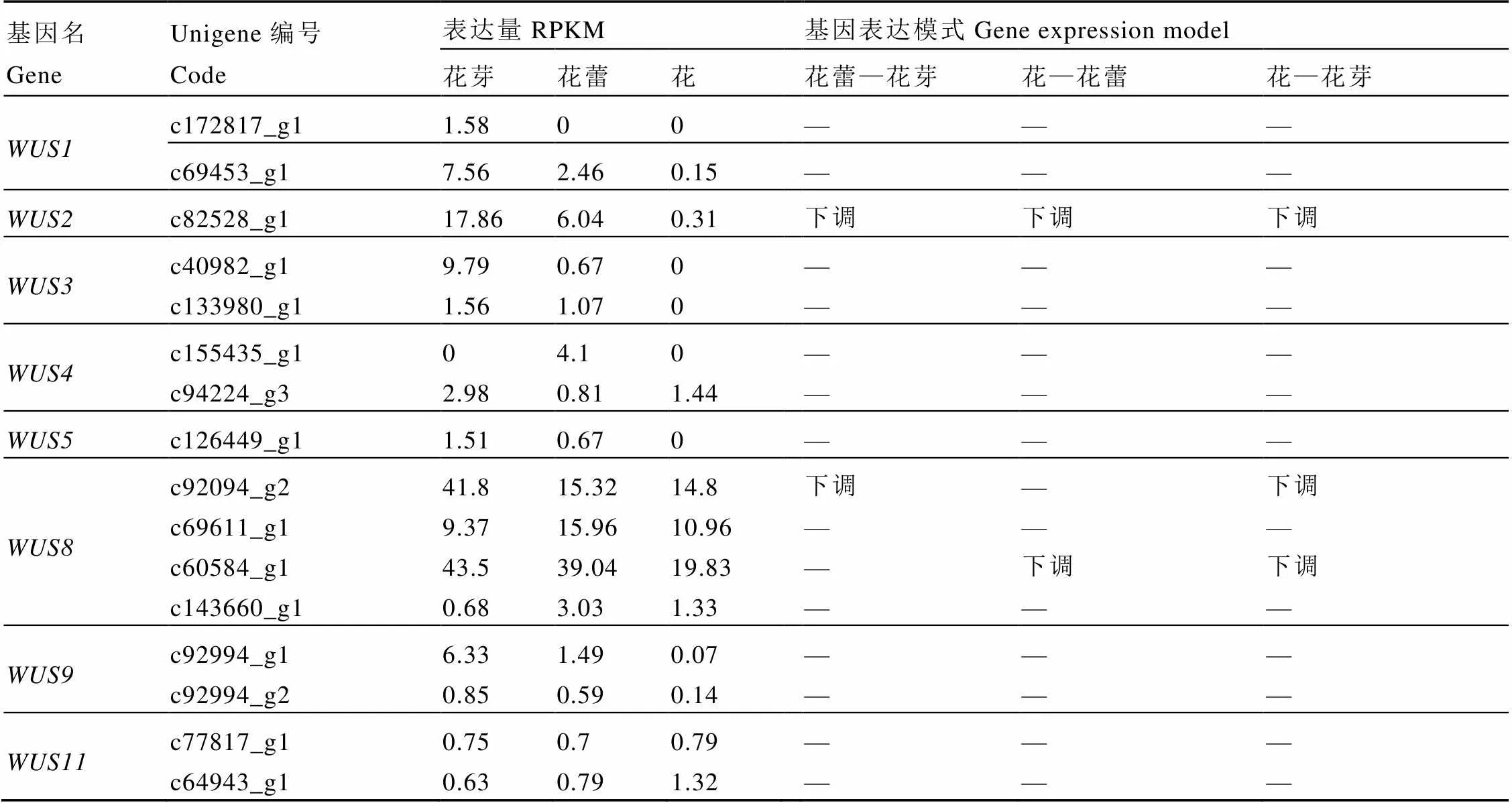
表4 WUS基因表达模式
2.3.4家族关键基因表达模式分析
家族分为(Class I KNOTTED-like homeobox)类和(ClassⅡ KNOTTED-like homeobox)类。主要在茎顶端分生组织中特异表达,是分生组织发生与维持所必需的关键基因,调控与器官发生相关的细胞分化[20];在所有组织中都表达,基因的功能研究还缺乏必要的实验数据,但它们在发育过程中可能扮演某种未知的功能[21]。在雌蕊缺失茶树花3个发育期的转录组中共发现29条unigene,共4条差异基因,其中有3条下调表达,1条上调表达(表5)。
对表5数据分析得出,类家族的差异基因c66694_g1在花芽和花中表达量较花蕾中高,c91417_g2、c93103_g2和c95476_g7都是在花芽中表达量较高,在花蕾和花中明显下降。
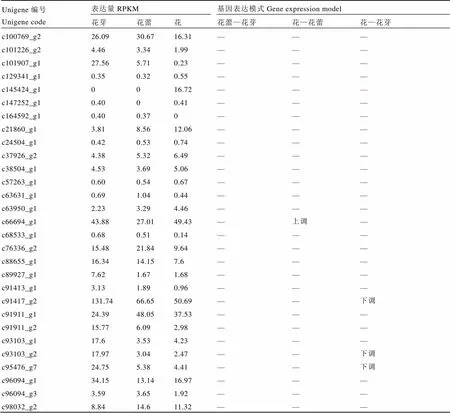
表5 KNOX家族关键基因表达模式
3 讨论
本研究利用雌蕊缺失茶树花的花芽、花蕾、花为材料。基于数字基因表达谱技术,发现了3 552条差异表达基因,其中花芽初期上调基因主要富集于植物激素信号转导途径,花蕾为生物合成和代谢活动的旺盛期,上调基因包括次生代谢物黄酮类、氨基酸等的生物合成等,花为呼吸代谢和碳水化合物的积累时期,上调基因富集于碳代谢作用,淀粉和蔗糖代谢等。从转录组中挖掘出34个与花器官发育相关的差异表达基因,其中生长素信号转导18个;花器官发育ABCDE模型相关基因9个,包括A功能基因3个,C功能5个,E功能1个;类基因3个;家族基因4个。说明在雌蕊缺失茶树花中同样存在部分与雌蕊发育相关的基因。
生长素作为一种重要的植物激素,参与调节植物生长发育的诸多过程[22-23]。在植物开花过程中,高含量的生长素有利于促进雄蕊和雌花的发育[24],雌花中,生长素在花芽分化初期含量较低,随着花的发育不断升高[25]。生长素信号转导过程中,通过与结合,促进早期基因转录,调控下游基因的表达[26-27],从而正向促进花器官的成熟[28-29]。在雌蕊缺失茶树花的花芽、花蕾和花3个时期转录组中发现同源基因c59897_g2和c96532_g3以及的同源基因c81183_g1和c99197_g1均在花芽初期表达量较高,随着花的发育表达量明显降低,说明在雌蕊缺失花的花蕾和花中和的低表达可能抑制了茶树花中下游基因的表达,从而影响了雌花的发育。为生长素早期应答基因,它的表达量与生长素浓度成正比[30];是生长素合成和运输的负调控因子,它的表达量与生长素浓度成反比[31]。检测到家族的差异基因为下调,而家族的差异基因呈上调,说明在茶树花发育的过程中,生长素浓度可能较低,从而导致茶树花雌蕊的缺失。基因是一种生长素输入载体,它能够引起不同部位间生长素浓度差异。是一种运输抑制剂响应蛋白,可以通过应答生长素来调控其它基因的转录。检测到的同源基因c71839_g1和c97091_g1均为上调,的同源基因c90769_g1为下调,推测茶树花中雄蕊的正常发育可能与和的正常调控相关。
在调控花器官形成的ABCDE模型理论中,A类和B类基因联合控制花瓣的发育,B类和C类基因共同决定雄蕊的形成,其中A类基因和C类基因相互抑制。在雌蕊缺失花中,A类差异基因下调表达,C类差异基因上调表达,B类基因表达无明显差异,推测茶树花中花瓣的缺失可能是C类基因对A类基因的抑制,使得低表达的A类基因无法与B类基因联合来促使花瓣发育。D类基因控制胚珠发育,本研究中未发现D类差异表达基因,并且D类基因中与促进心皮发育的基因未见表达。E基因决定着萼片的特性,它与ABC联合表达可以使叶片转化为完整的花器官[16],在雌蕊缺失茶树花中,E类基因仅发现()和基因,未见和。此外,()编码的蛋白质具有(APETALA2)蛋白结构域,它能促进雌蕊的发生和珠被生长[32]。及()的单基因缺失突变体都表现出微弱的雌蕊发育异常,但同时缺失这两个基因的突变体表现出非常严重的突变表型,隔膜、胎座及胚珠完全缺失[33]。本研究通过对雌蕊缺失花转录组中和的查找,不含基因,基因有11条。可见在雌蕊缺失的茶树花中,调控心皮和胚珠相关的基因大多出现异常,而调控花器官形成是一系列基因互相作用的结果,因此茶树花雌蕊的缺失可能是由于和D类基因的缺失,、和的少量表达等异常基因共同作用的结果。
研究表明,基因的缺失或者突变会导致雌蕊发育过程中花器官的缺失,基因是第三和第四轮花器官决定基因(C和E类基因)的主要调控因子,它的缺失会对第三、第四轮花器官的发育产生影响[17]。在茶树的雌蕊缺失花中,检测到基因同源的和的差异表达基因均为下调表达,说明基因的下调表达可能与雌蕊的缺失有关。
类家族分为类和类,在模式生物拟南芥中,类包括、、和,类包括、、和。类基因与心皮的启动和雌蕊边缘组织的生长密切相关,其中,在雌蕊的发育中与()、()及互作从而调控雌蕊边缘组织的发育[34],的异位表达可以诱导产生心皮状的珠心结构,诱导心皮产生并能将胚珠转化为心皮[35],同时作为和的转录调控因子,可能通过调控的表达来影响心皮发生,也能与其它相关因子作用来促进心他的发生。在该项研究中类家族共检测到29条基因,但是未检测到与雌蕊发育相关的、、基因,仅发现的1条下调表达基因c95476_g7,它在花芽中表达量较高,但是在之后的花蕾和花中明显降低。这些说明茶树花中雌蕊的缺失可能与类基因家族的缺失或者低表达相关。家族的另一类基因几乎在所有组织中都表达,但它们在发育过程中可能扮演某种未知的功能[20]。
茶树是重要的叶用经济作物,但是茶树的开花结实所用时间太长,不仅影响茶叶产量和品质,同时加速茶树的衰老。雌蕊缺失茶树是天然不结实茶树资源,前期的研究发现雌蕊缺失茶树内含物含量以及发芽密度都比正常茶树高,推测可能是雌蕊缺失茶树不结果使其生殖生长减弱,有利于茶树的营养生长,从而促使叶片中内含物的积累和提高发芽率。本研究对雌蕊缺失茶树花转录组中与花器官形成、发育相关的生长素信号转导途径、ABCDE模型、家族和家族基因进行分析,筛选出茶树花性别决定相关基因,可通过生物技术来调控茶树的开花结实。本研究结果为后期对雌蕊缺失茶树花雄蕊发育的研究、茶树花性别决定分子机制提供了理论依据。
[1] 吴淑平. 茶树营养生长与生殖生长的关系及调控方法[J]. 中国园艺文摘, 2011, 27(5): 182-183.
[2] 吴剑锋, 张海娟, 卢海宇, 等. 基因芯片分析芜菁雌蕊退化突变体tpa及野生型开放花的转录组差异[J]. 中国农业科学, 2011, 44(5): 972-981.
[3] Zhang J, Sun X L, Zhang L G, et al. Analysis of differential gene expression during floral bud abortion in radish (L.) [J]. Genetics and Molecular Research, 2013, 12(3): 2507-2516.
[4] Zhang J, Li Q F, Huang W W, et al. A vacuolar processing enzyme RsVPE1 gene of radish is involved in floral bud abortion under heat stress [J]. International Journal of Molecular Sciences, 2013, 14(7): 13346-13359.
[5] Pekker I, Alvarez J P, Eshed Y. Auxin response factors mediate Arabidopsis organ asymmetry via modulation of KANADI activity [J]. The Plant Cell, 2005, 17(11): 2899-2910.
[6] Carles C C, Lertpiriyapong K, Reville K, et al. The ULTRAPETALA1 gene functions early in Arabidopsis development to restrict shoot apical meristem activity and acts through WUSCHEL to regulate floral meristem determinacy [J]. Genetics, 2004, 167(4): 1893-1903.
[7] 宋维希, 陈林波, 李友勇, 等. 雌蕊缺失茶树的农艺性状与品质特征初步分析[J]. 湖北农业科学, 2016, 55(9): 2269-2271.
[8] 高暝, 陈益存, 杨素素, 等. 单性花植物性别分化研究进展[J]. 草业学报, 2015, 24(11): 206-217.
[9] 江昌俊, 王朝霞. 茶树中提纯总RNA的研究[J]. 茶叶科学, 2000, 20(1): 27-29.
[10] 史成颖, 宛晓春, 江昌俊, 等. 提取高质量茶树总 RNA的方法研究[J]. 安徽农业大学学报, 2007, 34(3): 360-363.
[11] Dinneny J R, Weigel D, Yanofsky M F. A genetic framework for fruit patterning in Arabidopsis thaliana [J]. Development, 2005, 132(21): 4687-4696.
[12] Balanzá V, Navarrete M, Trigueros M, et al. Patterning the female side of Arabidopsis: the importance of hormones [J]. Journal of Experimental Botany, 2006, 57(13): 3457-3469.
[13] Zahn L M, Leebens-Mack J, Ma H, et al. To B or not to B a flower: the role of DEFICIENS and GLOBOSA orthologs in the evolution of the angiosperms [J]. Journal of Heredity, 2005, 96(3): 225-240.
[14] Battaglia R, Brambilla V, Colombo L, et al. Functional analysis of MADS-box genes controlling ovule development inusing the ethanol-inducible alc gene-expression system [J]. Mechanisms of Development, 2006, 123(4): 267-276.
[15] Pinyopich A, Ditta G S, Savidge B, et al. Assessing the redundancy of MADS-box genes during carpel and ovule development [J]. Nature, 2003, 424(6944): 85-88.
[16] Pelaz S, Ditta G S, Baumann E, et al. B and C floral organ identity functions require SEPALLATA MADS-box genes [J]. Nature, 2000, 405(6783): 200-203.
[17] Carles C C, Lertpiriyapong K, Reville K, et al. The ULTRAPETALA1 gene functions early in Arabidopsis development to restrict shoot apical meristem activity and acts through WUSCHEL to regulate floral meristem determinacy [J]. Genetics, 2004, 167(4): 1893-1903.
[18] Xu Y Y, Wang X M, Li J, et al. Activation of the WUS gene induces ectopic initiation of floral meristems on mature stem surface in Arabidopsis thaliana [J]. Plant Molecular Biology, 2005, 57(6): 773-784.
[19] Sarkar A K, Luijten M, Miyashima S, et al. Conserved factors regulate signalling inshoot and root stem cell organizers [J]. Nature, 2007, 446(7137): 811-814.
[20] 李春苑, 阮美煜, 贾海燕, 等. 同源异型盒基因 I 类 KNOX 的表达调控及在植物形态建成中的作用[J]. 细胞生物学杂志, 2009, 31(5): 635-640.
[21] Di Giacomo E, Sestili F, Iannelli M A, et al. Characterization of KNOX genes in[J]. Plant Molecular Biology, 2008, 67(1/2): 135-150.
[22] Kepinski S, Leyser O. Plant development: auxin in loops [J]. Current Biology, 2005, 15(6): R208-R210.
[23] Bohn-Courseau I. Auxin: a major regulator of organogenesis [J]. Comptes Rendus Biologies, 2010, 333(4): 290-296.
[24] 杨传平, 刘桂丰, 姜静, 等. 白桦雄花发育过程中内源激素含量的变化[J]. 东北林业大学学报, 2002, 30(4): 1-4.
[25] 宋福南, 杨传平, 刘雪梅. 白桦雌花发育过程中内源激素动态变化[J]. 植物生理学通讯, 2006, 42(3): 465-466.
[26] Ramos J A, Zenser N, Leyser O, et al. Rapid degradation of auxin/indoleacetic acid proteins requires conserved amino acids of domain II and is proteasome dependent [J]. The Plant Cell, 2001, 13(10): 2349-2360.
[27] Gan D, Zhuang D, Ding F, et al. Identification and expression analysis of primary auxin-responsive Aux/IAA gene family in cucumber () [J]. Journal of Genetics, 2013, 92(3): 513-521.
[28] Tabata R, Ikezaki M, Fujibe T, et al. Arabidopsis auxin response factor6 and 8 regulate jasmonic acid biosynthesis and floral organ development via repression of class 1 KNOX genes [J]. Plant and Cell Physiology, 2010, 51(1): 164-175.
[29] Rademacher E H, Möller B, Lokerse A S, et al. A cellular expression map of the Arabidopsis AUXIN RESPONSE FACTOR gene family [J]. The Plant Journal, 2011, 68(4): 597-606.
[30] 吕剑, 喻景权. 植物生长素的作用机制[J]. 植物生理学通讯, 2004, 40(5): 624-628.
[31] Kant S, Bi Y M, Zhu T, et al. SAUR39, a small auxin-up RNA gene, acts as a negative regulator of auxin synthesis and transport in rice [J]. Plant Physiology, 2009, 151(2): 691-701.
[32] Krizek B A, Prost V, Macias A. AINTEGUMENTA promotes petal identity and acts as a negative regulator of AGAMOUS [J]. The Plant Cell, 2000, 12(8): 1357-1366.
[33] Skinner D J, Hill T A, Gasser C S. Regulation of ovule development [J]. The Plant Cell, 2004, 16 (suppl 1): S32-S45.
[34] 卢海宇, 曹家树, 余小林. 控制雌蕊发育的分子遗传调控机制[J]. 细胞生物学杂志, 2009, 31(1): 63-68.
[35] Ragni L, Belles-Boix E, Günl M, et al. Interaction of KNAT6 and KNAT2 with BREVIPEDICELLUS and PENNYWISE ininflorescences [J]. The Plant Cell, 2008, 20(4): 888-900.
Digital Gene Expression Analysis of Tea Flowers withoutPistil at Three Development Stages
LI Mei1, CHEN Linbo1, TIAN Yiping1, XIA Lifei1, SONG Weixi1, LIANG Mingzhi1, JIANG Changjun2*
1. Tea Research Institute, Yunnan Academy of Agricultural Sciences, Yunnan Engineering Research Center of Tea Germplasm Innovation and Matching Cultivation, Menghai 666201, China; 2. State Key Laboratory of Tea Plant Biology and Utilization, Anhui Agricultural University, Hefei 230036, China
The expression patterns of important genes in tea flowers without pistil at the flower bud, alabastrum and florescence stages was studied by the transcriptome and digital gene expression profiling technology. The results found that genes associating with biosynthesis and metabolic activities were highly expressed in tea flowers during the flowering process. Six genes involved in auxin signal transduction and the A、C and E genes of ABCDE model might be closely correlated with the stamen development and response to pistil deletion, which were under complex regulation. The down- regulation ofandinfamily might also regulate the C and E genes, and result in the deletion of pistil. In thegene family, none homologous gene ofwas detected. The down-regulation of 3 homologous genes of the KNOX I might weaken the initiation of carpels and the growth of marginal tissues.According to the digital gene expression analysis, a preliminary understanding of the network associating with the pistil deletion and stamen development in tea flower was obtained, which provides a theoretical basis for further research on pistil deletion, stamen development, molecular mechanism of sterility and sex determination in tea plant.
tea flowers, pistil deletion, digital gene expression profiling, transcriptome, floral organ regulation
S571.1;Q52
A
1000-369X(2017)01-097-11
2016-08-09
2016-10-17
国家自然科学基金(31560220、31460216)、云南省人才培养计划(2015HB105)、茶树生物学与资源利用国家重点实验室开放基金(SKLTOF20150105)。
李梅,女,研究实习员,主要从事茶树分子生物学研究。
jiangcj@ahau.edu.cn
