ToxCast化学品对CYP450异构酶抑制的QSAR研究
2017-02-22朱祥伟青岛农业大学环境科学系山东青岛66109浙江大学药学院浙江杭州310058
朱祥伟,陈 浮(1.青岛农业大学环境科学系,山东 青岛 66109;.浙江大学药学院,浙江 杭州 310058)
ToxCast化学品对CYP450异构酶抑制的QSAR研究
朱祥伟1*,陈 浮2(1.青岛农业大学环境科学系,山东 青岛 266109;2.浙江大学药学院,浙江 杭州 310058)
研究收集美国国立健康研究所化学基因组中心利用高通量筛选测定的1万多种化合物对5种细胞色素P450(CYP450)酶的抑制数据,利用随机森林法构建MOE 2D描述符的分类模型. 采用五折交叉验证建模策略保证模型的预测能力. 模型对1A2、2C9、2C19、2D6和3A4等酶抑制剂预测的正确分类率分别高达84.4%、82.5%、82.1%、78.4%和80.0%, 结构分析表明, 卤代(氯/氟)芳烃结构在抑制剂中出现概率显著高于非抑制剂. 最后, 利用上述模型对ToxCast项目954个化合物进行虚拟筛选. 实例化合物分析验证了模型预测与实际效应的吻合程度. 构建的模型可进一步对其它环境化学品的CYP450酶抑制活性进行预测, 加快对化学品健康风险的初步筛选.
环境化学品;定量构效关系;虚拟筛选;随机森林;细胞色素P450
随着工业化程度的发展,每年都有大量化学品排放到环境.中国环境保护部 2013年发布的“化学品环境风险防控“十二五”规划”显示我国现有生产使用的化学物质达4万多种[1].2015年版《危险化学品名录》收录的有害化学物质近3千余种.美国国立环境健康科学研究所(NIEHS)评估显示,环境化学品如持久性有机污染物、杀虫剂、卤代烃类等会导致多种健康风险如癌症、自身免疫性疾病[2-3].美国环境保护局(EPA)实施的ToxCast项目[4]即旨在利用计算毒理学手段研究环境中使用最广泛、接触最多的化学品的健康危害.欧盟REACH计划及美国EPA都支持以定量构效关系(QSAR)[5]为主要手段的计算毒理学在化学品毒理与药效预测、优先污染物筛选方面应用.
人体肝脏中50多种细胞色素P450(CYP450)异构酶,通过氧化、还原、水解及水合等 I类代谢反应等将底物生物转化.异构酶中以 1A2、2C9、2C19、2D6和3A4最为重要[6],代谢的化合物占所有酶代谢总量的 90%.非底物分子结合CYP450酶会引起酶活性抑制[7],增加健康风险.许多研究利用QSAR方法构建了CYP450异构酶抑制剂预测的分类模型.Zhou等[8]利用支持向量机(SVM)建立了基于826个CYP3A4抑制剂与873个非抑制剂的分类模型.对 166个抑制剂与677个非抑制剂的验证集的预测准确率达到83%.Yap等[9]利用 SVM 建立了 CYP3A4、CYP2D6和CYP2C9的底物与抑制剂分类模型,预测准确率大于90%.然而,很少有模型用于大规模化学品的筛选.Sun等[10]利用美国国立化学基因组中心(NCGC)测定的1万多个化合物对5种CYP450 (1A2、2C9、2C19、2D6和3A4) 酶活性抑制数据,构建了5组SVM分类模型.不同模型对各自占总化合物数目约 50%的检验集分子预测准确率都在 80%以上.然而该研究没有对所建分类模型采用严格的外部交叉验证.
本文收集 NCGC测定的化合物对 5种CYP450 酶活性抑制数据,将分子结构进行标准化校正[11],采用严格的外部交叉验证流程建立CYP450抑制剂预测的分类模型.以ToxCast项目化学品为对象进行虚拟筛选.着重考察其对CYP450酶的抑制特点,为评估这些环境化学品的健康风险提供数据支撑.
1 材料与方法
1.1 化合物结构校正标准化
下载NCGC测定的1万多种化合物对5组CYP450酶的抑制效应数据(https://pubchem.ncbi. nlm.nih.gov/bioassay/1851).针对每一组 CYP450异构酶数据,删除活性不明确(inconclusive)物质,保留活性与非活性数据.针对大量不适合计算化学描述符的有机盐、带电离子、混合物等分子,首先剔除有机重金属化合物、混合物.利用ChemAxon Standardizer(v.5.4)软件,通过分子中性化(去电荷)、消除互变异构体、芳香环化(六元环由单双键交替形式转化成环状结构)、结构平面化等步骤生成标准SMILES结构.最后,检测并剔除重复化合物.
1.2 分子结构描述符
为实现快速建模,采用数量较少的MOE 2D (http://chembench.mml.unc.edu)描述符,共184种,包括分子拓扑结构、物理性质、化学键信息.删除标准偏差小于0.001的描述符.若2个描述符间相关系数大于0.90,则随机删除一个.
1.3 建模方法与流程
随机森林(RF)算法[12]采取有放回的随机抽样,构造子数据集,描述符在逻辑节点处依阈值大小分叉,训练分类树,最终综合多颗分类树结果. RF具有学习速度快、分类准确率高、不易产生过拟合等优点.本文构建500棵分类树,随机选取描述符总量开方值(四舍五入)个描述符变量构建每棵分类树
[13],通过编译软件包“RandomForest”[14]在R语言平台(v.3.0.2)上建模.
描述分类模型好坏的标准有:1)敏感度,即真阳性率,描述模型对抑制剂预测的准确度;2)特异度,即真阴性率,描述模型对非抑制剂预测的准确度;3)正确分类率(CCR).CCR为敏感度和特异度的均值,用于矫正准确率表征分类模型对非均衡数据集(如2C9、2D6)的预测能力带来的偏差.采用 5折交叉验证确保模型的外部预测能力[15]即将数据随机分成5组大小相同的子集.选取4组作为建模数据集,余下1组外部验证.此过程重复5次,确保每个子集有一次作为外部验证集的机会.最终,每个化合物有四次机会参建模,有一次机会用于外部验证.
在深井直孔中的钻杆柱可视为受拉力作用的柔性杆体。正弯曲段钻杆柱摩阻力可简化为作用于该孔段中间点,摩阻力近似为:
Y随机化用于确定模型的稳健能力[18].它将训练集数据的活性值随机打乱,随后建立模型对外部验证集进行预测.Y随机化建模至少进行 5次.利用单尾t检验计算正常模型与Y随机化模型预测CCR的显著程度.若t检验大于P>0.05,即表明QSAR模型不稳健.
2 结果与讨论
2.1 5组CYP450酶活性抑制模型
2.1.1 建模数据集 剔除有机金属分子、混合物重复分子等不规范化合物后,5组CYP450酶抑制剂(阳性)与非抑制剂(阴性)数据如表1所示.除2C9与2D6酶抑制剂明显少于非抑制剂外,其他 3种酶抑制剂与非抑制剂数目相对均衡.5组酶用于QSAR建模的MOE描述符个数列于表1.
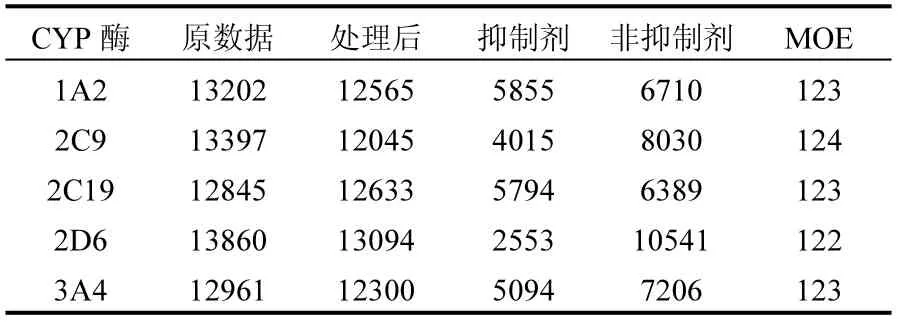
表1 CYP450酶抑制剂、非抑制剂及分子描述符Table 1 Chemical and descriptor information of the CYP450datasets
2.1.2 CYP450分类模型 利用RF[12]建立经5折交叉验证的分类模型. RF模型由500棵分类树组成.每棵分类树对化合物的预测有阳性(抑制剂,用1表示)或阴性(非抑制剂,用0表示).不同分类树的预测也不相同.依据模型中阳性预测总个数占总分类树(500棵)的比例,则化合物为抑制剂的的概率在0~1之间.根据一系列不同的分类阈值,以敏感度为纵坐标,以 1-特异度为横坐标绘制受试者工作特征曲线(ROC)(图1),对角线表示随机预测准确率(0.5).1A2、2C9、2C19、2D6和3A4等5组模型的ROC曲线下面积分别为0.92、0.89、0.89、0.86和0.87,表明所建模型具有良好的分类能力,与此前模型[10]相当(1A2、2C9、2C19、2D6及 3A4的ROC值分别为0.93、0.89、0.89、0.85及0.87).
对每组数据集的Y随机化后,采用相同手段建模,模型CCR值均在0.5左右,与二元分类模型随机预测准确率(0.5)相当.单尾t检验计算(随机取样20%,n=1000)得到Y随机化模型与正常模型预测CCR有显著性的差异(P<<0.001),表明模型稳健.
采用严格型应用域(Z=0.5),75%建模化合物及30%的ToxCast化合物在可预测范围内.宽松型应用域(Z=3),超过98%的建模化合物及90%的 ToxCast化合物在可预测范围内.表2为CYP450模型中最优CCR的分类阈值.如1A2模型的阈值为0.47,即预测值小于0.47为1A2非抑制剂,大于0.47为抑制剂.需要指出,2D6模型的分类阈值为0.25,CCR为 78.4%. 2D6抑制剂的比例低(仅19.5%)导致分类阈值小.
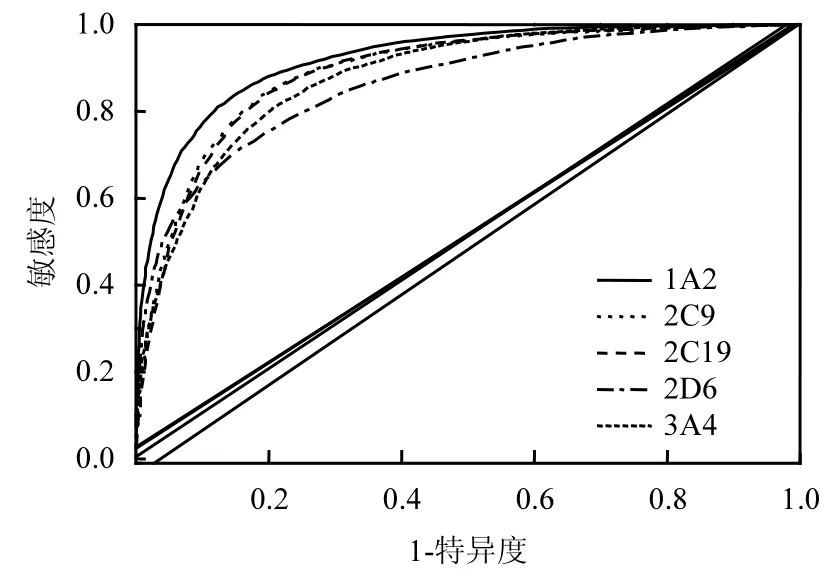
图1 5组CYP450模型的受试者工作特征曲线Fig.1 ROC curves of five CYP450 models
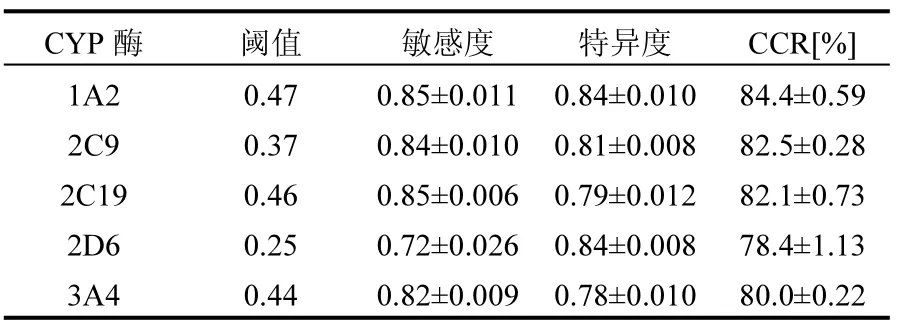
表2 五组模型的敏感度、特异度与CCRTable 2 Sensitivity, specificity, and CCR of the five models
对分子预测值排序发现,1A2、2C9、2C19、2D6和3A4等模型预测值排名前5%的分子为抑制剂的比例分别为100%、92.9%、95.3%、89.8%和97.3%.各组模型预测值后5%的分子为非抑制剂的比例分别为99.2%、98.8%、97.2%、99.1%和99.3%.表明,RF模型对接近阳性或阴性两极的分子预测正确率极高.
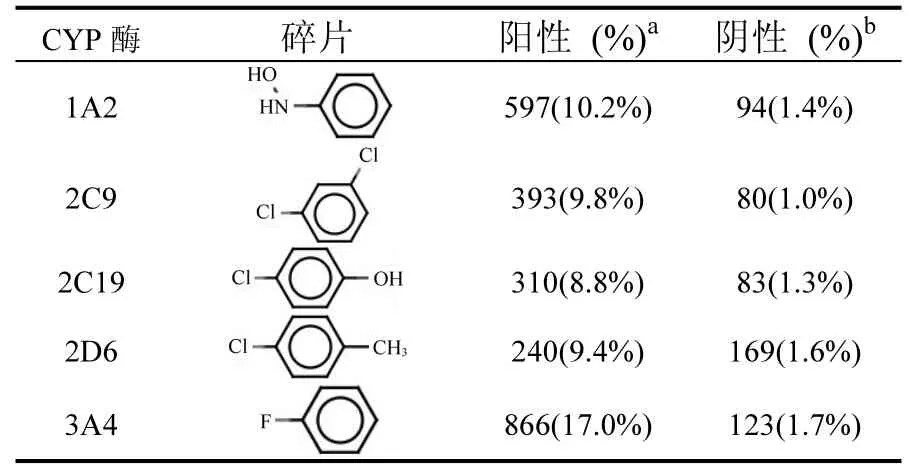
表3 MoSS模块中获取的毒性化合物亚结构碎片Table 3 Substructural alert of toxic chemicals obtained in MoSS
2.1.3 CYP450抑制剂的结构特征 通过KNIME软件[19]中分子碎片分析工具MoSS模块对抑制剂与非抑制剂的亚结构分析.筛选条件如下:①含某碎片(例如氯苯碎片)占抑制剂总数至少8%;②相同碎片(即氯苯碎片)占非抑制剂总数不高于 3%;③最小碎片非氢原子数大于 5;④最大碎片非氢原子数小于11.表3列出CYP450抑制剂与非抑制剂差异最显著的碎片信息. 1A2酶抑制剂与非抑制剂最显著差异是芳香胺类碎片,其它4种酶抑制剂主要结构特征是含有卤(氯/氟)代芳烃分子亚结构.
1A2酶抑制剂分子碎片除芳香胺碎片外,也包括间氯代芳烃,与2C9酶分子碎片一样.含间氯代芳烃碎片的分子占 1A2抑制剂总数的 8.4% (492个),占非抑制剂的1.4% (94个).每类CYP酶抑制剂较显著的亚结构都包括多种卤代芳烃、芳胺类碎片.例如,2C19酶抑制剂亚结构同样包括芳香胺类碎片、氟代芳烃碎片(与3A4相同).
2D6酶抑制剂最显著的碎片对氯甲苯同样也存在于 2C19酶的抑制剂中.以上芳香胺族化合物羟化、卤代芳烃还原脱卤反应过程中产生自由基离子可能对酶活性起抑制作用.同时,对重要描述符分析发现,logS (水中溶解度对数)、logP (辛醇-水分配系数对数)、SlogP (辛醇-水分配系数对数(包含H))对模型预测能力影响最大.
2.2 ToxCast项目化学品虚拟筛选
环境中大量化学品与肝脏 CYP450酶的相互作用形式仍然未知.如前所述,ToxCast I 期 II期项目旨在研究使用经济的方法获取约1000种化学品(包括杀虫剂、药物与护理品、食品添加剂等)的健康危害效应.利用5组CYP450模型对954个ToxCast化学品进行虚拟筛选.
2.2.1 虚拟筛选结果可信度 表 4统计了ToxCast化学品与CYP450酶化合物中约50个重复化合物.RF模型对这些分子活性的预测准确率都在98%以上.这间接反映了CYP450分类模型对ToxCast化合物预测的可信度.

表4 RF模型预测重复分子的敏感度、特异度与CCRTable 4 The Sensitivity, Specificity, and CCR of RF models against overlap chemicals
将实例分子虚拟筛选结果与实际生物效应对比,可进一步验证分类模型的可信度.图2 (饼状图)显示羟基丁二酸二乙酯(CAS RN:6915-15-7)、咖啡碱(83-67-0)、蔗糖(57-50-1)、柠檬酸(77-92-9)等食品添加剂的CYP450酶抑制效应预测.饼状图中代表每组CYP450酶的扇形面积大小与抑制效应大小成正比.这些食品添加剂对CYP450酶几乎没有任何抑制效应,与实际吻合.
禾草灵(51338-27-3)是内吸收性除草剂,对人淋巴细胞、小鼠及野鼠都有较强的毒性[18-19].模型预测显示禾草灵严重影响1A2、2C9和2C19的活性.啶酰菌胺(188425-85-6)是广谱类抗真菌剂,饼状图显示它对CYP450酶特别是1A2、2C9和2C19有较强的抑制效应.
辛伐他汀(CAS RN:79902-63-9)抑制内源性胆固醇的合成,是血酯调节药物.预测显示它仅强烈抑制3A4.3A4在CYP450酶中最为重要,主要负责药物和类固醇分子的代谢,可代谢药物占已知药物总量的50%[22].有实验显示辛伐他汀抑制3A4的活性[23],与模型预测结果吻合.综合分析发现,抗真菌剂、有机氯、有机磷杀虫剂或体内代谢物、失败药物或个人护理产品、持久性有机污染物如多环芳烃对 CYP450酶抑制能力较强.而一些人用或兽用药物、食品添加剂等对CYP450酶抑制较弱.

图2 部分ToxCast化学品(CAS号表示)虚拟筛选结果Fig.2 Virtual screening results of ToxCast chemicals
2.2.2 ToxCast化学品整体效应分析 采用表2中分类阈值对 ToxCast化学品分类,分别有40.1%、36.4%、37.0%、32.4%和 19.6%的化合物是1A2、2C9、2C19、2D6和 3A4酶的抑制剂.层次聚类分析发现,ToxCast化学品对2C19与2C9的抑制效应间聚类距离最小.化合物对 2C9与2C19的抑制效应间确定系数(R2)为0.78(图3).有研究表明, 2C9与2C19两个酶蛋白在一级序列上有91%的相似度[24].这说明在高度相似一级序列基础上折叠成的三级蛋白酶结构与外源分子的相互作用方式极其相似.
而1A2与其他4组CYP450酶的聚类距离都非常远,化合物对1A2预测抑制效应与对2C9、 2C19、2D6、3A4间R2分别为0.28、0.44、0.079、0.077(图3).暗示化合物对1A2的抑制模式不同于对其他酶的抑制模式有较大区别.同时,3A4同外源分子相互作用方式与2C9和2C19也有类同(R2分别为0.64与0.56).

图3 ToxCast化学品酶抑制效应相关图Fig.3 Correlation analysis of ToxCast chemicals
3 结论
3.1 研究构建了经严格外部验证的CYP450酶抑制剂分类QSAR模型,5折交叉验证显示模型具有较高的正确分类率.同时发现卤代芳烃分子亚结构是决定化合物对 CYP450酶抑制的重要结构特征.五个 CYP450模型已应用于 954个ToxCast化合物的虚拟筛选.通过对一些除草剂与食品添加剂分子的实例分析,间接证明了CYP450模型预测的可信度.
3.2 根据有机化合物结构对其CYP450酶抑制能力进行分类预测,筛选出了对 CYP450酶抑制性有较大贡献的分子碎片结构如芳香胺、卤代(氯/氟)芳烃.同时,构建的模型可进一步对其它环境化学品的 CYP450酶抑制活性进行预测,加快对化学品健康风险的初步筛选.
[1] 环境保护部.关于印发《化学品环境风险防控“十二五”规划》的通知.http://www.zhb.gov.cn/gkml/hbb/bwj/201302/t20130220_ 248271.htm. 2013.
[2] Thayer K A, Heindel J J, Bucher JR, et al. Role of environmental chemicals in diabetes and obesity: a national toxicology programworkshop review [J]. Environmental Health Perspective, 2012, 120(6):779-789.
[3] Alavanja M C R, Bonner M R. Occupational pesticide exposures and cancer Risk: a review [J]. Journal of Toxicology and Environmental Health, 2012,15(4):238-263.
[4] Dix D J, Houck K A, Martin M T, et al. The ToxCast program for prioritizing toxicity testing of environmental chemicals [J]. Toxicological Science, 2007,95(1):5-12.
[5] Cherkasov A, Muratov E N, Fourches D, et al. QSAR modeling: Where have you been? Where are you going to? [J]. Journal of Medicinal Chemistry, 2014,57(12):4977-5010.
[6] Kirchmair J, Williamson M J, Tyzack J D, et al. Computational prediction of metabolism: sites, products, SAR, P450 enzyme dynamics, and mechanisms [J]. Journal Chemical Information and Modeling, 2012,52(3):617-648.
[7] Ho H K, Chan J C Y, Hardy K D, et al. Mechanism-based inactivation of CYP450enzymes: a case study of lapatinib [J]. Drug Metabolism Reviews, 2015,47(1):21-28.
[8] Zhou D, Liu R, Otmani S A, et al. Rapid classification of CYP3A4inhibition potential using support vector machine approach [J]. Letters in Drug Design & Discovery, 2007,4(3): 192-200.
[9] Yap C W, Chen Y Z. Prediction of cytochrome P450 3A4, 2D6, and 2C9inhibitors and substrates by using support vector machines. [J]. Journal of Chemical Information and Modeling, 2005,45(4):982-992.
[10] Sun H, Veith H, Xia M, et al. Predictive models for cytochrome p450isozymes based on quantitative high throughput screening data. [J]. Journal of Chemical Information and Modeling, 2011, 51(10):2474-2481.
[11] Fourches D, Muratov E, Tropsha A. Trust, but verify: on the importance of chemical structure curation in cheminformatics and QSAR modeling research [J]. Journal of Chemical Information and Modeling, 2010,50(7):1189-1204.
[12] Breiman L. Random forests [J]. Machine Learning, 2001,45(1): 5-32.
[13] Svetnik V, Liaw A, Tong C, et al. Random forest: A classification and regression tool for compound classification and QSAR modeling [J]. Journal of Chemical Information Computational Science, 2003,43(6):1947-1958.
[14] Liaw A, Wiener M. Classification and Regression by random Forest [J]. R News, 2002,2(3):18-22.
[15] Zhu X-W, Sedykh A, Zhu H, et al. The Use of pseudoequilibrium constant affords improved QSAR models of human plasma protein binding [J]. Pharmacutical Research, 2013,30(7): 1790–1798.
[16] Golbraikh A, Shen M, Xiao Z Y, et al. Tropsha A. Rational selection of training and test sets for the development of validated QSAR models [J]. Journal of Computer-Aided Molecular Design, 2003,17(2-4):241-253.
[17] Zhu X-W, Xin Y-J, Chen Q-H. Chemical and in vitro biological information to predict mouse liver toxicity using recursive random forests [J]. SAR QSAR in Environmental Research, 2016, 27(7):559-572.
[18] Rucker C, Rucker G, Meringer M. y-Randomization and its variants in QSPR/QSAR [J]. Journal of Chemical Information Modeling, 2007,47(6):2345-2357.
[19] KNIME V3.1. KNIME [Z]. 2016. http://www.knime.org/.
[20] Westlake GE, Tarrant KA, Hardy AR. Biochemical and histological effects of diclofop-methyl in mice and voles under laboratory conditions [J]. Bulletin of Environmental Contamination and Toxicology, 1988,40(1):153-158.
[21] Unal F, Yüzbaşıoğlu D, Yılmaz S, et al. Genotoxic effects of chlorophenoxy herbicide diclofop-methyl in mice in vivo and in human lymphocytes in vitro. [J]. Drug and Chemical Toxicology, 2011,34(4):390-395.
[22] Li H, Sun J, Fan X, et al. Considerations and recent advances in QSAR models for cytochrome P450-mediated drug metabolism prediction. [J]. Journal of Computer-Aided Molecular Design, 2008,22(11):843-855.
[23] Hansten P D. Possible risks to patients receiving statins combined with other medications [J]. Journal of the American College of Cardiology, 2003,41(3):519-520.
[24] Williams P A, Cosme J, Ward A, et al. Crystal structure of human cytochrome P450 2C9with bound warfarin. [J]. Nature, 2003, 424(6947):464-468.
致谢:本论文受到同济大学环境学院刘树深教授的指导意见与建议,在此表示感谢.同时感谢审稿人专业而详细的意见与建议.
QSAR studies on the inhibition of ToxCast chemicals to cytochrome p450 isozymes.
ZHU Xiang-wei1*, CHEN Fu2
(1.Department of Environmental Science, Qingdao Agricultural University, Qingdao 266109, China;2.College of Pharmaceutical Sciences, Zhejiang University, Hangzhou 310058, China). China Environmental Science, 2017,37(1):386~391
Several computational classifiers were developed using over 10000 screened compounds collected from NCGC against five major CYP450 isozymes of 1A2, 2C9, 2C19, 2D6, and 3A4. Random forest was used to develop models for these five isozymes using a set of MOE 2D descriptors. Five-fold cross-validation strategy was implemented to ensure the internal predictive ability of the models. The rigorously validated models exhibited outstanding predictive power for all five CYP450 isozymes with correct classification rates of 84.4%, 82.5%, 82.1%, 78.4%, and 80.0% for 1A2, 2C9, 2C19, 2D6, and 3A4, respectively. Structural analysis showed that halogenated (chlorine/fluorine) aromatics have significant higher frequency in CYP450 inhibitors than that in non-inhibitors. This comprehensive study yielded a compendium of validated QSAR models, which were then used to virtual screen a set of 954compounds in ToxCast project initiated by US EPA. Case studies confirmed the predictability of QSAR models through comparing the putative properties with the activities of several compounds. The proposal models carried with robustness and reliability and could be used to virtual screening other environmental chemicals for tentative risk assessment.
environmental chemicals;QSAR;virtual screening;random forest;cytochrome P450
X171
A
1000-6923(2017)01-0386-06
朱祥伟(1984-),男,山东临沂人,讲师,博士,主要从事环境毒理学研究.发表论文30余篇.
2016-05-04
国家自然科学基金资助项目(21407087)
* 责任作者, 讲师, xwzhunc@gmail.com
