刀鲚嗅觉受体基因MOR-51I2克隆、序列分析及组织表达
2017-02-15王晓梅朱国利唐文乔
王晓梅朱国利唐文乔
(1. 上海海洋大学鱼类研究室, 上海 201306; 2. 上海市海洋动物系统分类与进化重点实验室, 上海 201306)
刀鲚嗅觉受体基因MOR-51I2克隆、序列分析及组织表达
王晓梅1,2朱国利1,2唐文乔1,2
(1. 上海海洋大学鱼类研究室, 上海 201306; 2. 上海市海洋动物系统分类与进化重点实验室, 上海 201306)
为研究刀鲚的嗅觉受体(Olfactory receptor, OR)是否参与其生殖洄游过程, 利用基因组步移技术, 从洄游型刀鲚(Coilia nasus)中克隆出嗅觉受体基因MOR-51I2的基因序列全长。该基因为单外显子结构, 编码区长为999 bp。在3′ UTR区域具有一段微卫星序列, 以(AC)n为重复单位, 并夹有若干T或G碱基, 且在不同生态型间具有明显长度差异。MOR-51I2基因所编码的蛋白具有7次疏水性α-螺旋的跨膜结构, 为G-蛋白偶联受体。MOR-51I2基因与已报道的其他鱼类的OR基因所编码的氨基酸序列的同源性在51%以上, 其中与大西洋鲱(Clupea harengus)的OR51I2-like基因的同源性高达83%。经qRT-PCR分析显示, 在定居型刀鲚中, MOR-51I2基因主要在嗅囊和性腺中表达, 在肝脏、鳃、肌肉微弱表达, 在心脏和眼睛中几乎不表达。其中, 雌性嗅囊中的表达量约是雄性嗅囊中的2倍, 是精巢和卵巢中的80—100倍。在洄游型刀鲚中, 该基因在雄性嗅囊的表达量约是雌性嗅囊中的6倍。MOR-51I2基因在洄游型刀鲚的雌性嗅囊中的表达量约是定居型刀鲚的雌性嗅囊中的1/5, 而在洄游型刀鲚的雄性嗅囊中的表达量却是定居型刀鲚的雄性嗅囊中的3倍。这些结果表明, MOR-51I2基因不但参与刀鲚的嗅觉功能, 而且可能参与了刀鲚的性腺发育及生殖洄游过程, 同时也可能与其生态型的分化相关。
刀鲚; 嗅觉受体基因; 组织表达; 生殖洄游; 生态型分化
刀鲚(Coilia nasus)是鲱形目(Clupeiformes)、鳀科(Engraulidae)的一种中小型鱼类, 具有洄游和定居两种生态型[1—4]。洄游型平时栖息于中国、日本和朝鲜半岛的近海, 春季性成熟后进入河流的中、下游产卵, 孵化后的幼鱼顺流而下进入海中肥育。定居型主要分布于长江中下游干流及其附属湖泊, 可在淡水中完成整个生活史[1,2,4]。在长江中,定居型刀鲚的资源量丰富, 是低值渔品。但洄游型刀鲚的种群数量却呈急剧下降趋势, 已成为长江流域最名贵的水产品之一。刀鲚的洄游型和定居型形态相近, 生殖洄游是区别两者的主要表征之一。鉴别生态类型、阐明生殖洄游机制对保护和利用长江洄游型刀鲚这一珍贵渔业资源具有积极意义。已有学者尝试用外部形态的多元分析[5—7]、“寄生虫标记”[8,9]、耳石形态及元素成分[10—13]、肌肉同位素组分差异[14]以及某些基因差异[4,15]等鉴别刀鲚的不同生态类型, 但对刀鲚生殖洄游机制的研究才刚起步[16,17]。
鱼类生殖洄游的嗅觉定向(Olfactory orientation)假说自20世纪50年代被提出以来[18], 嗅觉在美洲鳗(Anguilla rostrate)、红大麻哈鱼(Oncorhynchus nerka)、大西洋鲑(Pacific salmon)等过河口生殖洄游鱼类的洄游中所起的重要作用已逐渐被证实[19—21]。鱼类嗅觉依靠水中气味分子如氨基酸、核苷酸、类固醇、前列腺素和胆汁酸等诱导作用于嗅觉受体蛋白而引起[16,22,23]。嗅觉受体主要在嗅觉上皮组织中表达, 气味分子与嗅觉受体蛋白结合、识别并引发化学信号, 通过嗅神经传送到神经中枢从而实现嗅觉识别[24], 嗅觉受体蛋白则由嗅觉受体基因所编码[25—27]。已发现哺乳动物嗅觉受体有在进化上相互独立的4个家族, 分别为主嗅觉受体(Main olfactory receptors, MORs)、犁鼻器受体(Vomeronasal receptors, VRs)、痕量胺相关受体(Trace-amine associated receptors, TAARs)和类甲酰肽受体蛋白(Formyl peptide receptor-like proteins)[28]。主嗅觉受体是数量最大的嗅觉受体基因亚家族, 最早发现于褐家鼠(Rattus norvegicus), 编码区长度一般650 bp左右, 无内含子[29,30], 可识别水溶性气味分子[26,29,31]。主嗅觉受体基因目前已在一些模式鱼类, 如斑马鱼(Danio rerio)、红鳍东方鲀(Takifugu rubripes)、绿斑河鲀(Tetraodon nigroviridis)、三刺鱼(Gasterosteus aculeatus)、青鳉(Oryzias latipes)等[22,26,30,32,33]开展过研究, 但在非模式物种中研究尚少。在对多个刀鲚嗅囊转录组进行测序分析的基础上, 我们已初步筛选出洄游型与定居型刀鲚差异表达的几个MOR基因[16,22]。本研究克隆出其中的一个MOR基因, 分析了基因结构及在不同组织中的表达, 旨在弄清其在刀鲚两个生态型中的序列差异, 为长江刀鲚生殖洄游的嗅觉定向机制和生态型演化研究提供一些线索。
1 材料与方法
1.1 样本采集
实验所用刀鲚定居型样本为2014年3月采自江西鄱阳湖都昌水域, 洄游型样本2014年5月采自长江靖江江段, 都是2龄、性腺发育Ⅲ期的个体, 标注为定居PY-1-PY-6, 洄游JJ-1-JJ-6。所有样本都用定置刺网捕获, 尚未死亡的个体立即包埋于-20℃医用冰袋中, 使其失去知觉。在现场迅速用剪刀剖开腹部, 检视性腺发育状况, 对性腺发育Ⅲ期的个体进行实验取样[31]。定居型样本取嗅囊、精巢、卵巢、肌肉、心脏、眼睛、胃壁、肝脏和腮等组织或器官, 洄游型样本仅取嗅囊, 保存于液氮。返回实验室以后, 根据刀鲚矢耳石重量与年龄的对应关系[10,13], 留用2龄个体作为实验样本。实验开始前取出, 置-80℃保存备用。
1.2 刀鲚基因组DNA及总RNA的提取
利用动物组织基因组DNA小量提取试剂盒(上海莱枫生物科技有限公司)并按说明书提取洄游(JJ-1-JJ-6)、定居(PY-1-PY-6)刀鲚肌肉基因组DNA, 保存于-20℃冰箱。用EastepTM总RNA提取试剂盒(上海普洛麦格生物产品有限公司)提取定居型刀鲚组织或器官及洄游型刀鲚(雌/雄)嗅囊的总RNA, 进行3个重复。提取后的基因组DNA和总RNA用1%琼脂糖凝胶电泳进行检测, 判断其完整性; Thermo Nandrop2000分光光度计测RNA A260/ A280值, 评价纯度, 估测浓度大小。产物置于-80℃冰箱保存备用。
1.3 刀鲚cDNA文库的建立
使用PrimeScriptTMII 1st Strand cDNA Synthesis Kit试剂盒及其方法(宝生物工程有限公司), 以Oligo dT Primer为引物对洄游型刀鲚嗅囊总RNA进行普通反转录, 获得第一链cDNA, 为后续判断MOR-51I2基因是否含有内含子做准备。使用PrimeScriptTMRT reagent Kit with gDNA Eraser (Perfect Real Time)试剂盒(同上), 以RT Primer Mix为引物获得反转录cDNA, 为后续qRT-PCR反应做准备。产物保存于-20℃冰箱备用。
1.4 刀鲚MOR-51I2基因序列的获取
依据本实验室前期从洄游型刀鲚嗅囊cDNA文库中获得的MOR-51I2基因序列片段, 参照Genome Walking Kit试剂盒(宝生物工程有限公司)中对特异性引物的设计要求, 设计出获取3′序列的特异性引物(3′-SP1、3′-SP2和3′-SP3, 表 1)和获取5′序列的特异性引物(5′-SP1、5′-SP2和5′-SP3, 表 1)(引物均由上海生工生物工程股份有限公司合成)。优化反应体系和反应条件, 利用试剂盒中AP2简并引物和特异性引物SP1/SP2/SP3进行3轮热不对称PCR反应。反应产物用1%琼脂糖凝胶电泳检测、切胶回收和克隆测序(序列均由上海迈浦生物科技有限公司测序)。最终获得基因片段的侧翼序列, DNAMAN 6.0软件拼接后获得目的基因序列。

表 1 引物序列Tab. 1 Primer sequences
1.5 刀鲚MOR-51I2基因开放阅读框的获取
(2)合理利用区块链技术。区块链技术是一种互联网数据库技术,其特点是去中心化、不可纂改性、公开透明,让每个人均可参与数据库记录。区块链技术凭借其去中心化的特点,能够建立一个基于网络的公共账本,每一个区块包含一次网络交易,从而构建一个更加可靠的网络系统,从根本上解决网络交易中欺诈和“寻租”现象。因此,相关机构应合理利用区块链技术,进一步降低当前电子支付存在的病毒侵袭、黑客攻击、支付数据更改、资金无故流失等风险,健全信用体系,保障用户权益。
利用NCBI网站(http://www.ncbi.nlm.nih.gov/)中Blastx工具, 对刀鲚MOR-51I2基因序列进行比对,寻找同源性最高的基因序列, 再用jellyfish软件预测该基因的开放阅读框区域。根据预测结果, 在MOR-51I2基因的开放阅读框两侧设计特异性引物(ORF-R/ORF-F, 表 1), 进行基因序列的验证, 判断是否具有内含子并获取基因的开放阅读框。以洄游型刀鲚基因组DNA和嗅囊cDNA为模板, 进行PCR反应。反应体系: 模板<1 μg, ORF-R (10 μmol/L) 0.5 μL, ORF-F (10 μmol/L) 0.5 μL, 10×Taq Plus Buffer 2.5 μL, dNTP Mixture (2.5 mmol/L) 2 μL, Taq Plus DNA Polymerase (2.5 U/μL) 0.5 μL, 加ddH2O至25 μL。反应条件: 95℃ 5min; 95℃ 45s, 62℃45s, 72℃ 1min 30s, 30个循环; 72℃ 10min; 10℃保存。对PCR反应产物进行1%琼脂糖凝胶电泳检测,合格产物外送测序。为获得具有完整开放阅读框的MOR-51I2基因, 利用ORF-R/ORF-F引物对洄游和定居刀鲚基因组DNA进行PCR反应, 反应体系和反应条件同上。检测后, 反应产物外送测序, 拼接后获得目的片段。
1.6 刀鲚MOR-51I2基因序列分析
利用VecScreen(http://www.ncbi.nlm.nih.gov/ tools/vecscreen/)在线去除上述测序结果的载体, 用DNAMAN6.0软件进行与引物的比对和拼接, Blastx软件进行基因序列的预测翻译及同源性分析。用Expasy网站(http://expasy.org/tools/)中的ProtParam工具(http://web.expasy.org/protparam/)分析蛋白质基本理化性质, ProtScale工具(http://ca.expasy. org/tools/protscale.html)分析蛋白质亲疏水性, TMpred工具(http://www.ch.embnet.org/software/ TMPRED_form.html) 分析蛋白质跨膜区, Predict-Protein工具(http://www.predictprotein.org/)预测蛋白质二维结构, SWISS-MODEL ((http://swissmodel. expasy.org)在线预测蛋白质的三级结构。最后通过NCBI中Nucleotide blast工具下载同源DNA序列、Protein Blast下载同源蛋白序列, 利用ClustalX软件进行序列比对, MEGA 6.0软件构建Neighbor-joining系统发育树。
1.7 刀鲚MOR-51I2基因的组织表达谱
用qRT-PCR反应分析MOR-51I2基因在定居型刀鲚嗅囊、精巢、卵巢、肌肉、心脏、眼球、胃壁、肝脏及鳃等10个组织或器官及洄游型刀鲚嗅囊的相对表达水平。在MOR-51I2基因编码区内按荧光定量PCR反应的要求, 设计一对特异性引物QOR-R/QOR-F (表 1)。内参基因引物为GAPDHR/GAPDH-F (表 1), 采用SYBR®Premix Ex TaqTM(Tli RNaseH Plus)试剂盒和Bio-rad CFX Connet实时定量PCR仪, 进行PCR反应。反应体系: SYBR®Premix Ex TaqTM(Tli RNaseH Plus)(2×) 10 μL, 正反向引物(10 μmol/L)各0.4 μL, ROX Reference Dye (50×) 0.4 μL, cDNA模板(<100 ng) 2.0 μL, 灭菌蒸馏水6.8 μL, 共20 μL。以内参基因引物作阳性对照,灭菌蒸馏水作阴性对照, 每个组织和对照样本均进行3个重复。反应条件: 95℃ 30s; 95℃ 5s, 60℃ 30s, 72℃ 32s, 40个循环; 并做溶解曲线。反应结束后获得并导出各样本的Ct (threshold cycle)值, 随后采用2-ΔΔCt法[34]进行数据分析和表达差异性分析。
1.8 刀鲚MOR-51I2基因中微卫星序列分析
由于测序所得的MOR-51I2基因序列发现在非编码区3′ UTR有一段微卫星重复序列, 因而依据微卫星片段两侧设计了引物(OR-R/OR-F, 表 1)。以洄游型和定居型刀鲚基因组DNA为模板, 进行PCR反应。反应体系: 模板<1 μg, OR-F (10 μmol/L) 0.5 μL, OR-R (10 μmol/L) 0.5 μL, 2×Taq Plus PCR MasterMix 12.5 μL, 加ddH2O至25 μL。反应条件: 95℃ 5min; 95℃ 45s, 63℃ 45s, 72℃ 30s, 30个循环; 72℃ 10min; 10℃保存。反应产物用1.5%琼脂糖凝胶电泳检测。
2 结果
2.1 刀鲚MOR-51I2的基因序列
2.2 刀鲚MOR-51I2的蛋白结构
经ProtParam和ProtScale在线分析, 发现刀鲚MOR-51I2基因序列所编码的蛋白质序列, 其理化性质为: 氨基酸残基332个、分子式为C1751H2724N430O443S21、相对分子质量为37560.7、理论pI值为9.45、正/负电荷残基数为29和15、蛋白质序列的N端为起始密码子所编码的Met残基、半衰期为30hours、不稳定系数(II)为31.27(<40, 表明此蛋白较稳定)以及总平均亲水性(GRAVY)为0.648。经Predict Protein在线预测工具, 发现刀鲚MOR-51I2蛋白具有9个蛋白质绑定位点、6个DNA绑定位点、7个核苷酸绑定位点(图 2)。功能区域主要集中在氨基酸序列的N端、TM5-TM6间非跨膜区以及羧基端。二级结构有55.72%为α-型螺旋、10.24% β-型螺旋以及34.04% β-型转角, 并无二硫键结构。经SWISS-MODEL在线预测, 该蛋白质的三级结构具有7个α-型螺旋跨膜结构(图 3)。
2.3 刀鲚MOR-51I2基因与其他物种相关基因的进化关系

图 1 洄游型刀鲚(JJ-5) MOR-51I2基因的核苷酸序列和氨基酸序列Fig. 1 The sequence of nucleotide and amino acid of MOR-51I2 gene in the migratory C. nasus (JJ-5)“=”. 起始密码子ATG、终止密码子TAA (“*”); TM1-7. 7个跨膜域; “-”. 为微卫星序列, 长120 bp“=”. The initiation codon ATG, the termination codon TAA (“*”); TM1-7. Seven transmembrane regions; “-”. Microsatellite sequence, 120 bp

图 2 MOR-51I2蛋白的功能区、二级结构类型和跨膜区Fig. 2 The protein function region, secondary structure type and transmembrane region of MOR-51I2 protein“Protein binding region”. 蛋白质绑定位点; “DNA binding region”. DNA绑定位点; “RNA binding region”. RNA绑定位点; “Helix”. α-型螺旋; “Strand”表示β-型螺旋; “Buried”. 镶嵌于生物膜内片段; “Exposed”. 非镶嵌于膜内的片段; “TM1-7”. 蛋白质的7个跨膜区域“Protein binding region”. Protein binding sites; “DNA binding region”. DNA binding sites; “RNA binding region”. RNA binding sites;“Helix”. α-helix; “Strand”. β-helix; “Buried”. The sequence that mosaiced in the biofilm; “Exposed”. The sequence that non-mosaiced in the biofilm; “TM1-7”. Seven transmembrane regions

图 3 MOR-51I2蛋白的三级结构预测图Fig. 3 The predicted three-dimensional structure of MOR-51I2 protein1—7表示蛋白质7个α-型螺旋跨膜结构1—7. Seven α-helix position

表 2 同源DNA序列Tab. 2 The homologous DNA sequences
利用NCBI中的nucleotide blast工具作在线预测, 发现刀鲚MOR-51I2基因序列与其他鱼类同源基因的同源性在66%—85% (表 2)。同一物种选取其中1个同源性最高的核酸序列进行FASTA格式下载, 用MEGA6.0构建Neighbour-joining进化树(图 4)。可见, 基于核酸序列所构建的进化树可分为两大支, 其中刀鲚MOR-51I2基因与大西洋鲱(Clupea harengus) OR51I2-like基因、墨西哥脂鲤(Astyanax mexicanus) OR4K15-like基因及斑点雀鳝(Lepisosteus oculatus) OR4K15-like基因处于同一大分支, 序列之间的同源性较高, 分别为85%、76%和74%。但采用刀鲚MOR-51I2基因所编码的氨基酸序列与其他鱼类的同源嗅觉受体蛋白的氨基酸序列(表 3), 所构建的NJ进化树(图 5)与基于核酸序列所构建的进化树(图 4)有较大的差别。
2.4 刀鲚MOR-51I2基因的组织表达
利用特异性引物QOR-R和QOR-F对定居型刀鲚的10种组织或器官及洄游型刀鲚(雌/雄)的嗅囊进行实时荧光定量PCR反应, 得出各样品的Ct值。以在胃壁中的表达量为参照值, 利用2-ΔΔCt法分析所得的结果见图 6。可见, MOR-51I2基因在肌肉、心脏和眼睛中几乎不表达; 在肝脏和鳃中的表达量也很低; 但在嗅囊和性腺中的表达量很高, 且嗅囊的表达量是性腺表达量的30—100倍。另外, 雌性嗅囊中的表达量约是雄性嗅囊的2倍, 而精巢中的表达量却约是卵巢中的2倍。
用同样方法对两种生态型刀鲚嗅囊中的表达量进行分析比较(图 7), 显示MOR-51I2基因在洄游型雄性嗅囊中的表达量约是其雌性嗅囊中的6倍、定居型雄性嗅囊中的3倍, 但洄游型雌性嗅囊中的表达量却仅是定居型雌性嗅囊中的1/5。
2.5 刀鲚MOR-51I2蛋白在两个生态型间的差异性分析
通过特异引物ORF-R/ ORF-F, 对洄游型刀鲚(JJ-1-JJ-6)和定居型刀鲚(PY-1-PY-6)基因组DNA进行PCR扩增。获得的DNA序列经DNAMAN软件整理, jellyfish软件预测翻译的蛋白质序列。经比对发现, 两个群体的MOR-51I2蛋白间的相似性为97.5%, 仅有若干氨基酸残基的变化。
2.6 刀鲚MOR-51I2基因的微卫星在不同生态型间的差异性
用特异引物OR-F/OR-R对洄游型(JJ-1-JJ-10)和定居型刀鲚(PY-1-PY-10)进行PCR扩增, 经1.5%琼脂糖凝胶电泳检测(图 8)并测序。结果表明, 定居型群体的微卫星片段长度长于洄游型群体, 但同一群体的不同个体间其长度也有一定的变化。分析显示, 洄游型群体内的一致性为86.91%, 定居型群体内为93.29%, 两个群体间仅82.66%。洄游型群体的微卫星组成平均为48.6(AC)、11.4(GC)、0.6(AG)、1.0(AA)及2.4(AT), 定居型群体则为57.0(AC)、12.6(GC)、1.8(AG)、1.0(AA)及4.4(AT), 可见定居型较洄游型多8.4组(AC)1.2组(GC)、1.2组(AG)和2.0组(AT)。

图 4 刀鲚MOR-51I2核酸序列及其他鱼类相关嗅觉受体基因序列的系统发育树Fig. 4 The phylogenetic tree based on nucleotide sequence of MOR-51I2 in Coilia nasus and related olfactory receptor in other fishes节点处的数值为1000次bootstrap检验的支持率; 系统树中刀鲚用▲标出; 下同The numbers at each node represent the popularity rating that was replicated after 1000 bootstrap iterations; ▲ in the figure represented C. nasus; the same applies below

表 3 同源蛋白序列Tab. 3 The homologous protein sequences
3 讨论
3.1 MOR-51I2基因结构及其进化关系
本研究通过染色体步移技术获得了一个刀鲚嗅觉受体基因, 经与NCBI比对, 发现该基因与大西洋鲱OR 51I2-like基因的同源性最高, 属于主嗅觉受体基因家族。由于目前对该类基因尚未有统一的命名方式, 因此我们将其命名为MOR-51I2。该基因序列长1653 bp, 开放阅读框区域长999 bp, 无内含子, 为单外显子结构, 可编码332个氨基酸残基。所编码的蛋白质有9个蛋白质绑定位点、6个DNA绑定位点和7个RNA绑定位点, 不含二硫键结构, 能够形成7个疏水性α-型螺旋跨膜结构, 是典型的G蛋白偶联受体。
核酸序列比较显示, 刀鲚MOR-51I2基因与大西洋鲱OR51I2-like基因、墨西哥脂鲤OR4K15-like基因及斑点雀鳝OR4K15-like序列之间的同源性较高, 亲缘关系较近, 这一结果基本符合鱼类目一级阶元的进化关系[35]。但刀鲚MOR-51I2基因所编码的氨基酸序列与其他同源氨基酸序列构建的系统树, 尽管与大西洋鲱及墨西哥脂鲤这一小分支上的亲缘关系并没有改变, 却与斑点雀鳝的亲缘关系发生了变化, 而进化上位于高等的红丽鱼却单独处于进化树的基部位置。因此, 基于核酸序列所构建的系统树可能更大程度地反映了物种之间的进化关系, 而基于氨基酸序列构建的系统树可能更多地体现了该基因在功能上的趋同关系。从基因的结构和长度、编码蛋白质的结构和性质看, 刀鲚MOR-51I2基因均与现有文献报道的鱼类MOR基因相符[30, 34]。

图 5 刀鲚MOR-51I2氨基酸序列与其他鱼类相关嗅觉受体蛋白的系统发育树Fig. 5 The phylogenetic tree based on amino acid sequence of MOR-51I2 in C. nasus and related olfactory receptor in other fishes
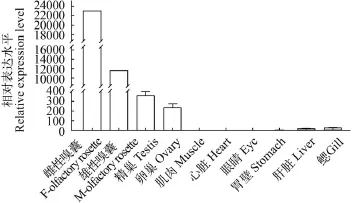
图 6 MOR-51I2基因在定居型刀鲚组织中的表达差异(n=3)Fig. 6 The expression of MOR-51I2 in organs/tissues of the settlement C. nasus (n=3)
3.2 MOR-51I2基因的组织表达
实时荧光定量PCR反应显示, MOR-51I2基因主要在嗅囊中表达, 在性腺中的表达量也很高, 而肌肉、心脏、眼睛、肝脏和鳃中几乎不表达或表达量很低。但雌性嗅囊中的表达量要高出雄性嗅囊约2倍, 而精巢中的表达量是卵巢中的约2倍。这表明MOR-51I2基因不但与嗅觉功能有关, 也可能参与了刀鲚的性腺发育, 在雌雄个体间又存在着差异。
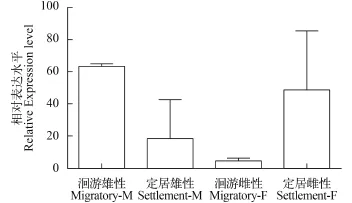
图 7 MOR-51I2基因在不同生态型刀鲚嗅囊中的表达差异(n=3)Fig. 7 The expression of MOR-51I2 in rosettes of the different ecotypes of C. nasus (n=3)
进一步分析发现, MOR-51I2基因在洄游型雄性嗅囊中的表达量约是同一生态型雌性嗅囊表达量的6倍, 但定居型雌性嗅囊中的表达量却约是同一生态型雄性嗅囊表达量的2.5倍。这预示着洄游型群体的雄性个体, 可能比雌性个体具有更强的识别水溶性气味分子的能力。定居型则相反, 雌性识别水溶性气味分子的能力可能明显强于雄性。分析还发现, 雄性嗅囊中的表达量洄游型要明显大于定居型, 而雌性则定居型要大于洄游型(图 7)。这是否预示着, 洄游型刀鲚的雄性个体在溯河生殖洄游中起着主导作用, 而定居型刀鲚的雌性个体在寻找产卵场时起主导作用?还需要克隆更多的基因加以证实。

图 8 刀鲚MOR-51I2基因中微卫星序列在不同生态型间的比较Fig. 8 Alignment of microsatellite sequence of olfactory receptor MOR-51I2 between the different ecotypes of C. nasusM代表Trans DNA MarkerⅠ; 泳道1—10代表洄游型刀鲚群体; 泳道11—20代表定居型刀鲚群体; 泳道21代表阴性对照(用ddH2O代替DNA模板)M. Trans DNA MarkerⅠ; 1—10. Migratory C. nasus; 11—20. Settlement C. nasus; 21. Negative control (ddH2O instead of DNA)
3.3 MOR-51I2基因的微卫星结构
此外, 在MOR-51I2基因紧邻终止密码子TAA的3′ UTR非编码区中有一段微卫星序列, 其中以(AC)n最丰富, 具有长度多态性, 在定居型和洄游型群体间具有明显的扩增差异带。前者的微卫星条带明显多于后者, 微卫星平均长度前者也高于后者。微卫星侧翼序列的突变和重复次数长度的变化, 包含着重要的物种进化信息[36,37]。本研究所显示的MOR-51I2基因表达量差异、微卫星序列变化以及核酸和蛋白构建的系统树差异, 是否与刀鲚定居型种群的形成和洄游习性的丧失相关, 还有待于进一步验证。
[1]Fish Research Laboratory Institute of Hydrobiology, Chinese Academy of Sciences. Fishes of Yangtze River [M]. Beijing: Science Press. 1976, 21—26 [湖北水生生物研究所鱼类研究室. 长江鱼类. 北京: 科学出版社. 1976, 21—26]
[2]Yuan C M, Lin J B, Qin A L, et al. The taxonomy history and current situation of fishes on Chinese Coilia and talking several realizations of reconstruction old taxonomy of fishes [J]. Journal of Nanjing University (Natural Sciences), 1976, (2): 1—12 [袁传宓, 林金榜, 秦安舲, 等. 关于我国鲚属鱼类分类的历史和现状-兼谈改造旧鱼类分类学的几点体会. 南京大学学报(自然科学版), 1976, (2): 1—12]
[3]Cheng Q Q, Lu D R. PCR-RFLP analysis of cytochrome b gene does not support Coilia ectenes taihuensis being a subspecies of Coilia ectenes [J]. Journal of Genetics, 2005, 84(3): 307—310
[4]Tang W Q, Hu X L, Yang J Y. Species validities of Coilia brachygnathus and C. nasus taihuensis based on sequence variations of complete mtDNA control region [J]. Biodiversity Science, 2007, 15(3): 224—231 [唐文乔, 胡雪莲, 杨金权. 从线粒体控制区全序列变异看短颌鲚和湖鲚的物种有效性. 生物多样性, 2007, 15 (3): 224—231]
[5]Cheng Q Q, Han J D. Morphological variations and discriminant of two populations of Coilia ectenes [J]. Journal of Lake Science, 2004, 16(4): 356—364 [程起群, 韩金娣. 鲚属两种群的形态变异及综合判别(英文). 湖泊科学, 2004, 16(4): 356—364]
[6]Cheng W X, Tang W Q. Some phenotypic varieties between different ecotypes of Coilia nasus in Yangtze River [J]. Chinese Journal of Zoology, 2011, 46(5): 33—40 [程万秀, 唐文乔. 长江刀鲚不同生态型间的某些形态差异. 动物学杂志, 2011, 46(5): 33—40]
[7]Wang D T, Yang J, Jiang T, et al. A comparative study of the morphology of different geographical populations of Coilia nasus [J]. Journal of Fisheries of China, 2012, 36(1): 78—90 [王丹婷, 杨健, 姜涛, 等. 不同水域刀鲚形态的分析比较. 水产学报, 2012, 36(1): 78—90]
[8]Hsu W N, Sun C B, Tong Y R, et al. The biological indicator of the spawning migration of anchovy (Coilia ectenes Joroan et Seale) in Yangtze River [J]. Journal of Nanjing University (Natural Sciences), 1978, 3: 85—91 [徐艿南, 孙超白, 童远瑞, 等. 长江流域刀鲚鱼生殖洄游的“生物指标”. 南京大学学报:自然科学版, 1978, 3: 85—91]
[9]Li W X, Wang G T. Helminth communities in Coilia nasus from anadromous, freshwater and landlocked stocks [J]. Chinese Journal of Zoology, 2014, 49(2): 233—243 [李文祥, 王桂堂. 洄游型、淡水型和陆封型刀鲚的寄生蠕虫群落结构. 动物学杂志, 2014, 49(2): 233—243]
[10]Guo H Y, Wei K, Tang W Q, et al. Sibling species discrimination for Chinese genus of Coilia fishes based on sagittal otolith morphology [J]. Acta Zootaxonomica Sinica, 2010, 35(1): 127—134 [郭弘艺, 魏凯, 唐文乔, 等.基于矢耳石形态特征的中国鲚属鱼类种类识别. 动物分类学报, 2010, 35(1):127—134]
[11]Yang J, Arai T, Liu H, et al. Reconstructing habitat use of Coilia mystus and Coilia ectenes of the Yangtze River estuary, and of Coilia ectenes of Taihu Lake, based on otolith strontium and calcium [J]. Journal of Fish Biology, 2006, 69(4): 1120—1135
[12]Jiang T, Zhou X Q, Liu H B, et al. Two microchemistry patterns in otoliths of Coilia nasus from Poyang Lake, China [J]. Journal of Fisheries of China, 2013, 37(2): 239—244 [姜涛, 周昕期, 刘洪波, 等. 鄱阳湖刀鲚耳石的两种微化学特征. 水产学报, 2013, 37(2): 239—244]
[13]Li Y X, He W P, Liu J S, et al. Annulus validation and age and growth estimation of anadromous Coilia ectenes in the Yangtze estuary [J]. Acta Hydrobiologica Sinica, 2010, 34(4):787—793 [黎雨轩, 何文平, 刘家寿, 等. 长江口刀鲚耳石年轮确证和年龄与生长研究. 水生生物学报, 2010, 34(4): 787—793]
[14]Wang L, Tang W Q, Dong W X. The signatures of stable isotopes delta N-15 and delta C-13 in anadromous and non-anadromous Coilia nasus living in the Yangtze River, and the adjacent sea waters [J]. Journal of Ocean University of China, 2015, 14(6): 1053—1058
[15]He W P, Cheng F, Li Y X, et al. Molecular identification of Coilia ectenes and Coilia mystus and its application on larval species [J]. Acta Hydrobiologica Sinica, 2011, 35(4): 565—571 [何文平, 程飞, 黎雨轩, 等. 刀鲚和凤鲚的分子鉴定及其在仔鱼种类鉴定中的应用. 水生生物学报, 2011, 35(4): 565—571]
[16]Zhu G L, Wang L J, Tang W Q, et al. De novo transcriptomes of olfactory epithelium reveal the genes and pathways for spawning migration in Japanese grenadier anchovy (Coilia nasus) [J]. PLoS One, 2014, 9(8): e103832
[17]Zhu G L, Tang W Q, Wang L J, et al. Identification of a uniquely expanded V1R (ORA) gene family in the Japanese grenadier anchovy (Coilia nasus) [J]. DOI: 10.1007/s00227-016-2896-9
[18]Hasler A D, Wisby W J. Discrimination of stream odors by fishes and its relation to parent stream behavior [J]. The American Naturalist, 1951, 85(823): 223—238
[19]Barbin G P, Parker S J, McCleave J D. Olfactory clues play a critical role in the estuarine migration of silverphase American eels [J]. Environmental Biology of Fishes, 1998, 53(3): 283—291
[20]Hino H, Iwai T, Yamashita M, et al. Identification of an olfactory imprinting-related gene in the lacustrine sockeye salmon, Oncorhynchus nerka [J]. Aquaculture, 2007, 273(2—3): 200—208
[21]Dittman A H, Quinn T P. Homing in Pacific salmon: mechanisms and ecological basis [J]. Journal Experiment Biology, 1996, 199(Pt 1): 83—91
[22]Zhu G L, Tang W Q, Liu D. Research progress of olfactory receptor genes in fishes [J]. Journal of Fisheries of China, 2015, 39(6): 916—927 [朱国利, 唐文乔, 刘东. 鱼类嗅觉受体基因研究进展. 水产学报, 2015, 39(6): 916—927]
[23]Lai X J, Hong W S, Zhang Q Y. Research progresses in fish olfactory system and sex pheromonal receptors [J]. Chinese Journal of Zoology, 2013, 48(2): 298—305 [赖晓健, 洪万树, 张其永. 鱼类嗅觉系统和性信息素受体的研究进展. 动物学杂志, 2013, 48(2): 298—305]
[24]Hino H, Miles N G, Bandon H, et al. Molecular biological research on olfactory chemoreception in fishes [J]. Journal of Fish Biology, 2009, 75(5): 945—959
[25]Liu D, Zhang Z L, Zhao Y H, et al. Advancements in the morphological and physiological research on the olfactory organ of fishes [J]. Chinese Journal of Zoology, 2005, 40(6): 122—128 [刘东, 张振玲, 赵亚辉, 等. 鱼类嗅觉器官的形态与生理研究进展. 动物学杂志, 2005, 40(6): 122—128]
[26]Niimura Y, Nei M. Evolutionary dynamics of olfactory receptor genes in fishes and tetrapods [J]. Proceedings of the National Academy of the Sciences, 2005, 102(17): 6039—6044
[27]Ngai J, Dowling M M, Buck L, et al. The family of genes encoding odorant receptors in the channel catfish [J]. Cell, 1993, 72(5): 657—666
[28]Zhou Y S, et al. Family structure and phylogenetic analysis of odorant receptor genes in the large yellow croaker (Larimichthys crocea) [J]. BMC Evolutionary Biology, 2011, 11(1717): 237
[29]Zhao H Q, Firestein S J. Vertebrate odorant receptors [J]. Cellular and Molecular Life Sciences, 1999, 56: 647—659
[30]Zhang X H, Firestein S. Genomics of olfactory receptors [J]. Results and Problems in Cell Differentiation, 2009, 47: 25—36
[31]Niimura Y. On the origin and evolution of vertebrate olfactory receptor genes: comparative genome analysis among 23 chordate species [J]. Genome Biology Evolution, 2009, 1: 34—44
[32]Alioto T S, Ngai J. The odorant receptor repertoire of teleost fish [J]. BMC Genomics, 2005, 6: 173
[33]Wellerdieck C, Oles M, Pott L, et al. Functional expression of odorant receptors of the zebrafish Danio rerio andof the nematode C-elegans in HEK293 cells [J]. Chemical Senses, 1997, 22(4): 467—476
[34]Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-∆∆Ctmethod [J]. Methods, 2001, 25(4): 402—408
[35]Nelson J.S. Fishes of the World [M]. Wiley-Blackwell. 2006, 624
[36]Liu Z J, Jensen J W. Aquaculture has Great Expectations of the Genome Revolution [C]. In: Celebration of the 50 Years of DNA, A Commemorative Book for the discovery of the DNA structure by Francis Crick and James Watson. London: Cambridge. 2003, 50—52
[37]Zardoya R, Vollmer D M, Craddock C, et al. Evolutionary conservation of microsatellite flanking regions and their use in resolving the phylogeny of cichlid fishes (Pisces: Perciformes) [J]. Proceedings of the Royal Society B-biological Sciences, 1996, 263(1376): 1589—1598
CLONING, SEQUENCE ANALYSIS AND TISSUES EXPRESSION OF COILIA NASUS OLFACTORY RECEPTOR GENE MOR-51I2
WANG Xiao-Mei1,2, ZHU Guo-Li1,2and TANG Wen-Qiao1,2
(1. Laboratory of Fishes, Shanghai Ocean University, Shanghai 201306, China; 2. Shanghai Key Laboratory of Marine Animal Taxonomy and Evolution, Shanghai 201306, China)
Olfaction is an important tool for fish to perceive the external environment. To study the role of olfactory receptors in the spawning migration of Coilia nasus, the full-length sequence of olfactory receptor gene MOR-51I2 was cloned from the migratory C. nasus using genome walking technology. The MOR-51I2 gene was composed of a single exon with the open reading frame of 999 bp. The 3′ untranslated region of this gene had a microsatellite sequence formed by (AC)n inserted by several T and G. In addition, the microsatellite sequences in different ecotype of C. nasus had significant length difference. The MOR-51I2 was a G-protein-coupled receptor with seven hydrophobic alphahelical transmembrane structures. The MOR-51I2 protein shared homology (>51%) with known related olfactory receptors from other fishes and it reached 83% homology with the olfactory receptor 51I2-like protein in Clupea harengus. The MOR-51I2 gene was highly expressed in the female olfactory rosettes of the settlement population of C. nasus, which is two times compared with that in male olfactory rosettes and 80 to 100 times compared with that in testis and ovary. The MOR-51I2 gene was expressed weakly in liver and gills and was almost not detected in the muscle, heart and eye. The expression level of female olfactory rosettes was 5 times higher than that in male olfactory rosettes in the migratory population. The expression level in female olfactory rosettes of the settlement population was 5 times higher than that in female olfactory rosettes in the migratory population, while the expression level in the male olfactory rosettes of the migratory population was 2 times higher than that in male olfactory rosettes in the settlement population. These results suggest that MOR-51I2 gene may not only regulate the olfactory function of C. nasus, but also mediate gonad development, spawning migration, and ecological differentiation of C. nasus.
Coilia nasus; Olfactory receptor gene; Tissues expression difference; Spawning migration; Ecotype differentiation
Q344+.1
A
1000-3207(2017)01-0033-10
10.7541/2017.5
2016-01-12;
2016-05-28
公益性行业(农业)科研专项(201203065); 国家自然科学基金(31172407, 31472280); 高等学校博士学科点科研专项(2012 3104110006); 上海高校水产学高峰学科资助 [Supported by the Special Fund for Agro-Scientific Research in the Public Interest (201203065); the National Natural Science Foundation of China (31172407, 31472280); the Ministry of Education's Doctoral Discipline Foundation (20123104110006); Shanghai Universities First-class Disciplines Project of Fisheries]
王晓梅(1989—), 女, 安徽亳州人; 硕士; 主要从事鱼类生殖洄游研究。E-mail: xmw0212@163.com
唐文乔, 教授, 博士生导师。E-mail: wqtang@shou.edu.cn
