微滴式数字聚合酶链式反应精准定量检测羊肉中掺杂猪肉
2017-02-08任君安邓婷婷黄文胜葛毅强
任君安,邓婷婷,黄文胜,葛毅强,陈 颖,*
微滴式数字聚合酶链式反应精准定量检测羊肉中掺杂猪肉
任君安1,2,邓婷婷2,黄文胜2,葛毅强1,3,陈 颖2,*
(1.中国农业大学食品科学与营养工程学院,北京 100083;2.中国检验检疫科学研究院,北京 100176;3.中国农村技术开发中心,北京 100045)
以羊和猪的单拷贝持家基因DNA复制蛋白A1为靶基因设计合成了适用于微滴式数字聚合酶链式反应的特异性引物和探针,通过理论推导获得了单位质量两种肉基因拷贝数之比的固定值,并进行了验证,据此将样品中羊肉和猪肉的拷贝数转换为相对质量分数,从而建立了羊肉中掺杂猪肉的精准定量检测方法。该方法可以很好地应用于羊肉中掺杂猪肉的含量检测,猪肉的最低定量限为1%,在5%~80%范围内绝对误差小于±1.30%,相对误差小于±10%,定量结果准确、重复性高,可以为肉类掺假的监管工作提供有力的技术参考。
羊肉;猪肉;精准定量;微滴式数字聚合酶链式反应
近年来,世界各地肉制品掺假情况频繁发生,2013年,欧洲多国在部分牛肉产品中检出马肉成分,爆发了所谓的马肉风波[1]。在我国,随着生活质量的提高,人们对动物蛋白的需求也日益增加,食用蛋白质含量相对较高的牛羊肉成了新的发展趋势[2]。与此同时,肉类产品标注信息与实际不符的情况也随之而来,如用猪肉等营养价值较低的廉价肉类掺入或代替羊肉等[3]。这种掺假行为不仅扰乱肉业市场秩序,损害消费者利益,甚至可能侵犯消费者的宗教信仰[4-5]。为保护消费者权益,全球不同国家和地区对肉制品的标识做了一系列规定。欧盟定量成分声明强制要求肉制品需要标识出所有成分及其净含量,并规定肉的主要成分为骨骼肌,结缔组织和脂肪等成分需单独列出且规定了最高含量,这是最严格的肉类成分标识制度[6]。目前,肉制品中动物物种成分的检测多集中在定性或半定量检测方面[7-10],然而随着掺假手段的不断变化,实际应用中对肉制品真实含量的检测需求却越来越多,定量检测逐渐成为了新的发展趋势。Iwobi等[11]通过构建哺乳动物和禽类的内标基因,以物种特异基因和通用内标基因的Ct值比值完成对不同物种的定量检测。López-Andreo等[12]通过构建等质量的多种肉混合样品,进行了混合样品中各种肉质量分数的定量。然而这些方法均采用的是实时荧光聚合酶链式反应(polymerase chain reaction,PCR)技术,通过扩增获得的Ct值推导出各个基因的拷贝数,并以此转化为肉的质量。但该Ct值受PCR扩增效率及DNA溶液中杂质的影响,由此值推导出的基因拷贝数与样品中的实际基因拷贝数偏差较大,从而加大了定量结果的偏差。
微滴式数字PCR(droplet digital PCR,ddPCR)是近几年在实时荧光PCR基础上发展起来的定量技术,可直接测得样品中目标基因的绝对拷贝数,目前已应用于基因表达量[13]、微生物丰度[14]、转基因产品含量[15]及食品成分的定量检测[16]。René等[17]以转基因大豆为样品,比较了多重实时荧光PCR反应与微滴数字PCR反应,发现微滴数字PCR定量的不确定度低于17%,效果优于实时荧光PCR。
目前,已有学者将ddPCR应用于肉类掺假的定量[18-19],然而,肉类定量检测难点在于如何将PCR扩增得到的基因拷贝数转换成肉的质量。这是因为不同种肉的细胞密度(每克组织中的细胞数量)和基因组大小不同,即便是等质量不同肉种其DNA得率(基因拷贝数)也不同。若直接将其拷贝数之比作为质量之比,结果会出现较大偏差[20]。Cai Yicun[18]和苗丽[19]等采用数字PCR方法,先将基因拷贝数转换为DNA质量,再从DNA质量换算为肉的质量。由于该方法需要建立两组标准曲线,实验步骤繁琐、工作量大,且进行两步转化,实验误差加大。
本实验以单拷贝持家基因D N A复制蛋白A(replication protein A,RPA1)为靶基因,开展了猪肉、羊肉掺假的ddPCR定量检测研究。通过理论推导获得单位质量两种肉的基因拷贝数之比的固定值,通过实验验证了不同含量肉种该值的稳定性,并利用该值实现了基因拷贝数比与质量比的转换,建立了羊肉中掺杂猪肉成分的精准定量检测方法,以期为肉制品掺假定量检测标准的制定,为肉制品市场监管提供技术支撑。
1 材料与方法
1.1 材料与试剂
羊、牛、驴、猪、鸡、鸭、鹅、火鸡、兔等肉类样品以及羊肉串、羊肉饺子、羊肉卷、酱羊蹄、羊肉肠和孜然羊肉粒 市售;狐狸、狗、马等样品来自本实验室。
扩增预混液(ddPCRTMSupermix for probes)、微滴生成专用油、微滴生成卡 美国Bio-Rad公司;引物和探针 英潍捷基(上海)贸易有限公司;其他常用化学试剂均为国产分析纯 北京化学试剂公司。
1.2 仪器与设备
Bio-Rad QX200 ddPCR生成仪和读数仪、T100 PCR仪美国Bio-Rad公司;高速离心机 美国Thermo公司;组织研磨机 美国Qiagen公司;紫外分光光度计 德国Eppendorf公司;电子天平 北京赛多利斯科学仪器有限公司;绞肉机 上海西贝乐电器公司。
1.3 方法
1.3.1 样品制备
猪羊混合样品制备:取羊和猪的背长肌,分别用绞肉机绞碎。称取猪肉20 g和羊肉5 g,将两者均匀混合配制成含有80%猪肉成分的羊肉样品;将上述样品与羊肉按照3∶1的比例混合配制成含有60%的猪肉成分的羊肉样品。同样地,依次配制成含有50%、30%、10%、5%和1%猪肉成分的羊肉样品,参照Wang Wei等[21]的方法提取DNA。
市售羊肉样品制备:称取羊肉串、羊肉饺子馅、羊肉卷、羊肉肠和孜然羊肉粒各20 g,用绞肉机绞碎,从中称取200 mg,参照Wang Wei等[21]的方法提取DNA。
1.3.2 引物与探针的设计
根据GenBank中公布的牛、驴、鸡、鸭、鹅、火鸡、兔、狐狸、狗、马、山羊、绵羊和猪等13个物种的持家基因RPA1基因DNA序列,进行同源序列比对,运用Primer Express 5.0软件,在羊和猪的特异性区域设计数字PCR引物和探针。引物和探针序列见表1。
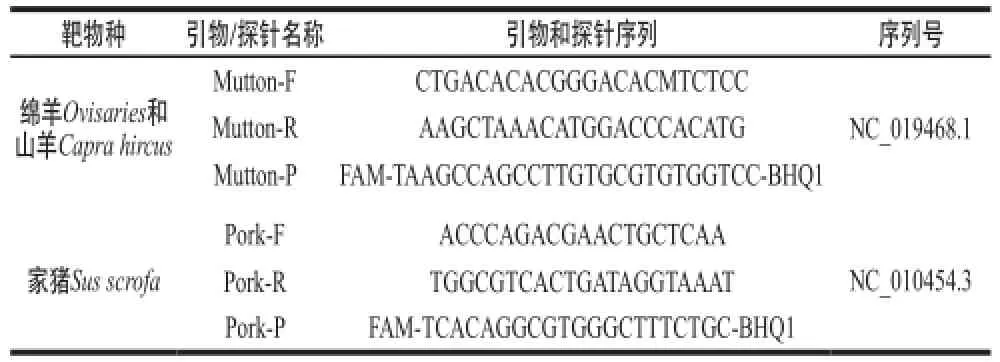
表1 引物和探针序列Table1 Primer and probe sequences for ddPCR assays
1.3.3 数字PCR检测
ddPCR反应体系为:ddPCRTMSupermix for Probes 10 μL,上、下游引物各1 μL(10 μmol/L),探针0.5 μL(10 μmol/L),DNA模板(10~100 ng/μL)5 μL,加双蒸水补足总体积至20 μL。反应体系充分混匀后加入微滴生成卡中,油包水微滴生成按照厂家的操作说明进行。
将生成的微滴全部转入96 孔PCR反应板中,进行扩增。PCR包括3 个步骤:95 ℃预变性10 min;94 ℃变性30 s,60 ℃退火1 min,45个循环;98 ℃固化微滴10 min。最后用微滴分析仪对扩增产物进行计数分析。
1.3.4 单位质量羊肉与猪肉基因拷贝数比值的确定和验证
肉的质量与样品DNA溶液中RPA1的基因拷贝数及单位质量的基因拷贝数有关,如式(1)所示:

式中:M为肉的质量;Q为基因拷贝数;C为单位质量肉的基因拷贝数。因此,不同肉种的质量比则应为式(2):

式中:Mp和Mm分别为猪肉和羊肉的质量;Qp和Qm分别为混合样品中猪肉和羊肉的基因拷贝数;Cp和Cm分别为单位质量猪肉和羊肉的基因拷贝数。
由于同一肉种的细胞密度和基因组大小是固定的,因此,对任何肉种而言,其Cm/Cp均为常数。只要测得混合样品中各成分的基因拷贝数,结合Cm/Cp这一常数,即可计算出混合肉样中各组分的比例。
为计算Cm/Cp,采用数字PCR分别定量检测猪肉含量为1%、10%、50%、60%和80%的羊肉样品中两种肉的RPA1基因拷贝数,并计算Cm/Cp和相对标准偏差(relative standard deviation,RSD)。
3.身体和环境是认知的构成。传统认知心理学并不否认环境在认知过程中的作用,但具身认知理论认为身体和世界在认知加工中扮演了某种构成性的(constitutive)的角色,而不仅仅是因果作用的角色[2],即身体和环境不仅仅是认知的因果关系,更是认知的构成部分,其造就了某种认知的结果。
为进一步确定Cm/Cp在单一质量下的准确性和稳定性,称取6 份50%猪肉含量的羊肉样品,根据1.3.1节进行样品处理并提取基因组DNA,通过建立的数字PCR扩增体系,测得猪肉和羊肉的RPA1基因拷贝数,每份混合样品重复3 次,分别取猪肉和羊肉基因拷贝数的平均值计算其比值,并以此计算Cm/Cp和RSD。
1.3.5 定量线性范围和方法的重复性和准确性验证
采用1.3.4节确定的单位质量下羊肉与猪肉基因拷贝数之比,将数字PCR测得的猪肉含量为1%~80%的样品中猪肉和羊肉的拷贝数分别转换为质量分数,并以测量值和真实值做线性范围关系曲线,并计算其相关系数R2。
以10%、5%和1%猪肉含量的羊肉样品为模拟样品,每个样品重复6 次,同时检测猪肉和羊肉RPA1基因拷贝数,并通过确定的Cm/Cp将其转化为质量分数,计算各样品6 次重复的平均值、RSD、绝对误差和相对误差,确定定量方法的准确性和重复性。
1.3.6 市售羊肉样品的检测
14 份市售羊肉样品(包括羊肉串、羊肉饺子、羊肉卷、羊肉肠、孜然羊肉粒)提取DNA,用建立的ddPCR扩增体系,测得样品中羊和猪RPA1基因拷贝数,并通过1.3.4节确定的单位质量下猪肉与羊肉拷贝数的比值计算其质量百分比。
2 结果与分析
2.1 引物探针特异性实验

图1 羊特异性引物探针数字PCR筛选Fig. 1 Screening of mutton- and goat-specific primers and probes
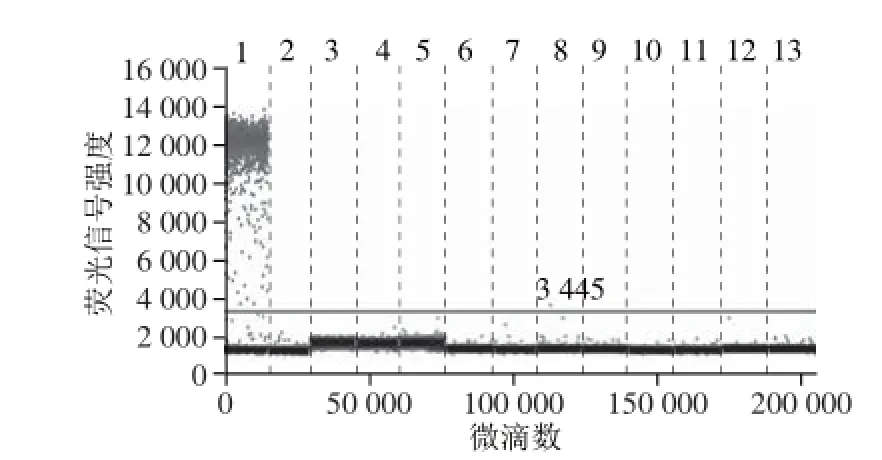
图2 猪特异性引物探针数字PCR筛选Fig. 2 Screening of pork-specific primers and probes
通过数字PCR对羊肉、猪肉等13 种肉类的DNA扩增结果显示,羊的引物探针特异性良好,可以同时扩增出绵羊肉和山羊肉,且其他肉类样品均没有扩增(图1);猪肉的引物探针只对目标样品猪肉有扩增,其余12 种肉类样品均没有扩增(图2)。以上结果说明表1中的引物和探针均能有效扩增靶标物种的RPA1基因,且对其他非目标物种无扩增。两组引物探针的特异性良好,可以用于后续的ddPCR定量检测实验。
分别检测猪肉含量为1%、10%、50%、60%和80%的5 个样品中猪、羊肉的RPA1基因拷贝数,并计算猪肉与羊肉二者的质量比与基因拷贝数之比的比值(表2)。结果表明,5 个不同猪肉含量样品的Cm/Cp平均值为1.84,RSD为3.50%,Cm/Cp的稳定性较高。因此实验证明,无论羊肉和猪肉含量如何变化,Cm/Cp一直为1.84稳定不变。
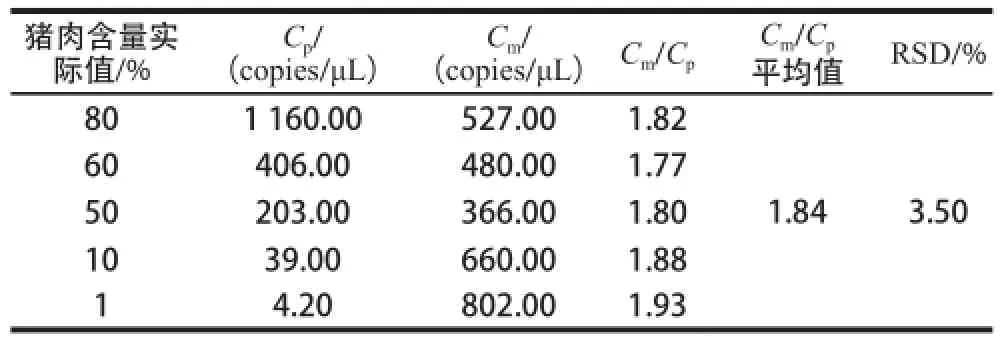
表2 不同猪肉含量时的Cm与Cp及Cm/CpTable2 The ratio of copy numbers of unit mass in different pork proporttiioonnss
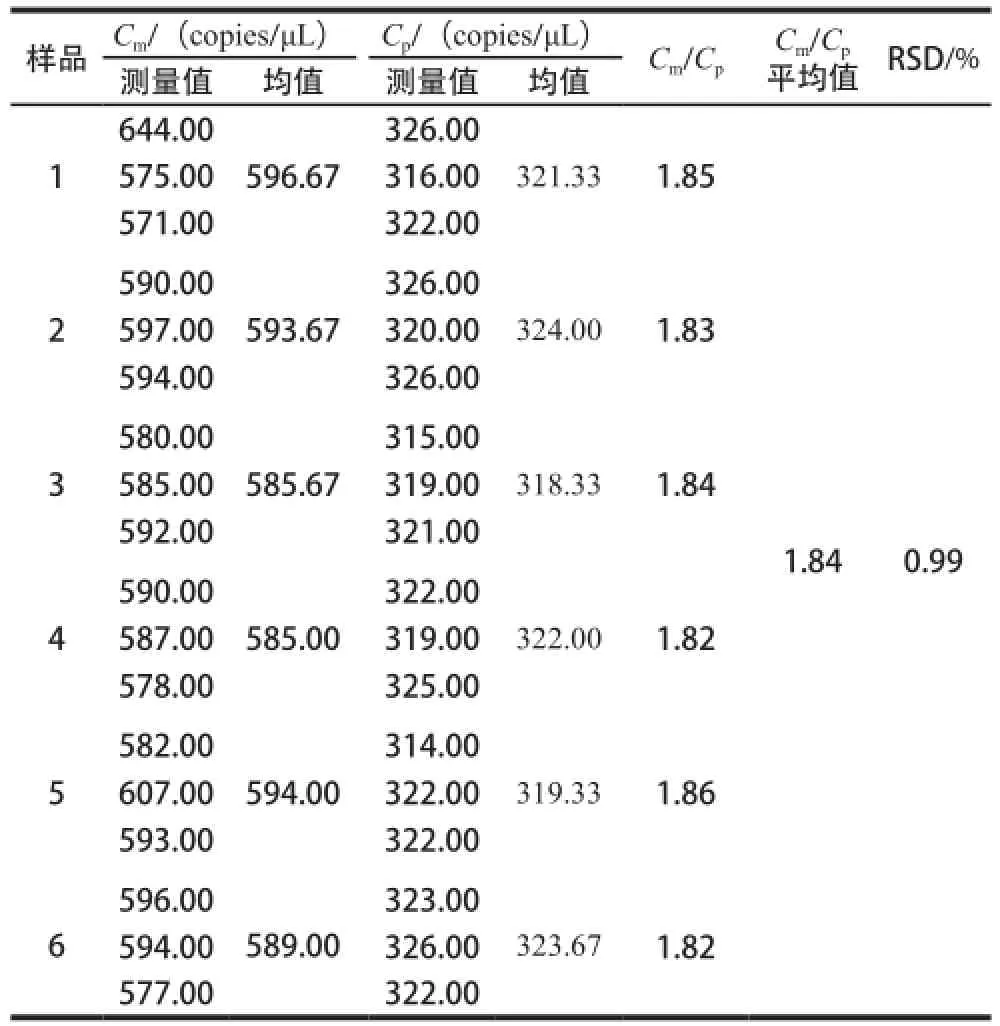
表 33 5500%猪肉含量时Cm和Cp及Cm/CpTable3 Copy numbers per unit mass of pork and mutton and their ratios att 5500% pork content
为进一步验证Cm/Cp的准确性和稳定性,采用建立的数字PCR体系,分别检测6 份50%猪肉含量的平行样品中猪肉和羊肉RPA1基因拷贝数的3 次均值,计算等质量条件下两种肉基因拷贝数的比值Cm/Cp,并计算6 份平行样品的Cm/Cp均值。结果显示,单位质量下羊肉与猪肉拷贝数之比为1.84,6份平行样品的结果稳定,RSD在1%以内,没有显著性差异(P>0.05)(表3)。因此,不论羊肉与猪肉含量产生变化还是单一质量多次重复,单位质量条件下Cm/Cp均为1.84,利用该比值可以将基因拷贝数转换为质量分数。
2.3 定量线性范围、准确度和重复性实验结果
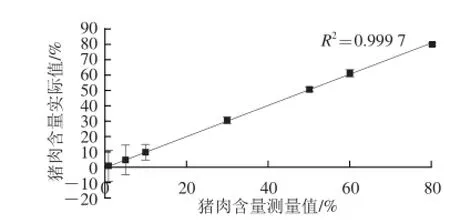
图3 猪、羊混合模拟样品中猪肉含量的定量线性范围Fig. 3 Linear quantitation range of pork content in binary mixture
为了验证方法的准确度和重复性,选择猪肉含量为10%、5%和1%的猪、羊低浓度混合样品进行定量准确度和重复性实验,每个样品重复检测6 次,结果见表4结果表明:猪肉含量为10%的样品实际测量平均值为9.96%,相对误差为-0.41%,RSD为4.59%;5%样品猪肉含量实际测量平均值为4.62%,误差为-7.51%,RSD为6.68%;1%样品猪肉含量实际测量平均值为1.17%,RSD为9.73%。猪肉含量为10%和5%的样品相对误差低于±7.51%,1%样品的相对误差为16.82%,由于目前掺假行为多是经济利益驱动型掺假,掺假含量较高,因此该方法符合常见掺假事件的定量检测。此外,3 组低含量样品中猪肉含量的RSD均小于10%,远低于国际食品法典委员会关于食品定性和定量检测方法标准指导方针中所规定的标准(<25%)[22],定量重复性符合食品打假执法的需要。
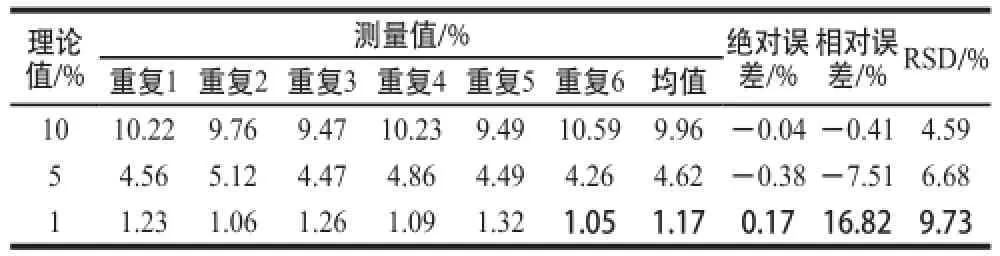
表4 羊肉中掺杂猪肉成分的定量准确度和重复性分析Table4 Quantitative accuracy and repeatability of pork content incorporated into mutton
2.4 市售羊肉制品的定量检测
采用本实验建立的方法检测市售羊肉串、羊肉馅、羊肉卷、羊肉馅饺子以及羊肉肠等14 份标识为纯羊肉制品中的羊肉和猪肉的相对含量,结果表明所有样品经DNA提取和微滴数字PCR扩增后,均能够检测出羊肉和/或猪肉含量(表5)。羊肉馅、羊肉串、羊肉馅饺子和羊肉肠中检测出猪肉成分,占样品总数的35.7%。其中,12号样品羊肉肠中未见羊肉成分,只有猪肉,1号和7号样品中除了羊肉,还添加有20%~60%的猪肉。11号和14号样品检出的猪肉含量低于本方法的定量低限,推断可能是污染导致。
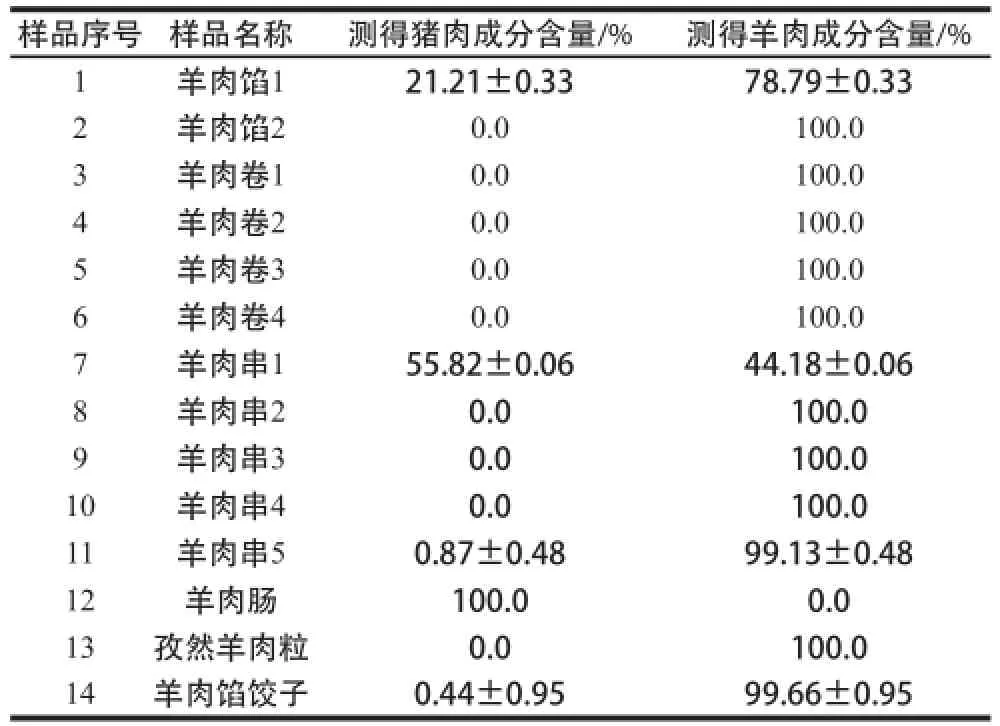
表5 市售羊肉制品中羊肉和猪肉成分相对含量的检测结果Table5 The relative contents of pork and mutton in commercial samples assayed by the ddPCR method
3 讨 论
数字PCR在定量检测方面精准度更高,与基于标准曲线的实时荧光PCR相比,数字PCR技术使用终点法检测,不依赖于Ct值(荧光强度达到指定值时的PCR循环数),是一种可以测定绝对拷贝数的DNA精确定量技术,避免了不同样品之间的扩增效率变化而导致的定量结果不准确[15]。Köppel等[23]报道的实时荧光PCR方法在最低含量为1%时的相对误差最高为65%。与之相比,本方法能准确获得靶基因的绝对拷贝数并转化为其质量分数,因此在最低检测限为1%时的相对误差更小,低于17%,有效提高了定量的检测低限及其准确性。
本研究选择单拷贝持家基因作为目的基因,其拷贝数不会因物种性别、年龄和部位发生变化,是良好的定量靶标,确保了定量的准确度和稳定性。Floren等[16]以ddPCR技术为基础,分别选用线粒体cyt b基因和单拷贝持家基因作为靶标设计引物探针,发现多拷贝的线粒体基因在同种肉的不同组织中差异为6 倍。而Rodriguez等[24]也发现,动物的性别、年龄或组织器官的不同也会影响线粒体DNA丰度,而单拷贝持家基因在所有细胞中拷贝数恒定,其数量与样品质量有较好的线性关系。
在肉类定量检测中,除了需要准确获得靶基因的绝对拷贝数外,将测得的靶基因相对丰度转换为相应的质量分数也是准确定量的关键之处。本研究通过理论推导结合实验验证后得到了单位质量下两种肉的基因拷贝数之比为一固定值,而Köppel等[23]学者通过实时荧光PCR得到的Ct值推测出各物种基因拷贝数与其所占质量分数呈正相关,与本实验研究结果吻合。苗丽等[19]建立的ddPCR方法也可将基因拷贝数转换为质量,但其为两步转换,加大了定量误差,最低可定量到5 mg(5%含量)样品时,误差最高达13.2%。本研究建立的方法利用两种肉基因拷贝数之比这一固定值,一步实现了基因拷贝数之比与质量之比的转化,在定量5%猪肉含量时的相对误差为-7.5%,且最低定量检测限为1%,既简化了实验步骤,又提高了定量的准确性和检测低限。
ddPCR为肉制品掺假精准定量提供了有力的技术手段,本研究表明通过理论推导并结合实验验证了单位质量下猪肉与羊肉拷贝数之比,使数字PCR测得的基因拷贝数可以直接转换为各组分的质量分数,定量结果准确。本实验所建立的方法不仅可以定量羊肉中掺杂猪肉,也可以应用于其他肉类及其他食品掺假的定量检测,为执法部门判定肉产品及其他食品中掺假还是无意污染、打击食品掺假现象及食品安全犯罪量刑等提供技术支撑。随着数字PCR技术的日益发展,定量检测食品中的动物源性成分将会成为我国行业标准发展的必然趋势,为我国肉制品掺假的市场监管和相关执法提供有力的技术保障。
[1] O’MAHONY P J. Finding horse meat in beef products: a global problem[J]. QJM: An International Journal of Medicine, 2013, 106(6): 595-597. DOI:10.1093/qjmed/hct087.
[2] 李志强, 王济民. 我国畜产品消费及消费市场前景分析[J]. 中国农村经济, 2000(7): 46-51.
[3] 金萍, 丁洪流, 李培, 等. 2013年苏州地区肉及其制品掺假情况调查[J].中国食品卫生杂志, 2014, 26(2): 168-172.
[4] NAKYINSIGE K, MAN Y B C, SAZILI A Q. Halal authenticity issues in meat and meat products[J]. Meat Science, 2012, 91(3): 207-214. DOI:10.1016/j.meatsci.2012.02.015.
[5] SENTANDREU M Á, SENTANDREU E. Authenticity of meat products: tools against fraud[J]. Food Research International, 2014, 60: 19-29. DOI:10.1016/j.foodres.2014.03.030.
[6] Europe: In off cial journal of the European communities. Method of analysis for the determination of constituents of animal origin for the off cial control of feed: EC152-2009[S]. Europe, 2009.
[7] LIU L, CHEN F C, DORSEY J L, et al. Sensitive monoclonal antibody-based sandwich ELISA for the detection of porcine skeletal muscle in meat and feed products[J]. Journal of Food Science, 2006, 71(1): M1-M6. DOI:10.1111/j.1365-2621.2006.tb12393.x.
[8] MONTOWSKA M, POSPIECH E. Species-specific expression of various proteins in meat tissue: proteomic analysis of raw and cooked meat and meat products made from beef, pork and selected poultry species[J]. Food Chemistry, 2013, 136(3): 1461-1469. DOI:10.1016/ j.foodchem.2012.09.072.
[9] CHOU C C, LIN S P, LEE K M, et al. Fast differentiation of meats from fifteen animal species by liquid chromatography with electrochemical detection using copper nanoparticle plated electrodes[J]. Journal of Chromatography B, 2007, 846(1): 230-239. DOI:10.1016/j.jchromb.2006.09.006.
[10] AMARAL J S, SANTOS C G, MELO V S, et al. Authentication of a traditional game meat sausage (Alheira) by species-specific PCR assays to detect hare, rabbit, red deer, pork and cow meats[J]. Food Research International, 2014, 60: 140-145. DOI:10.1016/ j.foodres.2013.11.003.
[11] IWOBI A, SEBAH D, KRAEMER I, et al. A multiplex real-time PCR method for the quantification of beef and pork fractions in minced meat[J]. Food Chemistry, 2015, 169(169): 305-313. DOI:10.1016/ j.foodchem.2014.07.139.
[12] LÓPEZ-ANDREO M, ALDEGUER M, GUILLÉN I, et al. Detection and quantification of meat species by qPCR in heat-processed food containing highly fragmented DNA[J]. Food Chemistry, 2012, 134(1): 518-523. DOI:10.1016/j.foodchem.2012.02.111.
[13] GUO G, HUSS M, TONG G Q, et al. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst[J]. Developmental Cell, 2010, 18(4): 675-685. DOI:10.1016/j.devcel.2010.02.012.
[14] DREO T, PIRC M, RAMŠAK Ž, et al. Optimising droplet digital PCR analysis appr oaches for detection and quantif cation of bacteria: a case study of fire blight and potato brown rot[J]. Analytical and Bioanalytical Chemistry, 2014, 406(26): 6513-6528. DOI:10.1007/ s00216-014-8084-1.
[15] MORISSET D, ☒TEBIH D, MILAVEC M, et al. Quantitative analysis of food and feed samples with droplet digital PCR[J]. PLoS ONE, 2013, 8(5): e62583. DOI:10.1371/journal.pone.0062583.
[16] FLOREN C, WIEDEMANN I, BRENIG B, et al. Species identif cation and quantification in meat and meat products using droplet digital PCR (ddPCR)[J]. Food Chemistry, 2015, 173(173): 1054-1058. DOI:10.1016/j.foodchem.2014.10.138.
[17] RENÉ K, THOMAS B, ANNA F, et al. Droplet digital PCR versus multiplex real-time PCR method for the detection and quantif cation of DNA from the four transgenic soy traits MON87769, MON87708, MON87705 and FG72, and lectin[J]. European Food Research Technology, 2015, 241(4): 521-527. DOI:10.1007/s00217-015-2481-3.
[18] CAI Y, XIANG L, RONG L, et al. Quantitative analysis of pork and chicken products by droplet digital PCR[J]. Biomed Research International, 2014, 2014(2014): 810209. DOI:10.1155/2014/810209.
[19] 苗丽, 张秀平, 陈静, 等. 微滴数字PCR法对肉制品中牛源和猪源成分的定量分析[J]. 食品科学, 2016, 37(8): 187-191. DOI:10.7506/ spkx1002-6630-201608033.
[20] BALLIN N Z, VOGENSEN F K, KARLSSON A H. Species determination-can we detect and quantify meat adulteration?[J]. Meat Science, 2009, 83(2): 165-174. DOI:10.1016/j.meatsci.2009.06.003.
[21] WANG Wei, HAN Jianxun, WU Yajun, et al. Simultaneous detection of eight food allergens using optical thin-film biosensor chips[J]. Journal of Agricultural and Food Chemistry, 2011, 59(13): 6889-6894. DOI:10.1021/jf200933b.
[22] Codex Committee on Methods of Analysis and Sampling. Guidelines on performance criteria and validation of methods for detection identification and quantification of specific DNA sequences and specific proteins in foods: CAC/GL740-2010[S]. Rome: Codex Alimentariusliment, 2010.
[23] KÖPPEL R, RUF J, RENTSCH J. Multiplex real-time PCR for the detection and quantification of DNA from beef, pork, horse and sheep[J]. European Food Research and Technology, 2011, 232(1): 151-155. DOI:10.1007/s00217-010-1371-y.
[24] RODRIGUEZ M A, GARCIA T, GONZALEZ I, et al. Identif cation of goose, mule duck, chicken, turkey, and swine in foiegras by speciesspecif c polymerase chain reaction[J]. Journal of Agricultural and Food Chemistry, 2003, 51(6): 1524-1529. DOI:10.1021/jf025784+.
A Precise Quantitative Assay for Measuring Pork Incorporated into Mutton Products by Droplet Digital PCR
REN Jun’an1,2, DENG Tingting2, HUANG Wensheng2, GE Yiqiang1,3, CHEN Ying2,*
(1. College of Food Science and Nutritional Engineering, China Agricultural University, Beijing 100083, China; 2. Chinese Academy of Inspection and Quarantine, Beijing 100176, China; 3. China Rural Technology Development Center, Beijing 100045, China)
The adulteration of meat products happened frequently in recent years, and authentication of meat products is necessary to protect consumers from an inferior product with a false description. Although at present, there are numerous qualitative methods for meat species identification, fewer quantitative detection methods have been reported. Herein, a droplet digital PCR method for the quantitative determination of pork incorporated in mutton products was developed. The single copy house-keeping gene encoding replication protein A1 (PRA1) was chosen as the target to design species-specific primers and probes for mutton and pig, respetively. Each assay was proved specific to the target species, respectively. The ratio constants between copy numbers and unit mass of pork and mutton were obtained by theoretical analysis and then verified by experiments. Consequently, the relative mass fractions of pork and mutton in the sample were measured based on the DNA copy numbers. The limit of quantitation (LOQ) of the method was confirmed to be 1%. The results showed that the absolute error was less than ±1.3%, and the relative error was less than ±10% in the range of pork proportion from 5% to 80%. The method developed in this paper was successfully applied to quantitate pork content incorporated into commercial mutton products and it may be useful for food administration laboratories to carry out meat species quantification in raw and processed foods.
mutton; pork; quantitative determination; droplet digital PCR (ddPCR)
10.7506/spkx1002-6630-201702049
TS207.3
A
1002-6630(2017)02-0311-06
任君安, 邓婷婷, 黄文胜, 等. 微滴式数字聚合酶链式反应精准定量检测羊肉中掺杂猪肉[J]. 食品科学, 2017, 38(2): 311-316. DOI:10.7506/spkx1002-6630-201702049. http://www.spkx.net.cn
REN Jun’an, DENG Tingting, HUANG Wensheng, et al. A precise quantitative assay for measuring pork incorporated into mutton products by droplet digital PCR[J]. Food Science, 2017, 38(2): 311-316. (in Chinese with English abstract)
10.7506/spkx1002-6630-201702049. http://www.spkx.net.cn
2016-05-14
中国检验检疫科学研究院基本科研业务费专项(2014JK027);“十三五”国家重点研发计划重点专项(2016YFD0401104)
任君安(1988—),女,博士研究生,研究方向为食品质量安全检测与控制。E-mail:renja126@126.com
*通信作者:陈颖(1972—),女,研究员,博士,研究方向为食品质量安全与控制。E-mail:chenyingcaiq@163.com
