含寡聚腺嘌呤序列DNA探针在金纳米粒子上的固定及杂交性能
2017-02-06张茜王宋蒙褚立强
张茜+王宋蒙+褚立强
摘 要 利用寡聚腺嘌呤序列(OAS)与金的强相互作用,在金纳米粒子(AuNPs)上固定不同密度DNA探针(DNA probe),详细探究不同条件 (OAS长度、AuNPs粒径、NaCl浓度等) 下单链DNA probe的固定效果,以及制备的纳米探针(Au.probe)与互补DNA目标分子(DNA target)的杂交性能。利用透射电子显微镜(TEM)、紫外可见分光光度计(UV.Vis)、激光粒度仪等对制备的AuNPs的形貌、粒径、表面DNA probe固定及杂交性能等进行了研究。结果表明,随OAS碱基数量由10增加到30和50,Au.probe上固定的DNA probe数量降低。对粒径为10.2和24.3 nm 的AuNPs,杂交效果最佳的NaCl浓度分别为300和25 mmol/L。随着AuNPs粒径增大,AuNPs单位面积上的DNA probe固定量及DNA target杂交量均呈下降趋势。
关键词 寡聚腺嘌呤序列; 金纳米粒子; DNA固定; DNA检测; 杂交性能
1 引 言
生物纳米分析技术是涵盖化学、物理学、生物学和医学等多学科技术的新兴技术。DNA和金纳米粒子(AuNPs)具有诸多独特、优良的分析特性,二者复合而成的新型纳米材料被广泛应用于微纳结构构建[1~3]、基因治疗[4~6]、生物传感[7~9]等领域。
DNA在AuNPs上的固定主要基于共价作用,包括AuS键[9~11]、AuN键[12]及AuC键[13]等。最常见的方法是利用AuS相互作用将含巯基的单链DNA固定到AuNPs表面,可通过DNA与小分子硫醇混合自组装,或后期利用小分子硫醇的取代反应,控制AuNPs表面DNA密度。但对AuNPs上固定的DNA密度的精确调控依然是研究的难点[14]。
最近研究表明,寡聚腺嘌呤序列(Oligo adenine sequence, OAS)可与Au表面优先吸附,亲和力与AuS键相当。Opdahl等[15~17]发现,可通过控制OAS长度精确控制单链DNA在Au基底上的接枝密度与构型,并系统研究了OAS与Au基底相互作用,以及DNA表面相互作用对其杂交稳定性的影响。Pei等[18]及Nourisaeid等[19]在比色法检测DNA研究中,实现了不经修饰、双嵌段DNA制备的AuNPs探针间的快速杂交。Jiang等[20]利用该方法,设计了一种基于RNA适配体的AuNPs传感器检测血液中的茶碱。Zhu等[21,22]利用OAS构建了以核.卫星结构的金/银硅基纳米杂化增强的SERS生物传感器,并用于Hg2+及Pb2+的高灵敏、特异性检测。与利用AuS相互作用相比,这种方法操作更简单,可控性更好,可以更方便控制DNA密度。这些研究均采用13 nm左右AuNPs,对不同尺寸AuNPs上含OAS单链DNA的固定及杂交行为的研究目前未见报道。
本研究考察了含OAS的单链DNA探针(DNA probe)在不同尺寸AuNPs上的固定及杂交行为。通过种子生长法制备了一系列不同粒径、分散性良好的AuNPs,利用OAS.Au间的相互作用制备了纳米探针(Au.probe)。利用透射电子显微镜(TEM)、紫外.可见分光光度计(UV.Vis)、激光粒度仪等对Au.probe进行了表征,并系统研究了不同离子强度、OAS长度及不同AuNPs粒径下,DNA probe的固定及杂交行为,为其在生物传感、基因诊断等领域的应用提供了实验基础。
2 实验部分
2.1 仪器与试剂
UV.2550型紫外可见分光光度计(日本岛津有限公司);HT7700透射电镜(日立仪器有限公司);ZS90激光粒度分析仪(英国马尔文仪器有限公司);XYF2.20.H超纯水机(北京湘顺源科技有限公司);THZ.100B恒温培养摇床(上海一恒科学仪器有限公司)。
氯金酸(HAuCl4·4H 2O,天津市金铂兰精细化工有限公司);含OAS单链DNA(上海生工生物工程有限公司,序列见表1);柠檬酸钠(C 6H 5Na 3O 7·2H 2O)、NaH 2PO4、Na 2HPO4、NaCl(阿拉丁试剂);所用实验试剂均为分析纯;实验用水为超纯水(电阻率18.25 MΩ·cm)。
2.2 实验过程
2.2.1 AuNPs制备 参照文献[23\]的方法,在三口烧瓶中加入150 mL 2.2 mmol/L柠檬酸钠溶液,快速搅拌,加热至沸腾。加入1 mL 25 mmol/L氯金酸溶液,煮沸10 min后,取30.2 mL,记为G0。将温度降至90 ℃并恒温,加入1.6 mL 25 mmol/L氯金酸和1.6 mL 60 mmol/L柠檬酸钠溶液,每隔30 min重复加入适量试剂并取出适量AuNPs样品(即不同代数的AuNPs,记为G1、G2……G8),在冰水混合物中降温后,利用UV.Vis、TEM和激光粒度仪进行表征。
2.2.2 Au.probe的制备 将330 μL AuNPs与过量DNA probe溶液混合, 置于恒温摇床,在35℃以120 r/min孵育16 h; 加入相应体积超纯水、PB缓冲液,恒温孵育1 h;再加入2 mol/L NaCl溶液,以调节最终NaCl浓度,恒温孵育10 h,并用UV.Vis进行表征。
2.2.3 Au.probe与DNA target的杂交 将DNA target按与Au.probe上DNA probe完全杂交的量,加入样品中,室温振荡孵育1 h,用UV.Vis进行表征。以DNA probe吸附前后吸收峰位置之差表征DNA probe吸附量变化;以DNA target杂交前后吸收峰位置之差表征DNA target杂交量变化[24,25]。
3 结果与讨论
3.1 AuNPs的制备
以种子生长法合成不同粒径AuNPs,其吸收光谱见图1。AuNPs种子(G0)最大吸收峰出现在518.5 nm处,峰型尖锐。随代次增加,最大吸收峰发生红移,峰形差别不大,说明其合成是按步增长,且不同代数的样品尺寸均匀。
TEM(图2)及激光粒度分析仪表征结果(表2)表明,AuNPs呈良好球状结构,分布均匀。随代次增加,粒径逐步增大。 Zeta电位测试表明, 其表面负电荷绝对值均达30 mV以上,稳定性好。
3.2 DNA probe的固定
图3为实验过程示意图,将含不同OAS长度的DNA probe固定到AuNPs上,形成Au.probe,再加入DNA target进行杂交。由于OAS与Au之间的强相互作用,DNA probe被直接吸附到AuNPs表面,通过裸眼观察即可初步判断Au.probe是否制备成功(图4)。对单纯AuNPs, 加入300 mmol/L NaCl后,溶液立即变为蓝灰色,UV.Vis图谱吸收峰变宽,出现大范围红移,即AuNPs发生团聚。而对Au.probe, 由于AuNPs表面固定了DNA probe,表面电荷密度增加,对AuNPs起到保护作用,加入300 mmol/L NaCl后,溶液稳定,仍呈粉红色。
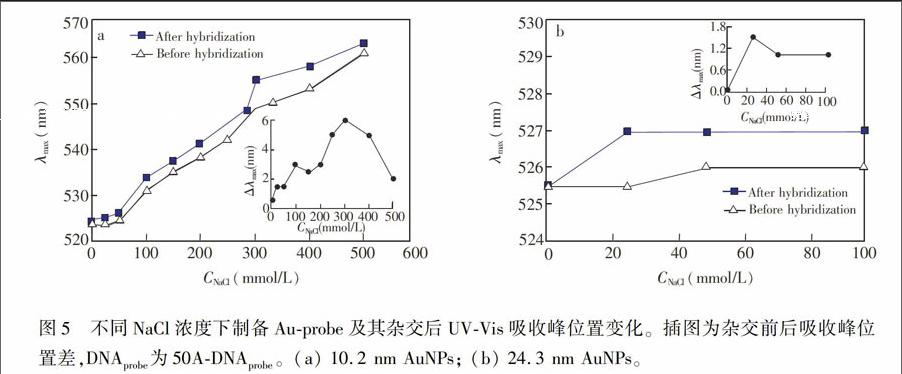
文献[26\]表明,可通过盐老化方法(即在一定浓度NaCl溶液中)增加Au.probe表面固定的DNA probe的密度,但不同粒径AuNPs所需NaCl浓度不同。由图5a可见,对于10.2 nm AuNPs,随NaCl浓度增加,最大吸收峰位置发生红移,300 mmol/L NaCl时差值达最大,杂交效果最佳。造成这种现象有两种可能的原因:一是加入NaCl导致溶液整体折光系数的变化。分别使用不含NaCl和含500 mmol/L NaCl的缓冲液作参比,对同一Au.probe样品进行测试,两者UV.Vis谱图基本一致,说明此因素不影响实验结果。其次,NaCl 起到静电屏蔽作用,减小了DNA probe间的静电排斥。随NaCl浓度从25 mmol/L开始增加,静电屏蔽作用增大,使DNA probe固定量增加,表面密度升高,Au.probe与DNA target杂交机会更大。同时,由于静电排斥的降低,DNA target更易与DNA probe杂交。但当NaCl浓度超过300 mmol/L,DNA probe表面密度过大,会形成空间位阻,导致杂交量下降。
24.3 nm AuNPs (图5b)的实验结果与10.2 nm基本一致,但最佳NaCl浓度为25 mmol/L。对大粒径AuNPs,需更高的表面电荷密度来保持稳定。但相同条件下,大粒径AuNPs制备的Au.probe上DNA probe固定量更少(见3.5节)。NaCl浓度较高时,DNA probe上的负电荷不足以保持Au.probe稳定而发生团聚。粒径越大,对NaCl浓度升高越敏感。因此,大粒径样品所需NaCl浓度通常较低。10.2 nm AuNPs制备的Au.probe,NaCl浓度为500 mmol/L仍可稳定存在;但24.3 nm AuNPs制备的Au.probe,NaCl浓度超过100 mmol/L即会聚集。
3.4 OAS长度的影响
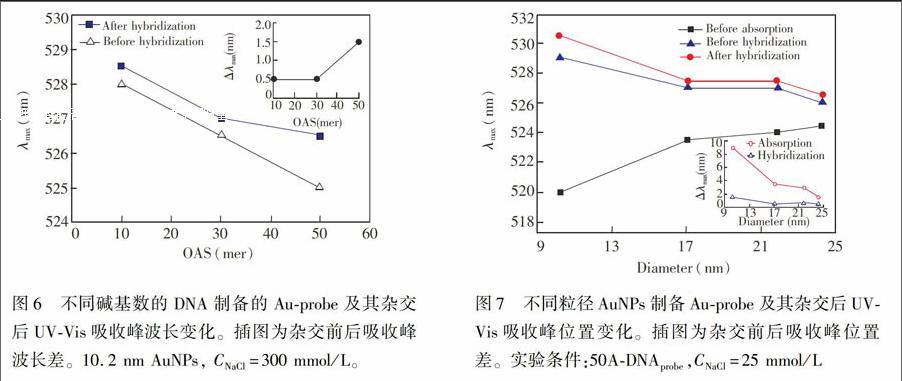
通过改变OAS碱基数,可以调控Au.probe表面DNA probe密度,并在纳米尺度上精确控制AuNPs表面DNA probe分子间距离。如图6所示,随OAS碱基数增长,其吸收峰蓝移,且与DNA target杂交量上升,表明随OAS链长度增加,Au.probe表面DNA probe量减少,密度降低,证明利用本方法可容易地实现对Au.probe 表面DNA probe密度的控制。同时,DNA probe间距离增大,使得其与DNA target的静电排斥及空间位阻降低,杂交更易进行,DNA target杂交量上升。
3.5 AuNPs粒径的影响
如图7所示,随AuNPs粒径增大,Au.probe上DNA probe量出现明显下降,DNA target杂交量则先下降然后基本保持不变。由于大粒径AuNPs表面积更大,因而随粒径增大,单位面积上的DNA probe固定量及DNA target杂交量均降低。
Au.probe表面DNA probe固定量及DNA target杂交量是由OAS与Au的吸附作用,以及DNA链间静电排斥和空间位阻共同作用的结果。小粒径AuNPs曲率更高,对相同长度OAS,Au.probe表面DNA probe间距离更大,静电排斥及空间位阻更小[27],DNA probe更易吸附到AuNPs表面,杂交也更易进行。因此,随AuNPs粒径增大,其单位面积上DNA probe固定量及DNA target杂交量均降低。
4 结 论
利用OAS.Au相互作用在AuNPs上固定单链DNA分子,系统研究了溶液NaCl浓度、OAS长度及AuNPs粒径对DNA probe固定及其与DNA target杂交性能的影响。结果表明,对10.2和24.3 nm AuNPs,杂交效果最佳的NaCl浓度分别为300和25 mmol/L。随着OAS碱基数量从10增加到30和50,Au.probe的DNA probe固定量降低。随粒径增大,AuNPs单位面积上DNA probe固定量及DNA target杂交量均降低。因此,DNA间静电排斥、位阻效应及溶液离子的静电屏蔽作用等均会影响Au.probe上DNA probe固定量及其与DNA target的杂交性能。本研究为AuNPs探针在生物传感、基因诊断、纳米材料构建等领域的应用提供了参考依据。
References
1 Sharma J, Chhabra R, Liu Y, Ke Y, Yan H. Angew. Chem. Int. Ed., 2006, 45(5): 730-735
2 Zheng J, Constantinou P E, Micheel C, Alivisatos A P, Kiehl R A, Seeman N C. Nano. Lett., 2006, 6(7): 1502-1504
3 Chao J, Lin Y, Liu H, Wang L, Fan C. Mater. Today, 2015, 18(6): 326-335
4 Dreaden E C, Alkilany A M, Huang X, Murphy C J, El.Sayed M A. Chem. Soc. Rev., 2012, 41(7): 2740-2779
5 Llevot A, Astruc D. Chem. Soc. Rev., 2012, 41(1): 242-257
6 Dykman L, Khlebtsov N. Chem. Soc. Rev., 2012, 41(6): 2256-2282
7 MA Li.Na, LIU Dian.Jun, WANG Zhen.Xin. Chinese J. Anal. Chem., 2010, 38(1): 1-7
马立娜, 刘殿骏, 王振新. 分析化学, 2010, 38(1): 1-7
8 Jans H, Huo Q. Chem. Soc. Rev., 2012, 41(7): 2849-2866
9 MA Qun, LI Yan.Le, GONG Nian.Chun, JIANG Xi, HUAN Shuang.Yan. Chinese J. Anal. Chem., 2015, 43(11): 1676-1681
马 群, 李艳乐, 龚年春, 江 西, 宦双燕. 分析化学, 2015, 43(11): 1676-1681
10 Mirkin C A, Letsinger R L, Mucic R C, Storhoff J J. Nature, 1996, 382(6592): 607-609
11 Alivisatos A P, Johnsson K P, Peng X, Wilson T E, Loweth C J, Bruchez M P J, Schultz P G. Nature, 1996, 382(6592): 609-611
12 Tang D, Tang J, Su B, Chen G. Biosens. Bioelectron., 2011, 26(5): 2090-2096
13 Laurentius L, Stoyanov S R, Gusarov S, Kovalenko A, Du R, Lopinski G P, McDermott M T. ACS Nano, 2011, 5(5): 4219-4227
14 Su S, Zuo X, Pan D, Pei H, Wang L, Fan C, Huang W. Nanoscale, 2013, 5(7): 2589-2599
15 Opdahl A, Petrovykh D Y, Kimura.Suda H, Tarlov M J, Whitman L J. Proc. Natl. Acad. Sci. USA, 2007, 104(1): 9-14
16 Schreiner S M, Shudy D F, Hatch A L, Opdahl A, Whitman L J, Petrovykh D Y.Anal. Chem., 2010, 82(7): 2803-2810
17 Schreiner S M, Hatch A L, Shudy D F, Howard D R, Howell C, Zhao J, Koelsch P, Zharnikov M, Petrovykh D Y, Opdahl A. Anal. Chem., 2011, 83(11): 4288-4295
18 Pei H, Li F, Wan Y, Wei M, Liu H, Su Y, Chen N, Huang Q, Fan C. J. Am. Chem. Soc., 2012, 134(29): 11876-11879
19 Nourisaeid E, Mousavi A, Arpanaei A. Physica E, 2016, 75: 188-195
20 Jiang H, Ling K, Tao X, Zhang Q. Biosens. Bioelectron., 2015, 70: 299-303
21 Zhu Y, Jiang X, Wang H, Wang S, Wang H, Sun B, Su Y, He Y. Anal. Chem., 2015, 87(13): 6631-6638
22 Shi Y, Wang H, Jiang X, Sun B, Song B, Su Y, He Y. Anal. Chem., 2016, 88(7): 3723-3729
23 Bastus N G, Comenge J, Puntes V. Langmuir, 2011, 27(17): 11098-11105
24 Camden J P, Dieringer J A, Zhao J, van Duyne R P. Acc. Chem. Res., 2008, 41(12): 1653-1661
25 Willets K A, van Duyne R P. Annu. Rev. Phys. Chem., 2007, 58(1): 267-297
26 Cutler J I, Auyeung E, Mirkin C A. J. Am. Chem. Soc., 2012, 134(3): 1376-1391
27 Hill H D, Millstone J E, Banholzer M J, Mirkin C A. ACS Nano, 2009, 3(2): 418-424
Abstract Oligo.adenine sequence (OAS) containing DNA probes (DNA probe) was immobilized on gold nanoparticles (AuNPs) with various surface densities via the strong interaction between OAS sequence and gold surface, and the influences of different conditions (e.g., length of OAS, size of AuNPs, concentration of NaCl, etc.) on the immobilization of DNA probe and hybridization performance of the resulting nanoparticle probe (Au.probe) were investigated. Transmission electron microscopy (TEM), ultraviolet.visible absorption spectroscopy (UV.Vis) and nanoparticle size analyzer were utilized to characterize the morphology and size of AuNPs and Au.probe, as well as the DNA probe immobilization on AuNPs surface and their hybridization with DNA target(DNA target). The results showed that, when the number of adenine in an OAS sequence increased from 10, to 30 and 50, the amount of the immobilized DNA probe decreased as expected. The optimal NaCl concentrations for the hybridization were 300 mmol/L for 10.2.nm AuNPs, and 25 mmol/L for 24.3.nm AuNPs, respectively. With increasing size of AuNPs, the amount of both immobilized DNA probe and hybridized DNA target decreased.
Keywords Oligo.adenine sequence ; Gold nanoparticles; Deoxyribonucleic acid immobilization; Deoxyribonucleic acid detection; Hybridization performance