葡萄OVATE基因家族生物信息学及表达
2016-12-23张亚光高世敏陶建敏
袁 月,张亚光,高世敏,陶建敏
葡萄基因家族生物信息学及表达
袁 月,张亚光,高世敏,陶建敏
(南京农业大学园艺学院,南京210095)
【目的】OVATE是一类调控植物生长发育的转录抑制因子,对葡萄基因家族()进行生物信息学和组织特异性表达分析,为该类基因的功能研究奠定基础。【方法】根据OVATE保守域蛋白序列(PF04844)对葡萄基因家族进行鉴定,利用生物信息学方法对葡萄基因家族染色体定位、基因结构、保守结构域、亚细胞定位等方面进行预测和分析,并分析葡萄和拟南芥基因家族的进化关系。采用实时荧光定量PCR技术检测组织表达特性。【结果】葡萄基因家族包含17个成员,不均匀地分布在11条染色体上,均没有内含子结构,编码115—444个氨基酸,等电点4.55—9.69,均为亲水蛋白;所有蛋白均包含完整的OVATE保守结构域,亚细胞定位主要在细胞核中。根据进化树拓扑结构,将葡萄和拟南芥OVATE蛋白家族聚为六类(I—VI),其中,Ⅱ、Ⅲ、Ⅴ类仅包含两个基因,Ⅰ、Ⅳ、Ⅵ类中和相互交错地聚类在一起;和共包含10个未知基序,保守元件1和2位于OVATE结构域区域,此外,在OVATE结构域外每个类别均包含特有基序。具有组织表达特异性,12个基因在根、茎、叶、花和果实中均可以检测到表达,其余5个基因仅在特定组织中表达。多数在根、嫩茎和花中表达量较高,在嫩叶和果实中仅检测到少数基因表达;在不同发育期表达量存在差异,通常在开花前1周和开花期表达量较高,而花后4周果实中表达量较低。1对旁系同源基因表达模式相似,3对旁系同源基因产生了新的表达模式。【结论】葡萄OVATE结构域序列比较保守,其在不同组织中呈现出多种表达模式,推测其可能参与了葡萄生长发育的调控。
葡萄;基因家族;生物信息学;表达
0 引言
【研究意义】OVATE是一类植物转录因子[1-5],调控植物生长发育的多个方面,包括胚囊和花粉发育[1]、次生细胞壁合成[5]以及果实形状[6-7]等。葡萄(L.)具有5 000多年的栽培历史[8],其保留着古老的开花植物的基因组结构,是非常有用的模式植物[9]。对葡萄基因家族进行生物信息学分析,对于葡萄功能基因组学的研究发展有重要意义。【前人研究进展】LIU等[6]首次在番茄中克隆出,研究发现其能够控制番茄果实形状,该基因的一个点突变导致翻译提前终止,使得野生番茄果实纵径增长,颈部生长受到限制,从而由圆形果实发育成梨形果实。编码一个亲水蛋白,该蛋白的C末端结构域DUF623在拟南芥、水稻和番茄中具有保守性,被命名为OVATE结构域,凡含有该结构域的蛋白,均属于OVATE蛋白家族。目前关于OVATE蛋白家族的研究主要集中在拟南芥、番茄和水稻中,其中,拟南芥基因组中共被鉴定出18个OVATE蛋白,功能分析表明这些蛋白作为转录抑制因子,调控植株生长发育的多个过程[3,5]。在酵母双杂交试验中,9个OVATE蛋白被发现与TALE同源异型盒蛋白相互作用,且AtOFP1和AtOFP5共同调控一个TALE同源异型盒蛋白BLH1的亚细胞定位,当这两个基因在烟草叶片中共表达时,BLH1蛋白从细胞核运输到细胞质中[1]。TALE同源异型盒蛋白包含一个三氨基酸残基环连接两个螺旋的结构[10],分为KNOX和BELL两个子类[11-13],KNOX和BELL蛋白相互作用形成异源二聚体,调控植物的生长发育[14-18],因此,拟南芥OVATE蛋白可以通过与TALE蛋白相互作用而间接调控植物生长发育,不仅如此,OVATE蛋白还可以直接调控靶基因的表达。是赤霉素合成关键酶基因,染色质免疫共沉淀表明该基因是AtOFP1蛋白的靶基因,AtOFP1通过抑制该基因的表达,调控赤霉素的生物合成,从而导致包括莲座叶、下胚轴、茎叶、花序梗、花器官和角果在内的地上部器官变短,该家族其他基因的过表达也具有相似的生理效应[3]。此外,沉默、、、、和中任一个基因,植株的表型并未发生变化,表明这些家族成员间存在功能冗余[4]。AtOFP1还与AtKu70相互作用,参与DNA非同源末端修复[19]。AtOFP4能够与KNAX7蛋白的相互作用,进而调控次生细胞壁的形成[5]。AtOFP5通过抑制BLH1-KNAT3复合物活性在胚囊的发育早期发挥作用[2]。在番茄全基因组中共预测了31个家族基因,其中和在生殖器官中的表达量高于营养器官,而和在苗期的根、幼叶和下胚轴中表达量较高,表明番茄家族成员在植株发育调控中发挥着不同的作用[20]。在水稻的基因组中共预测出31个家族基因,这些基因同样具有组织表达特异性,接近一半的基因在苗期表达量较高;外施油菜素内酯可以显著影响和的表达[21]。辣椒OVATE蛋白家族成员CaOVATE也是通过抑制表达,从而改变果实形状[7]。香蕉OVATE蛋白家族成员MaOFP1与香蕉MuMADS1蛋白存在互作,两者共同调控香蕉果实的发育,在香蕉果实发育前期表达量较高,后期表达则受到乙烯的抑制[22]。在最近的研究[23]中,包含拟南芥、水稻、番茄在内的13种植物基因家族被鉴定出来,共有265个OVATE蛋白序列,这些物种包含较古老的苔藓和石松门植物,生物信息学分析表明,基因家族存在不同的进化机制,主要为保守进化和分散扩增两种方式。【本研究切入点】目前,仅有一篇外文文献报道葡萄全基因组中包含9个家族成员,鉴定出的基因数目较少,且未详细地进行生物信息学和表达分析,因此,有必要对葡萄基因家族进行全面鉴定和分析。【拟解决的关键问题】在葡萄全基因组中搜索鉴定基因家族成员,全面分析基因结构、染色体定位、蛋白保守结构域及系统发育等信息,同时研究葡萄基因家族的组织表达特征,为进一步探究基因功能提供依据。
1 材料与方法
1.1 材料
供试品种为二倍体‘宝满’葡萄,种植于南京农业大学园艺学院汤山葡萄基地,以每个花序一半以上的小花开放作为开花期,分别于2015年5月5日(花前一周)采集花蕾,5月12日(开花期)采集开放的小花(含花冠和花药),6月12日(花后四周)采果实,采集新稍上完全展开的嫩叶和嫩茎。根取自组培苗,组培苗由南京农业大学园艺学院果树生物技术实验室培养,培养条件是光照时间16 h,光照强度2 000 lx,培养温度25℃。材料用液氮速冻后,保存于-80℃冰箱。
1.2 方法
1.2.1 葡萄和拟南芥基因家族的鉴定 拟南芥全基因组的注释序列在NCBI(http://www.ncbi. nlm.nih.gov/)中下载,葡萄全基因组注释序列分别在Grape Genome[9](http://genomes.cribi.unipd.it/grape/)和NCBI(http://www.ncbi.nlm.nih.gov/)中下载,在Pfam数据库[24](http://pfam.xfam.org/)中下载葡萄和拟南芥OVATE蛋白保守域序列(PF04844),利用Bioedit软件在葡萄和拟南芥的蛋白组中搜索这些序列的同源蛋白,删除重复序列,将得到的序列在Pfam上搜索,去除没有OVATE保守域的序列,最终获得葡萄和拟南芥家族基因。
1.2.2 葡萄基因家族的生物信息学分析 利用在线网站EXPASY(http://web.expasy.org/protparam/)中ProtParam工具[25]对葡萄OVATE家族蛋白的氨基酸组成、疏水性、等电点等理化性质进行分析;在NCBI上下载葡萄和拟南芥OVATE家族基因的CDS序列和基因组定位信息,利用在线软件GSDS2.0[26](http://gsds.cbi.pku.edu.cn/index.php)绘出基因结构图,并运用MapInspect工具进行染色体定位作图;利用序列分析软件 clustalX2.0,Jalview2.8和在线软件WebLogo3[27](http://weblogo.threeplusone.com/)分析葡萄OVATE家族蛋白的OVATE保守结构域序列;使用在线软件CelloV.2.5[28](http://cello.life.nctu.edu. tw/),WOLF PSORT(http://www.genscript.com/wolf- psort.html),以及在线网站Soft Berry(http://linux1. softberry.com/berry.phtml)中的PROTCOMP程序共同对葡萄OVATE家族基因进行亚细胞定位预测,利用MEGA5.0[29]软件对已鉴定的葡萄和拟南芥OVATE家族蛋白进行序列比对,并用邻位(Neighbor-Joining)算法构建系统进化树,进行Bootstrap测试,重复设置1 000;基序预测采用在线软件MEME[30](http://meme- suite.org/tools/meme),参数设置为:预测数目10,氨基酸数目4—70,位点数2—300。
1.2.3 葡萄基因家族表达分析 根据基因序列设计实时荧光定量PCR特异引物(表1),以葡萄(XM_002265440)作为内参基因,采用成都福际公司的多糖多酚植物组织总RNA提取试剂盒提取RNA,利用TaKaRa公司的反转录试剂盒PrimeScript™ 1st Strand cDNA Synthesis Kit反转录合成cDNA第一条链,稀释后的cDNA用于实时荧光定量PCR。PCR采用20 μL反应体系:cDNA 1 μL,2×SYBR Premix EX TaqTM(TaKaRa)10 μL,上、下游引物各0.2 μL,灭菌后ddH2O 8.6 μL;反应程序:95℃预变性4 min;95℃变性20 s,60℃退火20 s,72℃延伸40 s,40个循环。每个处理3次重复,采用ABI7300system软件和2-ΔΔCT[31]方法分析数据。

表1 引物序列
2 结果
2.1鉴定及蛋白质理化性质分析
通过搜索葡萄全基因组数据库,共鉴定出17个葡萄基因家族成员,根据其在染色体上的位置,依次命名为—。这些基因的编码区长度在348—1 335 bp,编码115—444个氨基酸,等电点在4.55—9.69,氨基酸序列的平均亲水系数在-1.032—-0.283,均为负值,表明这些蛋白均为亲水蛋白,但亲水程度不同,详见表2。
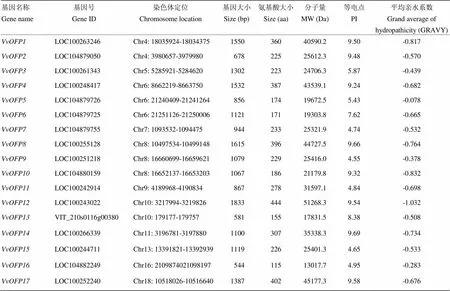
表2 葡萄OVATE家族基因信息和理化性质分析
2.2 VvOFPs的OVATE保守域的鉴定和分析
通过Pfam在线数据库对候选的葡萄OVATE家族蛋白进行鉴定,所有候选基因在C末端均含有OVATE保守结构域,下载这些蛋白的OVATE结构域序列,利用clustalX2.0软件进行序列比对,并使用Jalview2.8绘制并分析比对序列,WebLogo3创建保守域Logo,最终拼接得到图1,由图1可知,葡萄OVATE结构域序列比较保守,氨基酸数目在55—61,所有蛋白的OVATE结构域序列都比较完整,结合基序分析结果,基序1全部位点均在OVATE结构域区域,其中保守性强的氨基酸有亮氨酸(L35)、天冬酰胺(N43)、异亮氨酸(I51)及苯丙氨酸(F55);基序2大部分位点位于结构域区域,其中保守性强的氨基酸有丝氨酸(S1,S11)、脯氨酸(P4)、蛋氨酸(M12)。
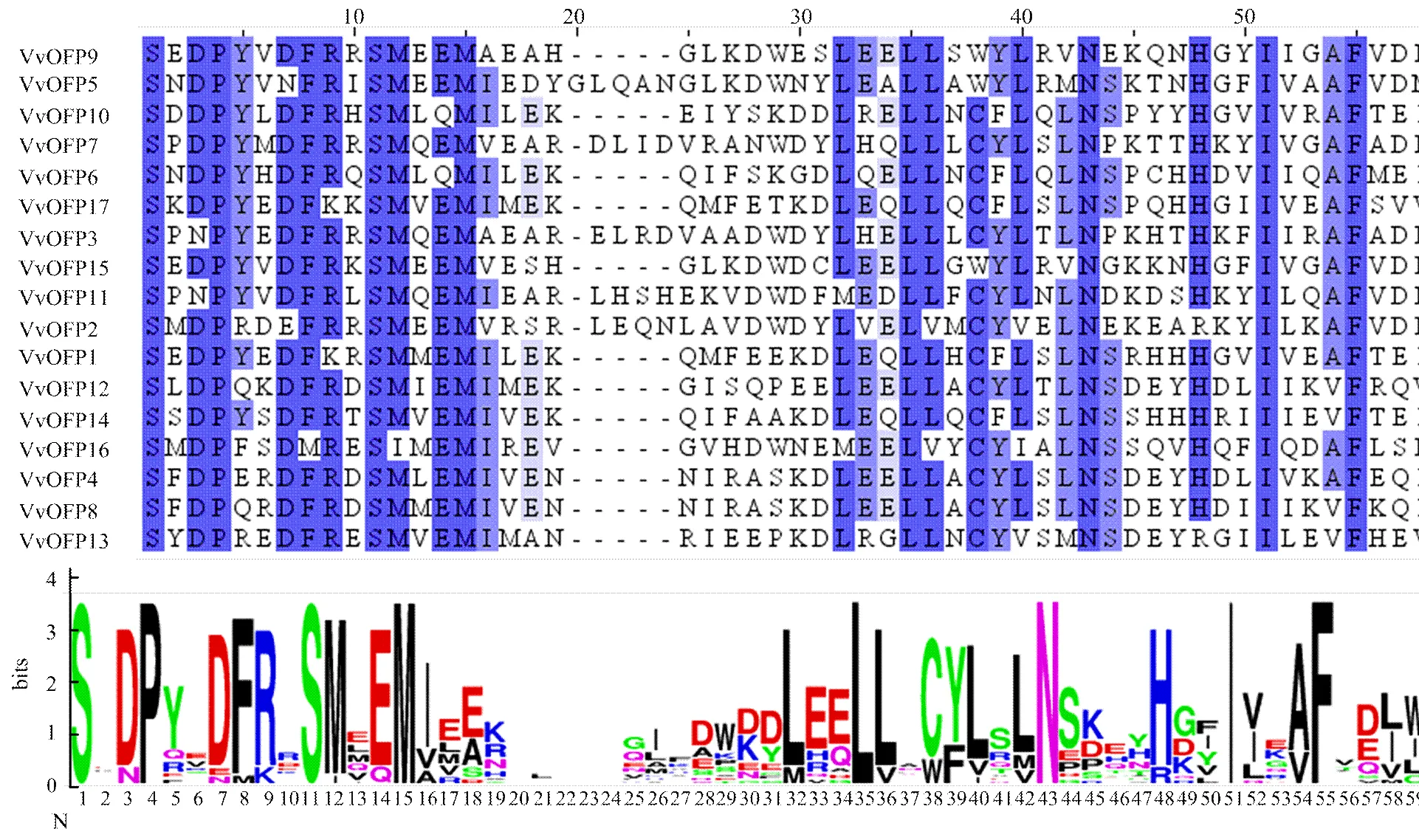
图1 葡萄OVATE蛋白家族的保守结构域序列比对
2.3的染色体定位分析
葡萄家族的17个基因不均等地分布在11条染色体上,1、2、3、12、14、15、17、19号染色体上均没有分布,其中,在6号和8号染色体上分布的基因最多,分别有3个基因;4号、10号染色体各分布2个基因;其余7条染色体均只含有1个基因。此外,6号染色体上的和及8号染色体上的和物理距离较近,分别为8.7和6.4 kb,以串联的方式排列在染色体上(图2)。

图2 葡萄OVATE基因家族的染色体定位
2.4 VvOFPs的进化树分析
构建葡萄和拟南芥OVATE蛋白家族系统进化树(图3-A),根据进化树的拓扑结构对两个物种OVATE家族蛋白进行聚类,可分为六类,分别是Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ和Ⅵ。其中,Ⅱ、Ⅲ、Ⅴ类所包含的基因较少,均只有两个基因,均包含1个和1个,说明该分支内基因进化速度相对缓慢,推测其基因功能相对保守。Ⅰ类包含2个和4个,Ⅳ类包含5个和4个,Ⅵ类包含7个和7个,在Ⅰ、Ⅳ、Ⅵ类中,和,和,和的亲缘关系较近,可能具有相似的功能,其余和相互交错地聚类在一起,形成了4对旁系同源基因对(),形成了4对旁系同源基因对(、、、)。
2.5 VvOFPs的基序分析
利用在线软件MEME对葡萄和拟南芥OVATE家族的蛋白序列进行基序预测,结果表明,葡萄OVATE蛋白家族共包含10个未知基序(图3-B)。每个蛋白序列的C末端均含有保守元件1和2,位于OVATE结构域区域,推测这两个基序可能与OVATE结构域的功能相关,结合进化树分析,Ⅴ类的基因仅有基序1和2,除Ⅰ和Ⅲ类之外,Ⅱ、Ⅳ和Ⅵ类中也分别存在1—2个基因仅有这两个基序。除基序1和2外,有些基序也在不同的类别中同时存在,如Ⅱ类中的1个基因与Ⅵ类中的4个基因共享基序7,Ⅳ类中的5个基因与Ⅰ类中的2个基因共享基序9;而有些基序是各类别所特有的,Ⅰ类中全部基因都共享保守元件3,该基序位于N末端DNA结合域中;Ⅲ类的两个基因共享基序10;Ⅳ类中3个基因共享未知基序8;Ⅵ类中6个基因同时共享基序4、5和6,基序分析结果和进化树分类结果基本一致。
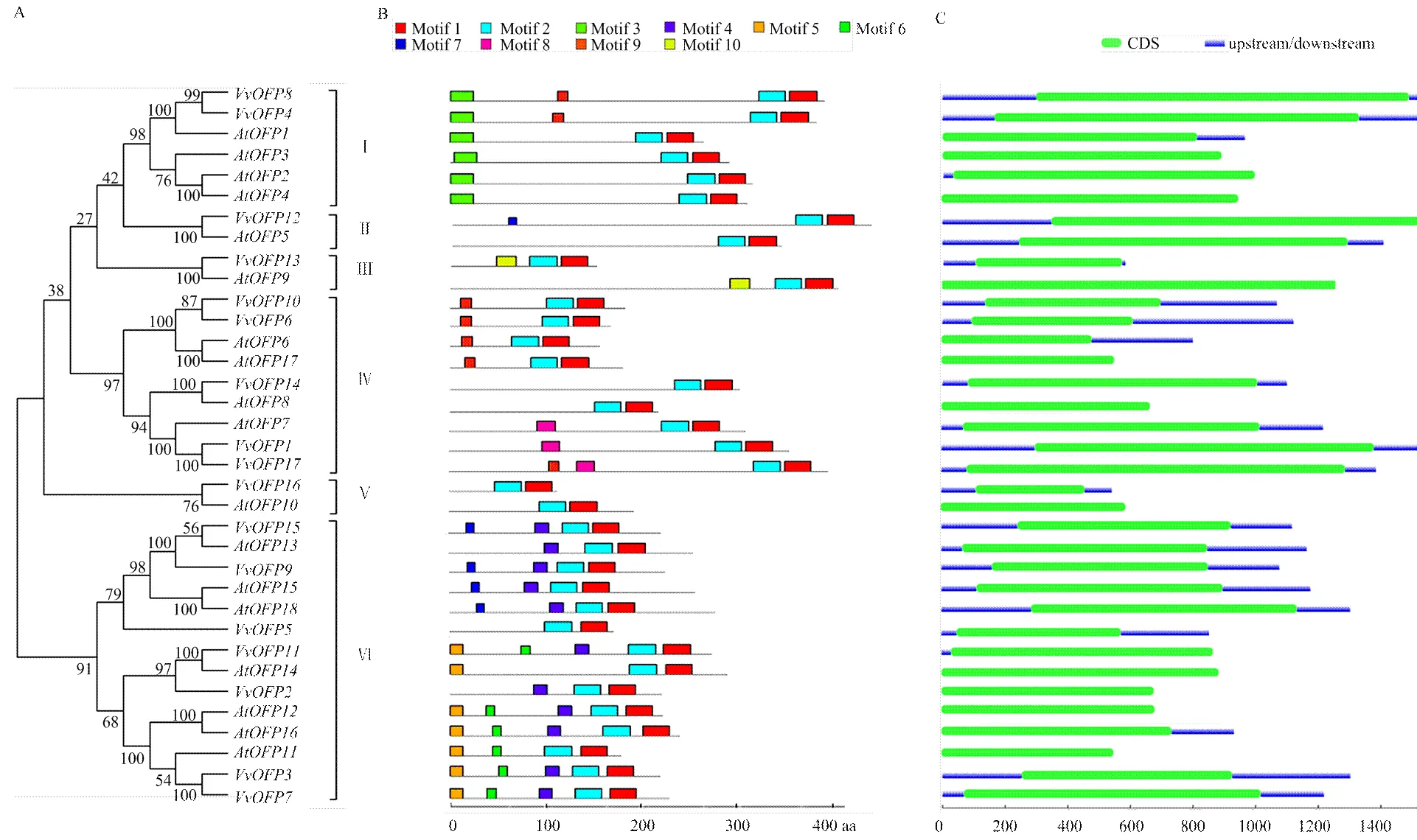
基因登录号分别是:AtOFP1,At5g01840;AtOFP2,At2g30400;AtOFP3,At5g58360;AtOFP4,At1g06920;AtOFP5,At4g18830;AtOFP6,At3g52525;AtOFP,At2g18500;AtOFP8,At5g19650;AtOFP9,T24H24.4;AtOFP10,At5g22240;AtOFP11,At4g14860;AtOFP12,At1g05420;AtOFP13,At5g04820;AtOFP14,At1g79960;AtOFP15,At2g36050;AtOFP16,At2g32100;AtOFP17,At2g36026;AtOFP18,At3g52540;系统发育树树枝上面或下面的数字代表自举值
2.6的内含子和外显子分析
利用Pfam数据库中OVATE保守结构域序列对拟南芥全基因组序列进行BLAST,并在植物转录因子数据库PlnTFDB(3.0)中搜索AtOFP9,共鉴定出18个家族基因,根据两个物种的基因组和CDS序列,绘制两个物种的基因结构图。图3-C表明拟南芥和葡萄家族基因均没有内含子,此外,葡萄家族有两个基因结构不完整,分别是缺少3′非编码区,既缺少非5′编码区,也没有3′非编码区;拟南芥家族只有6个基因包含完整的结构,、、、、、、和既缺少非5′编码区,也没有3′非编码区,、和缺少5′非编码区,缺少3′非编码区。
2.7 VvOFPs的亚细胞定位
在3个亚细胞定位网站上分析葡萄OVATE蛋白家族,预测的结果存在差异(表3),其中在CelloV2.5 预测结果中,15个蛋白被定位在细胞核,VvOFP9被定位在叶绿体和细胞外,VvOFP5被定位在细胞膜和细胞外;根据WOLF PSORT 预测结果,17个蛋白被定位在细胞核,12个在线粒体,11个在叶绿体,9个在三者中均被定位,还有少数蛋白被定位在细胞外、细胞膜、内质网和高尔基体上;在PROTCOMP预测结果中,8个蛋白被定位在细胞核,其余7个蛋白被定为在细胞外。综合分析预测结果,5个蛋白定位结果一致,定位在细胞核的分值最高,分别是VvOFP6、VvOFP7、VvOFP11、VvOFP13和VvOFP14,另外12个蛋白在两种或一种定位方法中被定为在细胞核,这符合转录因子在细胞核中调控基因表达的作用特点。

表3 葡萄OVATE蛋白家族的亚细胞定位预测
nucl:细胞核,plas:质膜,extr:细胞外,cyto:细胞质,mito:线粒体,E.R.:内质网,chlo:叶绿体,golg:高尔基体。括号内的数值代表预测的综合得分
nucl: nucleus, plas: plasma membrane, extr: extracellular, cyto: cytoplasm, mito: mitochondria, E.R.: endoplasmic reticulum, chlo: chloroplast, golg: golgi. The numbers in parenthesis mean the integral prediction scores of protein location
2.8家族基因在不同组织中表达分析
对葡萄基因家族在不同组织的表达结果(图4)进行分析,表明这些基因具有组织表达特异性,12个基因在各个组织中均可以检测到表达,其余基因、、、、仅在几个组织中检测到表达,且这5个基因集中在Ⅲ类和Ⅵ类。在根中表达量较高的是、、、、;在茎中表达量较高的是、、、、、;在花前1周的花蕾表达量较高的有、、、、、、、;在开花期的小花中表达量较高的有、、、。这些基因在叶片和花后4周的果实中表达量均较低。对比不同时期花和果实的表达量,可以发现全部基因在花后4周的果实中表达量均显著下降,大部分基因花前1周花蕾中的表达量高于开花期,这说明葡萄OVATE家族基因对果实发育的调控在开花期及其前1周。此外,和这对旁系同源基因表达模式相似,主要在生殖器官中表达;而和,和,和,这3对旁系同源基因表达模式不同。
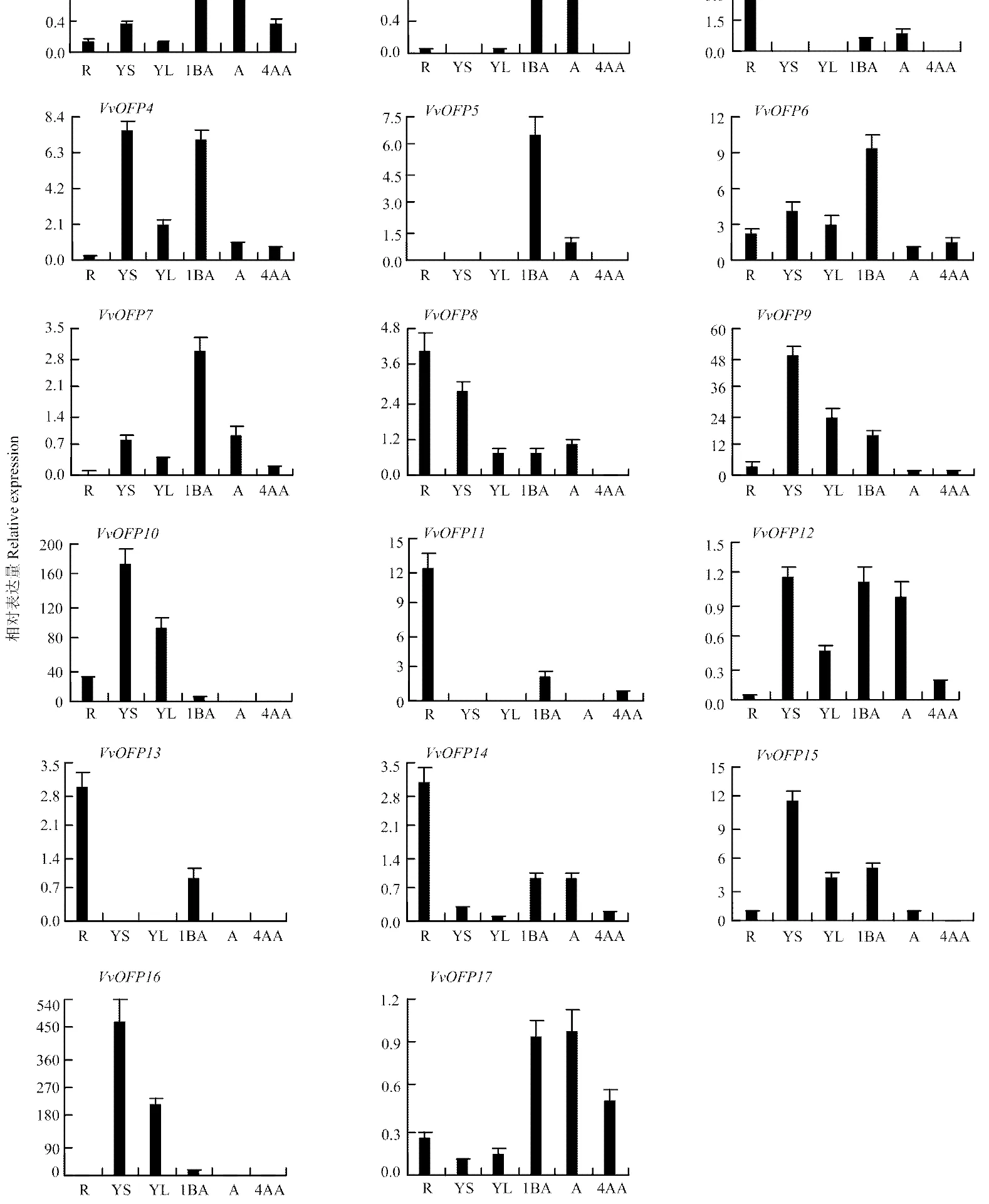
R:根,YS:嫩茎,YL:嫩叶,1BA:花蕾(花前1周),A:小花(开花期),4AA:果实(花后4周);垂直线表示的是3个生物学重复相对表达量之间的正偏差
3 讨论
本文利用Pfam数据库中OVATE保守结构域序列对葡萄全基因组序列进行BLAST,共获得葡萄基因家族成员17个,经过保守结构域分析鉴定,这些基因均属于基因家族。LIU等[23]根据已报道的拟南芥OVATE蛋白序列以及番茄OVATE蛋白(AAN17752),在Phyzome和Genoscope两个基因组网站上进行Blast,共获得9个葡萄基因家族成员,比本文获得的基因数少了8个。本文所鉴定的拟南芥基因家族成员与前人报道不尽相同,HACKBUSCH等[1]根据番茄OVATE蛋白结构域序列在拟南芥基因组数据库TAIR(http://www.arabidopsis.org/)中进行BLAST,获得18个,WANG等[4]发现(登录号At4g04030)编码的蛋白中缺少OVATE结构域,本文利用Pfam数据库中OVATE保守结构域序列对拟南芥全基因组序列进行BLAST,只获得了17个基因,缺少(登录号At4g04030),在植物转录因子数据库PlnTFDB(3.0)中查找到含有OVATE结构域的,将该蛋白序列在NCBI上BLAST,获得相似度100%的序列(登录号AF075598),该序列是通过细菌人工染色体克隆获得,氨基酸数目为411个,而氨基酸数目仅为128个,两个基因均位于4号染色体,存在部分重叠区域,因此,本研究认为即是。
葡萄和拟南芥均属于双子叶植物,前人研究[21,23]发现在单子叶植物的家族基因数目高于双子叶植物,高等植物家族多数基因缺少内含子结构,其中带有内含子的基因一般只有1个内含子,这可能是因为基因家族在进化过程中相对保守。本文对葡萄和拟南芥家族基因结构进行分析,发现两个物种均没有内含子结构。本研究根据葡萄和拟南芥基因家族的蛋白序列进行进化树构建,并根据进化树的拓扑结构分为6类,基序分析表明在OVATE结构域外,每个类别包含特有基序,这也同时验证了分类结果。在多项OVATE蛋白家族聚类分析研究中也采用了此类方法[4,20-21,23]。LIU等[23]提出基因家族包括保守进化和发散扩增两种进化机制,一方面有些分支每个物种仅有1—2个基因,这些基因在功能上相对保守,可能对植物的发育有着非常重要的作用;另一方面有些分支每个物种包含很多基因,这些基因在功能上发生了分化,拥有这个物种所特有的功能,本文也有类似的情况,如Ⅱ、Ⅲ、Ⅴ类中仅包含两个基因,可能发生了保守进化,产生相似的功能。
葡萄在生长发育过程中要经历营养生长和生殖生长两个时期,基因在不同发育期不同组织的表达特性反应了基因的功能,本研究表明葡萄基因家族在不同组织中特异表达,有些基因在某个组织中特异表达,例如只在花前一周和开花期检测到表达,说明该基因可能在花中发挥调控作用;有些基因在生殖器官或营养器官中表达量较高,例如,在花中表达量较高,而在根、叶中表达量极少,推测该基因可能参与了葡萄生殖生长的调控;有些基因在各组织中表达量差异不大,如在叶、茎,花前1周和开花期的花中表达量差异不大,推测该基因可能调控葡萄生长发育的多个过程。对比拟南芥基因家族组织表达研究结果[4],发现一些直系同源的葡萄和拟南芥家族基因也存在类似的表达特性,推测这些基因可能行使相似的功能,例如,和被检测到在嫩茎中表达量较高和在花蕾中表达量较高和在根中被检测到表达量较高。LIU等[23]研究发现番茄中旁系同源基因存在两种表达模式,一种是保持复制前的功能,两个基因具有相似的表达模式;另一类是两个基因的功能发生了变异,其中1个基因保留着原始的功能,而另1个基因则产生了新的表达模式。本研究中也有类似的发现,如和这对旁系基因在生殖器官中的表达量较高;、与属于直系同源基因,和在根中的表达量较高,而在根部的表达量很低,在花前1周的花蕾中表达量较高。
4 结论
通过分析葡萄基因家族的染色体定位、基因结构、亚细胞定位、保守域分析、系统进化和基序鉴定等多方面的生物学信息,较为系统地鉴定了葡萄基因家族,葡萄OVATE结构域序列比较保守;其在不同组织中呈现出多种表达模式,推测其可能参与了葡萄生长发育的调控。
References
[1] HACKBUSCH J, RICHTER K, MULLER J, SALAMINI F, UHRIG J F. A central role ofovate family proteins in networking and subcellular localization of 3-aa loop extension homeodomain proteins., 2005, 102(13): 4908-4912.
[2] PAGNUSSAT G C, YU H J, SUNDARESAN V. Cell-fate switch of synergid to egg cell inmutant embryo sacs arises from misexpression of the BEL1-like homeodomain gene., 2007, 19(11): 3578-3592.
[3] WANG S, CHANG Y, GUO J, CHEN J G.ovate family protein 1 is a transcriptional repressor that suppresses cell elongation., 2007, 50(5): 858-872.
[4] WANG S, CHANG Y, GUO J, ZENG Q, ELLIS B E, CHEN J G.ovate family proteins, a novel transcriptional repressor family, control multiple aspects of plant growth and development., 2011, 6(8): e23896.
[5] LI E, WANG S, LIU Y, CHEN J G, DOUGLAS C J. OVATE FAMILY PROTEIN4 (OFP4) interaction with KNAT7 regulates secondary cell wall formation in., 2011, 67(2): 328-341.
[6] LIU J, VAN ECK J, CONG B, TANKSLEY S D. A new class of regulatory genes underlying the cause of pear-shaped tomato fruit., 2002, 99(20): 13302-13306.
[7] TSABALLA A, PASENTSIS K, DARZENTAS N, TSAFTARIS A S. Multiple evidence for the role of angene in determining fruit shape in pepper., 2011, 11: 46.
[8] 陈杰忠. 果树栽培学各论. 北京: 中国农业出版社, 2011.
CHEN J Z.. Beijing: China Agriculture Press, 2011. (in Chinese)
[9] VITULO N, FORCATO C, CARPINELLI E C, TELATIN A, CAMPAGNA D, D'ANGELO M, ZIMBELLO R, CORSO M, VANNOZZI A, BONGHI C, LUCCHIN M, VALLE G. A deep survey of alternative splicing in grape reveals changes in the splicing machinery related to tissue, stress condition and genotype., 2014, 14: 99.
[10] BERTOLINO E, REIMUND B, WILDT-PERINIC D, CLERC R G. A novel homeobox protein which recognizes a TGT core and functionally interferes with a retinoid-responsive motif., 1995, 270(52): 31178-31188.
[11] HAY A, TSIANTIS M. A KNOX family TALE., 2009, 12(5): 593-598.
[12] HAMANT O, PAUTOT V. Plant development: a TALE story., 2010, 333(4): 371-381.
[13] HAY A, TSIANTIS M. KNOX genes: versatile regulators of plant development and diversity., 2010, 137(19): 3153-3165.
[14] BELLAOUI M, PIDKOWICH M S, SAMACH A, KUSHALAPPA K, KOHALMI S E, MODRUSAN Z, CROSBY W L, HAUGHN G W. TheBELL1 and KNOX TALE homeodomain proteins interact through a domain conserved between plants and animals., 2001, 13(11): 2455-2470.
[15] SMITH H M, HAKE S. The interaction of two homeobox genes,and, regulates internode patterning in theinflorescence.2003, 15(8): 1717-1727.
[16] SMITH H M, BOSCHKE I, HAKE S. Selective interaction of plant homeodomain proteins mediates high DNA-binding affinity., 2002, 99(14): 9579-9584.
[17] CHEN H, BANERJEE A K, HANNAPEL D J. The tandem complex of BELL and KNOX partners is required for transcriptional repression of., 2004, 38(2): 276-284.
[18] COLE M, NOLTE C, WERR W. Nuclear import of the transcription factor SHOOT MERISTEMLESS depends on heterodimerization with BLH proteins expressed in discrete sub-domains of the shoot apical meristem of., 2006, 34(4): 1281-1292.
[19] WANG Y K, CHANG W C, LIU P F, HSIAO M K, LIN C T, LIN S M, PAN R L. Ovate family protein 1 as a plant Ku70 interacting protein involving in DNA double-strand break repair., 2010, 74(4/5): 453-466.
[20] HUANG Z, VAN HOUTEN J, GONZALEZ G, XIAO H, VAN DER KNAAP E. Genome-wide identification, phylogeny and expression analysis of,andgene family in tomato., 2013, 288(3/4): 111-129.
[21] YU H, JIANG W, LIU Q, ZHANG H, PIAO M, CHEN Z, BIAN M. Expression pattern and subcellular localization of the ovate protein family in rice., 2015, 10(3): e118966.
[22] LIU J, ZHANG J, HU W, MIAO H, ZHANG J, JIA C, WANG Z, XU B, JIN Z. Banana Ovate family protein MaOFP1 and MADS-box protein MuMADS1 antagonistically regulated banana fruit ripening., 2015, 10(4): e123870.
[23] LIU D, SUN W, YUAN Y, ZHANG N, HAYWARD A, LIU Y, WANG Y. Phylogenetic analyses provide the first insights into the evolution of OVATE family proteins in land plants., 2014, 113(7): 1219-1233.
[24] FINN R D, COGGILL P, EBERHARDT R Y, EDDY S R, MISTRY J, MITCHELL A L, POTTER S C, PUNTA M, QURESHI M, SANGRADORVEGAS A. The Pfam protein families database: towards a more sustainable future., 2015, 44(Database issue): D279-D285.
[25] WALKER J M.. Humana Press, 2005.
[26] HU B, JIN J, GUO A Y, ZHANG H, LUO J, GAO G. GSDS 2.0: an upgraded gene feature visualization server., 2014, 31(8): 1296.
[27] CROOKS G E, HON G, CHANDONIA J M, BRENNER S E. WebLogo: a sequence logo generator., 2004, 14(6): 1188-1190.
[28] CHIN-SHENG Y, YU-CHING C, CHIH-HAO L, JENN-KANG H. Prediction of protein subcellular localization., 2006, 64(3): 643-651.
[29] TAMURA K, PETERSON D, PETERSON N, STECHER G, NEI M, KUMAR S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods., 2011, 28(10): 2731-2739.
[30] BAILEY T L, MIKAEL B, BUSKE F A, MARTIN F, GRANT C E, LUCA C, JINGYUAN R, LI W W, NOBLE W S. MEME SUITE: tools for motif discovery and searching., 2009, 37(Suppl2): W202-W208.
[31] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta delta C(T)) method., 2001, 25(4): 402-408.
(责任编辑 赵伶俐)
Bioinformatics and Expression of theGene Family in Grape
YUAN Yue, ZHANG Ya-guang, GAO Shi-min, TAO Jian-min
(College of Horticulture, Nanjing Agricultural University, Nanjing 210095)
【Objective】OVATE is a class of transcriptional repressor that regulates plant growth and development. In this study, the authors analyzed the grapegene family () from the aspects of bioinformatics and tissue-specific expression in order to lay a foundation for the functional studies in future. 【Method】Based on the conserved OVATE domain (PF04844),genes were identified from grape genome. Chromosome location, gene structure, conserved domain and subcellular localization were also analyzed by the bioinformatics methods. A phylogenetic analysis was conducted based on the alignment of the OVATE proteins from grape andgenomes. The expression patterns ofwere tested via quantitative real-time polymerase chain reaction (qRT-PCR). 【Result】Seventeen putativewere identified in the grape genome and they unevenly distributed on the eleven chromosomes. No intron was found in the gene structures of. Theencoded hydrophilic proteins, containing 115-444 amino acids, and their isoelectric points ranged from 4.55-9.69. In addition, all the VvOFPs contained the entire OVATE domain and were predicted to be located in the nucleus. According to the topology of phylogenetic tree, OVATE proteins from grape andwere divided into six groups (I-VI). Group II, III and V contained only two members, whilst members of groups I, IV and VI were clustered in the species-specific pattern. VvOFPs and AtOFPs contained 10 uncharacterized motifs. Motifs 1 and 2 were located in the OVATE domain region. Besides, group-specific motifs were also found outside the OVATE domain. qRT-PCR analysis showed thatexhibited different tissue-specific expression patterns. Interestingly, twelvewere found to be expressed in the detected organs of roots, stems, leaves, flowers and fruits while the others were expressed in the specific organ tissues. Most of thewere more highly expressed in roots, stems and flowers, whereas a little gene was detected with low level in leaves and fruits. Meanwhile,expressed differently at different developmental stage. Transcripts in flowers a week before or at anthesis were at a high level, while low in fruits four weeks after anthesis. In addition, one pair of paralogues exhibited the same expression patterns, whereas the other three pairs of paralogues exhibited divergent expression patterns. 【Conclusion】Results of this study showed that grape OVATE domain is conserved.exhibited different expression patterns and may be involved in regulation of plant growth and development.
grape;gene family; bioinformatics; expression
2016-03-16;接受日期:2016-06-07
国家现代农业葡萄产业技术体系项目(CARS-30)、国家“948”重点项目(2016-X19)、淮安科技局葡萄新品种引进及设施栽培技术研究与应用项目(HAC2014019)、江苏省科技厅设施条件下葡萄高效栽培新技术研究与应用项目(SBE2014030811)
袁月,Tel:15150536081;E-mail:2013104040@njau.edu.cn。通信作者陶建敏,Tel:13905160976,E-mail:tjm266@sina.com
